水稻是维持世界过半人口生存的重要粮食作物之一,广泛种植于世界各地,其中亚洲是最主要的水稻种植区域,占世界总种植面积的90%以上[1-2]。然而,自20世纪60-70年代以来,许多水稻病毒病的发生严重影响了水稻产量和粮食安全。截至目前,在水稻上已发现的病毒病害有15种,其中严重威胁水稻生产的有10种[3]。水稻草矮病是1963年在菲律宾首次被发现[4],而后在东亚、东南亚和南亚的许多国家和地区大面积爆发[5-8];2006-2007年间,该病害在越南南部爆发,危害面积超过48.5万hm2,造成大约82.8万t的粮食损失,致使数百万的农民蒙受巨大的经济损失[9]。
水稻草矮病的典型病害症状为病株矮化呈杂草状,分蘖急剧增多,叶片狭窄,叶片褪绿黄化且有许多形状不规则的褐色锈斑,感病水稻基本不抽穗,而且一旦感病很难治愈。目前,水稻草矮病仍在我国广东、广西、海南等省有零星分布[10]。水稻病毒病具有爆发性、迁移性、间歇性的发生特点,存在随时爆发危害的潜在可能,因此水稻草矮病毒依然受到国内外研究学者的关注。本文主要针对RGSV病毒粒子特性、常用检测方法、基因组结构及功能、病症形成机制研究和防治方法等进行了综述,以期为开展RGSV相关研究提供参考。
1 RGSV病毒粒子提纯RGSV病毒,在电镜下可以观察到大量直径6-8 nm的线状或长分枝丝状粒体(大部分长为950-1 350 nm),并能形成环状结构。病毒粒子是由细线状的核糖核蛋白(Ribonucleoprotein,RNP)、病毒正义链(viral-sense RNA,vRNA)、病毒负义链(viral-complementary RNA,vcRNA)、核衣壳蛋白和依赖RNA的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)组成[11-13]。
2 RGSV在介体昆虫内的增殖RGSV是纤细病毒属(Tenuivirus)的成员,由介体昆虫褐飞虱(Nilaparvata lugens)以持久增殖型方式传播[8]。Zheng等[14]通过免疫荧光技术研究了RGSV在褐飞虱体内的侵染路线,结果显示RGSV在褐飞虱中肠上皮细胞建立初侵染点,然后穿过基底膜进入中肠肌肉组织,而后扩散至血淋巴,进入唾液腺或者扩散至整个消化系统。这一研究成果对RGSV在褐飞虱体内的侵染路线进行了描述,为制定阻断病毒传播策略提供理论基础。
3 RGSV常用检测方法常用的RGSV检测方法有间接ELISA、非放射性分子杂交和RT-PCR等。张春嵋等[15]对这3种方法进行比较发现,运用自制的融合蛋白GST-NC进行抗血清检测,其灵敏度为1 mg鲜重的病株叶片或84 ng提纯病毒;地高辛(DIG)标记的DNA探针杂交法的灵敏度为50 μg病叶或6 ng提纯病毒;而RT-PCR的检测灵敏度为10 μg病叶或2 ng提纯病毒。其中RT-PCR和分子杂交检测RGSV的方法有很高的敏感性,特异性和可重复性。但考虑到费用,时间和实用性,ELISA用于大规模检测病毒样品是较好的选择。2012年,杨靓等[16]制备了RGSV p2的多克隆抗体,灵敏度达到1:8 192,可运用于RGSV的检测。
4 RGSV基因组结构及功能RGSV基因组是多组分的,最初被认为由4条ssRNA组成,基因组全长15.46 kb[17]。研究人员从提纯病毒中抽提的病毒中抽提病毒的基因组RNA,在经过1%琼脂糖凝胶电泳后发现了6条大小分别为10.0、4.0、3.0、2.9、2.7和2.6 kb的条带,同时还可以看见dsRNA[18]。这种情况在MStV、RSV、RHBV上也存在[19-21]。目前普遍认为RGSV至少由6条负单链RNA片段组成,分别命名为RNA1-6,均采用双义编码策略,即在vRNA和vcRNA靠近5' 端处都存在一个开放阅读框(Open read frame,ORF),至少能编码12个蛋白(图 1)[6, 18, 22-23]。
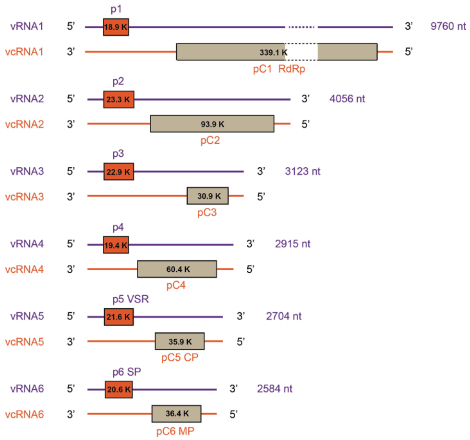
|
| 图 1 RGSV的基因组结构 |
碱基序列分析表明,RGSV所有片段的5' 端和3' 端分别有17和16个高度保守的碱基且能发生互补配对:5' -ACACAAAGUCCUGG(A/U)CA……UGCCCAGACUUUGUGU-3' 。另外通过序列比对分析发现RGSV的RNA1、RNA2、RNA5和RNA6分别对应于RSV的RNA1、RNA2、RNA3和RNA4,而RNA3和RNA4为RGSV基因组所独有,不与其它纤细病毒属成员相同[18, 22]。RGSV与水稻锯齿叶矮缩病毒(Rice ragged stunt virus,RRSV)复合侵染在田间感病水稻植株上普遍存在。Liu等[24-25]通过巢式RT-PCR及克隆测序方法研究发现RGSV能够利用prime-and-realign机制抓取RRSV基因组RNA的前导序列作为自身转录本的帽子结构,以此来维持自身转录本的完整性和稳定性。
RNA1:大小为9 760 nt,与其它纤细病毒属成员不同,它采用的是双义编码。vRNA1编码大小为18.9 kD的p1蛋白,功能未知;vcRNA1编码一个大小为339.1 kD的pC1蛋白,Toriyama等通过基因组序列比对分析发现,RGSV的vcRNA1与RSV的NS1存在很高的序列同源性,且与NS1一样具有病毒复制酶蛋白的特征性结构,如SDD三肽域等多个保守结构域[26],可在体外合成自身的ssRNA,所以推测其功能为RdRp[18, 22]。
RNA2:大小为4 056 nt,vRNA2编码大小为23.3 kD的p2蛋白,Chomchan等[27]通过Western-blot用p2的抗血清检测RGSV侵染后的水稻叶片组织得出,p2主要存在细胞质的可溶性组分中,也有部分在细胞壁中、多种细胞器和粗膜组分中被检测出,推测p2的功能可能为一个运动蛋白;而最近Nguyen等[28]研究发现RGSV越南分离株p2具有抑制RNA沉默途径的沉默抑制子(Viral suppressor of RNA silencing,VSR)活性;vcRNA2编码大小为93.9 kD的pC2蛋白。根据与白蛉热病毒属的相应蛋白之间氨基酸序列相似性分析,推测p2可能与膜结合组分相关,pC2可能是膜糖蛋白[19, 29-31],二者的生物学功能仍需进一步证实。
RNA3、4:大小分别为3 123 nt和2 915 nt,与其它纤细病毒属成员不同,为RGSV所独有。vRNA3编码大小为22.9 kD的p3蛋白,Zhang等[32]发现p3和p5存在互作关系,为研究蛋白的生物学作用,采用马铃薯X病毒(Potato virus X,PVX)系统通过农杆菌浸润法发现p3、p5单独或共注射都会增强马铃薯X病毒对本氏烟的致病性,且p3和p5共注射时导致PVX有更强的致病力;vcRNA3编码大小为30.9 kD的pC3蛋白,功能未知。vRNA4与vcRNA4分别编码大小为19.4 kD的p4蛋白和60.4 kD的pC4蛋白,功能未知。
RNA5:大小为2 704 nt,vRNA5编码大小为21.6 kD的p5蛋白,Zhang等[32]利用基于绿色荧光蛋白(Green fluorescent protein,GFP)的烟草瞬时表达实验,将RGSV p5与35S-ssGFP共注射16c本氏烟,注射后第2天发现所有叶片都能检测到很强的绿色荧光信号,但到注射后的第5天,阴性对照空载体与35S-ssGFP共注射样品中绿色荧光蛋白积累明显减弱,而阳性对照HC-Pro和RGSV p5与35S-ssGFP的共注射叶片上依然能够监测到较强的绿色荧光蛋白信号,证明RGSV p5具有沉默抑制的功能。同时通过酵母双杂交、双分子荧光互补实验等也验证了p5存在自身互作[32-33];vcRNA5编码大小为35.9 kD核衣壳蛋白pC5,是细丝状粒子的主要组成部分[27]。
RNA6:大小为2 854 nt,vRNA6编码大小为20.6 kD的p6蛋白,是病毒特异性蛋白(Disease-specific protein,SP),在病叶中大量积累,与病症的严重性密切相关[34];vcRNA6编码大小为36.4 kD的pC6蛋白,Hiraguri等[35]研究通过将pC6和融合绿色荧光蛋白的运动缺陷型烟草花叶病毒(Tobacco mosaic virus,TMV)共注射本氏烟叶片研究发现pC6可以协助TMV进行细胞间移动,表明pC6具有病毒运动蛋白的功能。
5 RGSV症状形成机制的探讨RGSV侵染水稻后的典型症状是:病株严重矮化,分蘖增多,呈现杂草状丛生,叶短而窄,灰绿色至灰黄色,并有许多不规则暗绿色小斑点,谷粒不实或有瘪粒,后期感染的水稻叶片呈黄褐色。国内外研究人员对RGSV病害症状形成机制进行了探索,本文主要从以下几个方面展开讨论。
5.1 分蘖增多分蘖增多是感染RGSV的水稻呈现出的主要症状之一,也是水稻矮缩病毒(Rice dwarf virus,RDV)和水稻锯齿叶矮缩病毒(Rice ragged stunt virus,RRSV)等矮缩类病毒侵染水稻后诱导产生的典型症状之一。在拟南芥中,MAX基因家族被证明具有调控茎分支的作用,同时也是调控独脚金内酯(Strigolactones,SL)生物合成的重要因子[36-37]。如Gomez-Roldan和Umehara等[36-37]研究发现拟南芥突变体max1、max3和max4具有茎分支增多的表型,而喷施GR24(SL类似物)会抑制茎分支增多过程,恢复为野生型,由此表明SL具有抑制植物分蘖增多的生物学功能。Ishikawa等[38]在2006年就发现水稻SL相关基因的突变体d10、d17/htd1、d27、d3和d14/htd2等都表现出分蘖增多和矮化的表型,但分子机制还不是很清楚。而后SL相关基因调控水稻分蘖增多的分子机制相继被阐释,其中D27、D17和D10是SL生物合成所必需的[39-41],D3和D14介导SL信号感知和信号转导[42-44],D53是SL信号转导的抑制因子[45]。这些研究发现说明了SL在控制植物分蘖过程中发挥着重要作用。在感染RGSV的水稻植株中,与SL信号传导相关的D3和D14/HTD2基因的表达水平受到抑制,而且SL的含量是减少的,意味着RGSV可能通过影响SL的合成及信号传导途径介导水稻分蘖增多的表型[46]。Xu等[47]研究发现,OsPIN1基因会编码一个生长素(Indoleacetic acid,IAA)转运蛋白PIN1,利用RNAi方法降低水稻OsPIN1基因的表达水平会抑制IAA向茎部输送,从而影响茎部IAA的积累量,最终导致OsPIN1-RNAi转基因水稻表现出分蘖增多的症状,这一结果显示生长素在调控植物分蘖过程中也具有重要作用。然而从Satoh等[46, 48]的转录组测序结果中可以看出,RGSV侵染后激活了OsPIN1,这与PIN1负调控水稻分蘖的结论不一致;与健康水稻相比,RGSV侵染水稻及RSV侵染水稻中IAA合成相关基因的表达被抑制,IAA降解相关基因的表达被诱导,推测RGSV和RSV侵染水稻中IAA含量可能降低了。因此,IAA是否参与RGSV诱导分蘖增多的致病过程仍值得深入研究。SPL14(Squamosa promoter binding protein-like 14)、RCN1(Reduced culm number 1)和Ostill1(Oryza sativa tillering 1)基因被报道能促进水稻分蘖增多[49-51]。RGSV会诱导RCN1和Ostill1的表达,但SPL14的表达却是受到抑制,意味着RCN1和Ostill1单独被激活可能有助于RGSV侵染水稻植株后过度分蘖的表型产生[46]。由此可见,SL、IAA等激素水平和信号传导途径的改变及性状基因的表达等都可能是RGSV诱导水稻分蘖增多的诱因。
5.2 植株矮化植株矮化是许多水稻病毒病主要表现的症状。Sakamoto等[52]研究发现赤霉素(Gibberellic acid,GA)合成相关酶类的水稻突变体cps1、ks1、ko2、kao、ga20ox2和ga3ox2等都有矮化的表型,而外源喷施GA会恢复这些突变体的表型。在感染RGSV的水稻植株中,与GA合成相关的CPS1和GA3ox2基因的表达水平受到抑制,意味着RGSV可能通过负调控GA的合成途径来介导水稻矮化的表型[46];Lo等[53]证明GA2ox是一个GA钝化基因,它会减少生物体内活性GA的含量,增加无活性GA的含量,而其过表达会使转基因水稻表现出矮化的症状。在感染RDV的水稻中,GA2ox被诱导表达[54],在感染RGSV的水稻中同样能诱导GA2ox的表达,进一步说明水稻病毒通过调控GA生物合成介导矮化症状的形成可能具有普遍性[46];油菜素内酯(Brassinosteroids,BR)是另外一类具有调控株高的植物内源激素。Yamamuro等[55]研究发现,水稻BRI1缺失突变体d61,对BR不敏感且会表现出矮化的表型,观察发现d61没有生长的节间细胞内的微管排布异常紊乱。然而,在感染RGSV的水稻植株中,OsBRI1基因表达水平没有明显的改变[46],表明OsBRI1可能与RGSV诱导水稻分蘖增多的表型不存在直接的关系。另外,细胞壁缺失也会引起植株矮化。如黄瓜花叶病毒Y(Cucumber mosaic virus strain Y,CMV-Y)和RDV会抑制细胞壁形成相关基因的表达,进而产生矮化的症状[56-57]。Choi等[58]研究发现OsEXP4基因编码一个扩张蛋白,OsEXP4-RNAi转基因水稻具有植株矮化的表型,它会影响细胞壁的结构。在感染RGSV或RSV的水稻植株中细胞壁合成相关基因和许多扩张蛋白基因的表达都受到明显抑制,表明RGSV侵染抑制水稻细胞壁的形成和细胞的伸长[46, 48],暗示细胞壁合成相关基因和扩张蛋白基因可能与RGSV介导水稻矮化症状的形成存在相关性。以上讨论说明GA等激素水平及其信号传导途径的改变、细胞壁合成和扩增蛋白基因的表达等可能是RGSV诱导水稻矮化的诱因。许多研究报道表明植株矮化和分蘖增多的表型往往偶联发生。例如,SL生物合成和信号转导相关基因的突变体均具有矮化及分蘖增多两种表型[39-44]。因此,RGSV诱导水稻矮化与分蘖增多的病害产生机制需结合两方面研究结果综合分析。
5.3 叶片黄化叶片黄化也是许多病毒病共有的病害症状,研究发现叶绿体受损会使植物叶片黄化,如RSV会引起寄主细胞缺失叶绿体,产生黄化萎蔫的症状[48]。番茄花叶病毒(Tomato mosaic virus,ToMV)、甘蔗花叶病毒(Sugar cane mosaic virus,SCMV)和TMV的编码蛋白通过与寄主叶绿体相关蛋白互作,破坏或扰乱寄主叶绿体的结构与功能,使其产生花叶的症状[59-61]。而不同的叶片黄化模式可能还会与植物体内叶绿素的合成与降解过程相关。例如,水稻突变体sgr、nyc1和nol会表现出叶片长久的保持绿色[62-64]。Park等[62]研究发现发现Sgr基因的过表达会使转基因水稻表现出叶片黄化,体外实验证明Sgr蛋白会与光捕获叶绿素绑定蛋白(Light-harves-ting chlorophyll binding protein Ⅱ,LHCPⅡ)发生互作,在类囊体膜(Thylakoid membrane)形成Sgr-LHCPll复合体,Sgr与LHCPll互作会促进LHCPll的降解,进而正调控叶绿素的降解,使水稻植株产生黄化症状;Sato等[63]发现NYC1和NOL互作且共定位在类囊体膜,在水稻中形成一个叶绿素b还原酶(Chlorophyll b reductase)的复合物,通过降解叶绿素b的含量使水稻植株产生黄化的表型;RGSV和RSV都会抑制叶绿素合成相关基因的表达,这意味着RGSV侵染导致水稻叶绿素的合成过程受到抑制[46, 48]。同时,在感染RGSV水稻中,Sgr和叶绿素还原酶两个与叶绿素降解相关的基因受到RGSV侵染的显著诱导。因此,RGSV诱导水稻产生黄化的表型可能也与Sgr等促进叶绿素降解过程相关[46]。值得一提的是,在感染RSV的水稻中,Sgr基因的表达水平却没有太大的变化,暗示着RGSV和RSV介导水稻出现叶片褪绿症状存在差异[46, 48]。以上研究报道说明叶绿体的结构和功能以及叶绿素水平的改变等因素是导致水稻叶片黄化的直接原因。然而,RGSV诱导水稻叶片黄化的致病过程受哪种因素调控尚无定论。
6 水稻草状矮缩病的防治措施水稻草矮病的爆发往往是随着介体昆虫褐飞虱的爆发而引起的。褐飞虱是水稻上主要害虫之一,广泛分布于南亚、东南亚和东亚各国,具有远距离迁飞能力[65]。研究发现,在春夏季,褐飞虱随着西南气流从中南半岛和广东、广西南部或菲律宾向我国北部迁飞,而在秋季顺东南风冷空气向南回迁[66],褐飞虱的迁飞可能是水稻草矮病交替流行发生于中国南部、越南、印度尼西亚和菲律宾等国家和地区的主要原因。由于缺少对水稻病毒致病机制的了解,至今尚无特效靶向药物的应用,是有效防控水稻病毒病的瓶颈。但根据水稻病毒病的流行特点,谢联辉教授提出了以“抗”、“避”、“除”、“治”四字原则为导向的的水稻病毒病害防控策略。首先,抗病品种的筛选和推广是最经济有效的病害防控策略。对此,国内外都做了大量的工作培育抗RGSV的水稻品种的工作,由于对RGSV的抗性机制研究缺乏,合适的抗RGSV的抗性基因还没有在自然水稻中被发现,因此还未能找到理想的抗病品种。随着分子生物学和遗传学的发展,基于RNA沉默的水稻抗病毒机制研究成果显著[67-70]。例如,Shimizu等[71]发现基于RNA干扰技术构建的RGSV的核衣壳蛋白pC5和运动蛋白pC6的RNAi转基因水稻对RGSV有很强的抗病性。Wu等[72-73]研究发现水稻病毒通过诱导基因沉默的核心元件AGO18的表达,使之作为分子锁扣结合miR168和miR528,正调控AGO1和活性氧水平介导水稻产生广谱的抗病性。以上抗病机制的揭示都将为未来抗病品种培育提供方向。病毒防控的另一策略即结合病虫测报,调节播种、插秧时间,适当早播或晚播,使感病率高、抗病性弱的秧苗期避开介体昆虫虫量高峰期,达到了较为理想的避病效果。另外,使用化学药剂杀虫防病也是有效防控病毒病的主要措施。通过药剂使用可有效控制虫口密度,降低传播介体数量和初侵染源。
综上所述,水稻草矮病的防治工作需要围绕RGSV的准确诊断,毒虫的预警与检测以及筛选理想的抗病品种等方面展开。根据水稻草矮病毒的致病特性及流行特点,再结合谢联辉院士提出的“四字”原则,综合防治水稻草矮病[74]。
7 展望尽管对RGSV的研究已经有半个多世纪了,但对该病毒的研究仍处于初级阶段,还存在许多未解答的问题。首先,RGSV基因组有12个ORF,编码12个蛋白,截至目前仅对pC1、p5、pC5和pC6的功能进行了验证,其它蛋白的功能及其与寄主水稻的互作机制尚不清楚;RGSV基因组与纤细病毒属其它成员存在差异,其RNA3和RNA4片段是RGSV所独有,那么RGSV与纤细病毒属的其它成员存在怎样的进化关系也是值得研究的问题;另外,虽然RGSV在介体昆虫体内的侵染路线已经确定[14],但是RGSV如何在介体昆虫体内进行复制、装配和增殖也缺乏系统的研究;Satoh等[46]通过转录组分析对RGSV介导病症形成机制做了一个初探,如何从遗传学角度进行深入验证是未来一个重要研究内容;如何挖掘抗病基因和抗病品种的培育及推广更是未来水稻病毒病害研究中的终极目标。以上所述均是未来RGSV研究的重要方向,对以上问题的解答将有助于我们深化对RGSV流行发病的认识,为防控RGSV策略的制定提供方向和理论依据。
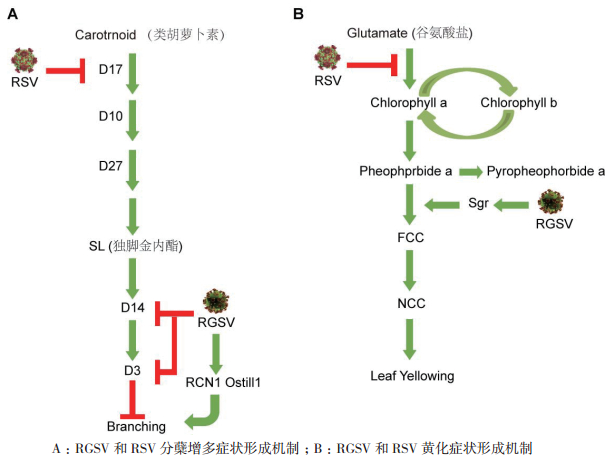
|
| 图 2 RGSV与RSV症状形成机制的可能工作模式 |
| [1] |
Normile D. Agricultural research.Reinventing rice to feed the world[J]. Science, 2008, 321(5887): 330-3. DOI:10.1126/science.321.5887.330 |
| [2] |
Zeigler RS, Barclay A. The relevance of rice[J]. Rice, 2008, 1(1): 3-10. DOI:10.1007/s12284-008-9001-z |
| [3] |
Sasaya T, Nakazono-Nagaoka E, Saika H, et al. Transgenic strategies to confer resistance against viruses in rice plants[J]. Frontiers in Microbiology, 2014, 4(2): 409. |
| [4] |
Rivera CT, Ou SH, Iida TT. Grassy stunt disease of rice and its transmission by the planthopper Nilaparvata lugens Stal[J]. Plant Disease Reporter, 1966, 50(7): 453-456. |
| [5] |
Hibino H. Biology and epidemiology of rice viruses[J]. Annu Rev Phytopathol, 1996, 34(1): 249-274. DOI:10.1146/annurev.phyto.34.1.249 |
| [6] |
Shirako Y, Falk BW, Haenni AL. Genus tenuivirus, in virus taxonomy: classification and nomenclature: Ninth Report of the International Commitee on Taxonomy of Viruses[M] //King AMQ, Lefkowitz EJ, Adams MJ, et al. San Diego: Elsevier Academic Press, 2011: 771-776.
|
| [7] |
谢联辉, 林奇英. 水稻品种对病毒病的抗性研究[J]. 福建农林大学学报:自然科学版, 1982, 2: 003. |
| [8] |
Hibino H. Rice grassy stunt virus[J]. Tropical Agriculture Research, 1986, 165-171. |
| [9] |
Cabauatan PQ, Cabunagan RC, Choi IR. Rice viruses transmitted by the brown planthopper Nilaparvata lugens Stål[M]. Planthoppers: New threats to the sustainability of intensive rice production systems in Asia, Internatrondl Rice Research Institute, 2009, 357-368.
|
| [10] |
吴祖建, 谢莉妍. 中菲水稻病毒病的比较研究:Ⅴ.水稻种质对病毒及其介体的抗性[J]. 福建农学院学报, 1994, 23(1): 58-62. |
| [11] |
Mayo M, De Miranda J, Falk B, et al. Genus Tenuivirus[M]. Academic Press, 2000, 904-908.
|
| [12] |
Pellegrini S, Bassi M. Ultrastructure alterations in rice plants affected by "grassy stunt" disease[J]. Journal of Phytopathology, 1978, 92(3): 247-250. DOI:10.1111/j.1439-0434.1978.tb03609.x |
| [13] |
Shikata E, Senboku T, Ishimizu T. Ishimizu T.The causal agent of rice grassy stunt disease[J]. Proceedings of the Japan Academy.Ser.B:Physical and Biological Sciences, 1980, 56(2): 89-94. DOI:10.2183/pjab.56.89 |
| [14] |
Zheng L, Mao Q, Xie L, Wei T. Infection route of rice grassy stunt virus, a tenuivirus, in the body of its brown planthopper vector, Nilaparvata lugens(Hemiptera:Delphacidae)after ingestion of virus[J]. Virus Research, 2014, 188: 170-173. DOI:10.1016/j.virusres.2014.04.008 |
| [15] |
张春嵋, 林奇英. 水稻草矮病毒血清学和分子检测方法的比较[J]. 中国病毒学, 2000, 15(4): 361-366. |
| [16] |
杨靓, 邓萍, 王开放, 等. 水稻草状矮缩病毒P2基因多克隆抗体的制备及应用[J]. 中国农学通报, 2012, 28(30): 1-5. |
| [17] |
Toriyama S. Purification and biochemical properties of rice grassy stunt virus[J]. Annals of the Phytopathological Society of Japan, 1985, 51: 59. |
| [18] |
Toriyama S, Kimishima T, Takahashi M. The proteins encoded by rice grassy stunt virus RNA5 and RNA6 are only distantly related to the corresponding proteins of other members of the genus Tenuivirus[J]. J Gen Virol, 1997, 78(9): 2355-2363. DOI:10.1099/0022-1317-78-9-2355 |
| [19] |
Ramirez BC, Macaya G, Calvert L A, et al. Rice hoja blanca virus genome characterization and expression in vitro[J]. J Gen Virol, 1992, 73(6): 1457-1464. DOI:10.1099/0022-1317-73-6-1457 |
| [20] |
Toriyama S, Watanabe Y. Characterization of single-and double-stranded RNAs in particles of rice stripe virus[J]. J Gen Virol, 1989, 70(3): 505-511. DOI:10.1099/0022-1317-70-3-505 |
| [21] |
Falk B, Tsai J. Identification of single-and double-stranded RNAs associated with maize stripe virus[J]. Phytopathology, 1984, 74(8): 909-915. DOI:10.1094/Phyto-74-909 |
| [22] |
Toriyama S, Kimishima T, Takahashi M, et al. The complete nucleotide sequence of the rice grassy stunt virus genome and genomic comparisons with viruses of the genus Tenuivirus[J]. J Gen Virol, 1998, 79(8): 2051-2058. DOI:10.1099/0022-1317-79-8-2051 |
| [23] |
Kormelink R, Garcia ML, Goodin M, et al. Negative-strand RNA viruses:the plant-infecting counterparts[J]. Virus Research, 2011, 162(1): 184-202. |
| [24] |
Liu X, Jin J, Qiu P, et al. Rice stripe tenuivirus has a greater tendency to use the prime-and-realign mechanism in transcription of genomic than in transcription of antigenomic template RNAs[J]. Journal of Virology, 2017, 92(1): e01414-e01417. |
| [25] |
Liu X, Xiong G, Qiu P, et al. Inherent properties not conserved in other tenuiviruses increase priming and realignment cycles during transcription of Rice stripe virus[J]. Virology, 2016, 496: 287. DOI:10.1016/j.virol.2016.06.018 |
| [26] |
Poch O, Sauvaget I, Delarue M, et al. Identification of four conserved motifs among the RNA-dependent polymerase encoding elements[J]. The EMBO Journal, 1989, 8(12): 3867. |
| [27] |
Chomchan P, Miranda G, Shiako Y. Detection of rice grassy stunt tenuivirus nonstructural proteins p2, p5 and p6 from infected rice plants and from viruliferous brown planthoppers[J]. Archives of Virology, 2002, 147(12): 2291-2300. DOI:10.1007/s00705-002-0893-4 |
| [28] |
Nguyen TD, Lacombe S, Bangratz M, et al. p2 of Rice grassy stunt virus(RGSV)and p6 and p9 of Rice ragged stunt virus(RRSV)isolates from Vietnam exert suppressor activity on the RNA silencing pathway[J]. Virus Genes, 2015, 51(2): 267-275. DOI:10.1007/s11262-015-1229-2 |
| [29] |
Takahashi M, Toriyama S, Hamamatsu C, et al. Nucleotide sequence and possible ambisense coding strategy of rice stripe virus RNA segment 2[J]. J Gen Virol, 1993, 74(4): 769-773. DOI:10.1099/0022-1317-74-4-769 |
| [30] |
De Miranda JR, Munozm, Wu R, et al. Sequence of rice hoja blanca tenuivirus RNA-2[J]. Virus Genes, 1996, 12(3): 231-237. DOI:10.1007/BF00284643 |
| [31] |
Estabrook EM, Suyenaga K, Tsai JH, et al. Maize stripe tenuivirus RNA2 transcripts in plant and insect hosts and analysis of pvc2, a protein similar to the Phlebovirus virion membrane glycoproteins[J]. Virus Genes, 1996, 12(3): 239-247. |
| [32] |
Zhang C, Liu XJ, Wu KC, et al. Rice grassy stunt virus nonstructural protein p5 serves as a viral suppressor of RNA silencing and interacts with nonstructural protein p3[J]. Archives of Virology, 2015, 160(11): 2769-2779. DOI:10.1007/s00705-015-2560-6 |
| [33] |
Chomchan P, Li SF, Shirako Y. Rice grassy stunt tenuivirus nonstr-uctural protein p5 interacts with itself to form oligomeric complexes in vitro and in vivo[J]. Journal of Virology, 2003, 77(1): 769-775. DOI:10.1128/JVI.77.1.769-775.2003 |
| [34] |
林丽明, 吴祖建. 水稻草矮病特异蛋白抗血清的制备及应用[J]. 植物病理学报, 1999, 29(2): 126-131. |
| [35] |
Hiraguri A, Netso O, Shimizu T, et al. The nonstructural protein pC6 of rice grassy stunt virus trans-complements the cell-to-cell spread of a movement-defective tomato mosaic virus[J]. Archives of Virology, 2011, 156(5): 911-916. DOI:10.1007/s00705-011-0939-6 |
| [36] |
Gomez-Roldan V, Fermas S, Brewer PB, et al. Strigolactone inhibition of shoot branching[J]. Nature, 2008, 455(7210): 189-194. DOI:10.1038/nature07271 |
| [37] |
Umehara M, Hanada A, Yoshida S, et al. Inhibition of shoot branching by new terpenoid plant hormones[J]. Nature, 2008, 455(7210): 195-200. DOI:10.1038/nature07272 |
| [38] |
Ishikawa S, Maekawa M, Arite T, et al. Suppression of tiller bud activity in tillering dwarf mutants of rice[J]. Plant and Cell Physiology, 2005, 46(1): 79-86. DOI:10.1093/pcp/pci022 |
| [39] |
Lin H, Wang RX, Qian Q, et al. DWARF27, an iron-containing protein required for the biosynthesis of strigolactones, regulates rice tiller bud outgrowth[J]. Plant Cell, 2009, 21(5): 1512-1525. DOI:10.1105/tpc.109.065987 |
| [40] |
Zou J, Zhang SY, Zhang SY, et al. The rice HIGH-TILLERINGDWARF1 encodingan ortholog of Arabidopsis MAX3 is required for negative regulation of the outgrowth of axillary buds[J]. The Plant Journal, 2006, 48(5): 687-698. DOI:10.1111/tpj.2006.48.issue-5 |
| [41] |
Arite T, Iwata H, Ohshima K, et al. DWARF10, an RMS1/MAX4/DAD1 ortholog, controls lateral bud outgrowth in rice[J]. The Plant Journal, 2007, 51(6): 1019-1029. DOI:10.1111/j.1365-313X.2007.03210.x |
| [42] |
Arite T, Umehara M, Ishikawa S, et al. d14, a strigolactone-insensitive mutant of rice, shows an accelerated outgrowth of tillers[J]. Plant Cell Physiolology, 2009, 50(8): 1416-1424. DOI:10.1093/pcp/pcp091 |
| [43] |
Gao ZY. Dwarf 88, a novel putative esterase gene affecting architecture of rice plant[J]. Plant Mol Biol, 2009, 71(3): 265-276. DOI:10.1007/s11103-009-9522-x |
| [44] |
Liu WZ, Wu C, Fu YP, et al. Identification and characterization of HTD2:a novel gene negatively regulating tiller bud outgrowth in rice[J]. Planta, 2009, 230(4): 649-658. DOI:10.1007/s00425-009-0975-6 |
| [45] |
Jiang L, Liu X, Xiong GS, et al. DWARF 53 acts as a repressor of strigolactone signalling in rice[J]. Nature, 2013, 504(7480): 401-405. DOI:10.1038/nature12870 |
| [46] |
Satoh K, Yoneyama K, Kondoh H, et al. Relationship between gene responses and symptoms induced by Rice grassy stunt virus[J]. Viruses Threatening Stable Production of Cereal Crops, 2013, 108. |
| [47] |
Xu M, Zhu L, Shou H, et al. A PIN1 family gene, OsPIN1, involved in auxin-dependent adventitious root emergence and tillering in rice[J]. Plant and Cell Physiology, 2005, 46(10): 1674-1681. DOI:10.1093/pcp/pci183 |
| [48] |
Satoh K, Kondoh H, Sasaya T, et al. Selective modification of rice(Oryza sativa)gene expression by rice stripe virus infection[J]. J Gen Virol, 2010, 91(1): 294-305. DOI:10.1099/vir.0.015990-0 |
| [49] |
Miura K, Ikeda M, Matsubara A, et al. OsSPL14 promotes panicle branching and higher grain productivity in rice[J]. Nature Genetics, 2010, 42(6): 545-549. DOI:10.1038/ng.592 |
| [50] |
Mao C, Ding W, Wu Y, et al. Overexpression of a NAC-domain protein promotes shoot branching in rice[J]. New Phytologist, 2007, 176(2): 288-298. DOI:10.1111/j.1469-8137.2007.02177.x |
| [51] |
Yasuno N, Takamure I, Kidou SI, et al. Rice shoot branching requires an ATP-binding cassette subfamily G protein[J]. New Phytologist, 2009, 182(1): 91-101. DOI:10.1111/j.1469-8137.2008.02724.x |
| [52] |
Sakamoto T, Miura K, Itoh H, et al. An overview of gibberellin metabolism enzyme genes and their related mutants in rice[J]. Plant Physiology, 2004, 134(4): 1642-1653. DOI:10.1104/pp.103.033696 |
| [53] |
Lo SF, Yang SY, Chen KT, et al. A novel class of gibberellin 2-oxidases control semidwarfism, tillering, and root development in rice[J]. Plant Cell, 2008, 20(10): 2603-2618. DOI:10.1105/tpc.108.060913 |
| [54] |
Satoh K, Shimizu T, Kondoh H, et al. Relationship between symptoms and gene expression induced by the infection of three strains of Rice dwarf virus[J]. PLoS One, 2011, 6(3): e18094. DOI:10.1371/journal.pone.0018094 |
| [55] |
Yamamuro C, Ihara Y, Wu X, et al. Loss of function of a rice brassinosteroid insensitive1 homolog prevents internode elongation and bending of the lamina joint[J]. Plant Cell, 2000, 12(9): 1591-1605. DOI:10.1105/tpc.12.9.1591 |
| [56] |
Shimizu T, Satoh K, Kikuchi S, et al. The repression of cell wall-and plastid-related genes and the induction of defense-related genes in rice plants infected with Rice dwarf virus[J]. Molecular Plant-Microbe Interactions, 2007, 20(3): 247-254. DOI:10.1094/MPMI-20-3-0247 |
| [57] |
Marathe R, Guan Z, Anandalashmi R, et al. Study of Arabidopsis thalianaresistome in response to Cucumber mosaic virus infection using whole genome microarray[J]. Plant Mol Biol, 2004, 55(4): 501-520. DOI:10.1007/s11103-004-0439-0 |
| [58] |
Choi D, Lee Y, Cho HT, et al. Regulation of expansin gene expression affects growth and development in transgenic rice plants[J]. Plant Cell, 2003, 15(6): 1386-1398. |
| [59] |
Lehto K, Tikkanen M, HiriarT JB, et al. Depletion of the photosystem Ⅱ core complex in mature tobacco leaves infected by the flavum strain of tobacco mosaic virus[J]. Molecular Plant-Microbe Interactions, 2003, 16(12): 1135-1144. DOI:10.1094/MPMI.2003.16.12.1135 |
| [60] |
Zhang C, Liu Y, Sun X, et al. Characterization of a specific interaction between IP-L, a tobacco protein localized in the thylakoid membranes, and Tomato mosaic virus coat protein[J]. Biochem Biophys Res Commun, 2008, 374(2): 253-257. DOI:10.1016/j.bbrc.2008.07.010 |
| [61] |
Cheng YQ, Liu ZM, Xu J, et al. HC-Pro protein of sugar cane mosaic virus interacts specifically with maize ferredoxin-5 in vitro and in planta[J]. J Gen Virol, 2008, 89(8): 2046-2054. DOI:10.1099/vir.0.2008/001271-0 |
| [62] |
Park SY, Yu JW, Park JS, et al. The senescence-induced staygreen protein regulates chlorophyll degradation[J]. Plant Cell, 2007, 19(5): 1649-1664. DOI:10.1105/tpc.106.044891 |
| [63] |
Sato Y, Morita R, Katsuma S, et al. Two short-chain dehydrogenase/reductases, NON-YELLOW COLORING 1 and NYC1-LIKE, are required for chlorophyll b and light-harvesting complex Ⅱ degradation during senescence in rice[J]. The Plant Journal, 2009, 57(1): 120-131. DOI:10.1111/tpj.2008.57.issue-1 |
| [64] |
Kusaba M, Ito H, Morita R, et al. Rice NON-YELLOW COLORING1 is involved in light-harvesting complex Ⅱ and grana degradation during leaf senescence[J]. Plant Cell, 2007, 19(4): 1362-1375. DOI:10.1105/tpc.106.042911 |
| [65] |
Usugi HT, Oraura T, Tsuchizaki T, et al. Rice grassy stunt virus:a planthopper-borne circular filament[J]. Phytopathology, 1985, 75: 894-899. DOI:10.1094/Phyto-75-894 |
| [66] |
张春嵋, 吴祖建, 林丽明, 等. 水稻草状矮化病毒沙县分离株基因组第六片段的序列分析[J]. 植物病理学报, 2001, 31(4): 301-305. |
| [67] |
Sanford J, Johnston SA. The concept of parasite-derived resistance-deriving resistance genes from the parasite's own genome[J]. Journal of Theoretical Biology, 1985, 113(2): 395-405. DOI:10.1016/S0022-5193(85)80234-4 |
| [68] |
Collinge DB, Jorgensen HJ, Lund OS, et al. Engineering pathogen resistance in crop plants:current trends and future prospects[J]. Annu Rev Phytopathol, 2010, 48: 269-291. DOI:10.1146/annurev-phyto-073009-114430 |
| [69] |
Mansoor S, Amin I, Hussain M, et al. Engineering novel traits in plants through RNA interference[J]. Trends in Plant Science, 2006, 11(11): 559-565. DOI:10.1016/j.tplants.2006.09.010 |
| [70] |
Prins M, Laimer M, Noris E, et al. Strategies for antiviral resistance in transgenic plants[J]. Mol Plant Pathol, 2008, 9(1): 73-83. |
| [71] |
Shimizu T, Ogamino T, Hiraguri A, et al. Strong resistance against Rice grassy stunt virus is induced in transgenic rice plants expressing double-stranded RNA of the viral genes for nucleocapsid or movement proteins as targets for RNA interference[J]. Phytopathology, 2013, 103(5): 513-519. DOI:10.1094/PHYTO-07-12-0165-R |
| [72] |
Wu JG, Yang ZR, Wang Y, et al. Viral-inducible Argonaute18 confers broad-spectrum virus resistance in rice by sequestering a host microRNA[J]. Elife, 2015, 4: e05733. |
| [73] |
Wu JG, Yang RX, Yang ZR, et al. ROS accumulation and antiviral defence control by microRNA528 in rice[J]. Nature Plants, 2017, 3: 16203. DOI:10.1038/nplants.2016.203 |
| [74] |
谢联辉, 林奇英. 中国水稻病毒病的诊断, 监测和防治对策[J]. 福建农业大学学报, 1994, 23(3): 280-285. |




