植物病原细菌利用高度保守的Ⅲ型分泌系统(Type Ⅲ secretion system,T3SS)向寄主细胞中直接分泌效应子蛋白(Effector),抑制或破坏寄主免疫系统,或诱导感病基因表达,以达到成功侵染和定殖的目的[1-2]。在某些情况下,细菌效应子可与植物抗病蛋白互作,引起寄主细胞过敏反应[3]。因此,T3SS在细菌毒性表达和诱导植物抗病反应方面均具有重要作用。
编码T3SS装置的hrp(Hypersensitive response and pathogenicity)基因簇一般由20多个基因组成,分成几个转录单元。其中部分基因编码了T3SS蛋白组份,其它编码了调控蛋白、效应子蛋白及其转运蛋白伴侣。植物病原细菌hrp基因的表达是被紧密调控的,通常在植物胞内被诱导表达,而在丰富营养培养基中被抑制表达[1]。
水稻白叶枯病是世界水稻产区最重要的细菌病害之一[4]。水稻白叶枯病菌(Xanthomonas oryzae pv.oryzae,Xoo)通过水稻叶缘水孔或伤口进入胞内,定殖木质部,产生胞外多糖(EPS),分泌胞外水解酶(如纤维素酶和木聚糖酶),导致病症产生[4]。此外,T3SS是关键致病因子之一,由9个hrp基因(hrpB1、hrpB2、hrpB4、hrpB5、hrpB7、hrpD5、hrpD6、hrpE和hrpF)、9个hrc基因(hrcC、hrcJ、hrcN、hrcQ、hrcR、hrcS、hrcT、hrcU和hrcV)和8个hpa(hpa1、hpa2、hpa3、hpa4、hpaA、hpaB、hpaF和hpaP)基因组成[5-7],其表达调控途径比较复杂[8]。
对T3SS基因表达及其调控网络的解析,对于阐明病原细菌的致病分子机制具有重要的科学理论意义。同时,目前可有效防治水稻细菌病害的农药种类较少,而hrp基因表达调控途径的相关蛋白可能是新药研发的新分子靶标[9-10]。因此,本文将结合本实验室的研究结果,综述Xoo的T3SS表达调控及其致病机理的研究进展,以期为水稻细菌病害发生机理的解析及其有效防控措施的研发提供一些新见解、思路和途径。
1 hrp基因的诱导性表达植物病原细菌hrp基因表达具有侵染特异性,在丰富营养培养基中表达受到抑制,但在特定的模拟植物体内环境的低pH贫瘠营养培养基中可被诱导。hrp诱导培养基己被有效地用于病原细菌基因表达调控和效应子分泌机制等研究中[11]。XOM2是Xoo hrp基因诱导培养基,其中碳源是基因表达诱导的重要因素之一;在测试的葡萄糖、蔗糖和果糖等碳源中,木糖对T3SS表达诱导效果最好[12-13]。由于水稻细胞壁富含木聚糖,不能分泌木聚糖酶的Xoo突变体丧失了致病性[14-15]。Xoo中与木糖利用有关的磷酸葡萄糖异构酶缺失突变体的致病性也下降,并且影响其在植物体内的生长[16]。因此,木糖可能不仅是Xoo在水稻内生长所需的关键糖源,也是hrp基因表达的主要诱导因子之一。
2 HrpG和HrpX是hrp基因表达的关键调控因子根据hrp基因簇调控系统、基因相似性及操纵元结构,可将病原细菌hrp基因簇分为两类:丁香假单胞、胡萝卜软腐病菌和欧文氏菌属于第一类;黄单胞和青枯病菌属于第二类[1, 17]。丁香假单胞3个关键蛋白HrpR、HrpS和HrpL调控了hrp基因表达[18]。HrpR/HrpS与σ54增强子结合蛋白同源,通过激活σ因子HrpL,进而激活其它hrp基因的表达[19-20]。HrpL可以活化启动子中含有hrp框(CGAACCNA-N14-CCACNNA)的基因[1]。蛋白酶Lon通过调控HrpR的降解、负向调控了T3SS的表达[21]。然而,HrpR/HrpS上游的调控路径至今仍尚不清楚。
黄单胞HrpG和HrpX是两个关键调控蛋白,hrpG和hrpX与hrp基因簇在基因组上的位置相距较远。HrpG属于OmpR家族双组分信号系统(TCS),在细菌对外界环境刺激感应中具有重要作用,同时调控了hrpX和hrpA(hrcC)的表达[22]。HrpG磷酸化是激活hrp基因表达的关键过程,但将磷酸基团转移至HrpG的组氨酸激酶(HK)至今尚未鉴定出来。HrpX属于AraC调控蛋白家族成员,对于hrp基因簇5个操纵元(hrpB、hrpC、hrpD、hrpE和hrpF)表达必不可少,而这些操纵元编码了T3SS装置所需的蛋白产物[6, 23]。
2.1 HrpX调控hrp基因启动子顺式作用元件受HrpX调控的基因启动子区通常是一个包含PIP-box的保守序列(TTCGB-N15-TTCGB;B:C、G或T),作为顺式作用元件控制基因表达[23-24]。对启动子区同源性进行分析,发现与RNA聚合酶σ70因子的-10结合元件类似的另外一个保守序列(YANNRT:Y,C/T;N,A/T/G/C;R,A/G,称为-10 box),位于PIP box下游30-31bp位置[24]。在HrpX调控的hrp基因操纵元(hrpB、hrpC、hrpD和hrpE)和hpa1启动子中均存在典型的PIP-box序列。通过寻找细菌基因组中PIP box,新鉴定了一批受HrpX调控的候选基因[24-25]。
受HrpX调控的hrpF启动子区有一个非典型的PIP-box(TTCGC-N8-TTCGC)能被HrpX结合[26]。Xoo中编码半胱氨酸蛋白酶的cysP2和编码酮戊二酸转运蛋白的kgtP基因,其启动子区域分别含有序列TTCGC-N12-TTCGC和TTCGA-N21-TTCGC,也受到HrpX的调控[27]。表明PIP-box序列多变,在Xoo中可能还存在其它更多受HrpX调控的基因。
2.2 HrpX调控效应子基因Xoo效应子蛋白分为转录激活因子类(TAL)和非转录激活因子类(non-TAL)。TAL效应子具有一个34氨基酸的中央重复区域、核定位信号和C端转录激活结构域;转录激活结构域作用于植物细胞,激活靶基因表达,从而提高植物感病性[28]。黄单胞TAL效应子较为保守,在Xoo中可多达26个。由于TAL效应子基因无PIP-box和-10 box,其表达并不受HrpX调控。
与TAL效应子不同,non-TAL效应子不具有结构上的相似性,但在其N端氨基酸组成上具有共同特征。大多数Xoo non-TAL效应子具有以下特征中的3个:(1)多于20%的Ser和Pro残基;(2)前50个氨基酸中Leu残基的比例少于6%;(3)在前12个氨基酸中没有或仅有一个酸性氨基酸残基(Asp或Glu);(4)第3或第4个氨基酸残基是Leu,Ile,Val或Pro。已经从MAFF311018菌株中鉴定了20个non-TAL效应子。尽管一些基因启动子区不存在PIP-box或-10 box,但已证实其中至少有17个受到HrpX调控[2]。
2.3 HrpX调控其它功能基因在Ⅱ型分泌系统(T2SS)装置缺失突变体中,许多HrpX依赖的分泌蛋白缺失或分泌量下降。通过N末端氨基酸分析,鉴定出一个T2SS分泌蛋白——半胱氨酸蛋白酶CysP2。cysP2启动子区有一个不典型的PIP-box,依赖于HrpX表达。因此,HrpX调控一些T2SS分泌蛋白编码基因的表达。
总之,在Xoo基因组中具有典型或非典型PIP-box和-10 box的基因分布比较分散,其中一部分的确受HrpX转录调控[25, 27]。目前还不清楚这类基因被HrpX调控的机制,推测有可能通过尚未鉴定、受HrpX调控的转录激活子而实现的。另外,大多数受HrpX调控基因的功能至今也尚不明了。
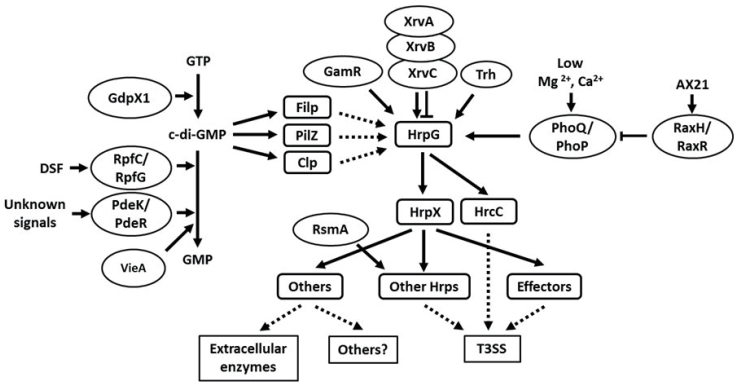
3 hrp基因表达的调控网络 3.1 双组分调控因子 3.1.1 RpfC/RpfG甘蓝黑腐病菌(X.campestris pv.campestris)TCS RpfC/RpfG对于致病性具有关键作用[29]。该TCS通过感应胞间可扩散性因子(DSF)信号分子,调节胞外酶、生物膜形成和运动性等毒性表型[30]。RpfG含有HD-GYP结构域,控制第二信使c-di-GMP的水解,从而调节毒性基因的表达等胞内过程[31]。c-di-GMP负向调控全局转录激活因子Clp表达,Clp通过2个受其调控的转录因子FhrR和Zur控制hrp基因表达[32-33]。Xoo的RpfC/RpfG和Clp也影响了hrp基因的表达[34-36]。
3.1.2 PhoP/PhoQXoo的PhoP/PhoQ通过调控hrpG的表达以应答低Ca2+浓度的环境条件。在低Ca2+浓度下,PhoP/PhoQ缺失突变导致hrpG和其它hrp基因表达下降,从而抑制细菌的毒性[37]。PhoP/PhoQ表达也受另一对TCS RaxR/RaxH负向调控,后者需要群体感应信号Ax21[38-39]。PhoP/PhoQ同时还应答低Mg2+浓度的环境条件,调控其它基因表达,包括corA1、groEL和dnaK,推测它们可能与Mg2+转运蛋白、蛋白折叠、细胞增殖/生存和自我调节有关。PhoP/PhoQ是Xoo抵抗抗菌肽和耐酸性环境所必须的,对于细菌的生存和致病性具有关键作用[37]。
3.1.3 ColR/ColS在双组分信号系统ColR/ColS缺失突变体中,hrpC和hrpE操纵元表达下降,但hrpG和hrpX表达量接近野生型水平。这使TCS与细菌生长、毒性、过敏反应及抗逆性有关[40]。
3.2 转录调控因子TrhhrpG基因转录激活是hrp基因表达的关键因素之一。Trh是已鉴定的hrpG激活子之一[41]。Trh属于GntR转录调控因子家族,其N末端有保守的DNA结合结构域,C末端有配体结合结构域。GntR蛋白通常作为基因表达的阻遏蛋白发挥功能,而Trh可能间接地正向调控hrpG基因的表达。但Trh的直接靶标以及其C末端结合分子尚未鉴定出来。
3.3 DNA/RNA结合蛋白 3.3.1 H-NS蛋白组蛋白类核结构(H-NS)蛋白是小的DNA结合蛋白,其在革兰氏阴性菌中高度保守[42-43]。这类蛋白是重要的全局调控因子,通常起转录阻遏蛋白的作用,调控毒性和环境信号响应基因的表达。Xoo有3个H-NS蛋白,其中XrvA激活hrpG基因表达[44],而XrvB阻遏hrpG基因表达,从而抑制了其它hrp基因表达[45],XrvC在Xoo侵染水稻过程中也调控其毒性[46]。
3.3.2 RsmARNA结合蛋白RsmA作为一个全局转录后修饰因子发挥作用,影响hrp基因表达[47]。该基因缺失降低了hrpA至hrpF操纵元表达,并且不依赖于HrpG和HrpX。RsmA(Xoo)还影响细菌毒性以及DSF的产生[48]。
3.4 糖代谢途径由于hrp基因表达依赖于糖源,在糖代谢与hrp基因调控间可能关联。Xoo依赖木糖[13],而细菌性条斑病菌(Xoc)则依赖蔗糖和果糖[11]。Xoo可能通过木糖抑制HrpX表达后的裂解,从而增加受HrpX调控的其它hrp基因的表达[49]。果糖二磷酸醛酸酶(FbaB)催化果糖-1,6-二磷酸与二羟基丙酮和甘油醛-3-磷酸盐间的可逆转换,对于糖水解和合成具有重要作用。FbaB除影响糖代谢外,还调控EPS产生、毒性及hrp基因表达[50]。与野生型相比,FbaB缺失突变体在水稻上的毒性降低、生长缓慢,在果糖、丙酮酸盐或苹果酸盐作为唯一碳源的培养基中EPS产量减少。同时,FbaB缺失后,hrpG和hrpX表达也受到抑制。有些hrp基因(hrcC、hrpE和hpa3)在FbaB突变体中表达量却上升。此外,FbaB本身也受到HrpG和HrpX的调控[47]。GamR是LysR类的半乳糖代谢调节因子,通过转录激活HrpG和HrpX;从而影响Xoo其它hrp基因的表达[51]。
3.5 c-di-GMP调控途径环二鸟苷酸(c-di-GMP)是在革兰氏阴性细菌中广泛存在的第二信使,参与调控细菌毒性、环境适应性、生物膜形成和运动性等[52-54]。双组分系统PdeK/PdeR调控了c-di-GMP的降解,正向调控了hrp基因表达,同时影响了EPS的产生和毒性[55]。VieA具有保守的c-di-GMP降解酶结构域,尽管其降解酶活性尚未被证实,但其对hrp表达具有正向调控作用[56]。GdpX1具有保守的c-di-GMP合成酶GGDEF结构域,对hrp基因表达具有负向调控作用[57]。应答调控因子PXO_02944的GGDEF/EAL结构域发生了突变,负向调控了hrpG基因的转录,抑制了病菌致病性[58]。
PilZ结构域蛋白是c-di-GMP的一类受体蛋白。Xoo有3个PilZ结构域蛋白,其中PXO_00049和PXO_02374均可作为受体与c-di-GMP分子发生结合,并对hrp基因表达具有负向调控作用[59]。Filp是一个退化的GGDEF/EAL结构域蛋白,可作为受体与c-di-GMP结合;同时它与另一个PilZ结构域蛋白PXO_02715发生互作,共同对hrp基因表达进行正向调控[60]。
4 结语自鉴定出HrpG和HrpX这两个关键调控因子以来,过去20年的研究已经初步构画出了Xoo hrp基因表达的复杂调控网络(图 1)。HrpG和HrpX不仅调控了hrp和效应子基因的表达,也调控其它毒性相关基因的表达。作为推测的TCS,HrpG磷酸化需要的组氨酸激酶是hrp基因表达的一个关键因素,但至今对该组氨酸激酶以及环境信号一无所知。Xoo存在通过TCS进行细胞密度相关的hrp基因表达的调控方式,如PhoP/PhoQ、RaxR/RaxH、RpfC/RpfG等。细胞密度感应开关可能是hrp基因表达的另一个关键调控因素。对于hrp基因表达的最初环节需要进一步的研究。此外,hrp基因级联调控可能与其它基因调控途径偶联,包括糖代谢和c-di-GMP信号途径等。这一复杂精细的调控网络调控了多个基因有序时空表达,从而帮助Xoo通过T3SS装置,将大量毒性效应子蛋白分泌到水稻细胞内,破坏寄主免疫系统,诱导感病基因表达,从而达到成功侵染、并在水稻寄主体内定殖的目的。
| [1] |
Alfano J, Collmer A. The type Ⅲ(Hrp)secretion pathway of plant pathogenic bacteria:trafficking harpins, Avr proteins, and death[J]. J Bacteriol, 1997, 179: 5655-5662. DOI:10.1128/jb.179.18.5655-5662.1997 |
| [2] |
Furutani A, Takaoka M, Sanada H. Identification of novel type Ⅲ secretion effectors in Xanthomonas oryzae pv.oryzae[J]. Mol Plant Microbe Interact, 2009, 22: 96-106. DOI:10.1094/MPMI-22-1-0096 |
| [3] |
Jones J, Dangl J. The plant immune system[J]. Nature, 2009, 444: 323-329. |
| [4] |
Ou S. Bacterial leaf blight[M]//. Ou S. Rice diseases. Kew: Commonwealth Mycological Institute, 1985: 70-74.
|
| [5] |
Furutani A, Tsuge S, Ohnishi K, et al. Evidence for HrpXo-dependent expression of type Ⅱ secretory proteins in Xanthomonas oryzae pv.oryzae[J]. J Bacteriol, 2004, 186: 1374-1380. DOI:10.1128/JB.186.5.1374-1380.2004 |
| [6] |
Ochiai H, Inoue Y, Takeya M, et al. Genome sequence of Xanthomonas oryzae pv.oryzae suggests contribution of large numbers of effector genes and insertion sequences to its race diversity[J]. Jpn Agric Res Q, 2005, 39: 275-287. DOI:10.6090/jarq.39.275 |
| [7] |
Oku T, Tanaka K, Iwamoto M, et al. Structural conservation of the hrp gene cluster in Xanthomonas oryzae pv.oryzae[J]. J Gen Plant Pathol, 2004, 70: 159-167. DOI:10.1007/s10327-004-0106-y |
| [8] |
Tsuge S, Furutani A, Ikawa Y. Regulatory network of hrp gene expression in Xanthomonas oryzae pv.oryzae[J]. J Gen Plant Pathol, 2014, 80: 303-313. DOI:10.1007/s10327-014-0525-3 |
| [9] |
Fan S, Tian F, Li J, et al. Identification of plant phenolic compounds that suppress the Xanthomonas oryzae virulence in rice via targeting type Ⅲ secretion system[J]. Mol Plant Pathol, 2017, 18: 555-568. DOI:10.1111/mpp.2017.18.issue-4 |
| [10] |
何晨阳, 吴茂森. 水稻—白叶枯病菌互作的功能基因组学研究[J]. 植物保护, 2007, 33: 95-96. DOI:10.3969/j.issn.0529-1542.2007.05.022 |
| [11] |
Wengelnik K, Marie C, Russel M, et al. Expression and localization of HrpA1, a protein of Xanthomonas campestris pv.vesicatoria essential for pathogenicity and induction of the hypersensitive reaction[J]. J Bacteriol, 1996, 178: 1061-1069. DOI:10.1128/jb.178.4.1061-1069.1996 |
| [12] |
Furutani A, Tsuge S, Oku T, et al. Hpa1 secretion via type Ⅲ secretion system in Xanthomonas oryzae pv.oryza e[J]. J Gen Plant Pathol, 2003, 69: 271-275. DOI:10.1007/s10327-003-0042-2 |
| [13] |
Tsuge S, Furutani A, Fukunaka R, et al. Expression of Xanthomonas oryzae pv.oryzae hrp genes in XOM2, a novel synthetic medium[J]. J Gen Plant Pathol, 2002, 68: 363-371. DOI:10.1007/PL00013104 |
| [14] |
Takeuchi Y, Tohbaru M, Sato A. Polysaccharides in primary cell walls of rice cells in suspension culture[J]. Phytochemistry, 1994, 35: 361-363. DOI:10.1016/S0031-9422(00)94764-2 |
| [15] |
Ray S, Rajeshwari R, Sonti RV. Mutants of Xanthomonas oryzae pv.oryzae deficient in general secretory pathway are virulence deficient and unable to secrete xylanase[J]. Mol Plant Microbe Interact, 2000, 13: 394-40. DOI:10.1094/MPMI.2000.13.4.394 |
| [16] |
Tsuge S, Ochiai H, Inoue Y, et al. Involvement of phosphoglucose isomerase in pathogenicity of Xanthomonas oryzae pv.oryzae[J]. Phytopathology, 2004, 94: 478-483. DOI:10.1094/PHYTO.2004.94.5.478 |
| [17] |
Gophna U, Ron EZ, Graur D. Bacterial type Ⅲ secretion systems are ancient and evolved by multiple horizontal-transfer events[J]. Gene, 2003, 312: 151-163. DOI:10.1016/S0378-1119(03)00612-7 |
| [18] |
Hutcheson S, Bretz J, Sussan T, et al. Enhancer binding proteins HrpR and HrpS interact to regulate hrp-encoded type Ⅲ protein secretion in Pseudomonas syringae strains[J]. J Bacteriol, 2001, 183: 5589-5598. DOI:10.1128/JB.183.19.5589-5598.2001 |
| [19] |
Wei Z, Beer S. hrpL activates Erwinia amylovora hrp gene transcription and is a member of the ECF subfamily of sigma factors[J]. J Bacteriol, 1995, 177: 6201-6210. DOI:10.1128/jb.177.21.6201-6210.1995 |
| [20] |
Xiao Y, Heu S, Yi J, et al. Identification of a putative alternate sigma factor and characterization of a multicomponent regulatory cascade controlling the expression of Pseudomonas syringae pv.syringae Pss61 hrp and hrmA genes[J]. J Bacteriol, 1994, 176: 1025-1036. DOI:10.1128/jb.176.4.1025-1036.1994 |
| [21] |
Bretz J, Losada L, Lisboa K, et al. Lon protease functions as a negative regulator of type Ⅲ protein secretion in Pseudomonas syringae[J]. Mol Microbiol, 2002, 45: 397-409. DOI:10.1046/j.1365-2958.2002.03008.x |
| [22] |
Laub M, Goulian M. Specificity in two-component signal transduction pathways[J]. Annu Rev Genet, 2007, 41: 121-145. DOI:10.1146/annurev.genet.41.042007.170548 |
| [23] |
Wengelnik K, Bonas U. HrpXv, an AraC-type regulator activates expression of five of the six loci in the hrp cluster of Xanthomonas campestris pv.vesicatoria[J]. J Bacteriol, 1996, 178: 3462-3469. DOI:10.1128/jb.178.12.3462-3469.1996 |
| [24] |
Tsuge S, Terashima S, Furutani A, et al. Effects on promoter activity of base substitutions in the cis-acting regulatory element of HrpXo regulons in Xanthomonas oryzae pv.oryzae[J]. J Bacteriol, 2005, 187: 2308-2314. DOI:10.1128/JB.187.7.2308-2314.2005 |
| [25] |
Furutani A, Nakayama T, Ochiai H, et al. Identification of novel HrpXo regulons preceded by two cis acting elements, a plant-inducible promoter box and a -10 box like sequence, from the genome database of Xanthomonas oryzae pv.oryzae[J]. FEMS Microbiol Lett, 2006, 259: 133-141. DOI:10.1111/fml.2006.259.issue-1 |
| [26] |
Koebnik R, Kruger A, Thieme F, et al. Specific binding of the Xanthomonas campestris pv.vesicatoria AraCtype transcriptional activator HrpX to plant-inducible promoter boxes[J]. J Bacteriol, 2006, 188: 7652-7660. DOI:10.1128/JB.00795-06 |
| [27] |
Guo W, Cai L, Zou H, et al. Ketoglutarate transport protein KgtP is secreted through the type Ⅲ secretion system and contributes to virulence in Xanthomonas oryzae pv.oryzae[J]. Appl Environ Microbiol, 2012, 78: 5672-5681. DOI:10.1128/AEM.07997-11 |
| [28] |
Doyle E, Stoddard B, Voytas D, et al. TAL effectors:highly adaptable phytobacterial virulence factors and readily engineered DNA-targeting proteins[J]. Trends Cell Biol, 2013, 23: 390-398. DOI:10.1016/j.tcb.2013.04.003 |
| [29] |
Tang J, Liu Y, Barber C, et al. Genetic and molecular analysis of a cluster of rpf genes involved in positive regulation of synthesis of extracellular enzymes and polysaccharide in Xanthomonas campestris pathovar campestris[J]. Mol Gen Genet, 1991, 226: 409-417. |
| [30] |
Dow J, Crossman L, Findlay K, et al. Biofilm dispersal in Xanthomonas campestrisis controlled by cell-cell signaling and is required for full virulence to plants[J]. Proc Natl Acad Sci USA, 2003, 100: 10995-11000. DOI:10.1073/pnas.1833360100 |
| [31] |
Dow J, Fouhy Y, Lucey J, et al. The HD-GYP domain, cyclic di-GMP signaling, and bacterial virulence to plants[J]. Mol Plant Microbe Interact, 2006, 19: 1378-1384. DOI:10.1094/MPMI-19-1378 |
| [32] |
He Y, Ng A, Xu M, et al. Xanthomonas campestris cell-cell communication involves a putative nucleotide receptor protein Clp and a hierarchical signaling network[J]. Mol Microbiol, 2007, 64: 281-292. DOI:10.1111/j.1365-2958.2007.05670.x |
| [33] |
Huang D, Tang D, Liao Q, et al. The Zur of Xanthomonas campestris is involved in hypersensitive response and positively regulates the expression of the hrp cluster via hrpX but not hrpG[J]. Mol Plant Microbe Interact, 2009, 22: 321-329. DOI:10.1094/MPMI-22-3-0321 |
| [34] |
管文静, 吴茂森, 何晨阳. 水稻白叶枯病菌核苷酸信号受体蛋白Clpxoo的分子鉴定及其功能分析[J]. 微生物学报, 2009, 49: 32-37. DOI:10.3321/j.issn:0001-6209.2009.01.007 |
| [35] |
霍欢, 孙蕾, 田芳, 等. 水稻白叶枯病菌群体感应系统对T3SS基因表达的调控作用分析[J]. 植物病理学报, 2012, 42: 620-625. DOI:10.3969/j.issn.0412-0914.2012.06.008 |
| [36] |
孙蕾, 吴茂森, 陈华民, 等. 水稻白叶枯病菌Δrpfxoo基因缺失突变体DSF信号产生和毒性表达[J]. 微生物学报, 2010, 50: 717-723. |
| [37] |
Lee S, Jeong K, Han S, et al. The Xanthomonas oryzae pv.oryzae PhoPQ two component system is required for AvrXA21 activity, hrpG expression, and virulence[J]. J Bacteriol, 2008, 190: 2183-2197. DOI:10.1128/JB.01406-07 |
| [38] |
Burdman S, Shen Y, Lee S, et al. RaxH/RaxR:a two-component regulatory system in Xanthomonas oryzae pv.oryzae required for AvrXa21 activity[J]. Mol Plant Microbe Interact, 2004, 17: 602-612. DOI:10.1094/MPMI.2004.17.6.602 |
| [39] |
Lee S, Han S, Bartley L, et al. Unique characteristics of Xanthomonas oryzae pv.oryzae AvrXa21 and implications for plant innate immunity[J]. Proc Natl Acad Sci USA, 2006, 103: 18395-18400. DOI:10.1073/pnas.0605508103 |
| [40] |
Zhang S, He Y, Xu M, et al. A putative colRXC1049-colSXC1050 two-component signal transduction system in Xanthomonas campestris positively regulates hrpC and hrpE operons and is involved in virulence, the hypersensitive response and tolerance to various stresses[J]. Res Microbiol, 2008, 159: 569-578. DOI:10.1016/j.resmic.2008.06.010 |
| [41] |
Tsuge S, Nakayama T, Terashima S, et al. Gene involved in transcriptional activation of the hrp regulatory gene hrpG in Xanthomonas oryzae pv.oryzae[J]. J Bacteriol, 2006, 188: 4158-4162. DOI:10.1128/JB.00006-06 |
| [42] |
Dorman CJ. H-NS:a universal regulator for a dynamic genome[J]. Nat Rev Microbiol, 2004, 2: 391-400. DOI:10.1038/nrmicro883 |
| [43] |
Fang FC, Rimsky S. New insights into transcriptional regulation by H-NS[J]. Curr Opin Microbiol, 2008, 11: 113-120. DOI:10.1016/j.mib.2008.02.011 |
| [44] |
Feng J, Song Z, Duan C, et al. The xrvA gene of Xanthomonas oryzae pv.oryzae, encoding an H-NS-like protein, regulates virulence in rice[J]. Microbiology, 2009, 155: 3033-3044. DOI:10.1099/mic.0.028910-0 |
| [45] |
Kametani-Ikawa Y, Tsuge S, Furutani A, et al. An H-NS-like protein involved in the negative regulation of hrp genes in Xanthomonas oryzae pv.oryzae[J]. FEMS Microbiol Lett, 2011, 319: 58-64. DOI:10.1111/fml.2011.319.issue-1 |
| [46] |
Liu Y, Long J, Shen D, et al. Xanthomonas oryzae pv.oryzae requires H-NS-family protein XrvC to regulate virulence during rice infection[J]. FEMS Microbiol Lett, 2016, 363(10). DOI:10.1093/femsle/fnw067 |
| [47] |
Chao N, Wei K, Chen Q, et al. The rsmA-like gene rsmAXcc of Xanthomonas campestris pv.campestris is involved in the control of various cellular processes, including pathogenesis[J]. Mol Plant Microbe Interact, 2008, 21: 411-423. DOI:10.1094/MPMI-21-4-0411 |
| [48] |
Zhu P, Zhao S, Tang J, Feng J. The rsmA-like gene rsmA(Xoo)of Xanthomonas oryzae pv.oryzae regulates bacterial virulence and production of diffusible signal factor[J]. Mol Plant Pathol, 2011, 12: 227-37. DOI:10.1111/mpp.2011.12.issue-3 |
| [49] |
Ikawa Y, Tsuge S. The quantitative regulation of the hrp regulator HrpX is involved in sugar-source-dependent hrp gene expression in Xanthomonas oryzae pv.oryzae[J]. FEMS Microbiol Lett, 2016, 363(10). DOI:10.1093/femsle/fnw071 |
| [50] |
Guo W, Zou L, Li Y, et al. Fructose-bisphosphate aldolase exhibits functional roles between carbon metabolism and the hrp system in rice pathogen Xanthomonas oryzae pv.oryzicola[J]. PLoS On, 2012, 7: e31855. DOI:10.1371/journal.pone.0031855 |
| [51] |
Rashid M, Ikawa Y, Tsuge S. GamR, the LysR-type galactose metabolism regulator, regulates hrp gene expression via transcriptional activation of two key hrp regulators, HrpG and HrpX, in Xanthomonas oryzae pv.oryzae[J]. Appl Environ Microbiol, 2016, 82: 3947-58. DOI:10.1128/AEM.00513-16 |
| [52] |
管文静, 吴茂森, 何晨阳. c-di-GMP信号途径对细菌致病性的调控作用[J]. 微生物学通报, 2009, 36: 427-431. |
| [53] |
薛丁榕, 田芳, 李海云, 等. 水稻白叶枯病菌第二信使c-di-GMP代谢酶基因的预测和分析[J]. 生物技术通报, 2015(11): 131-138. |
| [54] |
杨凤环, 田芳, 陈华民, 等. 病原细菌受体介导的c-di-GMP信号传导及其调控机制[J]. 植物保护, 2017, 43: 9-14. |
| [55] |
Yang F, Tian F, Sun L, et al. A novel two-component system PdeK/PdeR regulates c-di-GMP turnover and virulence of Xanthomonas oryzae pv.oryzae[J]. Mol Plant Microbe Interact, 2012, 25: 1361-1369. DOI:10.1094/MPMI-01-12-0014-R |
| [56] |
梁士敏, 杨凤环, 管文静, 等. 水稻白叶枯病菌EAL结构域蛋白VieAxoo基因缺失突变和功能分析[J]. 微生物学报, 2011, 51: 29-34. |
| [57] |
Yang F, Qian S, Tian F, et al. The GGDEF-domain protein GdpX1 attenuates motility exopolysaccharide production and virulence in Xanthomonas oryzae pv.oryzae[J]. J Appl Microbiol, 2016, 120: 1646-1657. DOI:10.1111/jam.2016.120.issue-6 |
| [58] |
李潇桐, 杨凤环, 梁士敏, 等. 水稻白叶枯病菌毒性表达的负调控因子PXO_02944的分子鉴定[J]. 中国农业科学, 2014, 47: 2563-2570. DOI:10.3864/j.issn.0578-1752.2014.13.008 |
| [59] |
Yang F, Tian F, Chen H, et al. The Xanthomonas oryzae pv.oryzae PilZ-domain proteins function differentially in cyclic di-GMP binding and regulation of virulence and motility[J]. Appl Environ Microbiol, 2015, 81: 4358-4367. DOI:10.1128/AEM.04044-14 |
| [60] |
Yang F, Tian F, Li X, et al. The degenerate EAL-GGDEF domain protein Filp functions as a cyclic di-GMP receptor and specifically interacts with the PilZ-domain protein PXO_02715 to regulate virulence in Xanthomonas oryzae pv.oryzae[J]. Mol Plant Microbe Interact, 2014, 27: 578-589. DOI:10.1094/MPMI-12-13-0371-R |