鱼类资源下滑、匮乏或者小型化是当今渔业资源面临的重大问题之一,究其原因主要有过度捕捞、水污染、栖息地退化、遗传污染、气候变化和入侵物种引进等[1-3],这些因素使得鱼类资源保护具有挑战性。水生态系统构成复杂多样,某些鱼类因个体小、数量少、善隐蔽等因素难以被捕获,且处于不同生长发育阶段形态上差异较大,极难鉴定,这使得鱼类资源调查极为困难。传统的调查主要利用拖网、围网、电捕鱼、钓鱼、水声和视觉等方法[4]来观察鱼类,不仅耗时费力、价格不菲,而且有的方法会对鱼体造成伤害[5],同时还可能会由于采样方式的缺陷导致数据不全面、不可靠。环境DNA(eDNA)是最近开发的一种高灵敏、高效率且无损伤的调查工具[6],能够为解决鱼类资源研究过程中存在的难题提供高效快捷的方法,且已经达到了成熟的科学水平[7-9]。eDNA是指从生物体生活环境中直接提取到的DNA片段总和,由不同物种的DNA混合而成,主要包含生物体细胞释放到水中的胞内遗传物质和细胞结构裂解或死亡后释放到水中的胞外DNA[10-11]。eDNA技术是指从环境样品中直接提取DNA片段,应用分子手段检测eDNA中所包含识别片段,结合目标物种或种群的特异性基因识别片段进行比对的一种手段,其结果可用于分析目标生物在生态系统中的分布特征。近几年来,eDNA技术在鱼类种类及多样性监测[12-15]、资源量估测[16-17]和种群分布[18]等方面都有一定的发展和应用。随着鱼类资源的深入研究,分子技术与传统的调查方法相结合的方式将成为未来该领域研究的一个发展趋势。本文主要对环境DNA技术在鱼类资源研究中的应用进行了综述,旨为下一步开展鱼类物种多样性、资源量估算和种群分布等研究提供参考。
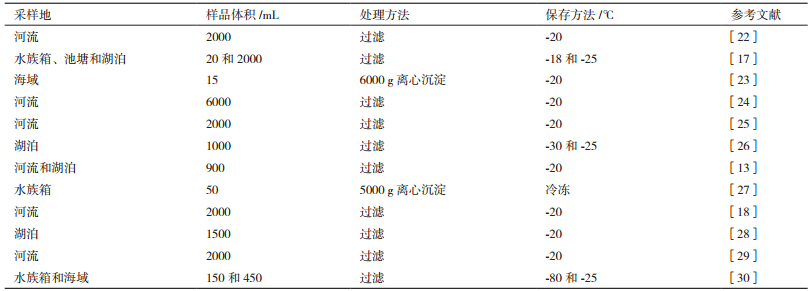
1 环境DNA技术的研究方法 1.1 样品采集依据选取的eDNA分析方法有必要选择合适的水样采集方式。一般的,使用采水器在不同深度的水位采集不同的水样,采水器等设备和储存容器应无菌处理,以避免来自除取样水体之外的其他来源的目标DNA污染样品。采集的水样体积在15 mL-10 L不等(表 1),而最常用的水样体积为1 L-2 L,且同一水体中需采集多个重复样品[19-20]。通过多次取样的方法以增加水样中的物种空间覆盖度,提高物种检出概率[10]。研究表明,水体沉积物中的eDNA比水体中的更加集中[21],有时为了提高检出概率,可选择采集水域中的底泥沉积物。
通常水体中eDNA主要有离心沉淀和真空过滤两种方法来分离(表 1)。水样采集后,应保持样品冷却[31],并尽量减少暴露于光下[32],且在24 h内进行过滤或沉淀[20],以减少DNA降解,降低物种检出率。过滤主要是通过滤膜来进行的,滤膜的孔径大小不一,有0.22 μm-1.5 μm、0.2 μm-10 μm[33]和0.45 μm-3 μm[10]不等。孔径越小,eDNA提取的量越大,但孔径小易堵塞滤膜,影响过滤速度[33]。一般使用2.5倍体积的无水乙醇或者0.6倍体积的异丙醇沉淀eDNA[34],也可通过离心的方法。eDNA可通过多种方法从过滤或沉淀物中提取,可利用氯仿[35-36]和二氧化硅萃取[37],或细胞的物理裂解[14, 22],DNA快速提取试剂盒[38-39]等提取。
1.3 eDNA分析研究表明,水样中的大部分eDNA可能是包含在线粒体或游离的细胞内的DNA,而不是游离DNA[33]。由于线粒体DNA拷贝数大、区分物种的保守序列较多,常优先于核DNA进行eDNA分析,以提高检测概率。与核DNA比较而言,线粒体DNA很大程度上避免了环境中的DNA易降解和浓度低等因素造成的不易检测问题[10]。
eDNA分析通常基于带有物种特异性遗传标记序列的线粒体DNA片段(80-250 bp)进行高通量测序[5],克隆测序分析时可以超出这个范围。eDNA用于研究鱼类资源方面,假如只调查研究某一种鱼类时,则只需针对该种设计特异性高的引物,确保目标物种的高靶特异性,没有碱基对错配,以及任何紧密相关或共存物种尽可能多的错配[24]。假如研究一鱼类群体时,则需要设计通用引物,主要包含16s、COI、Cytb1、Cytb4和D-loop等[40],使环境中鱼类群体的eDNA扩增的识别片段足够多,还需建立目标(A)与非目标(B)基因库,将扩增B超过A设定值的引物剔除,以得到具足够通用性且一定特异性的通用引物[41]。利用特异性引物进行PCR扩增,扩增的片段几十到几百bp不等(表 2),获得DNA识别片段,测序后可得到该片段的序列。PCR结果进行凝胶电泳,显示正确长度的独特PCR凝胶条带为阳性检测结果,阳性结果表明eDNA样品中包含目标物种的DNA,代表取样水域中有该鱼类生存;阴性结果说明不含有来自目标物种的DNA,代表取样水域中没有目标鱼类或者取样量不足于检测到。为了确认阳性检测结果,至少重复两次试验[25, 42]。为避免影响试验结果,应在DNA样品采集、提取和扩增的试验过程中设置阴性对照组以避免交叉污染[22, 39]。
国内外运用eDNA技术监测鱼类种类和物种多样性调查方面报道不少(表 3)。主要包含濒危种、稀有种和入侵种的监测。常见鱼类是水域中鱼类群落的组成,应用eDNA了解其资源状况,有利于保护和开发鱼类资源,有利于当地的渔业增殖放流工作,有利于水生态系统的稳定。而濒危或稀有鱼类由于自身或人为或自然等因素导致其野生种群易面临绝灭的危险。一个关键种的灭绝可能破坏水生态系统中食物链,造成水生态系统的不稳定。濒危或稀有鱼类不易捕获且生物量少,而eDNA技术可在生物量较小的情况下进行检测,对其监测具有重要意义。入侵鱼类易造成生物多样性丧失,早发现早处理是应对生物入侵的关键环节[44]。而在入侵前期,入侵种生物量较小,未形成种群,易低估入侵程度。因此,应用eDNA技术对入侵种进行早期检测,可为生物入侵预防工作提供支持。很大程度上,水体中提取到的DNA量影响着水生系统中的生物多样性检测[45]。
在渔业资源评估上,资源量基于传统方法采样很难进行准确估计的[10],而对于那些处于受威胁和濒危物种来说,准确和精确的资源量估计显得至关重要[50],这对于保护和挽救工作具有重要意义。水声学可以估算一个水域中鱼类的平均密度,但也很难得到准确和精确的数据。因此,eDNA浓度与资源量之间的关系是一个相当大的研究课题,但也存在eDNA技术对资源监测的准确性如何保证的问题。近年来研究表明,可以从eDNA在水样中的丰度来估计鱼类的相对资源量,并发现资源量与eDNA浓度之间存在正相关关系[16, 27],卢珊[51]发现eDNA与相应的水生动物间存在密切的关系,但这种关系可能不适用于所有物种和环境条件[52]。可见,模拟实验可了解某个种类的丰度与eDNA浓度的关系,从而推测其资源量与eDNA浓度的关系方程。由此,对某个自然水域中的eDNA进行采样分析,监测某个种类的eDNA浓度,据模拟实验所得的方程来推测其在某个自然水域中的资源量。由于eDNA的来源,脱落率,降解速率,转运和沉降等因素,这些因素都会影响eDNA的提取,从而不表现出这种关系[53]。也就是说,eDNA在水体中存在具有实时性,易影响与鱼类资源量的生物关系。除此之外,在样品处理期间错误引用也可能会导致数量关系的分离。运用eDNA研究鱼类生物量时要注意近缘物种干扰、鱼类的活动周期以及水流速度方向等问题。在今后的一段时间内,资源量和eDNA浓度之间的关系可能是一个热点的研究领域。
2.3 种群分布传统的研究方法很难精确的划分鱼类种群的分布范围,而eDNA技术不受条件的限制,具灵敏度高、无损伤性的特点,只需采集水体样本获得DNA即可分析鱼类的存在。运用eDNA技术来分析鱼类入侵种和濒危种种群分布的报道不少,美国密歇根湖及其周边河流中的鲢(H.molitrix)和鳙(H.nobilis)的扩散分布范围已经超出了传统调查方法研究划定的区域[22],这对入侵种的防治工作具有提示作用[25]。Takahara等[25]分析了蓝鳃太阳鱼(L.macrochirus)在日本本土及周边岛屿水域的扩散分布趋势情况。欧洲泥鳅(Misgurnus fossilis)不仅在丹麦现有分布区有分布,且在以前调查发现的分布区中也有其的分布踪迹[54]。另外,入侵种克氏原鳌虾(Procambarus clarkii)在梯田中分布情况也有报道[55]。可见,在种群分布确定方面,eDNA技术优于传统的调查方法,其发展和应用将有助于本地种的监测,也有助于对入侵种和濒危种部分范围进行监测。
2.4 食性分析与“三场一通道”监测eDNA技术可运用于鱼类的食性分析,近年来,eDNA也开始应用于动物食性的研究,但鱼类食性分析的鲜有报道。食性分析的传统方法主要有消化残留物观察法和肠道解剖法,它们都存在着局限性,依赖于观察者对肠道内未消化物和消化残留物的准确判断,对于生活在水下的微小型鱼类难以通过视觉观察来了解。eDNA技术在动植物[56-57]、浮游生物[51]、虾类[51, 55]和鱼类等[22-25]监测都有应用的报道。因此,在食性分析时,对水生植物、浮游生物及其饵料鱼类都可以应用eDNA进行监测,可作为一种便捷的检测方法;而肉食性鱼类与作为饵料鱼类可通过设计特异性引物加以区分开来。可见,可以应用eDNA的方法来监测鱼类肠道中饵料生物的种类,且具有无限潜力,再结合传统方法,或许在全面了解鱼类食性方面可取得巨大的进展。
目前,应用eDNA技术对定居性鱼类的产卵场和洄游性鱼类“三场一通道”监测工作鲜有报道,鱼类的“三场一通道”主要是指鱼类产卵场、索饵场、越冬场和洄游通道,是鱼类栖息生活生存的重要场所。渔业资源调查工作者可在鱼类种群分布探究的基础上,eDNA技术结合传统的调查方法可否更快捷有效的对定居性鱼类种群产卵场和对洄游性鱼类“三场一通道”进行监测,传统方法的耗费人力物力和时间较大,应用eDNA技术监测“三场一通道”将是今后渔业资源工作者值得探索的方向。
3 展望与基于捕获的方法相比,eDNA技术具有高灵敏、无损伤、低成本、快捷方便等特点,在鱼类种类监测、鱼类多样性监测、资源量估测和种群分布等方面已得到应用,而将来是否可应用于食性分析与“三场一通道”监测的工作中值得探讨。但目前eDNA技术还存在不足之处,如不能提供鱼类的生理状态、生长发育阶段等生物学特征以及鱼类的种群结构;不能提供鱼类目标种存在或不存在的实时数据;环境中保存的线粒体eDNA不能在将“纯种”与杂交种区分开来。虽然存在这些缺点,但将eDNA技术作为一种鱼类资源调查评估的工具未免不可,结合传统的采样方法,如电动捕捞、网捕、水声调查等,可以有效的摸清鱼类资源状况,但eDNA技术还不能完全取代经过时间验证的鱼类资源评估方法。
| [1] |
Coryd S, Stevenj C. Conservation of aquatic resources through the use of freshwater protected areas:opportunities and challenges[J]. Biodiversity & Conservation, 2007, 16(7): 2015-2029. |
| [2] |
Dudgeon D, Arthington AH, Gessner MO, et al. Freshwater biodiversity:importance, threats, status and conservation challenges[J]. Biological Reviews, 2006, 81(2): 163-182. DOI:10.1017/S1464793105006950 |
| [3] |
Revenga C, Campbell I, Abell R, et al. Prospects for monitoring freshwater ecosystems towards the 2010 targets[J]. Philosophical Transactions of the Royal Society of London, 2005, 360(1454): 397-413. DOI:10.1098/rstb.2004.1595 |
| [4] |
Order TO. Standard methods for sampling North American freshwater fishes:opportunities for international collaboration[J]. Fisheries, 2017, 42(3): 7. |
| [5] |
Bohmann K, Evans A, Gilbert MTP, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends in Ecology & Evolution, 2014, 29(6): 358-367. |
| [6] |
姜维, 赵虎, 邓捷, 等. 环境DNA分析技术—一种水生生物调查新方法[J]. 水生态学杂志, 2016, 37(5): 1-7. |
| [7] |
Evans NT, Shirey PD, Wieringa JG, et al. Comparative cost and effort of fish distribution detection via environmental DNA analysis and electrofishing[J]. Fisheries, 2017, 42(2): 90-99. DOI:10.1080/03632415.2017.1276329 |
| [8] |
Laramie MB, Pilliod DS, Goldberg CS. Characterizing the distribution of an endangered salmonid using environmental DNA analysis[J]. Biol Conserv, 2015, 183: 29-37. DOI:10.1016/j.biocon.2014.11.025 |
| [9] |
Mckelvey KS, Young MK, Knotek WL, et al. Sampling large geographic areas for rare species using environmental DNA:a study of bull trout Salvelinus confluentus occupancy in western Montana[J]. Journal of Fish Biology, 2016, 88(3): 1215-1222. DOI:10.1111/jfb.2016.88.issue-3 |
| [10] |
Rees HC, Maddison BC, Middleditch DJ, et al. The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology[J]. Journal of Applied Ecology, 2014, 51: 1450-1459. DOI:10.1111/1365-2664.12306 |
| [11] |
Taberlet P, Coissac E, Hajibabaei M, et al. Environmental DNA[J]. Molecular Ecology, 2012, 21: 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [12] |
Evans NT, Olds BP, Renshaw MA, et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding[J]. Molecular Ecology Resources, 2015, 16(1): 29-41. |
| [13] |
Keskin E. Detection of invasive freshwater fish species using environmental DNA survey[J]. Biochemical Systematics & Ecology, 2014, 56: 68-74. |
| [14] |
Thomsen PF, Kielgast J, Iversen LL, et al. Detection of a diverse marine fish fauna using environmental dNA from seawater samples[J]. PLoS One, 2012, 7: e41732. DOI:10.1371/journal.pone.0041732 |
| [15] |
Thomsen PF, Kielgast J, Iversen LL, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21: 2565. DOI:10.1111/j.1365-294X.2011.05418.x |
| [16] |
Lacoursière-Roussel A, Côté G, Leclerc V, et al. Quantifying relative fish abundance with eDNA:a promising tool for fisheries management[J]. Journal of Applied Ecology, 2016, 53(4): 1148-1157. DOI:10.1111/1365-2664.12598 |
| [17] |
Takahara T, Minamoto T, Yamanaka H, et al. Estimation of fish biomass using environmental DNA[J]. PLoS One, 2012, 7: e35868. DOI:10.1371/journal.pone.0035868 |
| [18] |
Yamanaka H, Minamoto T. The use of environmental DNA of fishes as an efficient method of determining habitat connectivity[J]. Ecological Indicators, 2016, 62(1): 147-153. |
| [19] |
Jerde CL, Chadderton WL, Mahon AR, et al. Detection of asian carp DNA as part of a Great Lakes basin-wide surveillance program[J]. Canadian Journal of Fisheries & Aquatic Sciences, 2013, 70(4): 522-526. |
| [20] |
Pilliod DS, Goldbergcaren S, Arklerobert S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples[J]. Canadian Journal of Fisheries & Aquatic Sciences, 2013, 70(8): 1123-1130. |
| [21] |
Turner CR, Uy KL, Everhart RC. Fish environmental DNA is more concentrated in aquatic sediments than surface water[J]. Biol Conserv, 2015, 183: 93-102. DOI:10.1016/j.biocon.2014.11.017 |
| [22] |
Jerde CL, Mahon AR, Chadderton WL, et al. "Sight-unseen" detection of rare aquatic species using environmental DNA[J]. Conservation Letters, 2011, 4(2): 150-157. DOI:10.1111/conl.2011.4.issue-2 |
| [23] |
Foote AD, Thomsen PF, Sveegaard S, et al. Investigating the potential use of environmental DNA(eDNA)for genetic monitoring of marine mammals[J]. PLoS One, 2012, 7(8): e41781. DOI:10.1371/journal.pone.0041781 |
| [24] |
Wilcox TM, Mckelvey KS, Young MK, et al. Robust Detection of rare species using environmental DNA:the importance of primer specificity[J]. PLoS One, 2013, 8: e59520. DOI:10.1371/journal.pone.0059520 |
| [25] |
Mahon AR, Jerde CL, Galaska M, et al. Validation of eDNA surveillance sensitivity for detection of Asian carps in controlled and field experiments[J]. PLoS One, 2013, 8(3): e58316. DOI:10.1371/journal.pone.0058316 |
| [26] |
Takahara T, Minamoto T, Doi H. Using environmental DNA to estimate the distribution of an Invasive fish species in ponds[J]. PLoS One, 2013, 8(2): e56584. DOI:10.1371/journal.pone.0056584 |
| [27] |
Klymus KE, Richter CA, Chapman DC, et al. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix[J]. Biol Conserv, 2015, 183: 77-84. DOI:10.1016/j.biocon.2014.11.020 |
| [28] |
Keskin E, Unal EM, Atar HH. Detection of rare and invasive freshwater fish species using eDNA pyrosequencing:Lake Iznik ichthyofauna revised[J]. Biochemical Systematics & Ecology, 2016, 67: 29-36. |
| [29] |
徐念, 常剑波. 长江中下游干流环境DNA样本鱼类物种检测的初步研究[J]. 水生态学杂志, 2016, 37(5): 49-55. |
| [30] |
赵梦迪.利用环境DNA分析冬季中国东黄海水域的鱼类多样性[D].上海: 上海海洋大学, 2017.
|
| [31] |
Takahara T, Minamoto T, Doi H. Effects of sample processing on the detection rate of environmental DNA from the Common Carp(Cyprinus carpio)[J]. Biol Conserv, 2015, 183: 64-69. DOI:10.1016/j.biocon.2014.11.014 |
| [32] |
Strickler KM, Fremier AK, Goldberg CS. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms[J]. Biol Conserv, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [33] |
Turner CR, Barnes MA, Xu CCY, et al. Particle size distribution and optimal capture of aqueous macrobial eDNA[J]. Methods in Ecology & Evolution, 2014, 5(7): 676-684. |
| [34] |
范圣第, 权春善. 环境DNA的提取和纯化方法的研究[J]. 大连民族大学学报, 2005, 7(5): 1-4. DOI:10.3969/j.issn.1009-315X.2005.05.001 |
| [35] |
Kristy D, Florian A. Transport distance of invertebrate enviro-nmental DNA in a Natural River[J]. PLoS One, 2014, 9(2): e88786. DOI:10.1371/journal.pone.0088786 |
| [36] |
Renshaw MA, Olds BP, Jerde CL, et al. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction[J]. Molecular Ecology Resources, 2015, 15(1): 168-176. DOI:10.1111/men.2014.15.issue-1 |
| [37] |
Goldberg CS, Pilliod DS, Arkle RS, et al. Molecular detection of vertebrates in stream water:ademonstration using rocky mountain tailed frogs and idaho giant salamanders[J]. PLoS One, 2011, 6(7): e22746. DOI:10.1371/journal.pone.0022746 |
| [38] |
Collins RA, Armstrong KF, Holyoake AJ, et al. Something in the water:biosecurity monitoring of ornamental fish imports using environmental DNA[J]. Biological Invasions, 2013, 15(6): 1209-1215. DOI:10.1007/s10530-012-0376-9 |
| [39] |
Olson ZH, Briggler JT, Williams RN. An eDNA approach to detect eastern hellbenders(Cryptobranchus a.alleganiensis)using samples of water[J]. Wildlife Research, 2012, 39(7): 629. DOI:10.1071/WR12114 |
| [40] |
刘军, 赵良杰, 凡迎春, 等. 鱼类环境DNA研究中通用引物的筛选验证[J]. 淡水渔业, 2016, 46(1): 9-17. DOI:10.3969/j.issn.1000-6907.2016.01.002 |
| [41] |
马鸿娟, Stewart K, 马利民, 等. 环境DNA及其在水生生态系统保护中的应用[J]. 生态学杂志, 2016, 35(2): 516-523. |
| [42] |
Piaggio AJ, Engeman RM, Hopken MW, et al. Detecting an elusive invasive species:a diagnostic PCR to detect Burmese python in Florida waters and an assessment of persistence of environmental DNA[J]. Molecular Ecology Resources, 2014, 14(2): 374-380. DOI:10.1111/1755-0998.12180 |
| [43] |
Minamoto T, Yamanaka H, Takahara T, et al. Surveillance of fish species composition using environmental DNA[J]. Limnology, 2012, 13(2): 193-197. DOI:10.1007/s10201-011-0362-4 |
| [44] |
Pyšek P, Richardson DM. Invasive species, environmental change and management, and health[J]. Annual Review of Environment & Resources, 2010, 35(1): 25-46. |
| [45] |
Deiner K, Walser JC, Mächler E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J]. Biol Conserv, 2015, 183(9): 53-63. |
| [46] |
Eva B, Harmony P, Thomas G, et al. Trails of river monsters:detecting critically endangered mekong giant catfish pangasianodon gigas using environmental DNA[J]. Global Ecology & Conservation, 2016, 7(C): 148-156. |
| [47] |
Song JW, Small MJ, Casman EA. Making sense of the noise:The effect of hydrology on silver carp eDNA detection in the Chicago area waterway system[J]. Science of the Total Environment, 2017, 15: 605-606, 713-720. |
| [48] |
Karahan A, Douek J, Paz G, et al. Employing DNA barcoding as taxonomy and conservation tools for fish species censuses at the southeastern Mediterranean, a hot-spot area for biological invasion[J]. Journal for Nature Conservation, 2017, 36: 1-9. DOI:10.1016/j.jnc.2017.01.004 |
| [49] |
Yamamoto S, Masuda R, Sato Y, et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea[J]. Scientific Reports, 2017, 7: 40368. DOI:10.1038/srep40368 |
| [50] |
Briers R. Ecology:from individuals to ecosystems[J]. Freshwater Biology, 2006, 51(9): 1787-1788. DOI:10.1111/fwb.2006.51.issue-9 |
| [51] |
卢珊.常见水生动物与其环境DNA的定性与定量关系[D].南京: 南京师范大学, 2015.
|
| [52] |
Spear SF, Groves JD, Williams LA, et al. Using environmental DNA methods to improve detectability in a hellbender(Cryptobranchus alleganiensis)monitoring program[J]. Biol Conserv, 2015, 183: 38-45. DOI:10.1016/j.biocon.2014.11.016 |
| [53] |
Barnes MA, Turner CR. The ecology of environmental DNA and implications for conservation genetics[J]. Conservation Genetics, 2016, 17(1): 1-17. |
| [54] |
Sigsgaard EE, Carl H, Møller PR, et al. Monitoring the near-extinct European weather loach in Denmark based on environmental DNA from water samples[J]. Biol Conserv, 2015, 183: 46-52. DOI:10.1016/j.biocon.2014.11.023 |
| [55] |
马竹欣.利用环境DNA技术调查入侵种克氏原螯虾在元阳梯田的分布[D].昆明: 云南大学, 2016.
|
| [56] |
徐浩, 罗茜, 李云, 等. 环境DNA研究技术及其在生态学领域的应用[J]. 生物技术通报, 2014(10): 49-55. |
| [57] |
于水强, 王文娟, Li BL, 等. 环境DNA技术在地下生态学中的应用[J]. 生态学报, 2015, 35(15): 4968-4976. |