2. 吉林正方农牧有限公司,梅河口 135000
2. Jilin Zhengfang Agriculture and Animal Husbandry Co., Ltd., Meihekou 135000
在畜禽生产上,脂质代谢一直备受关注,肝脏作为机体脂类代谢的最大器官,探究其脂质代谢机制一直为研究热点,在水禽通过超饲生产肥肝等研究中更是重中之重。在肝脏脂质代谢的调控网络中,过氧化物酶体增殖物激活受体(Peroxisome proliferator-activated receptors,PPARs)是其中一个关键枢纽[1]。PPARs是一类需要配体激活的核转录因子,属于核受体超家族(Nuclear-hormone receptor superfamily,NR),目前已知有PPARα、PPARβ/δ和PPARγ三种亚型[2-4],在动物机体能量代谢过程中均发挥重要作用[5-6]。PPARα自19世纪末被发现以来,其在脂类代谢中的作用机制一直备受关注,PPARα基因在动物肝脏中广泛控制着与游离脂肪酸转运和β-氧化有关联的蛋白质的表达,在脂类代谢和维持葡萄糖稳态方面具有重要意义[7]。
1 PPARα结构特点及作用机理 1.1 PPARα基因和蛋白的结构特点不同物种的PPARα基因结构有明显差异。鸭的PPARα基因cDNA全长1 430 bp,最长开放阅读框为1 407 bp,可编码468个氨基酸[8]。山羊PPARα基因CDS区全长1 413 bp,结构稳定,共编码470个氨基酸[9]。猪PPARα基因全长1 488 bp,可编码468个氨基酸[10]。藏鸡PPARα基因序列1 430 bp,开放阅读框1 404 bp,也可编码468个氨基酸[11]。而人类PPARα(NR1C1)基因位于第22号染色体22q12-q13.1位置,跨度大约93.2 kb碱基,同山羊、蔵鸡等一样,人类PPARα基因也可编码468个氨基酸[12]。小鼠PPARα基因位于15E2染色体,也可编码包括468个氨基酸的蛋白[2],其PPARα基因转录的mRNA均有8个外显子编码,包括外显子1,2和部分外显子3编码的5'非编码区,外显子3其余部分和外显子4-8编码的PPARα编码区[13],外显子8最后232 bp碱基编码PPARα基因的3'端非编码区[14]。
PPARα蛋白结构特点同NR其他成员相似,由A/B、C、D和E/F几个结构域组成[15],如图 1所示。A/B域位于PPARα蛋白N末端,该区域包含激活功能区,其转录因子活性低且不受配体约束,可通过磷酸化作用及与其他结构域互作调节PPARα功能,其基因转录特异性由受体中A/B结构域的功能特异性序列决定[16]。C结构域为DNA结合区(DNA-binding domain,DBD),之后为柔性铰链区(Hinge)即结构域D,其连接着DBD区和配体结合区E(Ligand-binding domain,LBD)。D结构域上的柔性铰链区可识别辅抑制蛋白即阻遏蛋白上的阻遏功能域,并与其结合,在辅抑制区与辅激活区有部分重叠,能够使PPARα、DNA、配体及蛋白质相互结合[17]。
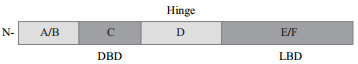
|
| 图 1 PPARα蛋白结构示意图 |
鸭PPARα蛋白中α-螺旋较多,该蛋白质含有1段短的核定位信号序列KKNRNKC,在引导蛋白质进入细胞核过程中发挥作用,同时还有2个锌指结构组成的DBD区及1段LBD区。山羊PPARα蛋白是一种结构较为稳定的带负电的亲水性蛋白,以α-螺旋和无规则卷曲为主,无信号肽和跨膜蛋白,属于膜内蛋白,其蛋白序列中总共有49个磷酸化位点,9个糖基化位点。蛋白结构高度保守,保守结构域中含有明显的DBD区域和LBD区域。三级结构预测其蛋白主要为通过长链卷曲连接的2个结构区域,且均以helix-螺旋结构为主。藏鸡PPARα蛋白为不稳定酸性蛋白,存在27个磷酸化位点和4个糖基化位点,无信号肽和跨膜螺旋结构,其二级结构预测中α-螺旋占37.61%,β-折叠占16.67%,无规则卷曲比例占45.73%。
1.2 PPARα配体分类及其表达模式PPARα作为细胞核受体,能够与特异性配体结合并被激活,进而启动PPARα信号通路,发挥其信号转导途径的调控功能。其激动剂可以分为外源性配体和内源性配体两大类。外源性配体是合成化合物,又称过氧化物酶体增殖子(Peroxisome proliferators,PPs),内源性配体是生物分子类,为机体本身大分子物质或代谢生成的小分子中间产物,两者在PPARα表达中均发挥重要作用。
1.2.1 PPARα外源性配体PPs包括安妥明、非诺贝特、纳非烯、环丙贝特、Wy-14 643(哌啶酸)等降血脂药物[18],以及增塑剂邻酞酸二辛酯(Diethylhexyl phthalate,DEHP)、己二酸二辛酯[Di(2-ethylhexyl)adipate,DEHA]等工业试剂,另外某些食品调味剂、除草剂、杀虫剂和白三烯D4受体拮抗剂等也可作为PPARα配体[19]。Wy-14 643作为PPARα的外源性配体,具有良好的降血脂作用,有研究表明,经Wy-14 643处理过的鼠,其肝脏内脂质代谢重要途径β-氧化及ω-氧化的相关氧化代谢酶活性升高,过氧化物酶体数量增多体积增大,肝脏内脂肪酸氧化活动增强,而过氧化物酶体增殖及β-氧化代谢酶活性升高,又可引发DNA损伤,进而改变细胞增殖周期,诱导细胞凋亡[20]。其他外源性配体虽然结构各异,但其对鼠肝脏脂代谢作用机制跟Wy-14 643基本相同。
1.2.2 PPARα内源性配体充当PPARα配体的内源性分子包括脂肪酸及脂肪酸衍生物。PPARα大多数内源性配体已通过体外方法鉴定,膳食饱和脂肪酸(Saturated fatty acids,SFA)和不饱和脂肪酸(Unsaturated fatty acids,UFA)都可作PPARα直接配体,动物机体内脂肪酸分解和新脂肪酸合成过程中生成的某些中间产物可作为PPARα最终配体[21],此外,机体内某些酶如8-、12-、15-和5-脂肪氧合酶(Lipoxygenases,LO),环氧合酶(Cyclooxygenases,COX),细胞色素P450(Cytochrome P450,CYP450)以脂肪酸为底物生成的物质也可作为PPARα配体[22-24]。尽管动物机体内有许多合成配体能有效激活PPARα,但PPARα几乎主要起脂质感应作用以调节机体能量燃烧[25-26]。有研究表明,PPARα能感知某些内源性脂质代谢中间产物作为配体,并通过诱导下游脂质代谢相关基因参与其运作,脂肪酰基辅酶A氧化酶1(Acyl-CoA oxidase1,Acox1)是脂肪酸β-氧化体系的第一个酶,也是限速酶,Fransen和Yeldandi等[27-28]通过敲除小鼠体内Acox1基因探究激活PPARα对肝脏代谢作用的影响。结果表明,小鼠缺乏Acox1后肝细胞内极长链脂肪酸在积累,且极长链脂酰辅酶A因缺乏Acox1不能进入脂肪酸氧化途径,其未代谢底物便可作为内源性配体激活PPARα。此外,通过小鼠基因敲除实验证明,烯酰辅酶A水合酶/ L-3-羟酰基辅酶A脱氢酶(Enoyl-CoA hydratase/L-3-hydroxyacyl-CoA dehydrogenase,L-PBE/MFP1),D-3-羟基酰基辅酶A脱水酶/ D-3-羟基酰基辅酶A脱氢酶(D-3-hydroxyacyl-CoA dehydratase/D-3-hydroxyacyl-CoA dehydrogenase,D-PBE/MFP2)和甾醇载体蛋白x(Sterol carrier protein x,SCPx)是降解PPARα内源性配体所必须的[29-33],而其他如脂肪酸合成酶(Fatty acid synthase,FAS),脂肪乙酰辅酶A合成酶(Fatty acyl-CoA synthetase,FACS)和某些LO是生成PPARα配体所必须的[34-37]。
1.2.3 PPARα表达模式同NR家族许多非类固醇成员一样,PPARα可与另一种核受体类视黄醇X受体(Retinoid X receptor,RXR)组成异源二聚体[38]。如图 2,PPARα/RXR异二聚体可与靶基因启动子上游的PPRE反应元件(PPRE:AGGTCA N AGGTCA,N可是任意一个核苷酸)结合,调节其基因表达[39]。PPARα/RXR异二聚体不受PPARα配体约束,可募集共抑制因子复合物如核共抑制剂(N-CoR)并抑制靶基因转录[40]。在细胞质中被配体激活后,共抑制因子复合物从PPARα/RXR异二聚体释放,而共激活剂复合物如类固醇受体共激活因子-1(Steroid receptor coactivator-1,SRC-1)将被募集到靶基因启动子区以启动转录[41]。
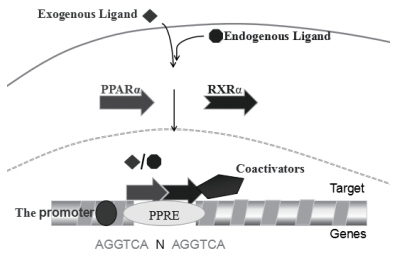
|
| 图 2 PPARα表达模式示意图 |
肝脏作为机体最大的能量代谢器官,可以协调脂肪酸和葡萄糖代谢,是全身能量稳态的中心参与者。肝脏内脂肪代谢主要包括脂肪生成、脂肪酸氧化代谢和脂质分泌3个方面[42]。核受体PPARs家族的3个亚型虽已被证明均可参与肝脏的脂质代谢过程,但PPARγ主要调节肝脏脂质合成,而PPARα主要功能为调节肝脏脂肪酸氧化代谢和能量消耗[43-45]。在动物肝脏内,PPARα主要通过被PPs、SFA、UFA及其衍生物即不同特异性配体激活,从而介导其靶基因调节脂肪酸合成代谢[46-47],对动物肝脏脂肪代谢乃至整个机体脂肪酸稳态具有重要意义。
2.1 PPARα基因影响肝脏脂肪合成肝脏内的脂肪生成主要包括新生脂肪酸合成和将新生脂肪酸转化为甘油三酯两个过程。在肝脏内,脂肪生成受转录因子固醇元件结合蛋白(Sterol regulatory element-binding protein,SREBP-1c),碳水化合物反应元件结合蛋白(Carbohydrate response-element binding protein,ChREBP)和PPARγ共同调控[48-51]。其中SREBP-1c可调节糖酵解和脂质生成基因表达,如硬脂酰辅酶A去饱和酶(Stearoyl CoA desaturase,Scd-1)和FAS,进而调控肝脏内脂肪生成过程[52]。
研究表明,PPARα可通过调节初级转录因子SREBP-1c和肝X受体α(Liver X receptor α,LXRα)来增加Scd-1和其他脂肪生成基因的转录从而影响脂肪生成。通过敲除小鼠体内PPARα基因发现,LXRα非甾体配体合成可有效诱导脂肪生成基因,证明PPARα可参与内源性LXRα配体生成[52]。PPARα在肝脏合成脂肪过程中的作用虽然不大,但其转录因子在脂肪酸氧化代谢过程中的调控作用似乎自相矛盾,推测PPARα参与脂肪生成可能是处于某种故障-安全补偿机制。
2.2 PPARα基因调控肝脏脂肪酸氧化代谢肝脏在脂肪酸氧化中起重要作用,其脂肪酸氧化过程受PPARα调控[53]。肝脏内脂肪酸氧化过程主要发生3个亚细胞器中,大部分β-氧化在线粒体和过氧化物酶体内进行,而由CYP4A催化的ω-氧化发生在内质网[54]。这三条脂肪酸氧化代谢通路中的一些关键酶均具有PPRE元件,且主要受PPARα调控。另外,PPARβ/δ也可以参与调控一些酶活性[55]。
线粒体β-氧化主要参与短链(< C8),中链(C8-C12)和长链(C12-C20)脂肪酸的氧化过程,可使脂肪酸转变成酮体进而作为肝外组织的能量氧化底物,也可在饥饿期间进入三羧酸循环以进一步氧化成水和二氧化碳,是脂肪酸生成能量的主要来源[53]。脂肪酸在氧化之前需转化为酰基辅酶A,线粒体β-氧化的第一步是脂肪酰基辅酶A酯通过直链酰基辅酶A脱氢酶家族进行α-β-脱氢;第二、第三和第四步主要有2-烯酰辅酶A水合酶、3-羟酰辅酶A脱氢酶和3-酮酰辅酶A硫解酶(Peroxisomal thiolase,PTL)活性的异源三聚体蛋白参与[25]。PPARα可调节长链酰基辅酶A合成酶(Long chainacyl-CoA syn-thetases,ACSL)和肉毒碱棕榈酰转移酶1(Carnitine palmitoyltransferase-1,CPT1),对于产生脂肪酰基辅酶A和促进脂肪酰肉毒碱进入线粒体至关重要[56]。此外,PPARα基因作为核受体,可通过脂质家族成员Lipin1的编码转录激活,并通过与Lipin1和PPARγ辅助激活因子-1α(PGC-1α)的直接协同相互作用激活参与线粒体脂肪酸氧化代谢的许多基因的表达[57]。PGC-1α-PPARα回路可在Lipin-1诱导型共激活因子的作用下,增加参与脂肪酸β-氧化,TCA循环和线粒体呼吸链的基因的表达[58]。
过氧化物酶体β-氧化是流线型朝向代谢,可参与极长链脂肪酸(> C20),2-甲基支链脂肪酸,前列腺素类化合物,二元羧酸和C27胆汁酸的中间体二羟基甾醇酸和三羟基甾醇酸的氧化代谢[59-60]。过氧化物酶体β-氧化的底物不够丰富且毒性相对较大,并且它们不能被线粒体β-氧化系统处理,然而,过氧化物酶体β-氧化可缩短极长链脂肪酸链长,链缩短的酰基辅酶A可被分流到线粒体完成β-氧化。过氧化物酶体β-氧化有诱导型和非诱导型两种,已有研究证明过氧化物酶体增殖物在过氧化物酶体系β-氧化系统中的协同诱导作用受PPARα调控[61]。在PPARα调控诱导型过氧化物酶体β-氧化途径中,ACOX1将极长链脂肪酰基辅酶A分解为相应的反式-2-烯酰辅酶A为第一反应、第二反应和第三反应中,L- PBE / MFP1经过水合脱氢反应生成酮脂酰辅酶A,再由PTL转化为乙酰辅酶A,成为比原始分子短两个碳原子的酰基辅酶A。缩短的酰基辅酶A重新进入β-氧化循环,该过程可重复约5个循环,能去除10个碳原子,最后缩短至适当长度的酰基辅酶A可转运至线粒体完成β-氧化[62]。在该氧化途径中,ACOX1、L-PBE / MFP1和PTL三种基因都受到PPARα严格调控[63]。在非诱导过氧化物酶体β-氧化系统中,2-甲基-支链脂肪酰辅酶A为第一步反应的底物,可在非诱导型ACOX2作用下生成支链-脂酰辅酶A,支链-脂酰辅酶A通过D-PBE / MFP2转化为3-酮酰辅酶A,最后经具有硫解酶活性的SCPx分解[62]。
微粒体ω-氧化由PPARα调控的CYP4A酶进行调节。SFA、UFA在内质网中经ω-羟基化作用可生成ω-羟基脂肪酸,再经脱氢后可在胞质溶胶中产生有剧毒的二羧酸,二羧酸被转化成二羧基辅酶A并进入过氧化物酶体诱导型β-氧化系统进一步代谢[64]。ω-氧化生成的二羧酸可以作为过氧化物酶体β-氧化体系的底物进行反应,也可以作为PPARα的配体,诱导PPARα活化进而调控肝脏内脂肪酸的3种主要氧化途径。
3 PPARα在畜禽方面的研究进展有关PPARs早期研究便证明其3种亚型主要作用于动物机体能量代谢方面,因此PPARα基因在畜牧学上现阶段的研究主要集中于PPARα对畜禽能量代谢调控机理的探索,不同畜禽品种PPARα基因结构及分子特性的分析以及PPARα基因与畜禽部分经济性状相关性的探究。
3.1 对PPARα调控畜禽能量代谢机理的探索近年来研究发现,PPARα的mRNA表达与畜禽动物能量代谢相关,可一定程度上调节畜禽机体的脂肪酸代谢及脂肪沉积等活动。陈兴勇等[64]发现PPARα于皖西白鹅育肥20 d高表达,育肥30 d低表达(P < 0.000 1),且其表达量上升伴随UFA/SFA含量上升,同时SFA含量下降,推测皖西白鹅PPARα基因的相对表达量与脂肪酸组成显著相关,可作为脂肪酸性状选育的候选基因[26]。林婄婄等[65]通过研究广灵大尾羊和小尾寒羊两种具有显著尾型差异的绵羊发现,PPARα和PPARγ基因在不同月龄个体的几种脂肪组织中mRNA均有表达,且浅层脂肪组织的表达量低于深层脂肪组织,二者在调节脂肪代谢方面的功能相反,但是存在协同作用。罗锦标等[66]研究发现在填饲前后朗德鹅的11种组织中,PPARα在除腹脂外其他10种组织中都有表达,较高表达于肝脏、肾脏等组织,填饲后发现其在肾脏中依然高表达,与PPARγ基因表达情况对比,证明PPARα与PPARγ表达模式并不一致,即PPARs亚型在不同组织的脂肪代谢调控中具有不同功能。贺綦等研究表明PPARα基因可显著促进鸡L-FABP基因的表达,进而调控脂肪酸的代谢活动[67]。徐凯等[68]通过检测在马身猪和大白猪不同月龄个体的肝脏内PPARα基因mRNA的表达量发现,这两种不同脂肪型猪肝脏组织PPARα基因的mRNA呈规律性变化且存在差异,说明猪PPARα可能是肝脏脂质代谢的调控因子,可进一步调控猪脂肪沉积。饶辽源等检测到山羊PPARα基因在肾脏和肝脏中表达量较高,在心脏中中度表达,在肺脏和脾脏中相对低表达,说明PPARα基因可能与体内脂肪氧化、脂质代谢和抗氧化应激等调控功能有关[9]。
3.2 对畜禽PPARα基因结构及分子特性的分析有关畜禽PPARα基因结构及分子特性的分析研究表明,不同物种间PPARα基因结构存在差异,其蛋白结构也有所不同。马云等[8]证明鸭PPARα基因核苷酸序列与所编码氨基酸序列与鸡、胸草雀、人、牛、家马等均具有同源性,且其进化关系与鸡最为密切,亲缘关系最近。李彦莹等[69]扩增出鸭PPARα启动子序列,其长2 526 bp,存在典型的CAAT-box、TATA-box顺式作用元件,预测存在特异性蛋白1(Specificity protein 1,Sp1)、核因子κB(Nuclear factor kappa B,NF-κB)和CCAAT增强子结合蛋白(CCAAT/enhancer bindingprotein alpha,C/EBP-α)转录因子结合位点。饶辽源等[9]扩增并克隆山羊PPARα基因CDS编码区,其氨基酸序列与绵羊和牛同源性最高。冯秉仁等[10]发现猪PPARα基因与猫、犬亲缘关系最近,可编码468个氨基酸,平均分子质量为52.17 kD,为不稳定的亲水蛋白,该蛋白不存在信号肽和跨膜结构域,包含9个潜在的O-糖基化位点和28个磷酸化位点二级结构由α螺旋、β转角、延伸链和随机卷曲组成,具有Zn-C4和HOLI两个功能结构域,系统进化分析显示。
3.3 与畜禽经济性状相关性的探究在探究PPARα与畜禽部分经济性状相关性方面,大多为PPARα基因多态性与畜禽的生长性状、脂肪性状、肉质性状、屠宰性状等经济性状的关联研究。武子寅等[70]检测鸡PPARα基因编码区SNPs发现,鸡PPARα基因编码区630 bp处有一个C→T突变,形成CC、CT和TT三种基因型,且试验鸡群不同基因型个体间在失水率、系水力及肌内脂肪含量等肉质性状上均出现显著差异(P < 0.05),证明PPARα基因可能参与鸡的糖脂代谢和能量代谢,可筛选为影响鸡肉质性状候选基因进行进一步研究。方文良等[71]通过扩增牛PPARα基因内含子3片段检测出SNP位点44087(G/A),并发现中国荷斯坦奶牛PPARα基因44 087(G/A)位点普遍表现为GG基因型频率最高,且GA基因型是优良基因型,其个体的体细胞评分(SCS)、乳蛋白率、耐热指标等均优于GG基因型个体,因此44 087(G/A)位点GA基因型可作为中国荷斯坦奶牛的人工选择依据,以降低奶牛热应激危害,提高牛奶品质和产量。李芬等[72]以中国地方黄牛为对象研究发现,试验牛PPARα基因第7外显子164 bp处存在SNP(C/T),且该位点CC和CT两种基因型个体之间在体斜长指标上有显著差异(P < 0.05),表明该位点可能作为黄牛生长性状辅助选择指标之一。田亚东等[73]以安卡×固始鸡F2资源群为试验材料发现,PPARα基因外显子存在SNP位点,且其多态性显著影响鸡腹脂重和腹脂率(P < 0.05),可以作为分子标记用于鸡脂肪性状的分子标记辅助选择。
4 小结随着转录组、代谢组及蛋白组多组学技术水平的提高和生物信息学大数据的发展,传统的畜牧研究已经逐渐深化到机体代谢过程中基因调控网络以及分子互作影响,在畜禽脂质代谢方面的研究也借助高通量测序和多组学结合分析等手段从原始的种间差异研究过度到个体间基因调控的差异,并逐渐完善畜禽脂代谢过程各关键基因和分子的互作机制。PPARα基因作为肝脏脂质代谢调控的关键枢纽,在畜牧研究上进一步探究其基因功能以及完善其信号网络,鉴定信号转导过程所涉及的转录因子、靶基因、相关配体及辅佐因子的种类及作用,以及明确相关通路互作机制等仍需要进一步努力探究。研究PPARα基因及其信号通路调控畜禽肝脂代谢机制无疑可促进畜牧生产上与脂代谢相关经济性状的选择,在畜牧生产应用方面有重要意义。
| [1] |
Grygiel-Górniak B. Peroxisome proliferator-activated receptors and their ligands:nutritional and clinical implications-a review[J]. Nutrition Journal, 2014, 13(1): 17. |
| [2] |
Issemann I, Green S. Activation of a member of the steroid hormone receptor superfamily by peroxisome proliferators[J]. Nature, 1990, 347(6294): 645-650. DOI:10.1038/347645a0 |
| [3] |
Bookout AL, Jeong Y, Downes M, et al. Anatomical profiling of nuclear receptor expression reveals a hierarchical transcriptional network[J]. Cell, 2006, 126(4): 789-799. DOI:10.1016/j.cell.2006.06.049 |
| [4] |
Wahli W, Michalik L. PPARs at the crossroads of lipid signaling and inflammation[J]. Trends in Endocrinology & Metabolism Tem, 2012, 23(7): 351-363. |
| [5] |
Sasaki T, Nakata R, Inoue H, et al. Role of AMPK and PPARγ1 in exercise-induced lipoprotein lipase in skeletal muscle[J]. American Journal of Physiology Endocrinology & Metabolism, 2014, 306(9): E1085-E1092. |
| [6] |
Lamichane S, Dahal BL, Kwon SM. Pivotal roles of peroxisome proliferator-activated receptors(PPARs)and their signal cascade for cellular and whole-body energy homeostasis[J]. International Journal of Molecular Sciences, 2018, 19(4): E949. DOI:10.3390/ijms19040949 |
| [7] |
Rajamani K, Colman PG, Li LP, et al. Effect of fenofibrate on amputation events in people with type 2 diabetes mellitus(FIELD study):a prespecified analysis of a randomised controlled trial[J]. Lancet, 2009, 373(9677): 1780-1788. DOI:10.1016/S0140-6736(09)60698-X |
| [8] |
马云, 王云云, 张晓婷, 等. 鸭PPARα基因结构及功能的生物信息学分析[J]. 浙江大学学报:农业与生命科学版, 2011, 37(4): 371-379. |
| [9] |
饶辽源, 周靖宣, 王林杰, 等. 山羊PPARα基因克隆、分子特性及组织差异表达分析[J]. 中国畜牧兽医, 2018, 45(3): 559-570. |
| [10] |
冯秉仁, 徐凯, 李步高, 等. 猪PPARα蛋白的生物信息学分析[J]. 当代畜牧, 2014(29): 93-96. |
| [11] |
林森, 徐亚欧, 林亚秋, 等. 藏鸡PPARα基因克隆与生物信息学分析[J]. 西南民族大学学报:自然科学版, 2015, 41(6): 661-666. |
| [12] |
Kersten S, Stienstra R. The role and regulation of the peroxisome proliferator activated receptor alpha in human liver[J]. Biochimie, 2017, 136: 75-84. DOI:10.1016/j.biochi.2016.12.019 |
| [13] |
Desvergne B, Wahli W. Peroxisome proliferator-activated receptors:nuclear control of metabolism[J]. Endocrine Reviews, 1999, 20(5): 649-688. |
| [14] |
Vohl MC, Lepage P, Gaudet D, et al. Molecular scanning of the human PPARα gene:association of the L162V mutation with hyperapobetalipoproteinemia[J]. Journal of Lipid Research, 2000, 41(6): 945-952. |
| [15] |
Pyper SR, Viswakarma N, Yu S, et al. PPARα:energy combustion, hypolipidemia, inflammation and cancer[J]. Nuclear Receptor Signaling, 2010, 8: e002. |
| [16] |
Hummasti S, Tontonoz P. The peroxisome proliferator-activated receptor N-terminal domain controls isotype-selective gene expression and adipogenesis[J]. Molecular Endocrinology, 2006, 20(6): 1261-1275. DOI:10.1210/me.2006-0025 |
| [17] |
Liu MH, Li J, Shen P, et al. A natural polymorphism in peroxisome proliferator-activated receptor-alpha hinge region attenuates transcription due to defective release of nuclear receptor corepressor from chromatin[J]. Molecular Endocrinology, 2008, 22(5): 1078-1092. DOI:10.1210/me.2007-0547 |
| [18] |
Chandran K, Goswami S, Sharmawalia N. Implications of a peroxisome proliferator-activated receptor alpha(PPARα)ligand clofibrate in breast cancer[J]. Oncotarget, 2015, 7(13): 15577-15599. |
| [19] |
Kusu R, Oishi A, Kakizawa K, et al. Effects of phthalate ester derivatives including oxidized metabolites on coactivator recruiting by PPARalpha and PPARgamma[J]. Toxicology in Vitro, 2008, 22(6): 1534-1538. DOI:10.1016/j.tiv.2008.05.010 |
| [20] |
Kim T, Wahyudi LD, Gonzalez FJ, et al. Nuclear receptor PPARα agonist Wy-14, 643 ameliorates hepatic cell death in hepatic IKKβ-deficient mice[J]. Biomolecules & Therapeutics, 2017, 25(5): 504-510. |
| [21] |
Georgiadi A, Kersten S. Mechanisms of gene regulation by fatty acids[J]. Advances in Nutrition, 2012, 3(2): 127-134. DOI:10.3945/an.111.001602 |
| [22] |
Chakravarthy MV, Lodhi IJ, Yin L, et al. Identification of a physiologically relevant endogenous ligand for PPARalpha in liver[J]. Cell, 2009, 138(3): 476-488. DOI:10.1016/j.cell.2009.05.036 |
| [23] |
Crisafulli C, Cuzzocrea S. The role of endogenous and exogenous ligands for the peroxisome proliferator-activated receptor alpha(PPAR-alpha)in the regulation of inflammation in macrophages[J]. Shock, 2009, 32(1): 62-73. DOI:10.1097/SHK.0b013e31818bbad6 |
| [24] |
Ng VY, HuangY, Reddy LM, et al. Cytochrome P450 eicosanoids are activators of peroxisome proliferator-activated eceptor α[J]. rug Metabolism & Disposition, 2007, 35(7): 1126-1134. |
| [25] |
Yan Y, Wang ZB, Tang CK. PPARs mediate the regulation of energy metabolism by long-chain fatty acids[J]. Progress in Physiological Sciences, 2016, 47(1): 1-6. |
| [26] |
Reddy JK, Hashimoto T. Peroxisomal beta-oxidation and peroxisome proliferator-activated receptor alpha:an adaptive metabolic system[J]. Annual Review of Nutrition, 2001, 21(21): 193-230. |
| [27] |
Fransen M, Nordgren M, Wang B, et al.Aging, age-related diseases and peroxisomes[M]// Peroxisomes and their Key Role in Cellular Signaling and Metabolism.Springer Netherlands, 2013: 45-65.
|
| [28] |
Yeldandi AV, Rao MS, Reddy JK. Hydrogen peroxide generation in peroxisome proliferator-induced oncogenesis[J]. Mutation Research/fundamental & Molecular Mechanisms of Mutagenesis, 2000, 448(2): 159-177. |
| [29] |
Baes M, Van Veldhoven PP. Hepatic dysfunction in peroxisomal disorders[J]. Biochim Biophys Acta, 2016, 1863(5): 956-970. DOI:10.1016/j.bbamcr.2015.09.035 |
| [30] |
Jia Y, Qi C, Zhang Z, et al. Overexpression of peroxisome proliferator-activated receptor-alpha(PPARalpha)-regulated genes in liver in the absence of peroxisome proliferation in mice deficient in both L-and D-forms of enoyl-CoA hydratase/dehydrogenase enzymes of peroxisomal beta-oxi[J]. J Biol Chem, 2003, 278(47): 47232-47239. DOI:10.1074/jbc.M306363200 |
| [31] |
Martens K, Ver Lv TE, van Batenburg MF, et al. Coordinate induction of PPAR alpha and SREBP2 in multifunctional protein 2 deficient mice[J]. Biochimica Et Biophysica Acta Molecular & Cell Biology of Lipids, 2008, 1781(11-12): 694-702. |
| [32] |
Ip E, Farrell GC, Robertson G, et al. Central role of PPARα-dependent hepatic lipid turnover in dietary steatohepatitis in mice[J]. Hepatology, 2003, 38(1): 123-132. DOI:10.1053/jhep.2003.50307 |
| [33] |
Rao MS, Reddy JK. Peroxisomal beta-oxidation and steatohepatitis[J]. Seminars in Liver Disease, 2001, 21(1): 43-55. DOI:10.1055/s-2001-12928 |
| [34] |
Chakravarthy MV, Lodhi IJ, Yin L, et al. Identification of a physiologically relevant endogenous ligand for PPARalpha in liver[J]. Cell, 2009, 138(3): 476-488. DOI:10.1016/j.cell.2009.05.036 |
| [35] |
Hines IN, Kremer M, Moore SM, et al. Impaired T cell-mediated hepatitis in peroxisome proliferator activated receptor alpha(PPARα)-deficient mice[J]. Biol Res, 2018, 51(1): 5. DOI:10.1186/s40659-018-0153-z |
| [36] |
Yu S, Reddy JK. Transcription coactivators for peroxisome proliferator-activated receptors[J]. Biochimica et Biophysica Acta, 2007, 1771(8): 936-951. DOI:10.1016/j.bbalip.2007.01.008 |
| [37] |
Montagner A, Polizzi A, Fouché E, et al. Original article:Liver PPARα is crucial for whole-body fatty acid homeostasis and is protective against NAFLD[J]. Gut, 2016, 65(7): 1202-1214. DOI:10.1136/gutjnl-2015-310798 |
| [38] |
Evans RM, Mangelsdorf DJ. Mangelsdorf DJ.Nuclear receptors, RXR, and the big bang[J]. Cell, 2014, 157(1): 255-266. DOI:10.1016/j.cell.2014.03.012 |
| [39] |
Meer DLMVD, Degenhardt T, Väisänen S, et al. Profiling of promoter occupancy by PPARα in human hepatoma cells via ChIP-chip analysis[J]. Nucleic Acids Research, 2010, 38(9): 2839-2850. DOI:10.1093/nar/gkq012 |
| [40] |
Feige JN, Gelman L, Michalik L, et al. From molecular action to physiological outputs:peroxisome proliferator-activated receptors are nuclear receptors at the crossroads of key cellular functions[J]. Progress in Lipid Research, 2006, 45(2): 120-159. DOI:10.1016/j.plipres.2005.12.002 |
| [41] |
Michalik L, Auwerx J, Berger JP, et al. International Union of Pharmacology.LXI.Peroxisome proliferator-activated receptors[J]. Pharmacological Reviews, 2006, 58(4): 726-741. DOI:10.1124/pr.58.4.5 |
| [42] |
Serviddio G, Bellanti F, Vendemiale G. Free radical biology for medicine:learning from nonalcoholic fatty liver disease[J]. Free Radical Biology & Medicine, 2013, 65(6): 952-968. |
| [43] |
Rui L. Energy metabolism in the liver[J]. Comprehensive Physiology, 2014, 4(1): 177-197. |
| [44] |
Evans RM, Barish GD, Wang YX. PPARs and the complex journey to obesity[J]. Nature Medicine, 2004, 10(4): 355-361. DOI:10.1038/nm1025 |
| [45] |
Misra P, Reddy JK. Peroxisome proliferator-activated receptor-α activation and excess energy burning in hepatocarcinogenesis[J]. Biochimie, 2014, 98(1): 63-74. |
| [46] |
Mei CL, He P, Cheng B, et al. Chlamydia pneumoniae induces macrophage-derived foam cell formation via PPAR alpha and PPAR gamma-dependent pathways[J]. Cell Biology International, 2009, 33(3): 301-308. DOI:10.1016/j.cellbi.2008.12.002 |
| [47] |
de la Rosa Rodriguez MA, Kersten S. Regulation of lipid droplet-associated proteins by peroxisome proliferator-activated receptors[J]. Biochimica Et Biophysica Acta, 2017, 1862(10): 1212-1220. DOI:10.1016/j.bbalip.2017.07.007 |
| [48] |
Linden AG, Li S, Choi HY, et al. Interplay between ChREBP and SREBP-1c coordinates postprandial glycolysis and lipogenesis in livers of mice[J]. Journal of Lipid Research, 2018, 59(3): 475-487. DOI:10.1194/jlr.M081836 |
| [49] |
Hostetler HA, Huang H, Kier AB, et al. Glucose directly links to lipid metabolism through high affinity interaction with peroxisome proliferator-activated receptor alpha[J]. J Biol Chem, 2008, 283(4): 2246-2254. DOI:10.1074/jbc.M705138200 |
| [50] |
Wang YX, Lee CH, Tiep S, et al. Peroxisome-proliferator-activated receptor δ activates fat metabolism to prevent obesity[J]. Cell, 2003, 113(2): 159-170. DOI:10.1016/S0092-8674(03)00269-1 |
| [51] |
Yu S, Matsusue K, Kashireddy P, et al. Adipocyte-specific gene expression and adipogenic steatosis in the mouse liver due to peroxisome proliferator-activated receptor gamma1(PPARgamma1)overexpression[J]. J Biol Chem, 2003, 278(1): 498-505. DOI:10.1074/jbc.M210062200 |
| [52] |
Balanarasimha M, Davis AM, Soman FL, et al. Ligand-regulated heterodimerization of peroxisome proliferator-activated receptor α with liver X receptor α[J]. Biochemistry, 2014, 53(16): 2632-2643. DOI:10.1021/bi401679y |
| [53] |
Baes M, Van Veldhoven PP. Hepatic dysfunction in peroxisomal disorders[J]. Biochim Biophys Acta, 2016, 1863(5): 956-970. DOI:10.1016/j.bbamcr.2015.09.035 |
| [54] |
Sanderson LM, Degenhardt T, Koppen A, et al. Peroxisome proliferator-activated receptor β/δ(PPARβ/δ)but not PPARα serves as a plasma free fatty acid sensor in liver[J]. Molecular & Cellular Biology, 2009, 29(23): 6257-6267. |
| [55] |
Nakamura MT, Yudell BE, Loor JJ. Regulation of energy metabolism by long-chain fatty acids[J]. Progress in Lipid Research, 2014, 53(1): 124-144. |
| [56] |
Ferdinandusse S, Denis S, Faust PL, et al. Bile acids:the role of peroxisomes[J]. Journal of Lipid Research, 2009, 50(11): 2139-2147. DOI:10.1194/jlr.R900009-JLR200 |
| [57] |
Rui LY. Energy metabolism in the liver[J]. Comprehensive Physiology, 2014, 4(1): 177-197. |
| [58] |
Bi L, Jiang Z, Zhou J. The role of lipin-1 in the pathogenesis of alcoholic fatty liver[J]. Alcohol & Alcoholism, 2015, 50(2): 146. |
| [59] |
Jia Y, Qi C, Zhang Z, et al. Overexpression of peroxisome proliferator-activated receptor-alpha(PPARalpha)-regulated genes in liver in the absence of peroxisome proliferation in mice deficient in both L-and D-forms of enoyl-CoA hydratase/dehydrogenase enzymes of peroxisomal beta-oxi[J]. J Biol Chem, 2003, 278(47): 47232-47239. DOI:10.1074/jbc.M306363200 |
| [60] |
Kliewer SA, Xu HE, Lambert MH, et al. Peroxisome proliferator-activated receptors:from genes to physiology[J]. Recent Prog Horm Res, 2001, 56(1): 239-263. DOI:10.1210/rp.56.1.239 |
| [61] |
Wolfrum C, Spener F. Fatty acids as regulators of lipid metabolism[J]. European Journal of Lipid Science & Technology, 2015, 102(12): 746-762. |
| [62] |
Reddy JK. Peroxisome proliferators and peroxisome proliferator-activated receptor alpha:biotic and xenobiotic sensing[J]. American Journal of Pathology, 2004, 164(6): 2305-2321. DOI:10.1016/S0002-9440(10)63787-X |
| [63] |
Antonenkov VD, Hiltunen JK. Transfer of metabolites across the peroxisomal membrane[J]. Biochimica Et Biophysica Acta, 2012, 1822(9): 1374-1386. DOI:10.1016/j.bbadis.2011.12.011 |
| [64] |
陈兴勇, 赵宁, 张燕, 等. 皖西白鹅育肥期肌肉脂肪酸组成及肝PPARα、FADS2和ME1基因表达规律的研究[J]. 畜牧兽医学报, 2017, 48(10): 1912-1919. |
| [65] |
林婄婄, 高中元, 袁亚男, 等. PPARα和PPARγ基因在不同脂尾型绵羊脂肪组织中的发育性表达研究[J]. 畜牧兽医学报, 2012, 43(9): 1369-1376. |
| [66] |
Luo JB, Tian JY, Tao ZR, et al. Regulatory expression of peroxisome proliferatoractivated receptors genes during fatty liver formation in geese[J]. Journal of Integrative Agriculture, 2010, 9(1): 113-120. |
| [67] |
贺綦, 史洪岩, 王海霞, 等. 鸡转录因子C/EBPα、KLF2、KLF3、KLF7、PPARα对L-FABP启动子活性的影响[J]. 中国畜牧杂志, 2014, 50(17): 13-17. DOI:10.3969/j.issn.0258-7033.2014.17.004 |
| [68] |
徐凯, 王效京, 刘宏, 等. 过氧化物酶体增殖物激活受体α在猪肝脏组织中发育性表达研究[J]. 中国畜牧兽医, 2014, 41(10): 178-182. |
| [69] |
李彦莹, 周天予, 杨振, 等. 鸭PPARs启动子扩增、序列比较及其转录因子表达模式的聚类分析[J]. 农业生物技术学报, 2016, 24(12): 1890-1899. |
| [70] |
武子寅, 温彦涛, 黎寿丰, 等. 鸡PPARα基因多态性与肉质性状关联的研究[J]. 中国家禽, 2013, 35(7): 9-12. DOI:10.3969/j.issn.1004-6364.2013.07.004 |
| [71] |
方文良, 黄金明, 王长法, 等. 中国荷斯坦牛PPARα基因多态性研究及其与耐热性能的关联分析[J]. 中国畜牧兽医, 2012, 39(8): 165-170. DOI:10.3969/j.issn.1671-7236.2012.08.040 |
| [72] |
李芬, 李荣荣, 张琼琼, 等. 中国地方黄牛PPARα exon7基因多态性与生长性状的关联研究[J]. 中国牛业科学, 2016, 42(1): 1-5. DOI:10.3969/j.issn.1001-9111.2016.01.001 |
| [73] |
田亚东, 亢娟娟, 孙桂荣, 等. PPARα基因对安卡×固始鸡资源群胴体品质的遗传效应分析[J]. 华北农学报, 2010, 25(6): 14-18. |




