2. 农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
环状RNA(circRNA)是在RNA成熟过程中一个叫反向剪接的环节中出现的。代替了传统的线性连接方式由上游的5'端的结合位点与下游的3'端结合位点反向连接形成环状。环状RNA是一类特殊的内源性非编码RNA,继长链非编码RNA和微小RNA之后的一类特殊群体,但是现在同样有研究称环状RNA构成了一类有趣的长链非编码RNA群体。虽然承认其在细胞内的存在性已经有30多年,但对其在细胞内的广泛性和高丰度是直到21世纪初期才被逐渐发现。环状RNA具有稳定的结构,可以不依靠蛋白单独存在。环状RNA作为一类特殊的RNA群体,由于其本身的环化结构而不带有正常RNA的3'和5'端,不能通过传统的检测技术检测而被忽略,直到新一代测序技术的出现和发展使其可以通过去除rRNA富集之后进行测序将它重新带入科学家的视野中[1-4]。
环状RNA由外显子反向剪接形成的ecircRNA、内含子来源的inciRNA以及外显子和内含子都参与的EIciRNA三大类组成。很多生物信息学的方法都证明了环状RNA的反向剪接方式,其标志性的剪切位点GT-AG成为检测其存在的一个重要标志,从来源上讲,其大部分来源于外显子和内含子,但同样也包含基因间的序列和非编码的UTR区域;这些分析同时也表明不同的环状RNA有可能来自于同一个基因座,同时出现一种替代环化的现象,这些环状RNA序列长度在大到25 000 nt以上小到小于100 nt不等,已有研究证实在病毒和拟病毒中最小的环状RNA为220 nt,同时其也是一个可以编码16 kD长度蛋白的环状RNA[1-3, 5-6]。环状RNA在很多发育过程中都起着重要的作用,如作为miRNA的海绵体,通过结合miRNA来调节miRNA的表达;与U1SnRNP结合来促进基因的转录等。一些研究报道称,环状RNA在不同的组织和不同的发育时期的表达量不同。这意味着环状RNA不仅是监管和拼接的副产品,同时也是在基因调控上有着重要作用的功能性基因[7]。与此同时,环状RNA对多种疾病都存在着调节作用并且可以作为其中一些疾病的生物标记物。因此,环状RNA的重要程度是不言而喻的。本文将对环状RNA的形成机制、功能、尤其是检测方法、novel环状RNA的鉴定以及疾病相关性一个认知程度内的概述。
1 环状RNA的发现及存在物种环状RNA是一类由3'端反向与5'端连接形成的共价闭合环状RNA分子,环状RNA与microRNA以及长链非编码RNA在转录的过程中占据了总RNA含量的95%左右。首次提出环状RNA的概念是在1976年,Sange[8]和他的同事提出高等植物中的一种类病毒RNA为环状,称为环状RNA。在1979年,在电子显微镜下发现了环状RNA为其存在性提供了有力的证据[9]。1991年,Nigro等[10]在研究RNA翻译表达的时候发现很多异常拼接的RNA分子,其中肿瘤抑制基因DCC在体外发生了异常拼接。在1993年,在动物的睾丸中发现性别决定基因sry是一种环状RNA[11]。但是环状RNA在从发现到被重视这30年间一直被视为RNA剪接成熟过程中的异常剪接物而被忽视,直到21世纪初随着高通量测序技术的发展和深入,环状RNA才重新走入科学家的视野。至今为止,随着研究的深入,已经在线虫、小鼠、大鼠、猪、猴子和人类等物种中发现了环状RNA的存在。其中Jeck等[1]在人成纤维细胞中检测到至少25 000种环状RNA;Memczak等[12]在线虫、小鼠和人体中分别找到了大约700种、1 900种和2 000种环状RNA。未发现的环状RNA仍有很多,同样在其他物种中也必然存在丰富的环状RNA,还有待进一步探究。
2 环状RNA的形成机制及其生物学特征 2.1 环状RNA的形成机制环状RNA是一类特殊的内源性非编码RNA具有高丰度、高保守性及高度的组织特异性等特点。环状RNA在哺乳动物的组织中是非常普遍的,并且可以通过和miRNA结合及其他的方式从转录和非转录的水平上调节基因的表达。环状RNA具有重要的调节作用,但是在刚发现其存在的时候,其被视为一种前体RNA成熟剪接过程中的非功能性产物,本质原因即为其形成方式的独特,并不是传统的线性连接方式,而是一种特殊的剪接方式—反向剪接。其形成的环状RNA的前体来源大体分为两类,即外显子和内含子。
2.1.1 外显子环状RNA的形成机制第一例被发现的内源性产生的环状RNA是20世纪90年代在一个对于人类细胞中DCC的转录研究中发现的。据这个实验的研究者称外显子的转录方式超出了预期,下游的3'跳跃到了上游与5'端连接,尽管是这种不正常的连接方式,其所用的连接位点和接受位点与正常的连接方式是一样的[13]。这种连接方式被称为外显子的“跳读”[1],由此外显子的形成方式已逐渐清晰。后来根据Jeck等[3]的研究,总结出外显子形成环状RNA的两种主流形式,第一种方式为直接反向剪接,由下游的3'端跳跃到上游与5'端结合形成环状RNA,由于其过程中发生的反向拼接现象,其中间部分的内含子序列随之一起折叠,并且在此过程中内含子与外显子连接的3'以及5'端连接形成套索结构,并随之被剪切去掉,剩下的外显子部分反向连接形成环状结构。第二种方式为内含子竞争性配对结合,此种方式中pri-mRNA两端的内含子由于竞争性的互补配对促使外显子发生反向折叠连接在一起,再将内含子部分剪切除去形成外显子部分连接的环状RNA,如图 1。已有研究证实直接反向剪接形成环状RNA的效率要高于内含子竞争性配对方式形成环状RNA,而且在机体内这种形成方式更为普遍[14]。外显子形成的环状RNA可以分为单个外显子、2个及2个以上外显子形成的环状RNA几种,根本在于其剪切位点的不同。环状RNA在体外的转录合成已经得到了证实[15]。
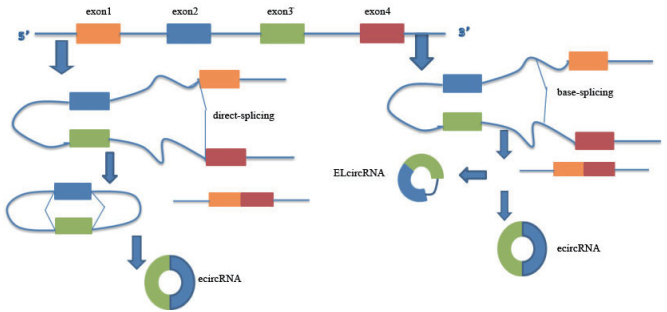
|
| 图 1 外显子环状RNA的形成 |
剪接在某种程度上就是intron splicing即内含子的剪接,而内含子的进化分类如果按剪接方式来分可以分为I类内含子、Ⅱ类内含子(group Ⅰ、Ⅱ intron)和核mRNA前体内含子(Spliceosomal introns),其中核mRNA前体内含子(Splicesomal intron)对于外显子形成的环状RNA是密切相关的,而group Ⅰ intron和group Ⅱ intron代表了内含子环状RNA形成的两种不同驱动方式[16]。
2.1.2.1 I类内含子环状RNA的形成方式一个外源的鸟苷作为亲核试剂去攻击5'结合位点并与内含子结合,经酯基转移后,5'端外显子被切除并且外源的鸟嘌呤与内含子产生联系,5'端外显子的终端3'-OH攻击3'端拼接点,连接的外显子和一个线性的内含子被切除释放。最终,由2'端羟基和5'端接受位点形成磷酸二酯键形成内含子环状RNA[17]。
2.1.2.2 II类内含子环状RNA的形成方式环化的发生需要3'外显子的提前释放,内含子末端的2'羟基集团攻击5'剪接位点,通过形成磷酸二酯键产生一个5'外显子和一个环状RNA,如图 2[18]。

|
| 图 2 内含子环状RNA的形成 |
据报道,环化的区域两侧必然存在着长度不一的重复侧翼内含子序列,这是一种天然并且广泛存在于细胞基因中的重复序列-Alu序列,其存在促进了环状RNA的形成,Alu序列的作用受到藤黄节杆菌(Arthrobacter luteus,ALU)限制性酶的调控。同时,有相关研究报道还有一些其他内含子互补序列同样对反向剪接起促进作用。除了一些内含子互补序列对反向剪接起促进作用之外,一些RNA结合蛋白如肌肉生长调节因子(Muscleblind,MBL)能够促进环状RNA的合成,可变剪切调节因子(Quaking,QKI)在上皮间质转化(Epithelial-mesenchymal transition,EMT)过程中可以调节环状RNA的合成以及RNA结合调节蛋白(RNA binding motif protein 20,RBM20)同样对环状RNA的形成起调节作用[19]。
2.2 环状RNA的生物学特征生物学特性一般是指形态、大小、结构、颜色等理化性质,而环状RNA的生物学特性是对其种类、分布、丰度等性质的概括。由于环状RNA反向剪接而成的特征,其存在很多独特的生物学特性。
环状RNA种类繁多,能够由一个外显子或者许多个外显子构成,环形的长度从小于100 nt到大于4 000 bp不等[3, 20]。存在范围非常广泛,在鼠类中已经发现近2 000种环状RNA,在线虫中发现超过700种。它在不同物种间有很好的保守性,有趣的是,已经证实69种环状RNA在人类与鼠类之间有着精准的保守性。环状RNA的表达量是非常少的,但是其在细胞中的丰度非常高。
3 环状RNA的功能 3.1 作为microRNA的海绵体MicroRNA作为非编码小RNA家族中的一员,其在基因表达的调控方面具有至关重要的作用。而研究发现环状RNA通过其具有microRNA海绵体的作用,可以通过与microRNA的结合来调控microRNA的作用以此间接调控着很多的生理状态。
3.1.1 ciRS-7在环状RNA研究中,对microRNA的研究最多的就是miR-7,它对其他癌症和疾病中的很多相关因素都具有调节的功能,如细胞发育、增殖和细胞凋亡[21-22]。在对许多恶性肿瘤研究发现,它能作为很多组织的癌症和肿瘤的抑制基因,包括乳房[23]、大脑[24]、头部和颈部[25]、肝脏[26]和肺[27]。小脑的退化相关蛋白1的反义链(CDR1as),亦称为miR-7海绵体,能够与micror-7结合并调节其功能,这提供了第一个环状RNA可以作为microRNA的海绵体的证据[28]。CiRS-7包含了70多个对mir-7的结合位点,并通过Argonaute蛋白质紧密联系在一起[29]。通过延伸和附近结合效应,这种环状RNA对不同的miRNA分别有一个单独的结合靶位点。而miR-671可以分裂ciRS-7/CDR1as来调节环状RNA对miR-7的抑制作用[30]。已有研究证明了环状RNA中ciRS-7/CRR1as的保守性和稳定性以及对miR-7活性的调节作用。通过与miRNA结合形成复合体以及重新释放它们来调节其活性,这是对miRNA活性研究的新发现。尽管这种竞争性内源RNA的想法并不新颖,并且饱含争议,但是环状RNA的稳定性足以支撑起其海绵效应的其余优势了。
3.1.2 Sry circRNA成熟的哺乳动物性别决定基因(Sex-determining region Y,Sry),能够形成一种特殊的环状序列[28]。这个长度在1.2kb的完全由外显子组成的环状RNA包含了16个miR-138的靶位点,也能起到类似于miRNA海绵体的作用。尽管这种作用和环状RNA ciRS-7/CDR1as的作用类型相似,都是竞争性内源RNA的方式,但是这种机制是建立在特殊的RNA序列和重复序列上的[31]。因此,这种机制是否是普遍存在的环状RNA调节机制还有待进一步的研究。
3.1.3 circHIPK3Zheng等[32]的进一步研究发现了一种环状RNA circHIPK3,这是一种来自于HIPK3基因外显子的环状RNA。体外研究显示,在癌细胞中沉默circHIPK3导致癌细胞的增长速度显著下降。通过荧光素酶报告基因的筛选,他们观察到circHIPK3可以结合多种microRNA,包括众所周知的肿瘤抑制因子miR-124[33]。这说明circHIPK3通过在人类细胞中调控多种microRNA来调节细胞的生长。
3.1.4 cirITCH最近发现的一种叫做cirITCH的环状RNA,同样有miRNA海绵的作用,通过调控mir-7和mir-20a来发挥作用。它增加了ITCH的表达水平,这是一种蛋白质编码基因,它会引发泛素介导的Dvl2退化并抑制常规的Wnt信号通路,具有整体的抗肿瘤效果[34]。
3.2 环状RNA的产生具有调节可变剪接的作用环状RNA的产生对可变剪接的调节作用几乎是已经确定的了。这种机制基于一个事实:pre-mRNA的剪接过程主要是3'端和5'端的竞争性结合[35],而在这个过程中一旦出现反向剪接现象,外显子将会出现“跳读”现象,这将使原本pre-mRNA的正常剪接过程发生变化,或者导致其被降解。在这种案例中,环状RNA的产生对于调节pre-mRNA的可变剪接过程有着重要的作用,尽管这将导致环状RNAs有可能失去功能性。同时反向剪接与正常的RNA剪接成熟在pre-mRNA的后加工过程中是存在竞争性的,环状RNA的产生意味着正常线性RNA的产量变少。
3.3 环状RNA的其他的一些可能性的功能很多不同的关于环状RNA的功能已经被发掘出来[36],包括其他影响因素的海绵体,例如RNA结合蛋白(RNA-binding proteins,RNPs)。环状RNAs能够通过结合和分离这些影响因素从而阻止它们发挥作用。在这方面强有力的证据就是其上含有的结合位点,如miR-7和ciRS-7/CDR1as的关系。然而由于大多数环状RNA的低含量,其持续影响目标分子的能力势必会大幅度降低[37]。
环状RNA可以作为一些混合物的“储藏室”,并且可以转移这些因素去一些特别的亚细胞位置,或者可以作为某些反应的支架。尽管环状RNA有起始密码子和合理的开放性阅读框,并且在一些功能性环状RNA上已经发生了翻译现象,如IRESes。但是其翻译性现阶段只在病毒中被证实了而在真核生物中还没有确实的答案,关于其翻译的证据到现在为止还是十分缺乏的
4 环状RNA的检测方法在1976年首次提出环状RNA的概念,1979年在显微镜下发现了类病毒中的环状RNA,1982-1988年研究者们用琼脂糖凝胶电泳和2D凝胶电泳中都检测到了环状RNA的存在,测序技术的发展使环状的检测变得越来越容易,从只能知道其存在到能确定是何种环状RNA,环状RNA的检测经历了长足的进步史。
4.1 分子生物学方法用northblot分离环状RNA,在琼脂糖凝胶电泳中,虽然环状RNA的迁移速率要比线性RNA的速率慢很多,但是实际上外显子环状RNA相比于其他来自于同一基因的全长线性RNA以及含有重复外显子序列的线性RNA所含有的核算数量要少的多,所以其在琼脂糖中的迁移速度反而要更快一些[38]。更具有决定性意义的实验是使用弱水或者靶向RNaseH进行降解处理,在琼脂糖凝胶电泳中未被处理的RNA迁移速率最慢,而被处理的线性RNA会出现两条条带,但是被处理后的环状RNA只有一条条带[11]。同时还可以用2D凝胶电泳,在此凝胶中线性RNA会跑出一条对角的直线,而环状RNA则是从斜对角一端到另一端的抛物曲线[30, 39]。但一种方法的检测必然存在不精准的地方,需要多种方法协同使用才能达到最佳效果。
环状RNA同样可以定量检测,环状RNA的pcr与普通的线性基因pcr不同,因为环状RNA在pre-mRNA的剪切过程中为反向剪接,其下游的3'位点与上游的5'位点结合导致其引物不是常规上游下游各一段引物相向扩增就可以得到结果,其junction位点处与线性RNA不同。但是其序列body处的碱基排列还是一致的,所以需要设计一种背对背引物(Divergent primer),可以反向扩增来实现环状RNA的定量验证。这种背对背引物的设计在环状RNA的数据库网站上有专门的设计专栏,如图 3[40-41]。
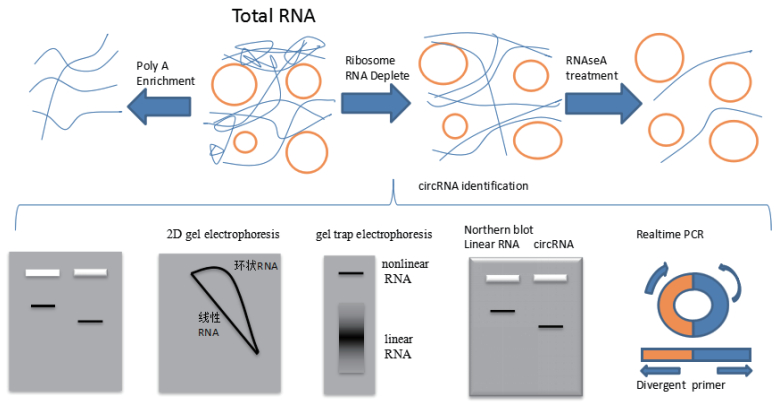
|
| 图 3 环状RNA检测图 |
环状RNA单独建库,其最关键的步骤在于去掉线性RNA及rRNA,单独留下环状RNA进行后续建库。
首先分离mRNA,通过磁珠法,利用磁珠上结合的oligo dT捕获带有poly A尾的mRNA,此时未与磁珠结合的环状RNA存在于缓冲体系中,收集上清,通过乙醇沉淀的方法,得到去除mRNA后的RNA,而磁珠捕获的mRNA可以用来构建转录组文库,为了后续与环状RNA信息进行联合分析。
下一步需要去除核糖体rRNA及其他线性RNA。首先采用rRNA清除试剂盒,用单链DNA探针ssDNA probe特异性的杂交rRNA,形成DNA-RNA双链产物,用RNase H对其进行降解,之后用DNase I降解探针,再用RNase R进一步降解线性RNA,乙醇沉淀得到最终的环状RNA。
最后通过片段化、反转录、尾端修复、加接头及PCR扩增等常规建库流程得到最终的环状RNA文库,采用PE150测序策略,一般5 G的数据量就可以达到分析要求。
4.2.2 全转录组建库全转录组包括mRNA、环状RNA、lncRNA等RNA的全部信息,其建库策略为只去除核糖体rRNA,不去除其余的线性RNA,通过构建一个全转录组文库,结合不同生物信息学的分析方法来获得全部的转录信息,并最终进行联合分析。
全转录组建库的关键在于去除核糖体rRNA,其大部分环节与环状RNA单组学建库是一致的,都是通过探针杂交并特异性酶解法去除DNA-RNA结构上的rRNA,并进行后续建库。测序策略仍为PE150,数据量要求10 G以上。
4.3 测序相关的检测算法在过去的20年里,人们对于环状RNA的研究已经取得了巨大的进步,但是起初对于环状RNA的忽视主要源于对其的检测技术不完整,并且没有特异性、有效的环状RNA序列信息,因此在检测技术不断发展的过程中出现了大量不同的检测算法。
2012年,Salzman等[2]提出了一种依赖于基因注释的算法,在这种算法中环状RNA能在已经比对基因组且进行外显子边界注释的标准数据库的基础上被检测出来,并且通过增加错误发现率(False discovery rate,FDR)的方法筛选出质量最优的比对片段来提升这种算法的准确率[20]。然而他们的算法主要依赖于已经注释的基因,对于那些还没有完全注释的基因不能精确的检测;并且这种建立在数据基础上的筛选有可能对那些低覆盖率的测序区域或者测序深度不够的区域起不到最好的检测效果。Memczak等[12]提出了利用GT-AG这段真核生物中特有的前体RNA剪切过程中的保守位点为信号来筛选出一些新的环状RNA;也有一些相似的算法用于寻找有miRNA海绵体功能的环状RNA[42]。但这些算法均采用了两部分序列比对的方式,这可能导致某些不具有此特征的环状RNA未被筛选出来,且这些算法在剔除假阳性方面可能无效。
Jeck等[1]采用了另外一种方式,比较RNase处理过和未处理的序列筛选出环状RNA的候选序列,并且剔除假阳性。这种方法对于环状RNA的检测更加敏感,并且能估算环状RNA的相对含量;然而这种方法在富集的过程中可能出现偏差,并且富集的过程是必不可少的。相较于环状RNA检测算法,序列比对算法发展历史久远,并且一些是被特定设计用于局部和分裂位点比对的。BWA-MEM利用空位延伸模型提出了一种可以提供最大准确率的局部比对位点,更加高效准确的算法。
Segemehl[43]用一种加强的后缀阵列针对保守位点检测的算法,且比其竞争对手在检测剪切位点上有更高的准确率。此后,Gao又提出了一种用sam格式中的CIGAR值进行分析,从sam文件中扫描PCC信号(paired chiastic clipping signals)。对于splicing信号(GT,AG)比较弱的,算法会从文件中抽取外显子边界位置,并用已知的边界来过滤假阳性。因为环状RNAs是环状拼接模式,在其拼接的过程中会出现不同的比对结果,这些成熟的比对算法有可能使环状RNA的检测有更高的准确率和效率,而如果没有这些算法,则假阳性的出现是不可避免的。
5 Novel环状RNA的鉴定及验证通过上述分子生物学与生物信息学以及不同检测算法的结合,我们能够得到准确率很高的环状RNA,这些环状RNA可以通过查找环状RNA数据库来确定其是否为新的环状RNA。例如,环状RNABase、deepBase、circBase、Circ2Traits等数据库。这些数据库中包含序列的查找,编号的确定以及引物的设计多种功能。确定其为新的环状RNA之后要对其功能进行实际验证。
5.1 环状RNA功能及活性的验证由于环状RNA较多情况下作为miRNA海绵体存在,可以采取AGO2免疫沉淀法分析环状RNA结合的miRNA。环状RNA本身结合着大量的AGO2蛋白,并且可以通过RIP确认。将环状RNA序列插入到萤火虫荧光素酶报道基因下游,通过环状RNA在结合miRNA后能够产生脱腺苷化,从而产生降解活性,抑制LUC酶活。并以此来判断环状RNA与哪些miRNA发生作用,进而对miRNA进行富集,通过miRNA靶基因预测来寻找其下游转录基因,并进行pcr和western blot验证基因的表达量以及蛋白的活性的变化,以此来判断环状RNA的功能。
鉴定环状结合的具体的miRNA,可以构建萤火虫荧光素酶的miRNA筛选文库。每个miRNA都和萤火虫荧光素酶报道基因一同转染进入细胞中。从而可以确定具体是哪些miRNA发生作用,并进行下一步验证。
5.2 环状RNA验证方法 5.2.1 环状RNA的Northern blot验证Northern blot是一种很传统的验证基因的手段,通过琼脂糖凝胶电泳针对特定的基因设计它的探针来检测其存在性,但是环状RNA的特殊性决定了其探针的设计不同于普通线性RNA。对于外显子环化环状RNA,建议探针尽可能跨backsplice junction位点;对内含子环化环状RNA,可围绕内含子区域设计探针。
5.2.2 环状RNA的过表达验证过表达验证在基因功能的验证是一种很流行的方法,环状RNA过表达思想主要源于环状RNA生物形成机制,已有多篇文章报道环状RNA成环机制,目前比较公认的成环机制为环状RNA侧翼序列的碱基互补配对,称之为Alu结构。基于环状RNA侧翼Alu序列特征,PCR扩增含侧翼Alu序列的目标DNA序列,随后依据对应限制性内切酶位点进行酶切,进而连接pEGFP-C1载体。连接载体进而转染对应细胞样本,定量PCR检测转染效率[44]。基于divergent primer验证环状RNA过表达倍数[45]。
过表达策略:(1)扩增目标区域包含环状RNA侧翼Alu序列或内部碱基互补序列,侧翼上下游1kb处过表达效率更佳;(2)目标区域扩增基于基因组DNA为模版。
5.2.3 环状RNA的敲除环状RNA敲除思路主要针对环状RNA反向剪接接口处序列信息设计siRNA,对于内含子环化环状RNA,也可针对内含子区域设计相应siRNA进行干扰。背对背引物验证环状RNA敲除倍数。
敲除策略:(1)外显子环化环状RNA,针对反向剪接连接位点前后序列设计siRNA。(2)内含子环化环状RNA,除针对反向剪接连接位点前后序列设计siRNA序列以外,也可针对内含(3)子区域序列设计siRNA。每个siRNA设计对应的对照,反向剪接连接位点一端互补配对,另一端错配。
6 环状RNA与疾病的相关性 6.1 环状RNA与疾病的相关性 6.1.1 环状RNA与心血管疾病的关系缺血性心脏病是发达国家致死率和发病率居高不下的疾病之一。第一个被验证的环状RNA cANRIL,其与单核苷酸多态性(Single nucleotide polymorphism,SNP)关系密切,有猜测这些改变了cANRIL的剪切拼接过程导致INK4A/ARF位点的表达,从而使动脉粥样硬化的发病率增高[5]。同时缺氧也是致使动脉粥样硬化发病的关键因素之一,而此因素同样受到环状RNA的调控[46]。Greene等[47]也表明环状RNA cZNF292是受到内皮细胞缺氧因素的调控并控制血管的生成,环状RNA在人类心脏组织中高度表达,与关键心脏基因TTN、RYR2、DMD都有联系。
6.1.2 环状RNA与阿耳滋海默氏病的关系最初的研究表明环状RNA在脑组织中的表达量很高[32],并且有可能参与到突触功能和神经可塑性的调控当中[48]。Lukiw等[49]的研究支持了这种说法,他们发现了老年痴呆症(AD)患者大脑的海马区是错误调控的,患者的海马区的miR-7/ciRS-7调控系统是错乱的,miR-7的上调导致了ciRS-7的缺失以及其它一些与此AD疾病相关的靶基因表达量的下调,包括泛素接合酶(UBE2A)蛋白。UBE2A是泛素循环中的一个重要因素,它通过吞噬作用帮助清除淀粉样肽。它在AD患者大脑中的缺失会导致淀粉状蛋白的生成,促使阿耳滋海默氏病即老年痴呆症发病。
6.1.3 环状RNA与糖尿病的关系糖尿病是长期不健康的生活状态造成的后果,早期检测和更好的治疗方法是必要的。Xu等[50]的研究表示过表达miR-7基因的转基因小鼠β细胞导致糖尿病的发生。同时该研究也表明过表达ciRS-7会抑制miR-7功能,进而改善胰岛素分泌。MiR-7的潜在靶基因已通过生物信息学分析确定,包括Myrip(调节胰岛素分泌颗粒基因)和Pax6(增强胰岛素转录基因)。
6.2 环状RNA与癌症的关系 6.2.1 环状RNA与胃癌的关系尽管许多发达国家胃癌的发病率显著下降,但胃癌仍是世界癌症中死亡率最高的疾病之一。Li等[51]的研究已经确定了环状RNA hascirc002059是一种与胃癌有关的环状RNA。其发现与健康对照组相比,患者的胃组织中此环状RNA的表达量是下调的。此外,在胃癌患者的血浆样本中发现了hsacirc002059,与健康对照组相比,其含量明显降低。类似地,Chen等[52]的研究已经确定了circPVT在胃癌组织中被上调,并通过调控miR-125家族的成员来促进细胞增殖。
6.2.2 环状RNA与膀胱癌的关系膀胱癌起源于膀胱的上皮内层,是全球排在第9位的最常见的癌症之一。在膀胱癌中,使用高通量微阵列技术同样发现了环状RNA[53]。通过这种方法,Zhong等[53]的研究发现与邻近的非肿瘤组织相比,在膀胱癌发现2种下调的环状RNA circFAM169A,circTRIM24和4个明显被上调的环状RNA circTCF25、circZFR、circPTK2和circBC048201。此外,在癌组织中,circTCF25可以通过调节miR-103a-3p和miR-107,从而提高CDK6基因的表达。这与癌症的发展密切相关。
6.2.3 环状RNA与肝细胞癌的相关性肝细胞癌(HCC)是肝脏疾病中的一种主要的恶性肿瘤,有慢性肝病和肝硬化的患者患病率居高。Zheng等[32]的研究表明与正常组织相比,在HCC中发现的环状RNA出现异常表达的现象。Qin等[54]在HCC中发现了hsacir0001649,并发现与相邻的正常肝组织相比,这一环状RNA的表达明显下降。与此形成对照的是,Shang等[55]已经确定了另一种环状RNA hsacir0005075,与正常组织相比在HCC中其表达量是明显下调的。与此类似,Yu等[56]在HCC中检测了先前发现的ciRS-7,发现它的表达在HCC肿瘤组织中与邻近的非肿瘤组织相比是显著下调的。由于ciRS-7对mir-7有海绵作用,进一步的验证中包括对ciRS-7的敲除和mir-7的过度表达,发现ciRS-7作为一种致癌基因,一定程度上是通过对miR-7的调控来影响HCC进行的。
7 展望迄今为止,我们所知道的环状RNA的种类以及功能仍然只占总环状RNA的一小部分,还有很多环状RNA待人们去发掘,环状RNA作为一类有调节功能的RNA,其在对人体研究中必然占据重要位置。高效快捷准确的检测及鉴定技术将成为重点发展的领域,从RNA序列数据库中对环状RNA进行预测及鉴定首先是要了解和抓住环状RNA在细胞中广泛存在的特点;其次在各种检测技术和算法中去除假阳性以及对环状RNA的富集是关键。环状RNA具有稳定的结构和高度的保守性,这些都是其能在不同的生物中发挥重要作用的依据,而除了miRNA海绵体、调节基因表达以及与RNA蛋白结合等作用外,其还有一些未证实的作用需要人们继续对环状RNA的功能进行更全面和广泛的鉴定。也因为环状RNA的高度保守性以及稳定性使其能够成为癌症的典型标志物,现阶段发现的环状RNA都是可以不依赖蛋白独立存在的,环状结构使其不容易被RNA酶类降解,所以只在某些特定癌症中存在的环状RNA的表达量变化将直观体现出一些特定癌症的变化情况,可以作为癌症的标志物,能够用于如肝细胞癌、乳腺癌、胃癌等一些特定癌症的诊断标志物,以及早期预防和后期治疗的有效手段,通过过表达或者抑制特定环状RNA可以达到抑制癌症的恶化的目的,而对于环状RNA的快速检测以及特定癌症环状RNA作用的探索将因此成为一个重要的研究方向;随着单细胞环状RNA测序技术的逐渐完善,可以从单细胞层面了解不同细胞间环状RNA的区别,有些环状RNA只在某些特定的细胞中存在,因此环状RNA同样可以作为标志物来区分同一组织不同区域的细胞;同时环状RNA无论是作为miRNA海绵体对基因表达的调控,还是其反向剪切的方式与正常线性RNA生成的剪切方式存在竞争关系,其都有调控生命进程的作用,环状RNA可以作为调控细胞生存状态的关键物质。因此,对于环状RNA自身结构功能性以及其转录过程中竞争性机制的研究也将成为重要议题。
同时环状RNA的研究还存在着很多问题需要解决。环状RNA的具体合成机制,其合成过程中涉及到的酶及剪切过程等详细环节需要研究完善;环状RNA参与生长发育过程中的许多环节,但是环状RNA的移动触发信号还不是很清楚;环状RNA可以作为miRNA的海绵体,但是其无论与一个miRNA反应还是多个miRNA反应,以及miRNA、环状RNA、目的基因之间的具体调控机制需要进一步完善;如何预测环状RNA的三级结构以及它们之间的相互作用需要更系统的探讨。这些都预示着环状RNA在将来很长一段时间里都会是研究的重点。
| [1] |
Jeck WR, Sorrentino JA, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. |
| [2] |
Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733 |
| [3] |
Zhang XO, Wang HB, Zhang Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. |
| [4] |
Gao Y, Wang J, Zhao F. CIRI:an efficient and unbiased algorithm for de novo circular RNA identification[J]. Genome Biology, 2015, 16(1): 4. |
| [5] |
Burd CE, Jeck WR, Liu Y, et al. Expression of linear and novel circular forms of an INK4/ARF-associated non-coding RNA correlates with atherosclerosis risk[J]. PLoS Genetics, 2010, 6(12): e1001233. DOI:10.1371/journal.pgen.1001233 |
| [6] |
AbouHaidar MG, Venkataraman S, Golshani A, et al. Novel coding, translation, and gene expression of a replicating covalently closed circular RNA of 220 nt[J]. Proceedings of the National Academy of Sciences, 2014, 111(40): 14542-14547. DOI:10.1073/pnas.1402814111 |
| [7] |
Hansen TB, Venø MT, Damgaard CK, et al. Comparison of circular RNA prediction tools[J]. Nucleic Acids Research, 2015, 44(6): e58-e58. |
| [8] |
Sanger HL, Klotz G, Riesner D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proceedings of the National Academy of Sciences, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 |
| [9] |
Hsu MT, Coca-Prados M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells[J]. Nature, 1979, 280(5720): 339-400. DOI:10.1038/280339a0 |
| [10] |
Nigro JM, Cho KR, Fearon ER, et al. Scrambled exons[J]. Cell, 1991, 64(3): 607-613. |
| [11] |
Capel B, Swain A, Nicolis S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-1030. DOI:10.1016/0092-8674(93)90279-Y |
| [12] |
Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [13] |
Greene J, Baird AM, Brady L, et al. Circular RNAs:biogenesis, function and role in human diseases[J]. Frontiers in Molecular Biosciences, 2017, 4: 38. DOI:10.3389/fmolb.2017.00038 |
| [14] |
Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs[J]. Nature Biotechnology, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890 |
| [15] |
Chen C, Sarnow P. Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs[J]. Science, 1995, 268(5209): 415-417. DOI:10.1126/science.7536344 |
| [16] |
Petkovic S, Müller S. RNA circularization strategies in vivo and in vitro[J]. Nucleic Acids Research, 2015, 43(4): 2454-2465. DOI:10.1093/nar/gkv045 |
| [17] |
Stoddard BL. Homing endonucleases from mobile group I introns:discovery to genome engineering[J]. Mobile DNA, 2014, 5(1): 7. DOI:10.1186/1759-8753-5-7 |
| [18] |
Costa M, Walbott H, Monachello D, et al. Crystal structures of a group Ⅱ intron lariat primed for reverse splicing[J]. Science, 2016, 354(6316): aaf9258. DOI:10.1126/science.aaf9258 |
| [19] |
Hsiao KY, Sun HS, Tsai SJ. Circular RNA-new member of noncoding RNA with novel functions[J]. Experimental Biology and Medicine, 2017, 242(11): 1136-1141. DOI:10.1177/1535370217708978 |
| [20] |
Salzman J, Chen RE, Olsen MN, et al. Cell-type specific features of circular RNA expression[J]. PLoS Genetics, 2013, 9(9): e1003777. DOI:10.1371/journal.pgen.1003777 |
| [21] |
Li Z, Rana TM. Therapeutic targeting of microRNAs:current status and future challenges[J]. Nature Reviews Drug Discovery, 2014, 13(8): 622-638. DOI:10.1038/nrd4359 |
| [22] |
Zhao J, Tao Y, Zhou Y, et al. MicroRNA-7:a promising new target in cancer therapy[J]. Cancer Cell International, 2015, 15(1): 103. |
| [23] |
Reddy SDN, Ohshiro K, Rayala SK, et al. MicroRNA-7, a homeobox D10 target, inhibits p21-activated kinase 1 and regulates its functions[J]. Cancer Research, 2008, 68(20): 8195-8200. DOI:10.1158/0008-5472.CAN-08-2103 |
| [24] |
Kefas B, Godlewski J, Comeau L, et al. microRNA-7 inhibits the epidermal growth factor receptor and the Akt pathway and is down-regulated in glioblastoma[J]. Cancer Research, 2008, 68(10): 3566-3572. DOI:10.1158/0008-5472.CAN-07-6639 |
| [25] |
Kalinowski FC, Giles KM, Candy PA, et al. Regulation of epidermal growth factor receptor signaling and erlotinib sensitivity in head and neck cancer cells by miR-7[J]. PLoS One, 2012, 7(10): e47067. DOI:10.1371/journal.pone.0047067 |
| [26] |
Ning BF, Ding J, Liu J, et al. Hepatocyte nuclear factor 4α-nuclear factor-κB feedback circuit modulates liver cancer progression[J]. Hepatology, 2014, 60(5): 1607-1619. DOI:10.1002/hep.v60.5 |
| [27] |
Ch ou, Yu-Ting, et al. EGFR promotes lung tumorigenesis by activating miR-7 through a Ras/ERK/Myc pathway that targets the Ets2 transcriptional repressor ERF[J]. Cancer Research, 2010, 70(21): 8822-8831. DOI:10.1158/0008-5472.CAN-10-0638 |
| [28] |
Hansen, Thomas B, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [29] |
Hansen TB, Kjems J, Damgaard CK. Circular RNA and miR-7 in cancer[J]. Cancer Research, 2013, 73(18): 5609-5612. DOI:10.1158/0008-5472.CAN-13-1568 |
| [30] |
Hansen TB, Wiklund ED, Bramsen JB, et al. miRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA[J]. The EMBO Journal, 2011, 30(21): 4414-4422. DOI:10.1038/emboj.2011.359 |
| [31] |
Hacker A, Capel B, Goodfellow P, et al. Expression of Sry, the mouse sex determining gene[J]. Development, 1995, 121(6): 1603-1614. |
| [32] |
Zheng Q, Bao C, Guo W, et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs[J]. Nature Communications, 2016, 7: 11215. DOI:10.1038/ncomms11215 |
| [33] |
Wang Y, Chen L, Wu Z, et al. miR-124-3p functions as a tumor suppressor in breast cancer by targeting CBL[J]. BMC Cancer, 2016, 16(1): 826. DOI:10.1186/s12885-016-2862-4 |
| [34] |
Li F, Zhang L, Li W, et al. Circular RNAITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway[J]. Oncotarget, 2015, 6(8): 6001-6013. |
| [35] |
Ashwal-Fluss R, Meyer M, Pamudurti NR, et al. circRNA biogenesis competes with pre-mRNA splicing[J]. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 |
| [36] |
Hentze MW, Preiss T. Circular RNAs:splicing's enigma variations[J]. The EMBO Journal, 2013, 32(7): 923-925. DOI:10.1038/emboj.2013.53 |
| [37] |
Denzler R, Agarwal V, Stefano J, et al. Assessing the ceRNA hypothesis with quantitative measurements of miRNA and target abundance[J]. Molecular Cell, 2014, 54(5): 766-776. DOI:10.1016/j.molcel.2014.03.045 |
| [38] |
Hurowitz EH, Brown PO. Genome-wide analysis of mRNA lengths in Saccharomyces cerevisiae[J]. Genome Biology, 2003, 5(1): R2. |
| [39] |
Schindler CW, Krolewski JJ, Rush MG. Selective trapping of circular double-stranded DNA molecules in solidifying agarose[J]. Plasmid, 1982, 7(3): 263-270. DOI:10.1016/0147-619X(82)90007-5 |
| [40] |
Cocquet J, Chong A, Zhang G, et al. Reverse transcriptase template switching and false alternative transcripts[J]. Genomics, 2006, 88(1): 127-131. |
| [41] |
McManus CJ, Duff MO, Eipper-Mains J, et al. Global analysis of trans-splicing in Drosophila[J]. Proceedings of the National Academy of Sciences, 2010, 107(29): 12975-12979. DOI:10.1073/pnas.1007586107 |
| [42] |
Guo JU, Agarwal V, Guo H, et al. Expanded identification and characterization of mammalian circular RNAs[J]. Genome Biology, 2014, 15(7): 409. DOI:10.1186/s13059-014-0409-z |
| [43] |
Hoffmann S, Otto C, Doose G, et al. A multi-split mapping algorithm for circular RNA, splicing, trans-splicing and fusion detection[J]. Genome Biology, 2014, 15(2): R34. |
| [44] |
Darbani B, Noeparvar S, Borg S. Identification of circular RNAs from the parental genes involved in multiple aspects of cellular metabolism in barley[J]. Frontiers in Plant Science, 2016, 7: 776. |
| [45] |
Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature Structural & Molecular Biology, 2015, 22(3): 256. |
| [46] |
Boeckel JN, Jaé N, Heumüller AW, et al. Identification and characterization of hypoxia-regulated endothelial circular RNA[J]. Circulation Research, 2015, 117(10): 884-890. DOI:10.1161/CIRCRESAHA.115.306319 |
| [47] |
Greene J, Baird AM, Brady L, et al. Circular RNAs:biogenesis, function and role in human diseases[J]. Frontiers in Molecular Biosciences, 2017, 4: 38. DOI:10.3389/fmolb.2017.00038 |
| [48] |
You X, Vlatkovic I, Babic A, et al. Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity[J]. Nature Neuroscience, 2015, 18(4): 603-610. DOI:10.1038/nn.3975 |
| [49] |
Lukiw W. Circular RNA(circRNA)in Alzheimer's disease(AD)[J]. Frontiers in Genetics, 2013, 4: 307. |
| [50] |
Xu H, Guo S, Li W, et al. The circular RNACdr1as, via miR-7 and its targets, regulates insulin transcription and secretion in islet cells[J]. Scientific Reports, 2015, 5: 12453. DOI:10.1038/srep12453 |
| [51] |
Li P, Chen S, Chen H, et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer[J]. Clinica Chimica Acta, 2015, 444: 132-136. DOI:10.1016/j.cca.2015.02.018 |
| [52] |
Chen J, Li Y, Zheng Q, et al. Circular RNA profile identifies circPVT1 as a proliferative factor and prognostic marker in gastric cancer[J]. Cancer letters, 2017, 388: 208-219. DOI:10.1016/j.canlet.2016.12.006 |
| [53] |
Zhong Z, Lv M, Chen J. Screening differential circular RNA expression profiles reveals the regulatory role of circTCF25-miR-103a-3p/miR-107-CDK6 pathway in bladder carcinoma[J]. Scientific Reports, 2016, 6: 30919. DOI:10.1038/srep30919 |
| [54] |
Qin M, Liu G, Huo X, et al. Hsa_circ_0001649:a circular RNA and potential novel biomarker for hepatocellular carcinoma[J]. Cancer Biomarkers, 2016, 16(1): 161-169. DOI:10.3233/CBM-150552 |
| [55] |
Shang X, Li G, Liu H, et al. Comprehensive circular RNA profiling reveals that hsa_circ_0005075, a new circular RNA biomarker, is involved in hepatocellular crcinoma development[J]. Medicine, 2016, 95(22): e3811. DOI:10.1097/MD.0000000000003811 |
| [56] |
Yu L, Gong X, Sun L, et al. The circular RNACdr1as act as an oncogene in hepatocellular carcinoma through targeting miR-7 expression[J]. PLoS One, 2016, 11(7): e0158347. DOI:10.1371/journal.pone.0158347 |




