2. 内蒙古大学生命科学学院,呼和浩特 010021
2. School of Life Science, Inner Mongolia University, Hohhot 010021
DbpA是依赖ATP的3'-5'RNA解旋酶,分子量为49.188 kD,主要在核糖体50S亚基形成的晚期发挥作用[1-2]。DbpA有一个DEAD盒,这是依赖RNA的ATP酶/解旋酶的特征[3]。DbpA特异地与23S rRNA的92号发夹结合,并表现出依赖RNA的ATP酶(ATPase)活性,水解一分子ATP可以解开8 bp的双链RNA[4]。除了大肠杆菌外,dbpA基因也存在于许多其他细菌中,这表明DbpA可能在某些情况下,比如缺失其他某一个核糖体成熟因子时,对核糖体的组装至关重要[5]。研究发现R331A点突变的DbpA缺失了其ATPase活性后,引起45S(50S核糖体亚基的前体)不完整颗粒的聚集,45S不完整颗粒可以激活野生型DbpA的ATP酶活性来完成50S核糖体的组装[2]。如果细菌内也缺乏野生型DbpA蛋白,那么过度表达R331A点突变的DbpA会导致各种核糖体颗粒的累积,但最终可以转化为有功能的50S大亚基[6]。
dbpA作为核糖体成熟因子,促进50S大亚基的组装,但它对大肠杆菌DNA复制起始过程的影响还没有报道。本研究通过观察dbpA缺失突变体(ΔdbpA)的DNA复制式样、倍增时间、细胞大小等表型变化来探讨dbpA基因对大肠杆菌DNA复制起始的影响,并探讨其作用机制。
大肠杆菌的细胞周期分为B期、C期和D期,并且也受到精细的调控。与真核细胞不同的是,大肠杆菌可以在上一次细胞周期还未完成时,开始下一次的细胞周期,使大肠杆菌的DNA在复制完成之前可以启动新一轮的DNA复制,所以大肠杆菌细胞内可以有多条染色体。大肠杆菌的染色体DNA复制起始于复制原点oriC,主要参与蛋白有DnaA、DnaB和DnaC等。DnaA蛋白是复制起始蛋白,特异地与oriC位点结合,然后DnaC蛋白协助DnaB蛋白(解旋酶)进入起始位点,逐渐形成复制引发体(Primosome)并开始复制。由于细胞内DnaA蛋白的数量和浓度与复制起始的发生率呈正比关系[7]。因此,我们检测ΔdbpA细胞内总蛋白和DnaA蛋白的量,并与野生型细胞比较,探究作为核糖体成熟因子的DbpA是否通过影响核糖体的组装或功能状态影响细胞内蛋白质的合成,改变细胞内总蛋白,包括DnaA蛋白的含量,从而影响DNA复制起始的机理。
DbpA也可能通过与DNA复制起始相关蛋白DnaA、DnaB或DnaC相互作用影响细菌DNA复制起始,因此我们采用温度敏感实验来验证它们之间是否有相互作用。dnaA46、dnaB252和dnaC2都是DNA复制起始缺陷的温度敏感突变体,它们在30℃时可以生长,但在42℃时无法生长[8-10]。我们分别将dnaA46、dnaB252和dnaC2的等位基因通过P1转导ΔdbpA构建双突变体,通过观察双突变体在30℃、37℃和42℃的生长情况,即温度敏感性的改变,来探讨DbpA是否与DnaA、DnaB或DnaC蛋白有相互作用。
细菌的细胞大小受多种因素的影响,如培养基的营养条件、pH和离子浓度,以及细胞内部环境等。我们通过测量ΔdbpA突变体的细胞大小,探讨dbpA基因是否影响细菌的细胞大小。
1 材料与方法 1.1 材料 1.1.1 菌株野生型大肠杆菌BW25113由本实验室保存。∆dbpA菌株由Baba提供,它是BW25113的dbpA基因通过同源重组被卡那霉素(kan)基因替换而构建[11]。
1.1.2 试剂与仪器BCA试剂盒购自Pierce公司,Western blotting用的鼠DnaA单克隆一抗和山羊抗鼠IgG-HRP多克隆二抗购自全式金公司,Hoechst33258 DNA染色液购自MedChemExpress公司。蔡司荧光显微镜Axio Imager A2购自Carl Zeiss公司,BD LSRFortessa流式细胞仪购自BD公司。
1.1.3 培养基和条件细菌的培养基为LB或ABTGcasa(AB基础培养基添加10 μg/mL维生素B1、0.2%的葡萄糖、0.5%的Casamino Acids)[12],LB的营养高于ABTGcasa培养基,培养温度为37℃。进行细菌选择时添加终浓度为50 μg/mL的卡那霉素(Kan)和30 μg/mL的氯霉素(Cap)。
1.2 方法 1.2.1 ∆dbpA菌株的鉴定PCR验证dbpA基因是否删除及kan基因是否插入。引物由上海生工生物工程公司合成。dbpA基因的上游和下游引物分别为:5'-GATGACCACGAGAATAGATTGTG-3';5'-GATACACAACGTTGCCATTTCTG-3'。PCR模板为野生型BW25113和∆dbpA菌株,扩增条件为:94℃变性30 s,53℃退火30 s,72℃延伸60 s,30个循环;最后72℃延伸10 min。kan基因的上游和下游引物分别为:5'-TGCTCGACGTTGTCACTGAAG-3';5'-CACCATGATATTCGGCAAGCAG-3'。扩增条件为:94℃变性45 s,53℃退火30 s,72℃延伸120 s,30个循环;最后72℃延伸10 min。
1.2.2 计算细菌的倍增时间过夜复苏的细菌按1:5 000比例接种至LB或ABTGcasa培养基中,检测和记录细菌在对数生长阶段从OD600(LB)或OD450(ABTGcasa)为0.05-0.5时的吸光度值。吸光度值先进行Log2计算,以得数作为横坐标,间隔时间作为纵坐标绘制线性关系图,最后计算出细胞倍增时间。实验重复3次,计算平均值和标准差。
1.2.3 测量细胞大小在LB或ABTGcasa培养基中的细菌以指数形式生长至OD600/450= 0.15时取1 mL菌液,离心收集菌体,先用TE缓冲液洗涤一次,然后用70%的乙醇固定。使用蔡司显微镜观察细胞,测量并记录100个以上细胞长轴的长度,取平均值并计算标准差。
1.2.4 流式细胞技术分析LB或ABTGcasa培养基中对数生长期的细胞,在OD600或OD450= 0.15取1 mL菌液,加入终浓度为10 μg/mL的先锋霉素(CPX)和300 μg/mL的利福平(RFP)处理3-5个细胞周期。先锋霉素可抑制细胞分裂;利福平通过抑制DNA复制起始所需的转录而阻止新的DNA复制的启动,但允许已经开始的并正在进行的复制完成[13-14]。所以药物处理后的细胞其染色体数目为整数,并代表添加药物时细胞内的复制原点数目[13]。药物处理后的细胞固定在70%乙醇中。使用流式细胞仪检测时,细胞经Hoechst33258 DNA染色液染色30 min,然后使用流式细胞仪进行分析。
1.2.5 温度敏感实验温度敏感突变体dnaA46(Ts)∷tet、dnaB52(Ts)∷tet和dnaC2(Ts)∷tet的等位基因通过P1转导至ΔdbpA突变体构建ΔdbpA dnaA46、ΔdbpA dnaB252和ΔdbpA dnaC2共3个双突变体[15]。涂板并过夜培养后,从ΔdbpA、DnaA46、DnaB252、DnaC2单突变体和构建的3个双突变体琼脂平板上分别挑取8个单菌落,每个单菌落分别涂布在3个LB琼脂平板上,3个琼脂平板分别置于30℃、37℃和42℃的培养箱中过夜培养,观察并记录每个突变体的8个单菌落在每种温度下生长的数量。
1.2.6 测定单个细胞的总蛋白ABTGcasa培养基中的细菌生长至OD450= 0.15时,取9 mL菌液,4℃ 12 000 r/min离心2 min收集菌体,用1 mL TE缓冲液洗涤一次,再用200 μL含有1% SDS和甘油的TE缓冲液重悬,煮沸5 min。使用BCA比色法测定9 mL菌液的总蛋白量[16]。为了检测某一体积菌液中细胞的数量,即菌体密度,再取10 μL菌液用培养液稀释104和105倍,然后涂布在含有相应抗生素的LB琼脂平板上,在37℃过夜培养至出现单菌落。计数单菌落的数量,计算菌体密度并计算出9 mL菌液中细菌数量。使用9 mL菌液中细菌细胞内的蛋白量除以9 mL菌液中细菌数量计算出每个细胞内的总蛋白质含量。实验重复3次,计算平均值和标准差。
1.2.7 测定单个细胞的DnaA蛋白上面提到的细胞提取液还使用Western blot来测定单个细菌内DnaA蛋白的含量。先设定每种菌株细胞内的总蛋白质的量相等,然后根据计算出的单个细胞内的总蛋白质含量,计算每种细胞提取液的使用量。样品使用12.5%的凝胶进行SDS-PAGE后,经半干式印迹将凝胶中的蛋白转移至聚偏二氟乙烯(PVDF)膜上,先后使用鼠DnaA的单克隆抗体(一抗)和山羊抗鼠的IgG-HRP(二抗)与膜孵育,接着进行显色和显影,根据条带颜色深浅确定DnaA蛋白的量[17]。实验重复3次,计算平均值和标准差。
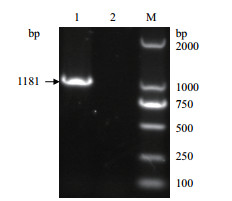
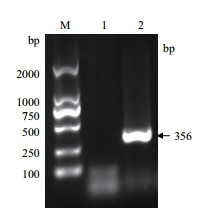
2 结果 2.1 ∆dbpA菌株的鉴定dbpA基因PCR扩增的电泳结果(图 1)显示野生型BW25113菌株在约1 200 bp处出现条带,与扩增的dbpA基因片段长度(1 181 bp)相仿;而∆dbpA菌株未扩增出dbpA基因条带,说明∆dbpA菌株中的dbpA基因缺失。kan基因PCR扩增的电泳结果(图 2)显示∆dbpA菌株在约350 bp处出现条带,与扩增的kan基因片段长度(356 bp)相仿;而野生型BW25113菌株未扩增出kan基因条带,说明kan基因插入∆dbpA菌株中,并替换了dbpA基因,即Baba提供的∆dbpA菌株构建成功。
|
| 图 1 PCR鉴定ΔdbpA菌株的dbpA基因是否删除 M:分子量标准;1:野生型BW25113;2:ΔdbpA |
|
| 图 2 PCR鉴定ΔdbpA菌株的kan基因是否插入 M:分子量标准;1:野生型BW25113;2:ΔdbpA |
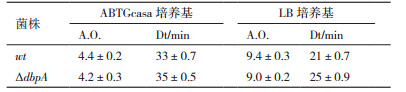
流式细胞技术检测结果(图 3,表 1)显示,在ABTGcasa培养基中,野生型BW25113细菌有2、4或8个染色体复制原点,细胞数目所占比例分别为10%、63%和17%,平均复制原点数目(AO)为4.4,倍增时间(Doubling time,Dt)是33 min。ΔdbpA含有2、4或8个染色体复制原点的细胞比例分别为18%、62%和10%,A.O.减少到4.2,倍增时间延长至35 min。在LB培养基中,野生型细胞有4、8或16个染色体复制原点,细胞所占比例分别为1%、58%和29%,A.O.是9.4,倍增时间是21 min。而ΔdbpA含有4、8或16个染色体复制原点的细胞比例分别为6%、66%和14%,A.O.减少到9.0,倍增时间延长至25 min。表明缺失dbpA基因使细菌DNA复制起始出现轻度延迟,生长速度也轻度减慢,并且培养基的营养越高,减慢也越多。
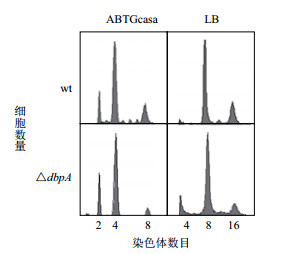
|
| 图 3 缺失dbpA基因导致细菌DNA复制起始发生轻度延迟 |
为了探讨dbpA基因是否影响细胞大小,我们使用显微镜测量野生型BW25113、ΔdbpA在ABTGcasa和LB培养基中的细胞大小。在ABTGcasa培养基中,野生型BW25113、ΔdbpA细胞的平均长度分别为2.69 μm和2.62 μm(图 4和表 2);在LB培养基中的平均长度分别为3.87 μm和3.49 μm(图 4和表 2),即分别减小3%和10%。上述结果表明缺失dbpA基因导致使细胞体积轻度减少,并且培养基的营养越高,减小程度也越大。
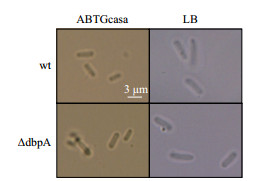
|
| 图 4 dbpA基因缺失导致细菌体积轻度减小 |
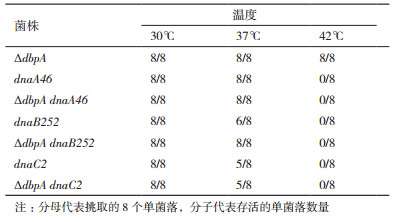
为了研究DbpA蛋白对DNA复制起始的影响是否通过与复制起始相关蛋白DnaA、DnaB或DnaC相互作用而导致的,我们使用温度敏感突变体dnaA46(Ts)、dnaB252(Ts)和dnaC2(Ts)构建ΔdbpA dnaA46、ΔdbpA dnaB252和ΔdbpA dnaC2共3种双突变体。我们发现dnaA46、dnaB252和dnaC2每种温度敏感突变体的8个单菌落在30℃时都能生长,37℃时绝大多数能生长,而42℃时都不能生长。ΔdbpA突变体的8个单菌落在30℃、37℃和42℃时都可以生长。3种双突变体的8个单菌落在30℃时都可以生长,37℃时绝大多数能生长,但42℃时都不能增长,即双突变体的dnaA46、dnaB252和dnaC2的温度敏感性都没有改变。表明DbpA影响DNA复制起始不是通过与DnaA、DnaB或DnaC蛋白相互作用导致的(表 3)。
DnaA蛋白是DNA复制的起始蛋白,并且细胞内DnaA蛋白的数量与染色体DNA复制的起始发生频率呈正相关。dbpA基因可能通过改变细胞内DnaA的数量影响细菌复制的起始。为了检测这个可能性,我们比较ΔdbpA和野生型BW25113单个细胞内的总蛋白和DnaA蛋白的含量。结果发现与较野生型细胞相比,在ABTGcasa培养基中,ΔdbpA单个细胞内的总蛋白和DnaA蛋白含量分别减少了8%和6%、(图 5-A);而在LB培养基中,分别减少17%和18%(图 5-B)。表明删除dbpA基因使细胞内的总蛋白和DnaA蛋白的量都轻度下降。并且培养基的营养越高,下降程度也越大。
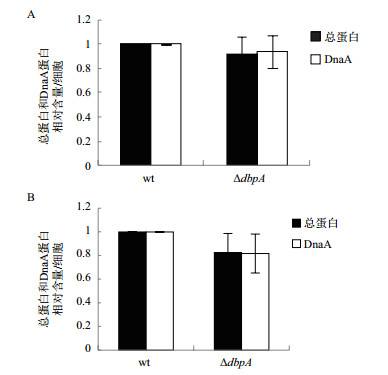
|
| 图 5 缺失dbpA基因降低ABTGcasa(A)和LB(B)培养基中单个细胞的总蛋白 |
大肠杆菌核糖体的组装是非常快速和高效的,且有多种装配途径,在2-3 min内(37℃)就能完成有功能的50S核糖体大亚基的组装[18]。大肠杆菌有5种DEAD蛋白,分别是CsdA、DbpA、RhlB、RhlE和SrmB,它们作用的底物都是RNA。DbpA作为依赖ATP的RNA解旋酶,特异地与23S rRNA结合,参与50S大亚基的装配,而它特异结合的23S rRNA的92号发夹还参与核糖体肽基转移酶中心(Peptidyltransferase center,PTC)的构成[2]。
对于dbpA是否影响细胞表型,不同的实验得出的结果也有很大差别。Iost等和Peil等[19-20]发现dbpA删除突变体的生长速度和核糖体轮廓在37℃和低温20℃或25℃时都没有变化。可能的原因:(1)突变或删除导致细菌缺失有功能的DbpA后,可以由其他核糖体成熟因子完成50S亚基组装所需的RNA异构化步骤。(2)在核糖体组装过程中缺少DbpA的作用,虽然出现了错误组装的中间粒子(45S),并累积到某一程度时,细菌通过其他机制纠正错误组装而形成有功能的50S大亚基。
Elles等[21]发现dbpA在R331A的定点突变导致其解旋酶活性和ATP酶活性降低;细菌还表现出明显的生长缓慢和冷敏感的表型。她们分别将含有野生型、K53A和R331A点突变dbpA基因的重组质粒转入TunerTM(DE3)pLacI RecA-菌株后,发现转入K53A和R331A点突变dbpA重组质粒的菌株生长速度在37℃时比野生型慢;在22℃低温时,它们的倍增时间分别为113 min、134 min和251 min;即R331A点突变的倍增时间在37℃是野生型的1.6倍,在22℃时是2.2倍,并且还发现细菌从生长缓慢期到对数生长期的用时也较长。说明dbpA突变使细菌在37℃和低温22℃时的生长速度都减慢。可能原因:(1)DpbA作为核糖体成熟因子参与50S大亚基的装配和成熟,缺失DpbA的功能导致45S颗粒聚集,50S核糖体无法形成或形成速度减慢,细胞内有功能的核糖体数量减少,降低翻译效率。(2)缺失DpbA的功能导致细胞内形成错误组装的、无功能的核糖体,降低翻译活性。(3)DbpA不仅与50S核糖体亚基组装有关,还与翻译活性有关,因为与它结合的23S核糖体的92号发夹还参与肽基转移酶中心(PTC)的组成,并且成熟核糖体的92号发夹被埋藏在PTC内。缺失DpbA的功能可能影响PTC的形成,进而影响翻译过程的进行。
本实验结果与Elles等[21]的结果相同,即与野生型BW25113比较,ΔdhpA的倍增时间在ABTGcasa培养基中增加6%,在LB培养基中增加19%左右。表明dbpA缺失的突变体在两种培养基中的生长速度均减慢,且培养基营养越高,生长速度减慢越多。出现这种现象的原因可能是培养基的营养越高,细菌生长速度越快,蛋白质的合成速度也越快,需要的核糖体也越多。但dbpA的缺失导致核糖体无法组装、组装速度慢及组装的核糖体无功能等原因,使细胞内有功能的核糖体数量下降,翻译活性降低,导致细胞内蛋白质合成减少。此外,缺失dpbA还可能影响PTC的形成,影响翻译过程,并导致细胞内蛋白质合成减少。以上因素引起细胞内总蛋白量降低,包括DnaA蛋白,并导致细菌代谢、生长和繁殖速度减慢,出现DNA复制起始延迟、倍增时间延长、细胞体积减小等表型变化。
4 结论dbpA影响大肠杆菌染色体复制的起始,缺失dbpA导致细菌出现DNA复制的延迟启动,生长速度的减慢及细胞体积的减小等变化,并且培养基的营养越高,变化程度也越大。主要原因可能是dbpA的缺失导致核糖体组装缺陷及无法形成,降低了细菌蛋白质的合成,包括DnaA蛋白,出现DNA复制延迟等表型变化。
| [1] |
Diges CM, Uhlenbeck OC. Escherichia coli DbpA is a 3'-> 5'RNA helicase[J]. Biochemistry, 2005, 44(21): 7903-7911. DOI:10.1021/bi050033x |
| [2] |
Sharpe Elles LM, Sykes MT, Williamson JR, et al. A dominant negative mutant of the E. coli RNA helicase DbpA blocks assembly of the 50S ribosomal subunit[J]. Nucleic Acids Res, 2009, 37(19): 6503-6514. DOI:10.1093/nar/gkp711 |
| [3] |
Iggo R, Picksley S, Southgate J, et al. Identification of a putative RNA helicase in E. coli[J]. Nucleic Acids Res, 1990, 18(18): 5413-5417. DOI:10.1093/nar/18.18.5413 |
| [4] |
Henn A, Cao W, Licciardello N, et al. Pathway of ATP utilization and duplex rRNA unwinding by the DEAD-box helicase, DbpA[J]. Proc Natl Acad Sci USA, 2010, 107(9): 4046-4050. DOI:10.1073/pnas.0913081107 |
| [5] |
López-Ramírez V, Alcaraz LD, Moreno-Hagelsieb G, et al. Phylogenetic distribution and evolutionary history of bacterial DEAD-box proteins[J]. J Mol Evol, 2011, 72: 413-431. DOI:10.1007/s00239-011-9441-8 |
| [6] |
Gentry RC, Childs JJ, Gevorkyan J, et al. Time course of large ribosomal subunit assembly in E. coli cells overexpressing a helicase inactive DbpA protein[J]. RNA, 2016, 22(7): 1055-1064. DOI:10.1261/rna.055137.115 |
| [7] |
Skarstad K, Boye E. The initiator protein DnaA:evolution, properties and function[J]. Biochim Biophys Acta, 1994, 1217(2): 111-130. DOI:10.1016/0167-4781(94)90025-6 |
| [8] |
Wechsler JA, Gross JD. Escherichia coli mutants temperature-sensitive for DNA synthesis[J]. Mol Gen Genet, 1971, 113: 273-284. DOI:10.1007/BF00339547 |
| [9] |
Hiraga S, Saito T. Initiation of DNA replication in Escherichia coli. Ⅱ. Effect of rifampicin on the resumption of replication of F episome and chromosome upon the returning of dna mutants from a non-permissive to a permissive temperature[J]. Mol Gen Genet, 1975, 137: 239-248. DOI:10.1007/BF00333019 |
| [10] |
Zyskind JW, Smith DW. Novel Escherichia coli dnaB mutant:direct involvement of the dnaB252 gene product in the synthesis of an origin ribonucleic acid species during initiaion of a round of deoxyribonucleic acid replication[J]. J Bacteriol, 1977, 129: 1476-1486. |
| [11] |
Baba T, Ara T, Hasegawa M, et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants:the Keio collection[J]. Molecular Systems Biology, 2006, 2: 1-11. |
| [12] |
Morigen, Molina F, Skarstad K. Deletion of the datA site does not affect once-per-cell-cycle timing but induces rifampin-resistant replication[J]. J Bacteriol, 2005, 187: 3913-3920. DOI:10.1128/JB.187.12.3913-3920.2005 |
| [13] |
Skarstad K, Boye E, Steen HB. Timing of initiation of chromosome replication in individual Escherichia coli cells[J]. EMBO J, 1986, 5: 1711-1717. DOI:10.1002/embj.1986.5.issue-7 |
| [14] |
Boye E, Løbner-Olesen A. Bacterial growth control studied by flow cytometry[J]. Res Microbiol, 1991, 142: 131-135. DOI:10.1016/0923-2508(91)90020-B |
| [15] |
Miller RV, Ripp S, Replicon J, et al. Virus-mediated gene transfer in freshwater environments[C]. In Gene Transfers and Environment. New York: Springer, 1992: 51-62. https://link.springer.com/chapter/10.1007%2F978-3-642-77450-8_7
|
| [16] |
Morigen, Løbner-Olesen A, Skarstad K. Titration of the Escherichia coli DnaA protein to excess datA sites causes destabilization of replication forks, delayed replication initiation and delayed cell division[J]. Molecular Microbiology, 2003, 50: 349-362. DOI:10.1046/j.1365-2958.2003.03695.x |
| [17] |
Morigen, Boye E, Skarstad K, et al. Regulation of chromosomal replication by DnaA protein availability in Escherichia coli:effects of the datA region[J]. Biochim Biophys Acta, 2001, 1521: 73-80. DOI:10.1016/S0167-4781(01)00292-5 |
| [18] |
Jain C. The E. coli RhlE RNA helicase regulates the function of related RNA helicases during ribosome assembly[J]. RNA, 2008, 14: 381-389. |
| [19] |
Iost I, Dreyfus M. DEAD-box RNA helicases in Escherichia coli[J]. Nucleic Acids Res, 2006, 34(15): 4189-4197. DOI:10.1093/nar/gkl500 |
| [20] |
Peil L, Virumäe K, Remme J. Ribosome assembly in Escherichia coli strains lacking the RNA helicase DeaD/CsdA or DbpA[J]. FEBS J, 2008, 275(15): 3772-3782. DOI:10.1111/j.1742-4658.2008.06523.x |
| [21] |
Elles LM, Uhlenbeck OC. Mutation of the arginine finger in the active site of Escherichia coli DbpA abolishes ATPase and helicase activity and confers a dominant slow growth phenotype[J]. Nucleic Acids Res, 2008, 36(1): 41-50. |