2. 中国科学技术大学 生命科学学院,合肥 230026
2. School of Life Sciences, University of Science and Technology of China, Hefei 230026
在高等生物中,DNA是遗传信息的主要载体。真核生物的DNA并不是裸露的,而是与细胞内的四种富含碱性氨基酸的组蛋白结合形成直径大约10 nm的核小体。核小体是处于动态的变化中,它不断的组装与去组装,同时形成不同的变体核小体[1]。在核小体上存在着许多翻译后修饰,包括甲基化、泛素化、乙酰化、SUMO化和糖基化等多种修饰,这些修饰的写入和擦除是基因调控的重要方式[2-3]。核小体重塑与去乙酰化酶(Nucleosome remodeling and deacetylation,NuRD)复合物是一种具有核小体重塑及组蛋白去乙酰化功能的多亚基蛋白质复合物,其核心组分包括CHD3/4、HDAC1/2、MBD2、MBD3、MTA1/2/3、GATAD2A/B和RBBP4/7[4-6]。NuRD广泛参与基因转录调控,信号转导,细胞发育以及细胞周期调控等一系列生命活动[7-9]。NuRD参与细胞活动的过程中,能够与不同的结合底物相互作用共同完成调节功能。NuRD的核心组分GATAD2A/B和RBBP4/7作为媒介能介导NuRD与结合底物之间的相互作用。其中,RBBP4也是多梳抑制复合物2(Polycomb repressive complex 2,PRC2)的组分之一。作为NuRD和PRC2两种复合物中的共同亚基,RBBP4已经鉴定出多种结合底物帮助二者实现调节功能[10-13]。在这些结合底物中,有一类结合底物可以通过其自身的一段保守的富含精氨酸和赖氨酸的区域参与RBBP4的相互作用,这段富含碱性氨基酸的序列通常以RXXP模块的形式出现[10]。RBBP4全长425个氨基酸,属于WD40蛋白质,由氮端的长α螺旋和一个七螺旋桨组成[14],如图 1-A所示。上述的RXXP模块与RBBP4之间存在一种保守的相互作用模式,模块中的一个精氨酸将其侧链伸入RBBP4顶端的口袋之中,与RBBP4上的残基形成氢键和疏水性的相互作用。
|
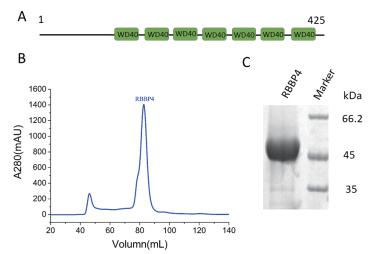
| 图 1 RBBP4的体外纯化与鉴定 A:RBBP4的结构域分布;B:RBBP4的Superdex 200凝胶过滤柱层析洗脱曲线;C:RBBP4的SDS-PAGE凝胶电泳图 |
ZMYND8包含一个PHD结构域,一个BROMO结构域,一个PWWP结构域和一个碳端的MYND结构域,如图 2-A所示。在细胞内,ZMYND8主要参与调节同源重组相关的DNA损伤修复以及癌细胞分化与侵袭[15]。最初的研究发现,它在T细胞淋巴瘤中的胚胎神经分化过程中发挥着重要功能[16]。在癌症相关的生命活动中,ZMYND8是全反式维甲酸(All-trans-retinoic acid,ATRA)介导的抗肿瘤细胞增生过程中的底物蛋白[17-18]。ZMYND8还可以与H3K4me3的特异性去甲基化酶KDM5C结合来共同调控增强子的活性,从而抑制部分癌症相关基因的过表达[19]。作为H3K4去甲基转移酶JARID1D转录的共抑制因子,ZMYND8可以通过其PHD和BROMO结构域组合而成的识别元件识别H3K4me1-H3K14ac来抵抗与癌细胞转移相关基因的表达[20]。ZMYND8的多个串联结构域构成刚性的元件,识别组蛋白的多种修饰[20-21]。在体内,ZMYND8相较于变体H3能够优先识别传统的H3上的K36me2。此外,ZMYND8还能够识别的H4K16ac[17]。在转录过程中,ZMYND8响应DNA损伤后,通过其BROMO结构域结合在H4K16ac上,进而招募NuRD至损伤区域抑制转录的进行,促进DNA双链断裂的修复[22]。已经发现的ZMYND8参与招募NuRD的相互作用方式是ZMYND8能够通过自身碳端的MYND锌指结构域与NuRD亚基GATAD2A中的PPPL模块相互作用来确保二者共定位于DNA损伤位点[23]。ZMYND8和NuRD之间是否还存在更广泛的相互作用来实现二者的共定位有待于进一步的研究。
本实验通过对ZMYND8一级序列的分析,发现ZMYND8上存在5段可能与RBBP4相互作用的富含碱性氨基酸残基的区域。重组蛋白RBBP4与体外合成的小肽的等温量热滴定实验结果显示RBBP4可以特异性地结合ZMYND843-54区域,亲和力为7.9 μmol/L。根据已知的RBBP4与其他小肽底物的复合物模型,对RBBP4上可能参与相互作用的残基进行了点突变实验,初步确定了ZMYND43-54与RBBP4的相互作用界面。结合以往的研究成果,我们提出了ZMYND8和NuRD在体内发挥功能时的结合模型。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和细胞克隆宿主菌Top10,感受态DH10ac,重组质粒pFastBacTM,来自粉纹夜蛾(Trichopulsia ni)的High Five细胞,上述材料均由本实验室保存。
1.1.2 培养基大肠杆菌的培养采用LB培养基;昆虫细胞的培养基采用ESF921培养基,购于上海生工生物工程股份有限公司。
1.1.3 试剂PCR的引物合成和重组质粒的测序均有南京金斯瑞公司完成;DNA聚合酶PimerSTAR MAX和连接酶混合体系Solution Ⅰ购于TaKaRa公司;DNA限制性内切酶,DNA Marker和蛋白质Marker购于Thermo fisher scientific公司;DNA胶回收试剂盒E.Z.N.A购于美国Omega Bio-tek公司;质粒抽提试剂盒购于Axygen公司;其他实验过程中使用到的试剂均为进口或国产的分析纯试剂。
1.2 方法 1.2.1 RBBP4的昆虫细胞表达利用PCR技术从人脑cDNA文库中扩增出RBBP4的全长基因。目的基因片段经Nde Ⅰ和Xho Ⅰ双酶切后亚克隆至pFastBac质粒中,将重组的质粒转化至DH10Bac感受态细胞中。准备三抗(50 μg/mL卡那霉素,7 μg/mL庆大霉素,10 μg/mL四环素)平板,平板上添加40 μL X-gal(20 mg/mL)和4 μL IPTG(200 mg/mL)。将转化液涂布到平板上,避光37℃培养36-48 h。待平板上的蓝白斑区分明显之后,挑取白斑接种至三抗的液体LB培养基中。鉴定片段插入成功后,抽提Bacmid转染High Five细胞。逐步扩增获得滴度高的P3代杆状病毒。悬浮培养High Five细胞至浓度1.0×106 cells/well,添加适合体积的P3代病毒,27℃,115 r/min摇床培养3-4 d,收集细胞。
1.2.2 RBBP4的体外纯化收集的昆虫细胞,超声破碎,超速离心后收集上清液。上清液转移至Ni-NTA柱纯化。纯化流程如下:缓冲液A(20 mmol/L Tris pH 8.0,500 mmol/L NaCl)平衡Ni-NTA柱。上清液流穿后,用含有20 mmol/L到100 mmol/L咪唑的缓冲液梯度清洗杂蛋白,最后用500 mmol/L的咪唑洗脱目的蛋白。洗脱液浓缩后,进一步Hiload 16/60 Superdex 200(GE Healthcare)柱纯化。目的蛋白峰收集透析至缓冲液B(20 mmol/L Tris pH 7.4,150 mmol/L NaCl)备用。
1.2.3 小肽合成实验中所使用的蛋白质小肽均由吉尔生化(上海)有限公司合成,纯度在95%以上。
1.2.4 等温量热滴定实验ITC使用的仪器为ITC2000(GE Healthcare)。实验中,小肽的浓度在1-3 mmol/L,蛋白质浓度为20-60 μmol/L。小肽和蛋白质均在缓冲液B中,实验温度20℃,滴定次数为20次。实验数据的处理使用的是ITC2000自带的Origin软件。
2 结果 2.1 RBBP4的体外纯化与鉴定在大肠杆菌表达体系中,无法在上清中获得可溶的RBBP4蛋白质。本实验利用昆虫细胞表达系统,能够在上清溶液中检测到目标蛋白质的表达。随后的Superdex 200凝胶过滤柱层析纯化过程中,RBBP4在波长280 nm的层析图谱显示(图 1-B),在82.59 mL处出现重组蛋白质的单一峰。纯化后的RBBP4经SDS-PAGE检测,结果(图 1-C)显示为均一的条带,表明目的蛋白质已经达到电泳纯,其相对分子质量大约在45 kD左右。这与理论计算的重组RBBP4的分子量相近,结合RBBP4在层析图中的出峰位置,表明RBBP4在溶液中呈现单体的状态。
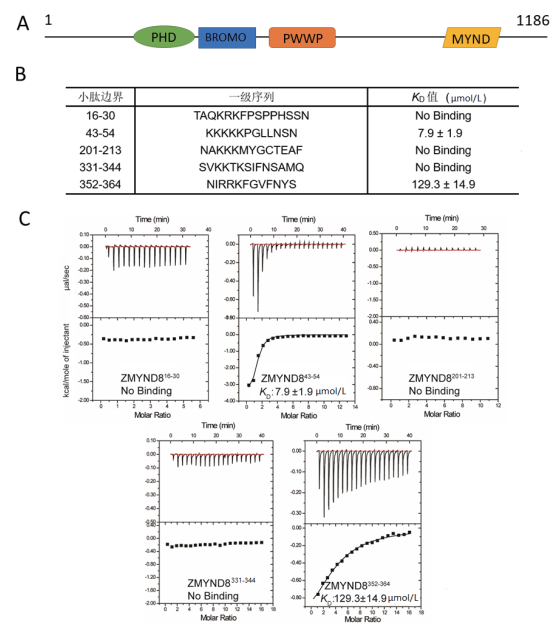
2.2 筛选ZMYND8上与RBBP4相互作用区域ZMYND8的全长序列分析结果(图 2-B)显示,5段富含精氨酸和赖氨酸的碱性区域具有与RBBP4结合的潜力。这些区域包括氨基酸残基:16-30(ZMYND816-30),43-54(ZMYND843-54),201-213(ZMYND8201-213),331-344(ZMYND331-344)和352-364(ZMYND8352-364)。基于上述序列,化学合成相应的小肽,利用ITC技术来检测小肽与RBBP4的相互作用。
|
| 图 2 鉴定ZMYND8上与RBBP4相互作用的区域 A:ZMYND8结构域分布图;B:不同小肽的序列及与RBBP4的结合常数(KD);C:RBBP4与各小肽的ITC滴定结果 |
ITC的滴定实验结果(图 2-B和2-C)显示:RBBP4不结合小肽ZMYND816-30,ZMYND8201-213和ZMYND8331-344。ZMYND843-54对RBBP4表现出强的结合能力,拟合的解离常数(KD)为7.9±1.9 μmol/L。此外,ZMYND8352-364滴定RBBP4的KD值为129.3±14.9 μmol/L,表明其对RBBP4呈现非常弱的亲和力。ZMYND843-54对RBBP4结合能力与已经报道的体内的结合底物对RBBP4的亲和力一致,量级在10 μmol/L左右。
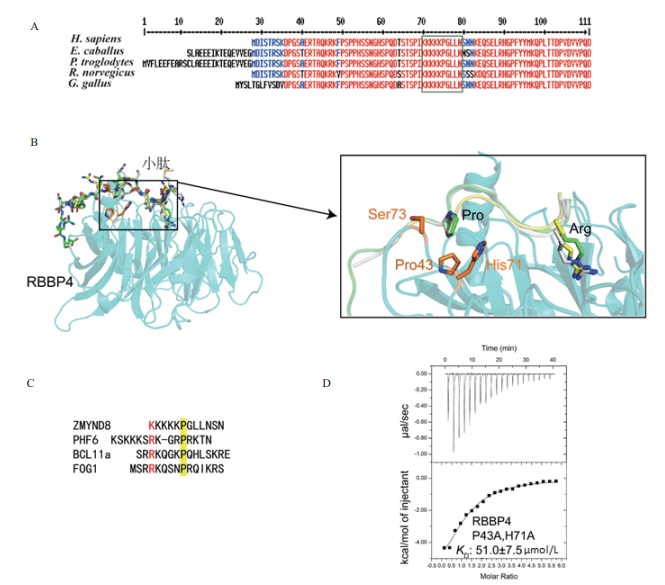
2.3 ZMYND843-54与RBBP4结合界面的初步鉴定ZMYND843-54位于蛋白质氮端的无序区域,与RBBP4的结合能力体现了此区域对ZMYND8行使功能的重要性。将不同种属的ZMYND8氮端序列进行比对,结果(图 3-A)显示从低等生物到高等生物,不同种属的ZMYND8氮端区域都对人源的ZMYND843-54表现出非常强的保守性。这也暗示了这段区域在不同种属中都能介导保守的相互作用。分析已经报道的RBBP4与底物小肽的晶体结构,二者之间存在两种保守的相互作用方式。如图 3-B显示:第一种:不同的结合小肽包含一个保守的精氨酸,其侧链深入RBBP4顶端的口袋内,与RBBP4上的181位的络氨酸和321位的苯丙氨酸形成π键,同时还能与231位的谷氨酸和277位的天冬酰胺形成氢键和静电相互作用;第二种:不同的结合小肽中均包含另一个保守的脯氨酸结合在RBBP4上的43位的脯氨酸、71位的组氨酸以及73位的丝氨酸构成的疏水口袋中。
|
| 图 3 ZMYND843-54与RBBP4结合界面的初步鉴定 A:不同种属(Homo sapiens、Equus caballus、Pan troglodytes、Rattus norvegicus和Gallus gallus)的ZMYND8的氮端序列的一级序列比对,绿色方框内残基为RBBP4相互作用区域;B:RBBP4与PHF6(绿色)、BCL11a(灰色)和FOG1(黄色)的复合物结构比对;C:ZMYND8、PHF6、BCL11a和FOG1参与RBBP4结合的小肽序列比对;D:RBBP4突变体(P43A/H71A)与ZMYND843-54小肽的ITC滴定结果 |
以此种方式与RBBP4结合的底物包括PHF6、BCL11a和FOG1。ZMYND843-54与这些结合底物的序列比对结果(图 3-C)显示,ZMYND843-54可能属于此类结合模式,特别是ZMYND8上第48脯氨酸表现出严格的保守性。为了进一步通过实验确定其结合位点,我们对RBBP4上可能与ZMYND8的48位脯氨酸存在相互作用的第43位的脯氨酸及第71位的组氨酸进行了双点突变实验。ZMYND843-54对RBBP4(P43A/H71A)突变体的亲和力为51.0±7.5 μmol/L,与野生型的RBBP4的亲和力相比下降了约6.5倍左右(图 3-D)。上述的实验结果表明ZMYND843-54结合在RBBP4的顶端区域。
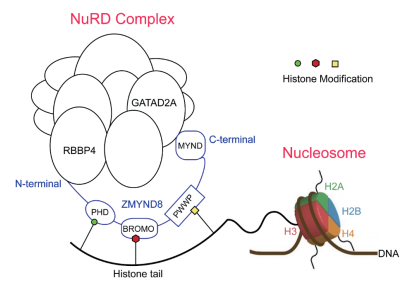
2.4 体内ZMYND8与NuRD结合模型ZMYND8是一个具有多个串联结构域的蛋白质。近期的研究显示,ZMYND8能够通过自身碳端的锌指MYND结构域与NuRD复合物的GATAD2A中的PPPL模块相互作用来帮助NuRD招募至DNA损伤位点。我们的实验结果发现ZMYND8氮端的43-54氨基酸残基能够与NuRD的亚基RBBP4结合,且结合界面与已经发现的结合底物PHF6,BCL11a和FOG1一致。综合所有的研究发现,ZMYND8与NuRD的结合模型,如图 4所示,ZMYND8通过氮端的43-54氨基酸残基与亚基RBBP4结合,同时其碳端的MYND结构域与亚基GATA2A结合。这样的相互作用模式能够使得ZMYND8以舒展的方式缠绕在NuRD的表面。此后,位于ZMYND8中间的PHD、BROMO和PWWP结构域能够进一步识别串联排列的各种组蛋白修饰,如H3K4me1、H3K14ac和H4K16ac等。
|
| 图 4 ZMYND8与NuRD体内结合模型 |
以往的研究表明NuRD复合物与DNA损伤有关,它能够辅助DNA损伤的响应[24-26]。然而对于NuRD如何定位到损伤的DNA区域的研究还不够充分。ZMYND8是一个既能结合染色质又能与NuRD相互作用的重要媒介。它与NuRD一起形成复合物来帮助损伤修复的响应。前期的研究表明ZMYND8中的MYND结构域能够识别NuRD亚基GATA2A中的PPPL模块,来实现二者的共定位[23]。然而上述的研究并未充分的阐述ZMYND8与NuRD二者之间广泛的相互作用模式。RBBP4是NuRD的一个重要的核心亚基,本实验体外验证了它能够通过自身β桶结构的顶端区域结合位于ZMYND8氮端的43-54位残基。这样的相互作用可能与DNA损伤响应过程中NuRD的招募有关。总结已经报道的实验结果,NuRD与底物蛋白的结合是通过多个亚基实现的。在DNA损伤修复的过程中,ZMYND8通过其氮端43-54位残基和碳端的MYND结构域来介导与NuRD之间的相互作用,这样的结合方式能够使得ZMYND8更加刚性和牢固的结合在NuRD的周围。而上述两种相互作用的功能意义还有待于我们进一步的探索研究。
分析RBBP4与底物小肽的结合特点,保守的结合模式为小肽中的一个精氨酸伸入RBBP4的顶端口袋中,同时距离其3到4位存在一个保守的脯氨酸,这种序列特点可被归纳为RXXP模块。这种保守的结合模式也为寻找新的RBBP4结合底物提供一定的依据。然而,此次研究鉴定出ZMYND8上与RBBP4的相互作用区域的43-54残基中并不包含保守的精氨酸,而是含有一个替代精氨酸的赖氨酸。这一结果也说明传统的RXXP模块并不是RBBP4识别的唯一模块类型,替代的赖氨酸同样能够完成识别RBBP4的作用。
4 结论本研究利用Bac-to-Bac昆虫杆状病毒表达系统,重组表达了人源NuRD的核心亚基RBBP4蛋白。经过镍柱以及分子筛纯化,获得了电泳纯的重组目的蛋白。同时,通过ITC实验我们鉴定出ZMYND843-54是介导与RBBP4相互作用的区域。利用氨基酸残基的点突变实验,初步确定了二者相互作用的界面。新发现的结合区域完善了ZMYND8与NuRD复合物相互作用的模式。上述的研究发现有助于我们更加全面的理解NuRD复合物如何与ZMYND8一起共同参与到转录调控和DNA损伤修复等一系列生命活动中。
| [1] |
Rhee HS, Bataille AR, Zhang LY, et al. Subnucleosomal structures and nucleosome asymmetry across a genome[J]. Cell, 2014, 159(6): 1377-1388. DOI:10.1016/j.cell.2014.10.054 |
| [2] |
Rodriguez-Paredes M, Esteller M. Cancer epigenetics reaches mainstream oncology[J]. Nature Medicine, 2011, 17(3): 330-339. DOI:10.1038/nm.2305 |
| [3] |
Tan MJ, Luo H, Lee S, et al. Identification of 67 histone marks and histone lysine crotonylation as a new type of histone modification[J]. Cell, 2011, 146(6): 1015-1027. |
| [4] |
Lai AY, Wade PA. Cancer biology and NuRD: a multifaceted chromatin remodelling complex[J]. Nature Reviews Cancer, 2011, 11(8): 588-596. DOI:10.1038/nrc3091 |
| [5] |
Low JKK, Silva APG, Sharifitabar M, et al. Architecture of the nucleosome remodeling and deacetylase(NuRD)complex[J]. Faseb Journal, 2017, 31. |
| [6] |
Allen HF, Wade PA, Kutateladze TG. The NuRD architecture[J]. Cellular and Molecular Life Sciences, 2013, 70(19): 3513-3524. DOI:10.1007/s00018-012-1256-2 |
| [7] |
Denslow SA, Wade PA. The human Mi-2/NuRD complex and gene regulation[J]. Oncogene, 2007, 26(37): 5433-5438. DOI:10.1038/sj.onc.1210611 |
| [8] |
Ahringer J. NuRD and SIN3 - histone deacetylase complexes in development[J]. Trends in Genetics, 2000, 16(8): 351-356. DOI:10.1016/S0168-9525(00)02066-7 |
| [9] |
Luo M, Ling T, Xie WB, et al. NuRDBlocks reprogramming of mouse somatic cells into pluripotent stem cells[J]. Stem Cells, 2013, 31(7): 1278-1286. DOI:10.1002/stem.v31.7 |
| [10] |
Liu ZH, Li FD, Zhang BB, et al. Structural basis of plant homeodomain finger 6(PHF6)recognition by the retinoblastoma binding protein 4(RBBP4)component of the nucleosome remodeling and deacetylase(NuRD)complex[J]. J Biol Chem, 2015, 290(10): 6630-6638. DOI:10.1074/jbc.M114.610196 |
| [11] |
Moody RR, Lo MC, Meagher JL, et al. Probing the interaction between the histone methyltransferase/deacetylase subunit RBBP4/7 and the transcription factor BCL11A in epigenetic complexes[J]. J Biol Chem, 2018, 293(6): 2125-2136. DOI:10.1074/jbc.M117.811463 |
| [12] |
Chen SM, Jiao LY, Shubbar M, et al. Unique structural platforms of Suz12 dictate distinct classes of PRC2 for chromatin binding[J]. Molecular Cell, 2018, 69(5): 840-852. DOI:10.1016/j.molcel.2018.01.039 |
| [13] |
Lejon S, Thong SY, Murthy A, et al. Insights into association of the NuRD complex with FOG-1 from the crystal structure of an RbAp48. FOG-1 complex[J]. J Biol Chem, 2011, 286(2): 1196-1203. DOI:10.1074/jbc.M110.195842 |
| [14] |
Xu C, Min JR. Structure and function of WD40 domain proteins[J]. Protein & Cell, 2011, 2(3): 202-214. |
| [15] |
Gong FD, Miller KM. Double duty: ZMYND8 in the DNA damage response and cancer[J]. Cell Cycle, 2018, 17(4): 414-420. DOI:10.1080/15384101.2017.1376150 |
| [16] |
Zeng WL, Kong QH, Li CC, et al. Xenopus RCOR2(REST corepressor 2)interacts with ZMYND8, which is involved in neural differentiation[J]. Biochemical and Biophysical Research Communications, 2010, 394(4): 1024-1029. DOI:10.1016/j.bbrc.2010.03.115 |
| [17] |
Adhikary S, Sanyal S, Basu M, et al. Selective recognition of H3. 1K36 dimethylation/H4K16 acetylation facilitates the regulation of all-trans-retinoic acid(ATRA)-responsive genes by putative chromatin reader ZMYND8[J]. J Biol Chem, 2016, 291(6): 2664-2681. DOI:10.1074/jbc.M115.679985 |
| [18] |
Basu M, Khan MW, Chakrabarti P, et al. Chromatin reader ZMYND8 is a key target of all trans retinoic acid-mediated inhibition of cancer cell proliferation[J]. Biochimica Et Biophysica Acta-Gene Regulatory Mechanisms, 2017, 1860(4): 450-459. DOI:10.1016/j.bbagrm.2017.02.004 |
| [19] |
Shen HJ, Xu WQ, Guo R, et al. Suppression of enhancer overactivation by a RACK7-Histone cemethylase complex[J]. Cell, 2016, 165(2): 331-342. DOI:10.1016/j.cell.2016.02.064 |
| [20] |
Li N, Li YY, Lv J, et al. ZMYND8 Reads the dual histone mark H3K4me1-H3K14ac to antagonize the expression of metastasis-linked genes[J]. Molecular Cell, 2016, 63(3): 470-484. DOI:10.1016/j.molcel.2016.06.035 |
| [21] |
Savitsky P, Krojer T, Fujisawa T, et al. Multivalent histone and DNA engagement by a PHD/BRD/PWWP triple reader cassette recruits ZMYND8 to K14ac-Rich chromatin[J]. Cell Reports, 2016, 17(10): 2724-2737. DOI:10.1016/j.celrep.2016.11.014 |
| [22] |
Gong FD, Chiu LY, Cox B, et al. Screen identifies bromodomain protein ZMYND8 in chromatin recognition of transcription-associated DNA damage that promotes homologous recombination[J]. Genes & Development, 2015, 29(2): 197-211. |
| [23] |
Spruijt CG, Luijsterburg MS, Menafra R, et al. ZMYND8 co-localizes with NuRD on target genes and regulates poly(ADP-Ribose)-dependent recruitment of GATAD2A/NuRD to sites of DNA damage[J]. Cell Reports, 2016, 17(3): 783-798. DOI:10.1016/j.celrep.2016.09.037 |
| [24] |
Smeenk G, Wiegant WW, Vrolijk H, et al. The NuRD chromatin-remodeling complex regulates signaling and repair of DNA damage[J]. Journal of Cell Biology, 2010, 190(5): 741-749. DOI:10.1083/jcb.201001048 |
| [25] |
Chou DM, Adamson B, Dephoure NE, et al. A chromatin localization screen reveals poly(ADP ribose)-regulated recruitment of the repressive polycomb and NuRD complexes to sites of DNA damage[J]. Proc Natl Acad Sci USA, 2010, 107(43): 18475-18480. DOI:10.1073/pnas.1012946107 |
| [26] |
Li DQ, Kumar R. Mi-2/NuRD complex making inroads into DNA-damage response pathway[J]. Cell Cycle, 2010, 9(11): 2071-2079. DOI:10.4161/cc.9.11.11735 |




