2. 亚热带农业生物资源保护与利用国家重点实验室,南宁 530005
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources, Nanning 530005
甘蔗是我国重要的糖料作物和新型能源作物,甘蔗对低温胁迫较为敏感,低温寒害常给甘蔗生产带来了巨大的经济损失[1]。选育高产优质抗寒性强的品种,可提高甘蔗抵御寒害的能力,降低蔗糖产业生产中的经济损失。目前甘蔗杂交育种是获得优良品种的主要途径[2],但甘蔗常规育种技术所需时间长,盲目性大。随着现代生物技术的飞速发展,从分子水平改良甘蔗新品种成为了可能[3]。由于甘蔗是高度复杂的多倍体,遗传背景十分复杂,目前甘蔗全基因组信息匮乏,现代生物技术辅助甘蔗常规育种的能力和幅度受限,甘蔗现代分子育种急需加强。转录组是一个生物体中所有基因表达后产物的综合[4],包括转录本的数量、特定发育阶段的表达动态、转录后的修饰以及非编码RNA的调控表达情况等,它可以从整体水平上研究基因的功能和结构,挖掘优良性状相关基因。转录组测序技术,是通过深度测序后进行转录组分析的技术[5-6],转录组测序能够在单核苷酸水平上对任意物种进行检测[7],它可依赖或不依赖于物种的参考基因组,因而为甘蔗等非模式生物的转录组研究提供了新的手段[8]。本研究在前期的抗寒形态变化、抗寒光合生理等的研究基础之上[9],选取抗寒性具有一定差异的桂糖08-1180和ROC22为材料,采用新一代高通量Illumina转录组测序技术,对2个不同抗性甘蔗基因型在低温胁迫和常温处理下的转录组进行分析,研究和比较2个甘蔗品种低温胁迫下基因表达的差异,分析与甘蔗抗寒相关的基因,为甘蔗抗寒基因挖掘、抗寒分子育种奠定基础。
1 材料与方法 1.1 材料以桂糖08-1180和ROC22号为材料,每个品种种植4桶,每桶种植4个蔗芽,淋足水分,待苗长出后,每桶留甘蔗4株。
1.2 方法 1.2.1 实验处理当甘蔗长至10片叶龄时,进行实验处理。实验设4个处理:桂糖08-1180常温(G1180_CW)、桂糖08-1180低温(G1180_DW)、ROC22常温(ROC22_CW)、ROC22低温(ROC22_DW),每处理设2个生物学重复。常温处理为甘蔗在自然条件下的温度处理,日温度变化幅度在23.0℃至35.0℃之间。低温处理为甘蔗在人工气候箱中进行4℃低温处理,常温和低温的处理时间均为36 h。处理完毕后,立即剪取+1叶叶片,进行液氮速冻处理。
1.2.2 RNA提取及样品检测采用RNAprep pure多糖多酚植物总RNA提取试剂盒(天根)进行提取,琼脂糖凝胶电泳检测RNA的降解及污染程度,Nanodrop检测RNA纯度,Qubit Fluorometer定量RNA浓度,Agilent 2100检测RNA的完整性。
1.2.3 cDNA文库构建及测序用带Oligo(dT)的磁珠富集mRNA,之后加入fragmentation buffer溶液将mRNA打断,以打断的mRNA为模板,用六碱基随机引物合成一链cDNA,然后加入缓冲液、dNTPs和DNA polymerase Ⅰ合成二链cDNA,得到cDNA文库。库检合格后,由北京诺禾致源生物公司进行Illumina HiSeq测序。
1.2.4 转录组原始数据的读取及拼接对Illumina高通量测序获得的原始图像数据,经CASAVA碱基识别分析后,转化为原始测序序列,称之为原始读子(Raw reads)。对原始读子去除接头、去除N(N表示无法确定碱基的信息)和去除低质量reads,获得clean reads。由于目前甘蔗的全基因组测序信息尚未见公布,本研究在获得clean reads后,采用Trinity软件进行de novo从头拼接。对拼接得到的转录本序列,取最长的转录本作为该基因的Unigene,对所有转录本及Unigene进行统计,并以此为基础进行后续的生物信息分析。
1.2.5 基因功能注释使用Blast方法,在Nr、Nt、KOG/COG、Swiss-prot、Pfam、GO、KEGG数据库对所有的Unigene进行注释。若在这些数据库中未能比对上的Unigene,采用estscan(3.0.3)软件预测其ORF。
1.2.6 基因表达水平的分析采用RSEM软件,以Trinity拼接获得的转录组作为参考序列(ref),将每个样品的clean reads往参考序列(ref)上做映射(mapping),并对其进行FPKM转换,分析各基因的表达水平。本研究采用DESeq进行基因差异表达分析,设定的筛选阈值为:padj < 0.05。
1.2.7 基因GO富集和KEGG富集分析采用GOseq进行GO富集分析。使用KOBAS(2.0)进行KEGG Pathway富集分析。
2 结果 2.1 测序数据及质量情况从表 1可以看出,本实验8个样品共获得394 907 890条raw reads,过滤后共获得382 684 356条clean reads,测序产生的clean reads的碱基数合计为57.41 G。碱基错误率均在0.02%的较低水平,Q20值和Q30值均较高,GC含量在53.28%-55.16%之间。
从表 2可以看出,对8个样品拼接后,共获得360 404条转录本和183 515个Unigenes,平均长度分别为1 073 bp和741 bp,N50长度分别为1 903 bp和1 284 bp。
从表 3可以看出,在NR、NT、KO、Swiss-Prot、PFAM、GO和KOG数据库中,对所有unigenes进行比对,有110 021个unigenes能在7个数据库中的至少1个数据库里显著匹配,注释成功率为59.95%。10 825个unigenes在7个数据库中均能匹配,占总unigenes的5.89%。
从图 1可以看出,低温胁迫下桂糖08-1180有8 965个上调表达基因,7 180个下调表达基因。ROC22有10 898个上调表达基因,9 419个下调表达基因。
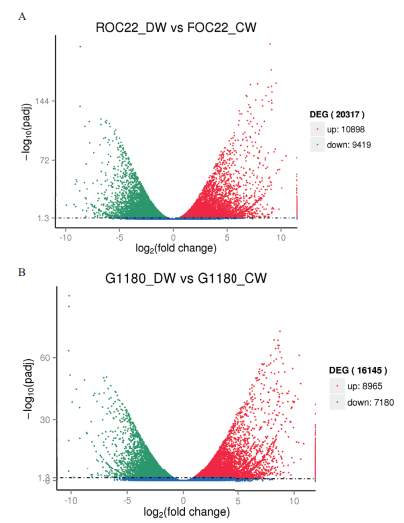
|
| 图 1 基因差异表达的火山图分析 图中散点为各个基因,蓝色圆点为差异不显著基因,红色圆点为差异显著的上调基因,绿色圆点为差异显著的下调基因 |
本研究进一步对2个品种的特有差异表达基因及共有差异表达基因进行分析,结果如图 2,可以看出,有13 113个差异表达基因为2个品种共有。7 204个差异表达基因只在ROC22品种中出现,为ROC22特有。而3 032个差异表达基因只在桂糖08-1180品种中出现,为桂糖08-1180特有。
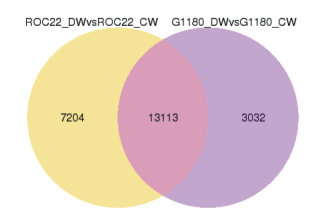
|
| 图 2 所有差异基因维恩图 ROC22_DWvsROC22_CW表示以常温为对照,ROC22低温胁迫下的差异表达基因组。G1180_DWvsG1180_CW表示以常温为对照,桂糖08-1180低温胁迫下的差异表达基因组 |
对桂糖08-1180和ROC22共有的差异表达基因进行GO分析,结果如图 3,可以看出,共有差异表达基因富集在生物过程大类中的2个小类,分别是非生物刺激反应和低温反应。
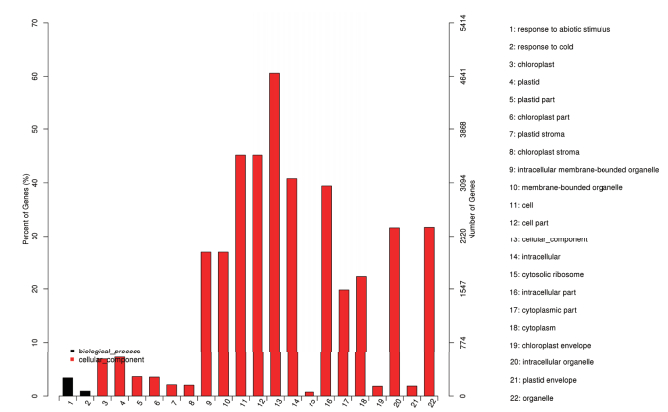
|
| 图 3 桂糖08-1180和ROC22共有差异表达基因GO富集分析 生物过程:1:非生物刺激反应;2:低温反应。细胞成分:3:叶绿体;4:质体;5质体组分;6:叶绿体组分;7:质体间质;8:叶绿体间质;9:细胞膜内的细胞器;10:膜结合细胞器;11:细胞;12:细胞部件;13:细胞组分;14:细胞内;15:胞质核糖体;16:细胞内组分;17:胞浆部分;18:细胞浆;19:叶绿体膜;20:胞内细胞器;21:质膜;22:细胞器 |
共有差异表达基因还富集在细胞成分大类中的20个小类,其中细胞组分、细胞、细胞部件中富集的差异基因较多。与光合作用相关的叶绿体、质体等富集的基因较多。
对共有的差异表达基因进行KEGG Pathway分析,共有的差异表达基因富集在前20条代谢通路的结果如图 4,可以看出,富集差异基因数最多的途径是核糖体,光合作用天线蛋白、光合作用等与光合作用相关的pathway中富集的差异基因也较多,说明低温对光合作用的代谢途径产生了较大的影响。

|
| 图 4 桂糖08-1180和ROC22共有的差异表达基因的KEGG富集结果 |
对桂糖08-1180和ROC22低温胁迫下特有的差异表达基因进行GO富集分析,结果如图 5和图 6,可以看出,桂糖08-1180的特有差异表达基因在生物过程大类的17个小类得到富集,其中DNA代谢过程、DNA整合、DNA重组中富集的差异基因较多;细胞成分大类的4个小类也得到富集,其中,以RNA为目标的RNA聚合酶复合物中富集的差异基因较多;分子功能大类的9个小类得到富集,其中,在ADP结合、天冬氨酸型内肽酶活性、天冬氨酸型肽酶活性中富集的差异基因较多。

|
| 图 5 桂糖08-1180特有差异表达基因的GO富集分析 生物过程:1:精氨酸生物合成过程;2:趋药性;3:DNA整合;4:RNA和蛋白质共价交联;5:排序;6:转位,DNA介导;7:转位;8:核苷酸代谢过程;9:磷酸核苷分解过程;10:细胞部件形态;11:细胞突起形态发生;12:DNA重组;13:DNA代谢过程;14:老化;15:乙醛酸代谢过程;16:谷氨酰胺家族氨基酸生物合成方法;17:精氨酸代谢过程。细胞成分:18:叶绿体核酮糖二磷酸羧化物;19:核酮糖二磷酸羧化酶复合物;20:以RNA为目标的RNA聚合酶复合物;21:中心体。分子功能:22:核酮糖二磷酸羧化酶活性;23:ADP结合;24:乙酰辅酶A:L-谷氨酸N-乙酰基转移酶活性;25:核糖核酸聚合酶活性;26:烟酸-核苷酸-二甲基苯并咪唑-磷酸核糖转移酶活性;27:CDP-甘油二酯二磷酸酶活性;28:天冬氨酸型内肽酶活性;29:天冬氨酸型肽酶活性;30:转座酶 |
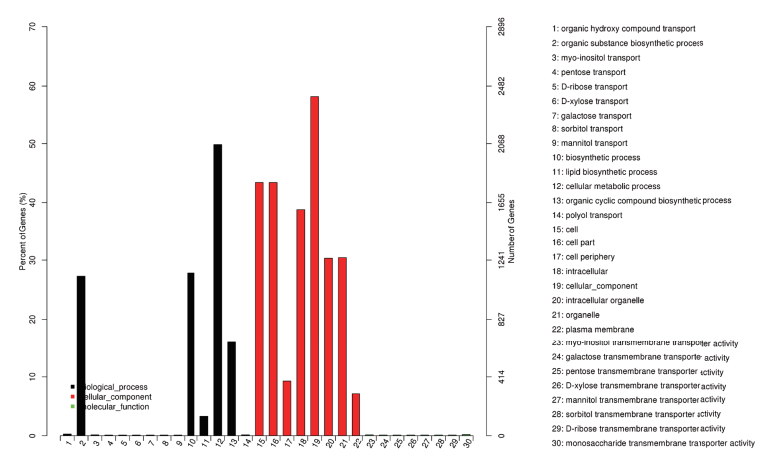
|
| 图 6 ROC22特有差异表达基因的GO富集分析 生物过程:1:有机羟基化合物转运;2:有机物生物合成过程;3:肌醇运输;4:戊糖运输;5:D-核糖的运输;6:木糖运输;7:半乳糖运输;8:山梨醇运输;9:甘露醇运输;10:生物合成过程;11:脂质的生物合成过程;12:细胞代谢过程;13:有机环化合物生物合成方法;14:多元醇的运输。细胞成分:15:细胞;16:细胞组分;17:细胞周边;18:细胞内;19:细胞内成分;20:胞内细胞器;21:细胞器;22:质膜。分子功能:23:肌醇跨膜转运蛋白活性;24:半乳糖跨膜转运蛋白活性;25:戊糖的跨膜转运活性;26:D-木糖的跨膜转运活性;27:甘露醇跨膜转运蛋白活性;28:山梨醇跨膜转运活性;29:D-核糖的跨膜转运活性;30:单糖跨膜转运活性 |
ROC22的特有差异表达基因富集在生物过程大类的14个小类中,其中细胞代谢过程、生物合成过程、有机物生物合成过程富集的差异基因较多;细胞成分大类的8个小类也得到富集,其中细胞内成分、细胞、细胞组分富集的差异基因较多;分子功能大类的8个小类得到富集,但富集的差异表达基因均较少。
2.5.3 桂糖08-1180和ROC22特有差异表达基因KEGG富集分析从图 7可以看出,桂糖08-1180特有差异表达基因富集的前20条KEGG通路中,植物激素信号转导中富集的差异基因数最多,淀粉与蔗糖的代谢、苯丙素生物合成富集的基因较多。而ROC22特有差异表达基因富集的前20条KEGG通路中,植物激素信号转导、甘油磷酯代谢、嘧啶代谢、半胱氨酸和蛋氨酸代谢等富集的差异基因数较多。
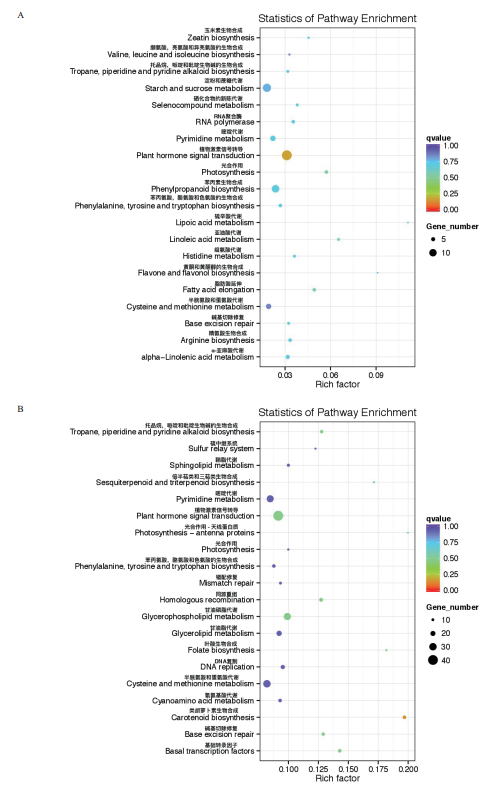
|
| 图 7 桂糖08-1180和ROC22特有差异表达基因的KEGG富集结果 |
RNA-seq是研究生物体转录组信息的有效方法,测定生物体在不同处理条件下的表达信息,可以帮助我们发掘相关的功能基因[5, 7]。转录组学是功能基因组学研究的一个重要内容,利用转录组数据库研究非模式植物的冷调节基因将是基因克隆的一个重要手段,也是特殊抗性植物基因发掘的重要方法[10]。目前转录组测序技术在甘蔗的抗旱[11-12]、抗黑穗病[13]、分蘖习性方面[14]应用均有报道,且均筛选到了大量具有相应功能的差异表达基因。本研究对低温胁迫下2个不同甘蔗品种(系)进行转录组测序及基因差异表达分析,获得了大量的具有一定功能的差异表达基因,这有助于从整体上了解不同甘蔗品种对低温胁迫的不同反应机制,可为甘蔗品种的抗寒性鉴定提供理论依据。
低温首先发生在细胞膜及膜系统相关系统中[16-17],细胞膜最先感知低温胁迫,通过信号传导,启动冷响应基因和转录因子,以缓解低温胁迫的破坏[18],植物在低温胁迫下,通过膜运输和感知系统稳定膜结构,维持细胞内环境的稳定[19]。但这种缓解能力是有限度的,一旦超出植株的忍受范围,体内的保护系统受到破坏,低温引起膜系统的透性和活性变化,从而使光合作用减弱,呼吸速率大起大落,同时细胞间冰晶对细胞膜和细胞壁产生机械损伤,水分平衡受到破坏,最终导致植物死亡[20]。Li等[21]选用抗寒性强的甘蔗品种GT28和抗寒性弱的品种YL6,研究了低温胁迫下甘蔗微管与叶绿体膜超微结构的变化,认为抗寒性强的品种GT28的微管和叶绿体膜结构稳定且正常,而抗寒性较弱的YL6微管数显著减少,叶绿体膜模糊降解,GT28具有更高的光合能力。在本研究中,2个品种中共有差异表达基因均在膜系统功能小类中得到了显著的富集。在与光合作用相关的叶绿体、质体、膜系统和细胞器等相关的GO条目和类胡萝卜素生物合成、光合作用天线蛋白、光合作用、吡啉与叶绿素代谢等KEGG代谢通路上,富集到了大量的差异基因。说明低温胁迫对桂糖08-1180和ROC22的光合作用相关代谢途径产生了重要的影响。笔者曾以桂糖08-1180和ROC22等9个育种材料,进行低温胁迫和常温处理,测定了各品种的形态指数、光合及叶绿素荧光等指标,结果表明随着低温胁迫处理时间的延长,各品种的冷害指数不断增大,叶片叶绿素含量均显著降低,叶片净光合速率、气孔导度在常温与低温处理之间差异显著,Fv/Fm、ΦPSⅡ均显著下降,且其变化大小因品种不同表现不一,桂糖08-1180的冷害指数及光合特性参数显示其抗寒性均优于ROC22[9]。
植物抗寒性是一种受微效多基因控制的数量性状,此类基因是一种诱发性基因,只有在特定的条件下才能表达,多个基因的共同表达才能提高植物的抗寒性[15]。冷胁迫中根据基因产物的不同分为2大类:一类在胁迫过程中直接发挥作用的功能基因编码的产物,包括脯氨酸等调节物质和抗冻蛋白等功能蛋白;另一类为调节基因,其基因产物参与调控下游基因表达与信号传导[22]。植物的抗寒生理反应主要来自于冷诱导基因(COR)的表达,COR基因的表达受多种转录因子的控制,其中,CBF/DREB通路是诱发其表达的一种主要方式[23]。植物激素在冷胁迫中对控制依赖CBF基因通路途径和不依赖CBF/DREB通路途径中均为重要的信号因子[24]。甘蔗是一种起源于热带的喜温性植物,其抗寒能力和机制不同于起源于低温带的抗冻性植物,不同品种之间的抗性机制也必然不同。本研究使用转录组测序的方法,对2个抗寒性不同的品种进行的差异基因的表达谱分析,结果表明了2个品种具有一定相同的抗寒性机制,同时,2个品种又独具各自的抗性机制。桂糖08-1180表现在DNA代谢过程、DNA整合、DNA重组、ADP结合、天冬氨酸型内肽酶活性、天冬氨酸型肽酶活性、淀粉与蔗糖的代谢、苯丙素生物合成途径中,富集的差异基因较多。而ROC22表现在细胞代谢过程、生物合成过程、有机物生物合成过程、甘油磷酯代谢、嘧啶代谢、半胱氨酸和蛋氨酸代谢等途径中,富集的差异基因数较多。但对不同品种富集的差异表达基因的功能、代谢途径及相关生理特性,还需做进一步深入研究。
4 结论通过转录组测序技术,共获得了大小为57.41 G的clean bases,进行从头拼接后,获得了183 515个Unigenes。基因差异表达分析表明,低温胁迫下,桂糖08-1180有16 145个基因的表达发生了变化,ROC22有20 317个基因的表达发生了变化,13 113个差异表达基因为2个品种所共有,7 204个差异表达基因为ROC22品种特有,3 032个差异表达基因为桂糖08-1180品种特有。
对桂糖08-1180和ROC22共有差异表达基因的功能富集分析表明,低温胁迫下,ROC22与桂糖08-1180,在对寒害逆境反应、膜系统、光合作用方面具有许多相同的功能小类,富集了大量的差异基因,说明对低温胁迫的应急响应、低温胁迫对膜系统的伤害及光合作用的下降是2个品种的共性。
对桂糖08-1180和ROC22特有的差异表达基因功能富集分析表明,ROC22在有机化合物的合成、运输和转运蛋白活性相关的条目富集得较多,而桂糖08-1180在DNA整合、RNA聚合酶、ADP结合及代谢酶中富集的差异表达基因较多。
| [1] |
位明明, 李维国, 高新生, 等. 中国热带作物抗寒育种研究进展与展望[J]. 热带作物学报, 2015, 36(4): 821-828. DOI:10.3969/j.issn.1000-2561.2015.04.030 |
| [2] |
杨荣仲, 周会, 王伦旺, 等. 甘蔗家系农艺性状遗传力分析[J]. 南方农业学报, 2016, 47(3): 337-342. DOI:10.3969/j:issn.2095-1191.2016.03.337 |
| [3] |
徐超华, 李纯佳, 苏火生, 等. 甘蔗非生物胁迫抗性研究进展[J]. 植物遗传资源学报, 2017, 18(3): 483-493. |
| [4] |
Coasta V, Angelini C, Defeis I, et al. Uncovering the complexity of transcriptomes with RNA-Seq[J]. J Biomed Biotechnol, 2010, 853916. |
| [5] |
祁云霞, 刘永斌, 荣威恒. 转录组研究新技术及其应用[J]. 遗传, 2011, 33(11): 1191-1202. |
| [6] |
Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10(1): 57-63. DOI:10.1038/nrg2484 |
| [7] |
王跃, 毛开云, 王恒哲, 等. 转录组学测序技术应用与市场分析[J]. 生物产业技术, 2017, 5(9): 11-17. |
| [8] |
刘红亮, 郑丽明, 刘青青, 等. 非模式生物转录组研究[J]. 遗传, 2013, 35(8): 955-970. |
| [9] |
唐仕云, 杨丽涛, 李杨瑞. 低温胁迫下不同甘蔗品种(系)光合特性的变化及其与耐寒性的关系[J]. 广西植物, 2012, 32(5): 679-685. DOI:10.3969/j.issn.1000-3142.2012.05.021 |
| [10] |
李洋, 曲彦婷, 周爱民, 等. 针叶福禄考(Phlox subulata)抗寒基因的克隆及分析[J]. 基因组学与应用生物学, 2015, 34(9): 1917-1927. |
| [11] |
刘洪博, 刘新龙, 等. 干旱胁迫下割手密根系转录组差异表达分析[J]. 中国农业科学, 2017, 50(6): 1167-1178. |
| [12] |
李穆, 程志远, 何丽莲, 等. 甘蔗印度种响应干旱胁迫的数字基因表达谱[J]. 分子植物育种, 2018, 16(7): 2099-2106. |
| [13] |
苏亚春.甘蔗应答黑穗病菌侵染的转录组与蛋白组研究及抗性相关基因挖掘[D].福州: 福建农林大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10389-1014323078.htm
|
| [14] |
丘立航, 罗含敏, 陈荣发, 等. 基于RNA-Seq的甘蔗主茎和分蘖茎转录组建立及初步分析[J]. 基因组学与应用生物学, 2018, 37(3): 1271-1279. |
| [15] |
姚晓华. 植物抗寒相关功能与调控基因研究进展[J]. 中国农学通报, 2013, 29(27): 141-147. |
| [16] |
Palta JP, Whitaker BD, Weiss IS. Plasma membrane lipids associated with genetic variability in freezing tolerance and cold acclimation of Solanum species[J]. Plant Physiology, 1993, 103(3): 793-803. DOI:10.1104/pp.103.3.793 |
| [17] |
Orvar BL, Sangwan V, Omann F, et al. Early stepsin cold sensing by plant cells: the role of actin cytoskeleton and membrane fluidity[J]. Plant J, 2000, 23(6): 785-794. DOI:10.1046/j.1365-313x.2000.00845.x |
| [18] |
Sudesh KY. Cold stress tolerance mechanisms in plants. A review[J]. Agronomy for Sustainable Development, 2010, 30(3): 515-527. DOI:10.1051/agro/2009050 |
| [19] |
Yukio K, Matsuo U. Mass spectrometric approach for identifying putative plasma membrane proteins of Arabidopsis leaves associa-ted with cold acclimation[J]. Plant J, 2003, 36: 141-154. DOI:10.1046/j.1365-313X.2003.01864.x |
| [20] |
王庆杰, 王立. 植物抗寒性基因研究综述[J]. 农村经济与科技, 2018, 29(8): 23-25. DOI:10.3969/j.issn.1007-7103.2018.08.017 |
| [21] |
Li SL, Li ZG, Yang LT, et al. Differential effects of cold stress on chloroplasts structures and photosynthetic characteristics in cold-sensitive and cold tolerant cultivars of sugarcane[J]. Sugar Tech, 2018, 20(1): 11-20. DOI:10.1007/s12355-017-0527-5 |
| [22] |
刘贝贝, 陈利娜, 等. 果树响应低温胁迫的分子机制及抗寒性鉴定方法研究进展[J]. 北方园艺, 2017(15): 174-179. |
| [23] |
Fu JJ, Geng JC, Miao YJ, et al. Transcriptomic analyses reveal genotype- and organ-specific molecular responses to cold stress in Elymus nutans[J]. Biologia Plantarum, 2018(6): 1-14. |
| [24] |
Eremina M, Rozhon W, Poppenberger B. Hormonal control of cold stress responses in plants[J]. Cellular and Molecular Life Sciences, 2016, 73: 797-810. DOI:10.1007/s00018-015-2089-6 |







