水体富营养化是指水中的氮、磷含量超标, 引起藻类等水生植物疯长, 导致水中溶解氧水平下降、水生生物出现大量死亡的现象[1]。相比氮元素, 磷元素在水体富营养化中发挥的作用更加明显, 一般认为当水体中总磷含量超过0.015 mg/L时就会造成水体富营养化[2-3]。为解决水体富营养化问题, 通常是对污水先处理再排放, 目前对污水处理普遍采用活性污泥法。活性污泥法除磷通常包含两种方式:一种是传统活性污泥法, 只对污水中的碳源有好的去除效果; 另一种是强化生物除磷(Enhanced biological phosphorus removal, EBPR)[4]。EBPR具体是指除磷污泥中特定微生物在厌氧、好氧交替条件下, 成为优势菌种, 并超量吸磷储存在体内, 最终以沉淀的形式排出, 达到除磷效果, 上述具有超强除磷能力的微生物称为聚磷菌(Phosphate accumulating organisms, PAOs)。随着对聚磷微生物的不断探索, 发现一种新型PAOs—积磷小月菌(Microlunatus phosphovorus), 其代谢方式与典型PAOs有所不同。本文将通过比较典型PAOs和积磷小月菌在强化生物除磷系统中的作用和机理, 以期为后续研究提供参考。
1 PAOs在强化生物除磷中的作用及机理传统活性污泥法只能吸收微生物正常生长所需的磷, 随污泥沉淀而去除, 如在常规的生物二级处理中, 污泥含磷量为污泥干重的1.5%-2.3%, 以污泥排除方式只能获得10%-30%的去除效果, 而EBPR的除磷效果是常规生物除磷的3-7倍[5], EBPR中污泥含磷量基本在4%-6%左右, 有些能达8%-12%, 最高可达15%, 系统除磷效率达80%-95%, EBPR具有除磷效率高、剩余污泥少、剩余污泥肥效高、经济可靠的优点[6]。EBPR系统得以实现, PAOs在其中扮演重要角色, 且在活性污泥众多微生物中占有一定比例。在厌氧条件下, PAOs能通过Poly-P水解获取吸收有机底物的能量, 在与其他好氧细菌竞争中获得优势[7]。例如, β变形菌门-2亚纲中的红环菌属, 简称“Accumulibacter”, 是一种典型的PAOs[8]。
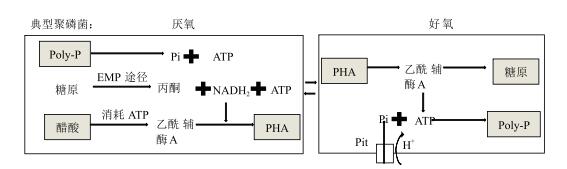
典型PAOs的代谢机理是在厌氧条件下分解Poly-P产生ATP, 同时吸收水体中存在的挥发性脂肪酸(Volatile fatty acid, VFA)如乙酸等, 随后乙酸被乙酰化成乙酰辅酶A, 在这过程中消耗ATP, 乙酰辅酶A聚合后和还原性物质烟酰胺腺嘌呤二核苷酸(NADH2)发生还原反应生成中间产物, 最后经聚合反应生成聚羟基烷酸(Polyhydroxyalkanoates, PHA)[6]。PHA作为还原产物, 反应过程中所需还原性物质来自糖降解途径(EMP), EMP不仅提供ATP, 而且为电子传递链提供NADH2, EMP途径作为还原力的来源被广泛接受[6, 9], 在好氧条件下, 分解厌氧条件下储存的PHA产生糖原和ATP, NADH2失电子产生NADH+、H+, 将PO4-运输到体内合成Poly-P[10](图 1)。
|
| 图 1 典型PAOs除磷过程中代谢方式 |
随着对PAOs的不断探索, 其含义逐渐拓展, 后延伸至一切超越自身生长所需、能够过量吸磷的微生物, 厌氧阶段合成PHA不再是必要条件[10]。积磷小月菌(Microlunatus phosphovorus)代谢方式与典型PAOs在不同培养条件下有所差异, 其在强化生物除磷系统中的作用和机理如下文所述。
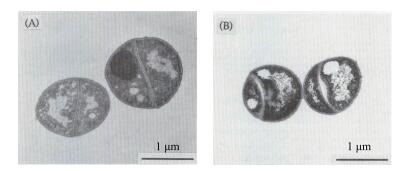
2 积磷小月菌及其在强化生物除磷中的作用积磷小月菌作为新的PAOs首次从实验室规模的除磷污泥中分离出来[11]。实验中发现这种菌具有超强除磷能力, 但是不具备典型PAOs的关键代谢特征, 不能利用醋酸, 厌氧条件下不合成PHA[12]。积磷小月菌是革兰氏阳性细菌, 细胞形态为球形, 单一、成对或成簇存在, 如图 2所示, 生长至2-4 d时, 球形细胞直径约为0.5-2.5 μm, 不形成孢子, 菌落圆形凸起表面光滑无光泽[13-14]。该菌最适生长pH值为7.0, 最适培养温度为25℃, 在最适条件下培养的倍增时间约为13 h[14-15]。Kawaharasaki等[16]通过16S rRNA寡核苷酸探针和荧光染色的方法发现积磷小月菌约占EBPR活性污泥中总细菌的3%, PAOs约占活性污泥总细菌的10%。由此看出积磷小月菌在PAOs中占有一定比例, 菌体内磷含量所占比例也是该菌具有超强吸磷能力的重要原因。前人研究发现积磷小月菌中磷含量超过自身干重10%, Poly-P含量最大值约为细胞干重的48%, 积磷小月菌中Poly-P颗粒如图 3中光亮部分所示[14, 17]。积磷小月菌作为已发现PAOs的一种, 在纯培养的条件下, 其吸磷、释磷能力较其他PAOs(如Lampropedia spp.和Acinetobacter spp.)高一个数量级[18]。以上研究表明作为新型高效PAOs的一种, 积磷小月菌的生化代谢反应和遗传学特性研究对EBPR有重要意义。
3 积磷小月菌在强化生物除磷中的作用机理积磷小月菌超强生物除磷能力与其特殊生物除磷机理有关, 积磷小月菌在除磷过程中所需碳源、能量来源和磷转运能力, 以及Poly-P的合成与代谢能力与典型PAOs有所差异。积磷小月菌体内存在PHA, 但它可利用葡萄糖和Poly-P作为碳源和能量, 其超强除磷能力与菌体有效的磷转运能力和Poly-P合成代谢能力有关。
3.1 积磷小月菌中PHA的研究进展PHA是很多细菌都可以合成的胞内聚酯, 在生物体内主要作为细胞碳源和能量的储存物质[2]。关于积磷小月菌中PHA是否为碳源和能量储存物质的研究, Santos等[18]使用核磁共振方法, 用13C标记的葡萄糖为碳源在厌氧、好氧交替的SBR中进行研究, 发现在13C的转移过程中没有形成PHA(含13C)。后Akar等[19]采用纯培养方法, 用气相色谱的方法进行物质定量, 发现在各种碳源和不同厌氧、好氧条件下, 积磷小月菌胞内都能积累PHA, PHA合成酶很可能与Poly-P合成酶同为诱导型酶。Kawakoshi等[20]认为积磷小月菌在好氧条件下产生PHA, 且存在一种新的PHA合成系统, 典型PAOs中存在ActP和phaABC基因, 两者分别负责糖原吸收和PHA逐步合成, Kawakoshi等发现积磷小月菌中存在替代phaABC系统的基因:MLP_23080、MLP_23090、MLP_12780, 且与典型PAOs合成PHA的方式有明显区别。Akar等通过定量确定PHA数量后, 根据好氧、厌氧条件下PHA数量对代谢过程中所用碳源进行推断。Santos和Kawakoshi等的观点不谋而合, 分别从核磁共振和基因方面做出解释, 积磷小月菌中的PHA很可能只作为细胞中的物质存在, 而不作为磷去除过程中的碳源和能量发挥作用, 进一步研究确定积磷小月菌中碳源和能源显得尤为重要。
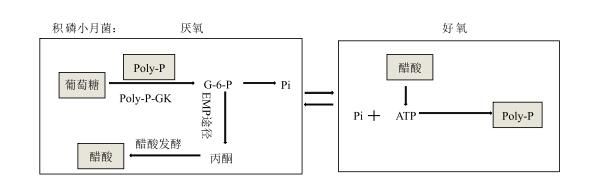
3.2 积磷小月菌中的糖代谢活性污泥中存在的非聚磷菌能发酵葡萄糖等物质产生VFA供给典型PAOs进行后续代谢反应, 而积磷小月菌可直接利用葡萄糖, 这是其碳源和能量供应的特殊之处。Tanaka等[17]研究发现积磷小月菌中存在磷酸依赖的葡萄糖激酶(Polyphosphate-dependent glucokinase, Poly-P-GK), 与ATP-葡萄糖激酶(ATP-glucokinase, ATP-GK)相比, Poly-P-GK是严格以Poly-P为能量的葡萄糖激酶, 在厌氧条件下, 以葡萄糖和Poly-P为底物, 在Poly-P-GK的作用下合成中间产物葡萄糖-6-磷酸(Glucose-6-phosphate, G-6-P)。之后代谢反应中, G-6-P中的磷被释放, 葡萄糖被发酵成醋酸, 这和厌氧条件下磷快速释放相符合[17, 20]。Santos等[18]研究发现积磷小月菌厌氧条件下将葡萄糖发酵成醋酸, 磷的释放会刺激葡萄糖吸收; 在好氧条件下, 积磷小月菌可以从厌氧阶段产生的醋酸中获取能量用于生长和合成Poly-P。积磷小月菌通过EMP途径发酵葡萄糖为醋酸, Poly-P-GK催化合成G-6-P是EMP途径的第一步, 研究发现积磷小月菌中存在两种ppgk基因(MLP_05430和MLP_26610)[20-21], 这是第一次在一种生物体内发现含两种ppgk基因, 利用Poly-P, 在两种ppgk基因作用下进行糖酵解, 如图 4所示。
|
| 图 4 M.phosphovorus除磷过程中糖代谢 |
以上研究可以看出积磷小月菌与典型PAOs的糖代谢方式不同, 在厌氧条件下, 可分别利用葡萄糖和Poly-P作为碳源和能量释放磷, 并为好氧条件下磷的吸收提供条件, 最终达到超量合成Poly-P。在Poly-P的分解合成过程中积磷小月菌体内磷的转运与代谢在生物除磷中起着举足轻重的作用。
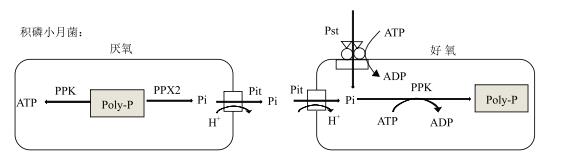
3.3 积磷小月菌中的磷代谢大肠杆菌中存在磷转运系统, 当外界磷过量时, 由低亲和力的转运系统(PitA)吸磷, 依靠质子动力获能, 并且PitA的合成不受PHO调控子控制, 当外界浓度低时, 高亲和性的转运系统(Pst)被PHO调节子诱导[22]。积磷小月菌中同样存在两种转运系统(Pit和Pst), Pit转运系统以质子为推动力, 而Pst转运系统需要消耗ATP, 积磷小月菌有效的磷吸收与释放能力, 可能与Pit基因的数量在已知的放线菌中数量最多有关, 在无需消耗ATP的情况下, Pit调节的可逆磷转运过程能够有效应对Ploy-P合成与降解所引起的细胞内磷水平急剧变化[20]。
Poly-P是由高能磷酸键连接正磷酸盐残基形成的线性聚合物, 主要作为能量储存器(低分子量Poly-P)和磷储存器(高分子量Poly-P), 它的合成与降解是PAOs能够除磷的原因之一[2, 23]。在大肠杆菌中, 多聚磷酸盐激酶(PPK)最终由一个可逆反应将ATP中的磷转变为Poly-P中的磷:
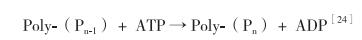
磷酸盐也能在外切聚磷酸酶(PPX)作用下从多聚磷酸盐中释放出来, 在PPX作用下, 能够使Poly-P分解, 这一过程伴随着由ADP向ATP的合成, 而ppk和ppx基因位于同一操纵子上[24]。积磷小月菌中好氧合成Poly-P与厌氧分解Poly-P同样需要PPK和PPX的参与。有研究认为积磷小月菌中存在4个假定的ppk基因, 其中包括一个ppk1(MLP_47700)和3个ppk2同系物(MLP_05750、MLP_50300、MLP_23310), 作为放线菌的一种, 积磷小月菌所含ppk基因的数量相对较多。已有研究表明多个数量的ppk基因与Poly-P的高转换率和积累能力有关[12, 20]。另外积磷小月菌中同样存在水解Poly-P末端磷酸盐的ppx基因, 但只含有一个ppx2同系物(MLP_44770)[20], 积磷小月菌有较少的PPX也有可能与其超强的聚磷能力有关, 其磷代谢过程如图 5所示。
|
| 图 5 M.phosphovorus磷的代谢方式 |
本课题组从污泥中分离出一株积磷小月菌JN459[25], 对JN459菌株好氧厌氧条件下的转录谱分析发现, JN459在好氧厌氧条件下与Poly-P代谢相关的基因ppk(MLP_47700, MLP_50300和ML-P_05750)、ppgk(MLP_05430和MLP_26610)、ppx(MLP_44770)、pap(MLP_23310)和ppnk(MLP_17420)表达水平存在显著差异, 厌氧条件下磷酸葡萄糖激酶(MLP_05430)与ATP依赖的磷酸NAD激酶(MLP_17420)的高表达揭示这两种酶在利用Poly-P作为生长所需能源中发挥着重要作用[26]。
4 展望EBPR活性污泥中积磷小月菌占有一定比例, 与典型PAOs相比, 积磷小月菌具有更强除磷能力, 在Poly-P代谢过程中糖原的利用方式不同, 且具有更有效的磷转运能力和合成能力, 加强积磷小月菌生化代谢反应和遗传学特性研究对污水除磷具有重要意义。未来对积磷小月菌的研究可以从以下几个方面入手:①已有文献中对积磷小月菌厌氧条件下碳源和能量供应有明显阐述, 但对好氧条件下物质转换和能量供应没有详尽说明, 可以深入研究; ②通过积磷小月菌的纯培养, 运用转录组学和蛋白质组学, 继续探索和鉴定mRNA与蛋白质, 研究其代谢机理, 从而更好地反应出不同蛋白质在厌氧、好氧条件下表达水平的差异, 以确定某些基因在积磷小月菌除磷过程中的作用; ③对代谢反应和遗传学特性深入研究, 找到在磷转运系统和Poly-P合成代谢过程中的关键基因, 构建除磷能力更强的工程菌, 以期达到更好的除磷效果; ④对积磷小月菌产PHA及其副产物的能力进行研究, 对将其作为生产PHA的工业菌种可行性进行探索。
| [1] |
Luo L, Duan N, Wang XC, et al. New thermodynamic entropy calculation based approach towards quantifying the impact of eutrophication on water environment[J]. Science of the Total Environment, 2017, 603-604: 86-93. DOI:10.1016/j.scitotenv.2017.06.069 |
| [2] |
李波.聚磷菌多聚磷酸盐激酶的分子生物学研究及高效除磷工程菌的构建[D].南京: 南京农业大学, 2008.
|
| [3] |
郎龙麒, 万俊锋, 王杰, 等. 生物除磷技术在水处理中的应用和研究进展[J]. 水处理技术, 2013, 39(12): 11-15. DOI:10.3969/j.issn.1000-3770.2013.12.003 |
| [4] |
蔡天明.高效聚磷菌的筛选、ppk基因的克隆及其应用研究[D].南京: 南京农业大学, 2005.
|
| [5] |
田淑媛, 杨睿, 顾平, 等. 生物除磷工艺技术发展[J]. 城市环境与城市生态, 2000(4): 45-47. |
| [6] |
邱立平, 王广伟, 张守彬, 等. 强化生物除磷系统中聚磷菌与聚糖菌的种群分析及代谢机制研究进展[J]. 环境污染与防治, 2010, 32(10): 66-71. DOI:10.3969/j.issn.1001-3865.2010.10.015 |
| [7] |
沈耀良, 玉宝贞. 废水生物除磷工艺中聚磷菌的作用机制及运行控制要点[J]. 环境科学与技术, 1995(2): 11-16. |
| [8] |
张明明, 王少坡, 于静洁, 等. 聚磷菌的代谢途径及其特性研究现状[J]. 工业水处理, 2013, 33(5): 4-8. DOI:10.3969/j.issn.1005-829X.2013.05.002 |
| [9] |
刘燕, 陈银广, 周琪, 等. 强化生物除磷系统中生化机理研究进展[J]. 水处理技术, 2006, 32(5): 1-4. DOI:10.3969/j.issn.1000-3770.2006.05.001 |
| [10] |
沈雨, 黄勇, 李大鹏, 等. 强化生物除磷系统主要微生物研究进展[J]. 水处理技术, 2010, 36(5): 14-17. |
| [11] |
Mino T. Microbial selection of polyphosphate-accumulating bacteria in activated sludge wastewater treatment processes for enhanced biological phosphate removal[J]. Biochemistry Biokhimiia, 2000, 65(3): 341-348. |
| [12] |
Gebremariam SY, Beutel MW, Christian D, et al. Research advances and challenges in the microbiology of enhanced biological phosphorus removal-a critical review[J]. Water Environment Research A Research Publication of the Water Environment Federation, 2011, 83(3): 195-219. DOI:10.2175/106143010X12780288628534 |
| [13] |
张长风, 余利岩, 张玉琴. 小月菌属放线菌的研究进展[J]. 微生物学报, 2017, 57(2): 179-187. |
| [14] |
Nakamura K, Hiraishi A, Yoshimi Y, et al. Microlunatus phosphovorus gen.nov.sp.nov.a new gram-positive polyphosphate-accumulating bacterium isolated from activated sludge[J]. International Journal of Systematic Bacteriology, 1995, 45(1): 17-22. DOI:10.1099/00207713-45-1-17 |
| [15] |
Nakamura K, Ishikawa S, Kawaharasaki M. Phosphate uptake and release activity in immobilized polyphosphate-accumulating bacterium Microlunatus phosphovorus, strain NM-1[J]. Journal of Fermentation & Bioengineering, 1995, 80(95): 377-382. |
| [16] |
Kawaharasaki M, Kanagawa T, Tanaka H, et al. Development and application of 16S RRNA-targeted oligonucleotide probe for detection of the phosphate accumulating bacterium microlunatus phosphovorus, in an enhanced biological phosphorus removal process[J]. Water Science & Technology, 1998, 37(4-5): 481-484. |
| [17] |
Tanaka S, Lee SO, Hamaoka K, et al. Strictly Polyphosphate-dependent glucokinase in a polyphosphate-accumulating bacterium, Microlunatus phosphovorus[J]. Journal of Bacteriology, 2003, 185(18): 5654-5656. DOI:10.1128/JB.185.18.5654-5656.2003 |
| [18] |
Santos MM, Lemos PC, Reis MA, et al. Glucose metabolism and kinetics of phosphorus removal by the fermentative bacterium Microlunatus phosphovorus[J]. Applied & Environmental Microbiology, 1999, 65(9): 3920-3928. |
| [19] |
Akar A, Akkaya EU, Yesiladali SK, et al. Accumulation of polyhydroxyalkanoates by Microlunatus phosphovorus under various growth conditions[J]. Journal of Industrial Microbiology & Biotechnology, 2006, 33(3): 215. |
| [20] |
Kawakoshi A, Nakazawa H, Fukada J, et al. Deciphering the gen-ome of polyphosphate accumulating actinobacterium Microlunatus phosphovorus[J]. DNA Research, 2012, 19(5): 383. DOI:10.1093/dnares/dss020 |
| [21] |
孙雪, 朱为静, 王亮, 等. 强化生物除磷系统主要微生物及其代谢机理研究进展[J]. 应用生态学报, 2014, 25(3): 892-902. |
| [22] |
管莉菠.Pseudomonas putida GM6聚磷相关基因的克隆和特性研究[D].南京: 南京农业大学, 2007.
|
| [23] |
Geissdörfer W, Ratajczak A, Hillen W. Transcription of ppk from Acinetobacter sp.strain ADP1, encoding a putative polyphosphate kinase, is induced by phosphate starvation[J]. Applied & Environmental Microbiology, 1998, 64(3): 896. |
| [24] |
石廷玉.结核分枝杆菌多聚磷酸盐外切酶Rv1026对耻垢分枝杆菌的菌落形态和生物膜的影响[D].重庆: 西南大学, 2011.
|
| [25] |
钟传青, 姜天翼, 王静, 等. 聚磷菌—JN459的分离和聚磷特性研究[J]. 山东建筑大学学报, 2015(1): 25-28. DOI:10.3969/j.issn.1673-7644.2015.01.005 |
| [26] |
Zhong CQ, Fu JF, Jiang TY, et al. Polyphosphate metabolic gene expression analyses reveal mechanisms of phosphorus accumulation and release in Microlunatus phosphovorus Strain JN459[J]. FEMS Microbiology Letters, 2018, 365(6). DOI:10.1093/femsle/fny034 |