环状RNA (circular RNA,circRNA)是一类新发现的内源性非编码RNA (non-coding RNA,ncRNA)。与传统的线性RNA (linear RNA)相比,circRNA不存在5'和3'端,为共价键连接在的环状闭锁结构,对RNA外切酶(RNase R)具有抗性,表达稳定,不易降解。近几年研究发现circRNA与人类疾病发生关联紧密,成为RNA领域研究的新热点。早期研究认为circRNA是标准RNA剪接的副产物,但随着研究发现,circRNA在真核生物中普遍存在,认为可能具有一定的生物学功能。circRNA来自RNA前体(pre-RNA)的反式剪接,即下游外显子同上游外显子反向结合形成地环形结构。不同序列来源的circRNA可发挥不同的生物学功能。有报导发现circRNA可吸附miRNA发挥miRNA海绵(miRNA sponges)作用,也有报导发现RNA环化与mRNA形成可竞争剪接因子,以此调控目的基因的表达。但是,circRNA的主要作用还未清晰。本文总结了circRNA的发现历程、特征及分类、环化生成及调控机制、主要功能及在畜禽中的研究进展,并对其在畜禽中的研究进展进行了展望,以期为进一步研究circRNA提供一定的理论依据和技术指导。
1 circRNA的发现历程circRNA是一类新发现具有环形结构的内源性RNA分子,广泛存在于真核生物体内[1]。如表 1,circRNA的发现历程有3个阶段:(1) circRNA真实存在性;(2)真核生物中circRNA的普遍性;(3)某个circRNA的功能性研究。
1976年,Sanger和Kolakofsky[2-3]在植物类病毒和副流感病毒颗粒中首次发现了circRNA。1979年,Capel等[14]在动物细胞和真菌酵母中发现circRNA,之后也在其他真核细胞中发现了circRNA,如在小鼠SRY[14],大鼠细胞色素P4502C24[5]、人的P4502C18中[6]等。但由于circRNA结构的特殊性及当时有限的研究手段,认为circRNA异常拼接产物。近年来高通量测序技术和生物信息学分析技术迅速发展为circRNA的研究提供了契机。2012年,Danan等[7]在古细菌发现circRNA的普遍存在性,并认为其可能具有生物学功能。Salzman等[8]通过RNA-Seq方法在人体中首次鉴定出约80个circRNA,至此circRNA的研究才正式进入人们的视野。
Jeck等[9]在人类的成纤维细胞中检测出超过25 000个circRNA,Memczak等[15]利用高通量测序结合人类白细胞数据库鉴定出1 950种人类circRNAs,1 903种小鼠circRNAs和724种线虫circRNAs,其中人和小鼠中共有81种circRNAs高度保守。随着大量研究表明,circRNA广泛存在真核生物等高等动物体内,并且在基因表达和机体生长发育中发挥重要作用。例如,circFUT10可抑制牛成肌细胞增殖,促进分化[11];circEIF3J和circPAIP2能促进母基因的线性转录本的表达[12, 16];circZNF609具有编码起始密码子和终止密码子的开放阅读框[17];circCDR1as和circSry可与AGO蛋白形成沉默复合体抑制翻译起始[10, 18]。目前circRNA被认为是一类新的功能大分子,参与基因表达调控,丰富了真核生物转录调控的多样性和复杂性[9, 18]。circRNA的发现为研究细胞发育和基因转录调控的分子机制掀开新的一页,对生命活动的探索具有重要意义。
2 circRNA的特征及分类 2.1 circRNA的特点目前发现,circRNA广泛存在于不同生物体内的各种类型的细胞和组织中。与线性RNA相比,circRNA是没有游离的5'帽子和3'Poly (A)尾巴的闭合环状结构。由于其闭锁的环状结构,circRNA对RNA外切酶RNaseR不敏感[19],比线性RNA更稳定,不易被降解,大多数circRNA的半衰期超过48 h,而线性RNA的平均半衰期只有约10 h[9, 20],这可能是circRNA可以抵抗分支酶和核酸外切酶RNase R的降解。但大部分circRNA在血清外泌体中并不稳定,半衰期小于15 s[21],且容易受其他RNA的干扰。癌细胞和癌组织中circRNA较线性转录本低[22],可能是线性转录本在细胞中易达到合成与降解的平衡,而circRNA更加稳定,在正常增殖的细胞中不断被“积累”,在癌细胞无限增殖过程中被“稀释”。circRNA的长度波动较大,但在大多数真核生物中circRNA具有高度的序列保守性[18, 23]。全基因组分析发现来源编码区外显子的circRNA具有高度的序列保守性,但是基因间和内含子来源的circRNA保守性相对较低[18]。
利用一种新的检测工具CIRI explorer2发现了同一基因中一些不保守的外显子只在circRNA中出现,而不存在于线性RNA中。这种只出现在circRNA中的外显子,是因为多个转录本在形成成熟线性RNA时,由于线性RNA的不稳定性而被降解掉,还是因为在形成circRNA时由选择性剪接而特意产生,到目前为止还不清楚。
研究报道发现circRNA在各种生物中表达丰富,其表达水平多数高于母基因的线性转录本[9, 15]。而且circRNA表达常具有一定的组织、时序和疾病特异性[15, 18]。已知大多数的circRNA属于非编码RNA (non-coding RNA,ncRNA),但是少数circRNA含有内部核糖体进入序列(Internal ribosome entry site,IRES)可能具有编码能力[24-25]。
2.2 circRNA的分类circRNA来源于母基因的外显子、内含子、非编码区、反义链转录本或基因间区。根据circRNA序列来源可以分为4大类(表 2) [16]:第一类是全部来源外显子的circRNA (Exonic circular RNA,EciRNA);第二类是全部来源于内含子的circRNA (Intronic circular RNA,ciRNA)第三类是来源于外显子和内含子的circRNA (Exon-intron circular RNA,EIciRNA);第四类包括来源于反义链转录本的反义circRNA (Antisense circular RNA)、来源于基因间序列或其他未注释基因组序列的circRNA (Intergenic circular RNA)。绝大多数circRNA属于EciRNA,其序列来源于外显子特别是编码区序列。
4大类circRNA除了序列来源不同外,还具有以下区别:(1) EciRNA和EIciRNA整个分子均是由3'-5'磷酸二酯键组成,但是ciRNA接合位点(junction)处由2'-5'磷酸二酯键连接[26];(2) EciRNA侧翼内含子的长度通常大于内含子的平均长度,并通常含有反向重复序列[9, 27];(3) ciRNA通常包含靠近5'剪接位点的7 nt GU富集碱基序列和靠近分支位点(Branchpoint,BP)的11 nt C富集碱基序列[28];(4) EciRNA主要存在于细胞质中[8, 23],而EIciRNA和ciRNA主要存在于细胞核中[26, 29];(5) EciRNA的序列保守性高于ciRNA和基因间circRNA的序列保守性[16]。
3 circRNA的生成和调控机制真核生物RNA是遗传信息从DNA到蛋白质传递的中间媒介,遗传信息在RNA水平上有着广泛且复杂的调控方式,基因在转录后的pre-RNA要经过复杂的加工才能形成成熟的RNA。单个基因位点可以通过经典可变反向剪接(Alternative back-splicing junctions)及经典可变剪接(Alternative splice junctions)产生多种类型的circRNA[30],因此不同来源的circRNA的生成机制也不同。
3.1 EciRNA的生成机制EciRNA是初级转录产物经过特殊的反向剪接反应(Back-splicing)形成的,是下游外显子序列5'剪接位点(5' splice site,5'SS)与上游外显子3'剪接位点(3'splice site,3'SS)反向连接生成了circRNA (图 1)。目前有两种模型解释了EciRNA反向剪接形成机制:(1)依赖于剪接体的套索驱动环化模型(外显子跳跃) (Lariat-driven circularization) (图 1-B);(2)内含子配对驱动环化模型(Intron-pairing-driven circularization) (图 1-C)。
套索驱动环化是由pre-mRNA在剪接过程中部分RNA发生折叠,拉近了非相邻外显子距离,发生外显子跳跃(Exon skipping)的经典剪接(图 1-B)。被跳过外显子的上游剪接受体(Splice acceptor,SA)和下游剪接供体(Splice donor,SD)共价结合形成包含外显子的套索前体(Exon-containing lariat precursor),该前体通过内部剪接去除内含子,形成EciRNA[31]。某些个circRNA中存在线性mRNA缺失的外显子,保护了这些外显子不被降解掉,这说明外显子跳跃是形成circRNA的合理机制[8-9]。许多EciRNA侧翼连接点有规范的剪接信号[9-10],经典的剪接位点决定了外显子环化效率[32],抑制经典剪接体会导致circRNA水平降低[32],这些都表明经典的剪接体机制是circRNA生成所必需的。酵母中大多数的circRNA都是由外显子套索前体产生[33]。
内含子驱动环化是位于侧翼内含子区域反向互补序列(Flanking intronic regions reverse complementary sequences,RCSs) (如Alu原件等) (图 1-C)互补配对,这会诱导下游外显子序列的5'SS反向接近上游外显子3’SS,有效地促进了反向剪接的发生[9],随后剪接体切除剩余内含子形成环形RNA分子[34]。当内含子没有被完全剪切掉,保留在新生成环状外显子之间,就会形成EIciRNA类型的分子。
“套索驱动”和“内含子配对驱动”的主要区别是先发生经典剪接还是反向剪接[35]。“套索驱动”是先发生经典剪接,产生一个线性RNA和一个包含外显子的内含子套索,随后内含子套索通过反向剪接生成circRNA[36]。“内含子配对驱动”是先发生反向剪接,并直接生成circRNA[9]。哺乳动物中大多数EciRNA是由内含子配对驱动生成的,而低级的真核生物中的EciRNA则是大多由套索驱动生成[33]。
3.2 EciRNA环化调控机制有研究发现外显子环化与线性剪接竞争剪接因子,进而与线性RNA存在竞争关系,circRNA的环化效率也受到剪接因子的调节[16],如一些顺式元件(cis-acting splicing regulatory elements)和反式原件(trans-acting splicing elements) (图 1) [16]。
研究发现单个外显子的circRNA (single exon circular RNA)长度通常大于多个外显子的circRNA (Multiple exon circular RNA)的平均长度[8]。Eci-RNA侧翼内含子含有倒置重复序列(如Alu等) (图 1-C) [16],并且通常含有多个重复序列[8]。这些序列存在竞争性配对,调节环化效率并造成可变环化,致使同一个母基因可以形成由不同外显子和内含子组成的多样型circRNA[27-28]。有研究发现只有30-40 nt短侧翼反向重复序列就足以促进环化[8],但这种侧翼区反向互补重复序列不一定有效诱导外显子环化,大多数的circRNA由短散在核重复序列(Short interspersed nuclear elements,SINEs)驱动形成,这说明内含子配对的稳定性是circRNA生成的必要条件[37]。
RNA结合蛋白(RNA-binding proteins,RBPs)可以通过与内含子中一些短的顺式元件结合发挥作用(图 1-D),促使下游外显子序列的5'SS反向接近上游外显子的3'SS,形成有利于反向剪接的结构,促进外显子环化[16]。在人和果蝇体内发现剪接因子盲肌蛋白(Muscleblind,MBL/MBNL1)可以结合到来源于第二外显子circMbl的pre-RNA内含子的MBL结合位点,使两个内含子靠近,从而促进circMbl生成[38]。震动蛋白(Quaking,QKI)能特异识别NA-CUAAY核心(core)及UAAY半位点(half-site) [39],牵拉结合点相互缩小空间距离,在人类上皮细胞-间质细胞转化(Epithelial-mesenchymal transition,EMT)的过程中诱导EciRNA生成[16]。脂肪瘤融合蛋白(Fused in sarcoma/Translocated in liposarcoma,FUS/TLS)可结合到内含子侧翼区,调控circRNA的形成[40]。有报道称提高RNA聚合酶Ⅱ转录延伸率(RNA-Pol II transcription elongation rate)可以增加初始circRNA的表达水平[41]。
有报道发现RNA编辑酶腺苷脱氢酶(Adenosine deaminase,ADAR)可以结合RNA双链,通过将腺苷酸转变成肌苷酸(A变成I) (图 1-E)。环化外显子侧翼区重复序列的双链产生U:I错配,使RNA双链结合能力减弱[34]。ADAR高表达会损伤mRNA前体环状配对序列,减少circRNA的生成[16]。在人和线虫中发现,ADAR的缺失造成circRNA的表达水平异常上升[34]。
EciRNA的生成是多个顺反调控原件共同调节的结果。例如,Laccase基因的环化受到内含子重复序列和RNA结合蛋白的调控[42],ADAR也会减弱RNA的内含子配对,控制circRNA的生成。
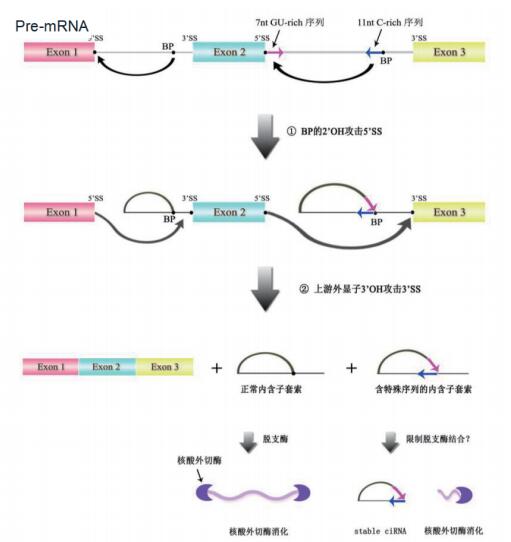
3.3 ciRNA的生成机制ciRNA是由含有特殊序列内含子在剪接过程中逃避脱支酶(Debranching enzyme,DBE)消化形成的circRNA[12]。这类内含子在3'SS端的分支位点(BP)附近含有11 nt C富集序列,在5'SS附近含有7 nt GU富集序列(图 2),这些序列形成的特殊结构会阻止脱支酶与之结合[16, 28]。
剪接过程中两个原件结合先形成包含外显子和内含子的套锁中间体(Lariat),再在脱支酶和核酸酶作用下形成ciRNA。其过程包括两次转酯反应(图 2):首先分支位点(Branchpoint,BP)的2'-OH攻击外显子5'SS,形成内含子套索结构,而后5'外显子的3'-OH攻击下游外显子3'SS,释放线性内含子并将外显子顺序连接,最后线性内含子释放3'尾巴后形成2'-5'连接的内含子来源的circRNA。正常内含子套索则是在脱支酶作用下被解开,再被核酸外切酶消化,而含有11 nt C和7 nt GU特殊序列的内含子套索会限制脱支酶与之结合,残余的部分线性RNA序列会被核酸外切酶降解[12]。不同于EciRNA,ciRNA对RNA脱支酶不敏感,并含有2'-5'接合点(Junction) [26]。ciRNA和EIciRNA主要存在于细胞核中调控基因表达[16]。
也有研究发现前体tRNA能剪切成环形成tricRNA (tRNA intronic circRNA) [43],tRNA剪接内切酶复合体通过识别前体tRNA内特殊的膨胀-螺旋-膨胀(BHB)的结构序列,切除反密码子环中的内含子[38],此部分内含子两端相连接形成tricRNAs[44]。有研究报道,在白血病患者中的PML/RARα基因上发现,了2个因染色体异位融合的circRNA (fusion circRNA,f-circRNA) [45]。因此,circRNA的生源机制有待进一步探索明确。
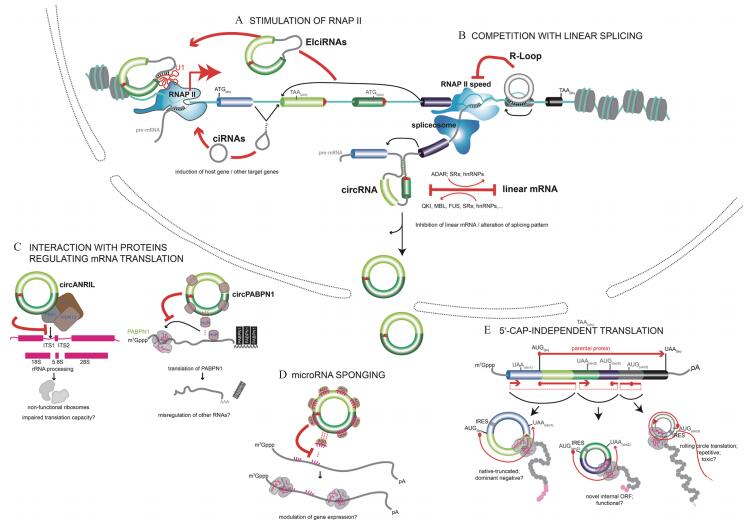
4 circRNA的功能circRNA广泛存在于各个物种中,大多数真核生物的circRNA具有序列保守性,表达呈现一定的组织特异性和发育阶段性,这表明circRNA是一种功能分子。有报道发现circRNA具有调控基因转录、调控蛋白质翻译、吸附miRNA等功能,也有报道发现一些circRNA具有编码多肽的能力(图 3)。
CircRNA可与转录因子结合调控基因转录(图 3-B)。EciRNA可以与细胞内某些蛋白质分子特异性结合,作为脚手架(Scaffold)与RNA或DNA结合,为RBP、RNA、DNA之间互作提供平台。这些转录因子包括RNA聚合酶Ⅱ、Quaking (QKI) [16]、EIF4A3[47]和MBL[38]等。例如,circMbl上含有MBL结合位点[32],而MBL会促进circMbl和Mbl mRNA的生成。因此circMbl作为自我调节分子会减少circMbl和Mbl mRNA的合成[48],提高果蝇和人细胞内线性剪接水平,会使circRNA的生成量明显减少[49-50]。细胞核中位于转录位点附近的内含子锚蛋白重复结构域52 (ci-ankyrin repeat domain 52,ci-ankrd 52)和内含子沉默信息调节因子7 (Silent information regulator 7,ci-sirt 7) [26],可联合RNA聚合酶Ⅱ复合体,顺式调节母基因的线性转录本表达。circEIF3J和circPAIP2会与U1小核核糖核蛋白(U1 small nuclear ribonucleoprotein,U1 snRNP)相互作用于基因转录起始位点上游300 bp处,顺式方式调控母基因线性转录本的转录[12, 16]。circRNA还可以通过与线性转录本竞争剪接的方式调控母基因的表达。线性剪接与反向剪接的竞争决定了circRNA和母基因线性转录本的表达水平。来源于小鼠甲酸精基因(Formin,Fmn)的circFmn包含了转录起始位点,遗留了一个非编码的线性转录物,因此circFmn可作为mRNA陷阱(mRNA trap)隔离转录起始位点,降低Fmn蛋白的表达水平[51]。
4.2 circRNA与蛋白质相互作用也有报导circRNA与RBP结合能发挥生物学功能(图 3-D)。circCDR1as和circSry可与miRNA的效应因子AGO相结合,形成RNA诱导沉默复合体(RNA-induced silencing complex,RISC) [10, 18],占据了mRNA的翻译起始位点,抑制翻译起始。CircANRIL可与pre-rRNA竞争性结合PES1蛋白,抑制核酸外切酶对结合PES1蛋白后的pre-rRNAs加工过程[52],表明circANRIL影响核糖体成熟,阻碍蛋白质翻译,进而导致细胞凋亡。circFOXO3与细胞周期依赖性激酶2 (Cyclin-dependent kinase 2,CD-K2)、P21形成circFOXO3-CDK2-P21三元复合物抑制细胞周期[53]。CDK2与细胞周期蛋白E (Cyclin E)结合形成cyclin E-CDK2复合物,促进细胞G1期向S期过度;CDK2也可与细胞周期蛋白A (Cyclin A)结合形成cyclin A-CDK2复合物,促使细胞完成S期。P21结合CDK2可抑制CDK2与cyclin A、cyclin E的结合,circFOXO3-CDK2-P21三元复合物增强了P21对CDK2的抑制作用,避免了cyclin E-CDK2复合物的形成,阻断细胞从G1期进入S期,同时也避免了cyclin A-CDK2复合物的形成,使细胞停滞在S期或发生凋亡。circFOXO3在细胞质中还可以与抗衰老相关蛋白分化抑制因子1 (Inhibitor of differentiation-1,ID1)、E2F1转录因子(E2F transcription factor 1,E2F1)以及抗应激黏着斑激酶(Facal adhesion-associated protein kinase,FAK)、HIF1α结合,阻碍这些蛋白进入细胞核或线粒体,从而促进心肌细胞衰老[54]。
4.3 circRNA作为miRNA海绵MicroRNA (miRNA)是一种大约22 nt的ncRNA分子,通过与目的基因mRNA的3’UTR结合降低目的基因的表达水平。circRNA含有大量的miRNA应答原件(miRNA response elements,MREs),可吸附miRNA (图 3-A),发挥miRNA海绵功能,降低miRNA对其靶基因的抑制作用。由此可见circRNA可作为竞争性内源RNA (Competing endogenous,ceRNA)在转录后水平调控靶基因的表达[16]。例如,来源于小脑退行性相关蛋白1反义转录本(Cerebellar degeneration-related 1 antisense transcript,CDR1as)的circRNA具有70个miR-7结合位点,可调控胰岛素受体底物1 (Insulin receptor substrate-1,IRS-1)等多个靶基因的表达[18]。有研究表明miR-671与CDR1as结合后会使得CDR1as裂解,并可释放与之结合的miR-7[55]。阿格蛋白(Argonature,AGO)可与CDR1as结合,使CDR1as免于降解及结合miR-7。circHIPK3可结合miR-124-3p和miR-338-3p调控β细胞Slc2a2、Akt1、Mtpn等关键基因表达[56]。来源于Y染色体性别决定基因(Sex-determining region Y,SRY)的circSry具有16个miR-138结合位点,可吸附miR-138调节肿瘤的侵袭、发展和转移[55, 57]。circFUT10可以结合miR-133a进而抑制牛成肌细胞增值、促进分化[11];circMTO1可以吸附miR-9抑制人肝癌细胞增值性和侵染性[58];circZFR可吸附miR-130a和miR-107抑制人胃癌细胞增殖和促进凋亡[59];circRBFOX2s可吸附miR-206促进鸡成肌细胞增殖[60-61]。对比lncRNA等线性miRNA海绵分子,circRNA不易被RNA外切酶降解,具有较高的稳定性和时效性[16, 58]。一些研究表明哺乳动物中的circRNA很少包含大于10个以上的miRNA结合位点,这可能与circRNA本身的稳定性有关[36]
4.4 circRNA可翻译功能蛋白质有些circRNA含有可编码多肽。目前报道的circRNA翻译机制有两种:(1)依赖内部核糖体嵌入位点(Internal ribosomal entry site,IRES) (图 3-C);(2)依赖N6-甲基腺苷(N6-methyladenosine,m6A)甲基化修饰。人circSHPRH可利用重叠密码子翻译功能蛋白质抑制胶质瘤生成[13]。丁型肝炎病毒(Hepatitis D virus,HDV)核心的负链circRNA可编码122个氨基酸的HDV抗原(Hepatitis D virus antigen,HDAg) [62]。人circ-FBXW7也含有IRES,可编码21 kD的蛋白质,抑制U251和U373癌细胞的增殖[63]。部分circRNA虽无IRES,仍能结合核糖体启动翻译。如在人和小鼠的成肌细胞中发现具有开放阅读框(Open reading frame,ORF) [17]的circZNF609可翻译蛋白质。Pamudurti等[64]在果蝇大脑组织中通过核糖体印迹鉴定出circMBL的终止密码子处有核糖体结合,并通过蛋白质谱分析得到了circRNA翻译的蛋白质。研究发现m6A甲基化修饰会促进circRNA的翻译[17],皆因此类circRNA起始密码子上含有RRACH (R=G或A;H=A、C或U),该序列中的A碱基可被甲基化酶复合体METTL3/14甲基化,促使YTHDF3可之结合,并招募eIF4G2和其他翻译起始因子,启动circRNA的翻译[17]。此cricRNA没有终止密码子,若翻译起始,circRNA的环状结构可使核糖体通过类似滚环扩增(Rolling circle amplification,RCA)的机制参与多肽延长。
4.5 circRNA参与细胞通讯和信号转导也有报道circRNA可调控细胞通讯和信号转导。血清等外泌体的circRNA与胞质中存在显著差异,表明circRNA进入外泌体是一个选择性过程,可能作为细胞间物质传递、信息交流的途径[29]。分泌小泡(含外泌体)中circRNA比线性RNA比例较高[65],表明分泌小泡是细胞选择性释放和清除内源性circRNA的一种机制。CircRNA也可调节mRNA的稳定性,例如mcircRasGEF1B可调控脂多糖(LPS)诱导的细胞间黏附因子(ICAM-1) mRNA的稳定性,敲低mcircRasGEF1B会影响LPS诱导的ICAM-1的表达[66]。
综上所述,circRNA是一类新发现的分子,具有不同的分子特点和形成机制,关于其功能研究还有待深入。circRNA在物种中具有较高的序列保守性,具有调控机体生长发育和新陈代谢的功能,在遗传品种改良和品系选育的工作中,有望作为分子育种的新靶点。
5 circRNA在畜禽中的研究进展畜禽的经济性状与疾病防控是动物科学关注的重点。近年来,circRNA在该领域的研究也是如火如荼,为挖掘农业动物重要经济性状遗传机制和调控网络提供了新思路。
5.1 circRNA在家畜中的研究进展家畜繁殖机能、生长发育、肌肉发育、脂肪沉积、产毛(绒)量、产奶量和疾病发生等是畜牧业关注的重要经济性状。研究发现,circRNA参与家畜大脑、卵巢、肌肉、脂肪、心脏、肝脏和乳腺等性状的发育和疾病发生过程。
在肌肉和脂肪发育方面,Liang等[67]从贵州小型猪脂肪、心肌等9种不同组织及3个发育阶段的骨骼肌中鉴定出了5 934个circRNA,绘制了猪circRNA的时空表达谱,确定了149个与肌肉生长发育、肌肉收缩、染色质修饰、阳离子平衡、ATP水解耦合的阳离子转运、细胞紧密连接及钙离子信号通路有关的circRNA。并构建了首个小型猪的circRNA数据库。Chen等[68]采集180 d和8年雅南母猪的心肌和背最长肌,通过RNA-seq发现了心肌样本中26个mRNA、4个lncRNA、22个miRNA和26个circRNA差异表达;骨骼肌样本中81个mRNA、5个lncRNA、79个miRNA和62个circRNA差异表达,由此可见心肌和骨骼肌的衰老过程中存在差异。在肌肉老化过程中,Chen等[68]还评估了多重共调控关系,构建了circRNA-miRNA-mRNA共表达网络,为研究肌肉老化的分子机制提供了良好的基础。Sun等[69]研究蓝塘猪和长白猪肌肉组织中差异表达的编码基因、lncRNA、circRNA及miRNA,结果在蓝塘猪中发现有1 401个circRNA高表达,2 959个circRNA低表达,其中有236个是与肌肉发育相关的,并发现有40个circRNA参与miRNA介导的ceRNA调控网络。
Li等[70]利用RNA-Seq方法研究胚胎期和成年期哈萨克羊背最长肌组织中的circRNA,共鉴定出6 113个circRNA,功能分析发现circRNA的母基因主要富集在肌肉生长和发育相关的信号通路,oar_circ_0000385、oar_circ_0000582和oar_circ_0001099具有肌肉发育相关miRNA的多重保守位点。Wei等[71]分析胚胎期和成年期秦川牛的背最长肌中差异表达的circRNA,共鉴定出12 981个circRNA,其中有624个在成年牛中高表达,204个低表达。典型的是circLMO7,过表达circLMO7会吸附miR-378a-3p抑制成肌细胞分化,使得细胞周期中S期细胞数量增加,G0/G1期细胞数量减少,促进细胞增殖,抑制细胞凋亡。同课题组Li等[11]发现circFUT10可以结合miR-133a促进成肌细胞分化,使得细胞周期中G0/G1期细胞数量增加,S期细胞数量减少,抑制细胞增值,促进细胞凋亡。Li等[72]发现circFGFR4可以吸附miR-107,促进Wnt3a基因表达,进而促进成肌细胞分化诱导细胞凋亡,对细胞增殖没有显著影响。
在大脑和生殖系统研究方面,VenØ等[73]对猪胚胎发育期大脑组织测序共发现4 634个circRNA具有时间表达差异,皮质部circRNA的表达丰度最高,这表明circRNA对于猪胚胎期脑部发育具有重要调控作用。Li等[74]利用RNA-seq研究产前、产后哈萨克羊脑垂体中差异表达的circRNA发现,大量的circRNA参与垂体激素分泌及相关通路,oar_circ_0000059具有58个与垂体相关的miRNA结合位点,并且40个circRNA包含至少1个IREs和ORF,这表明circRNA广泛参与垂体的发育和分泌功能。Tao等[75]通过麻城黑山羊和波尔山羊排卵前后卵泡组织RNA-seq分析,共检测出1 395个circRNA,其中有37个差异表达。GO和KEGG分析发现大量circRNA参与卵巢类固醇生成和P53信号通路。其中chi_circ_0008219可与3个卵泡相关的miRNA相结合,表明circRNA对母羊卵巢卵泡有潜在影响。
在泌乳性能研究方面,Zhang等[76]研究产后90 d和250 d荷斯坦奶牛乳腺组织中差异表达的circRNA,共鉴定出6 621个circRNA,其中有2 231个共表达。功能富集分析发现来源于4个酪蛋白编码基因(CSN1S1、CSN1S2、CSN2和CSN3)的circRNA在90 d奶牛乳腺组织中高表达,这些circRNA也具有miR-2284的结合位点,而miR-2284可与CSN1S1和CSN2的mRNA结合,由此猜测这些circRNA可能参与酪蛋白的表达调控。Zhu等[77]对分娩后1、7和21 d大鼠的乳腺组织高通量测序,发现并鉴定了来源于Tc2n基因Exon10和Exon11的circRNA (cTc2n)。定量检测发现cTc2n在乳腺中高表达。cTc2n在这三个时间点的表达变化呈现先显著下降、后轻微上升的趋势,而Tc2n mRNA呈现相反的趋势。生信分析发现cTc2n不存在miRNA结合位点,而Exon10和Exon11编码Tc2n蛋白的保守区域,推测cTc2n可抑制Tc2n mRNA的产生。
在动物疾病发生研究方面,小鼠作为畜牧学科研究的模式动物,在家畜研究中应用广泛,尤其是在疾病研究方面。Pei等[78]通过将小鼠暴露在氡辐射的条件中,通过对肺组织RNA-aeq发现107个circRNA上调,83个circRNA下调,其中30个高表达的circRNA具有多个miRNA结合位点,表明circRNA可能参与氡诱发的肺损伤的病理变化。Huang等[79]对水迷宫测试后的5月龄、10月龄SAMP8鼠和SAMR1鼠的海马体RNA-seq,发现mmu_circRNA_017963与自噬体组合、胞外分泌、细胞凋亡、RNA剪接、突触囊泡循环等多个过程,表明circRNA参与阿兹海默症的发生。Errichelli等[40]通过体外培养老鼠和人的胚胎干细胞,改变RNA结合蛋白FUS的表达水平,检测circRNA的变化,发现FUS可结合到侧翼内含子区,调节circRNA的形成。关于人肿瘤研究中,活体实验常在小鼠皮下异体移植人癌变的肿瘤,再通过注射过表达载体或干扰片段来改变特定circRNA的表达,研究发现circRPKCI[80]、circ-Amotl1[81]、circCDYL[82]、circHI-PK3[83-84]、hsa_circ_0007385[85]、hsa_circ_00126-73[86]、circCCDC66[87]和circRNA_100782等circR-NA分子可促进肿瘤生长,circ-FBXW7[63]、circ-IT-CH[88]、circC3P1[89]、circFoxo3[90]和cirZKSCAN1[91]等circRNA分子可抑制肿瘤生成。
5.2 circRNA在家禽中的研究进展在家禽的研究中,circRNA在生长发育、肌肉发育、产蛋量和疾病防治中的作用是人们主要关注的内容。
Ouyang等[60-61]采集11胚龄(E11)、16胚龄(E16)、1日龄(P1)杏花鸡母鸡的腿肌,通过RNA-seq共鉴定出13 377个circRNA,其中有1 470个差异表达,有946个circRNA具有一个或多个miRNA结合位点,共涉及150个已知miRNA分子。进一步分析发现鸡circRBFOX2.2-3和circRBFOX2.2-4可吸附miR-206,促进鸡成肌细胞增殖,抑制成肌细胞分化,鸡circSVIL.6-14可结合miR-203,促进成肌细胞增殖和分化[12, 92]。利用iTRAQ技术鉴定出差异表达蛋白,并与circRNA的数据联合分析,结果在E11和P1比较组发现23个差异蛋白基因具有差异表达的circRNA。包括肌肉收缩通路相关的基因DMD、TNNT3、TNN、TPM1、TPM2和TPM3,其中DMD和TNN基因含有多个差异的circRNA[60]。由此可见,在鸡胚胎期肌肉发育过程中,circRNA通过吸附miRNA或参与调控蛋白表达等方式调控肌肉发育。
此外,Zhang等[93]用鸡禽白血病J (ALV-J)亚型侵染ALV抗性和ALV敏感性鸡,20周后采集肝脏组织。RNA-seq鉴定出1 800多个circRNA,其中有32个circRNA差异表达。且发现在ALV抗性组中的12个circRNA存在多个miRNA分子结合位点,基因功能富集分析发现主要集中于免疫通路。这表明circRNA可能参与ALV-J引起的免疫过程。
6 展望目前对于circRNA的研究仍处于起步阶段,有大量技术问题需要突破。随着高通量测序和生物信息学的快速发展,将会有越来越多的circRNA被发现,其功能研究会越来越深入。circBase[94]、Circ2Traits[95]、CircNet[96]等数据库中收录了近10万circRNA测序结果,并能预测circRNA-miRNA-mRNA相互作用。circRNA过表达载体和干扰手段不断趋于成熟,使人工调控细胞内circRNA的表达成为可能,利于进一步探索circRNA的功能[18, 97]。CircRNA与疾病发生密切相关,可作为癌症等疾病监测的新型靶标分子[58]。在畜禽研究方面,主要集中在生长发育、肌肉发育等经济性状相关的circRNA的研究。对于circRNA的功能注释仅限于circRNA的来源编码基因或可能的miRNA结合位点进行预测分析,因此需进一步探索circRNA作用网络,研究circRNA在基因表达调控的作用,加深畜禽的经济性状相关circRNA的筛选与鉴定,更好的为畜禽分子育种提供理论依据和技术指导。
| [1] |
Koch L. RNA: Translated circular RNAs[J]. Nature Reviews Genetics, 2017, 18(5): 272. |
| [2] |
Kolakofsky D. Isolation and characterization of Sendai virus DI-RNAs[J]. Cell, 1976, 8(4): 547-555. DOI:10.1016/0092-8674(76)90223-3 |
| [3] |
Sanger HL, Klotz G, Riesner D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci USA, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 |
| [4] |
Capel B, Swain A, Nicolis S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-1030. DOI:10.1016/0092-8674(93)90279-Y |
| [5] |
Zaphiropoulos PG. Circular RNAs from transcripts of the rat cytochrome P450 2C24 gene: correlation with exon skipping[J]. Proc Natl Acad Sci USA, 1996, 93(13): 6536-6541. DOI:10.1073/pnas.93.13.6536 |
| [6] |
Zaphiropoulos PG. Exon skipping and circular RNA formation in transcripts of the human cytochrome P-450 2C18 gene in epidermis and of the rat androgen binding protein gene in testis[J]. Molecular & Cellular Biology, 1997, 17(6): 2985-2993. |
| [7] |
Danan M, Schwartz S, Edelheit S, et al. Transcriptome-wide discovery of circular RNAs in Archaea[J]. Nucleic Acids Research, 2012, 40(7): 3131-3142. DOI:10.1093/nar/gkr1009 |
| [8] |
Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733 |
| [9] |
Jeck WR, Sorrentino JA, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 |
| [10] |
Hansen TB, Wiklund ED, Bramsen JB, et al. miRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA[J]. Embo Journal, 2011, 30(21): 4414-4422. DOI:10.1038/emboj.2011.359 |
| [11] |
Li H, Yang J, Wei X, et al. CircFUT10 reduces proliferation and facilitates differentiation of myoblasts by sponging miR-133a[J]. Journal of Cellular Physiology, 2017, 233(11): 4643-4651. |
| [12] |
欧阳宏佳.环状RNA对鸡胚胎骨骼肌发育的影响[D].广州: 华南农业大学, 2017.
|
| [13] |
Zhang M, Huang N, Yang X, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J]. Oncogene, 2018, 37(13): 1805-1814. DOI:10.1038/s41388-017-0019-9 |
| [14] |
Capel B, Swain A, Nicolis S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-1030. DOI:10.1016/0092-8674(93)90279-Y |
| [15] |
Salzman J, Chen RE, Olsen MN, et al. Cell-type specific features of circular RNA expression[J]. PLoS Genetics, 2013, 9(9): e1003777. DOI:10.1371/journal.pgen.1003777 |
| [16] |
Bolha L, Ravnik-Glavač M, Glavač D. Circular RNAs: biogenesis, function, and a role as possible cancer biomarkers[J]. Int J Genomics, 2017. DOI:10.1155/2017/6218535 |
| [17] |
Yun Y, Fan X, Mao M, et al. Extensive translation of circular RNAs driven by N6-methyladenosine[J]. Cell Research, 2017, 27(5): 626. DOI:10.1038/cr.2017.31 |
| [18] |
Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [19] |
Li XF, Lytton J. A circularized sodium-calcium exchanger exon 2 transcript[J]. J Biol Chem, 1999, 274(12): 8153-8160. DOI:10.1074/jbc.274.12.8153 |
| [20] |
Schwanhäusser B, Busse D, Li N, et al. Global quantification of ma-mmalian gene expression control[J]. Nature, 2011, 473(7347): 337-342. DOI:10.1038/nature10098 |
| [21] |
Umekage S, Uehara T, Fujita Y, et al. In vivo circular RNA expression by the permuted intron-exon method[M]. InTech, 2012.
|
| [22] |
Bachmayrheyda A, Reiner AT, Auer K, et al. Correlation of circular RNA abundance with proliferation-exemplified with colorectal and ovarian cancer, idiopathic lung fibrosis, and normal human tissues[J]. Science Report, 2015, 5(8057): 8057. |
| [23] |
Guo JU, Agarwal V, Guo H, et al. Expanded identification and characterization of mammalian circular RNAs[J]. Genome Biology, 2014, 15(7): 409. DOI:10.1186/s13059-014-0409-z |
| [24] |
Perriman R, Ares M Jr. Circular mRNA can direct translation of extremely long repeating-sequence proteins in vivo[J]. RNA, 1998, 4(9): 1047-1054. DOI:10.1017/S135583829898061X |
| [25] |
Chen CY, Sarnow P. Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs[J]. Science, 1995, 268(5209): 415-417. DOI:10.1126/science.7536344 |
| [26] |
Zhang Y, Zhang XO, Chen T, et al. Circular intronic long noncoding RNAs[J]. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [27] |
Zhang XO, Wang HB, Zhang Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 1: 134-147. |
| [28] |
Zhang F, Deng B, Wen J, et al. PPARγ and MyoD are differentially regulated by myostatin in adipose-derived stem cells and muscle satellite cells[J]. Biochem Biophys Res Commun, 2015(2): 375-380. |
| [29] |
Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature Structural & Molecular Biology, 2015, 22(3): 256-264. |
| [30] |
Zhang XO, Dong R, Zhang Y, et al. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs[J]. Genome Research, 2016, 26(9): 1277-1287. DOI:10.1101/gr.202895.115 |
| [31] |
Filippenkov IB, Sudarkina OY, Limborska SA, et al. Multi-step splicing of sphingomyelin synthase linear and circular RNAs[J]. Gene, 2018, 645: 14-23. |
| [32] |
Starke S, Jost I, Rossbach O, et al. Exon circularization requires canonical splice signals[J]. Cell Rep, 2015, 10(1): 103-111. DOI:10.1016/j.celrep.2014.12.002 |
| [33] |
Barrett SP, Wang PL, Salzman J. Circular RNA biogenesis can proceed through an exon-containing lariat precursor[J]. Elife Sciences, 2015, 4: e07540. DOI:10.7554/eLife.07540 |
| [34] |
Ivanov A, Memczak S, Wyler E, et al. Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals[J]. Cell Rep, 2015, 10(2): 170-177. DOI:10.1016/j.celrep.2014.12.019 |
| [35] |
Lasda E, Parker R. Circular RNAs: diversity of form and function[J]. RNA, 2014, 20(12): 1829. DOI:10.1261/rna.047126.114 |
| [36] |
Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs[J]. Nature Biotechnology, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890 |
| [37] |
Rui D, Ma XK, Chen LL, et al. Increased complexity of circRNA expression during species evolution[J]. RNA Biol, 2017, 14(8): 1064-1074. DOI:10.1080/15476286.2016.1269999 |
| [38] |
Ashwal-Fluss R, Meyer M, Pamudurti NR, et al. CircRNA biogenesis competes with pre-mRNA splicing[J]. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 |
| [39] |
Galarneau A, Richard S. Target RNA motif and target mRNAs of the Quaking STAR protein[J]. Nature Structural & Molecular Biology, 2005, 12(8): 691-698. |
| [40] |
Errichelli L, Dini MS, Laneve P, et al. FUS affects circular RNA expression in murine embryonic stem cell-derived motor neurons[J]. Nature Communications, 2017, 8: 14741. DOI:10.1038/ncomms14741 |
| [41] |
Zhang Y, Xue W, Li X, et al. The biogenesis of nascent circular RNAs[J]. Cell Rep, 2016, 15(3): 611-624. DOI:10.1016/j.celrep.2016.03.058 |
| [42] |
Kramer MC, Liang D, Tatomer DC, et al. Combinatorial control of Drosophila circular RNA expression by intronic repeats, hnRNPs, and SR proteins[J]. Gen Dev, 2015, 29(20): 2168-2182. DOI:10.1101/gad.270421.115 |
| [43] |
Meng X, Li X, Zhang P, et al. Circular RNA: an emerging key player in RNA world[J]. Brief Bioinform, 2016, 18(4): 547-557. |
| [44] |
Lu Z, Filonov GS, Noto JJ, et al. Metazoan tRNA introns generate stable circular RNAs in vivo[J]. RNA, 2015, 21(9): 1554-1565. DOI:10.1261/rna.052944.115 |
| [45] |
Guarnerio J, Bezzi M, Jeong JC, et al. Oncogenic role of fusion-circRNAs derived from cancer-associated chromosomal translocations[J]. Cell, 2016, 165(2): 289-302. DOI:10.1016/j.cell.2016.03.020 |
| [46] |
Holdt LM, Kohlmaier A, Teupser D. Molecular roles and function of circular RNAs in eukaryotic cells[J]. Cell Mol Life Sci, 2018, 75(6): 1071-1098. DOI:10.1007/s00018-017-2688-5 |
| [47] |
Dudekula DB, Panda AC, Grammatikakis I, et al. CircInteractome: a web tool for exploring circular RNAs and their interacting proteins and microRNAs[J]. RNA Biol, 2016, 13(1): 34-42. DOI:10.1080/15476286.2015.1128065 |
| [48] |
Jakub W, Pedro M, Sara O, et al. Genome-wide analysis of Drosophila circular RNAs reveals their structural and sequence properties and age-dependent neural accumulation[J]. Cell Reports, 2014, 9(5): 1966-1980. DOI:10.1016/j.celrep.2014.10.062 |
| [49] |
Huang G, Li S, Yang N, et al. Recent progress in circular RNAs in human cancers[J]. Cancer Letters, 2017, 404: 8-18. DOI:10.1016/j.canlet.2017.07.002 |
| [50] |
Chen LL. The biogenesis and emerging roles of circular RNAs[J]. Nat Rev Mol Cell Biol, 2016, 17(4): 205-211. DOI:10.1038/nrm.2015.32 |
| [51] |
Chao CW, Chan DC, Kuo A, et al. The mouse formin (Fmn) gene: abundant circular RNA transcripts and gene-targeted deletion analysis[J]. Mol Med, 1998, 4(9): 614-628. DOI:10.1007/BF03401761 |
| [52] |
Holdt LM, Stahringer A, Sass K, et al. Circular non-coding RNA ANRIL modulates ribosomal RNA maturation and atherosclerosis in humans[J]. Nature Communications, 2016, 7: 12429. DOI:10.1038/ncomms12429 |
| [53] |
Du WW, Yang W, Liu E, et al. Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2[J]. Nucleic Acids Res, 2016, 44(6): 2846-2858. DOI:10.1093/nar/gkw027 |
| [54] |
Du WW, Yang W, Chen Y, et al. Foxo3 circular RNA promotes cardiac senescence by modulating multiple factors associated with stress and senescence responses[J]. European Heart Journal, 2016, 38(18): 1402-1412. |
| [55] |
Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [56] |
Stoll L, Sobel J, Rodriguez-Trejo A, et al. Circular RNAs as novel regulators of beta-cell functions in normal and disease conditions[J]. Mol Metab, 2018, 9: 69-73. DOI:10.1016/j.molmet.2018.01.010 |
| [57] |
Peng L, Yuan XQ, Li GC. The emerging landscape of circular RNA ciRS-7 in cancer[J]. Oncol Rep, 2015, 33(6): 2669-2674. DOI:10.3892/or.2015.3904 |
| [58] |
Han D, Li J, Wang H, et al. Circular RNA MTO1 acts as the sponge of miR-9 to suppress hepatocellular carcinoma progression[J]. Hepatology, 2017, 66(4): 1151. DOI:10.1002/hep.29270 |
| [59] |
Liu T, Liu S, Xu Y, et al. Circular RNA-ZFR inhibited cell proliferation and promoted apoptosis in gastric cancer by sponging miR-130a/miR-107 and modulating PTEN[J]. Cancer Res Treat, 2018, 2017: 537. |
| [60] |
Ouyang H, Chen X, Wang Z, et al. Circular RNAs are abundant and dynamically expressed during embryonic muscle development in chickens[J]. DNA Res, 2017, 25(1): 71-86. |
| [61] |
Ouyang H, Nie Q, Zhang X. P3052 Characterization of Circular RNAs in relation to embryonic muscle development in chicken[J]. J Anim Sci, 2016, 94(supplement4): 79. DOI:10.2527/jas2016.94supplement479x |
| [62] |
Kos A, Dijkema R, Arnberg AC, et al. The hepatitis delta (delta) virus possesses a circular RNA[J]. Nature, 1986, 323(6088): 558-560. DOI:10.1038/323558a0 |
| [63] |
Yang Y, Gao X, Zhang M, et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. Journal of the National Cancer Institute, 2018, 110(3): djx166. |
| [64] |
Pamudurti NR, Bartok O, Jens M, et al. Translation of CircRNAs[J]. Molecular Cell, 2017, 66(1): 9. DOI:10.1016/j.molcel.2017.02.021 |
| [65] |
Lasda E, Parker R. Circular RNAs co-precipitate with extracellular vesicles: a possible mechanism for circRNA clearance[J]. PLoS One, 2016, 11(2): e0148407. DOI:10.1371/journal.pone.0148407 |
| [66] |
Ng WL, Marinov GK, Liau ES, et al. Inducible RasGEF1B circular RNA is a positive regulator of ICAM-1 in the TLR4/LPS pathway[J]. RNA Biol, 2016, 13(9): 861-871. DOI:10.1080/15476286.2016.1207036 |
| [67] |
Liang G, Yang Y, Niu G, et al. Genome-wide profiling of Sus scrofa circular RNAs across nine organs and three developmental stages[J]. DNA Res, 2017, 24(5): 523-535. DOI:10.1093/dnares/dsx022 |
| [68] |
Chen J, Zou Q, Lv D, et al. Comprehensive transcriptional landscape of porcine cardiac and skeletal muscles reveals differences of aging[J]. Oncotarget, 2018, 9(2): 1524-1541. |
| [69] |
Sun J, Xie M, Huang Z, et al. Integrated analysis of non-coding RNA and mRNA expression profiles of 2 pig breeds differing in muscle traits[J]. J Anim Sci, 2017, 95(3): 1092-1103. |
| [70] |
Li CY, Li XY, Yao Y, et al. Genome-wide analysis of circular RNAs in prenatal and postnatal muscle of sheep[J]. Oncotarget, 2017, 8(57): 97165-97177. |
| [71] |
Wei X, Li H, Yang J, et al. Circular RNA profiling reveals an abundant circLMO7 that regulates myoblasts differentiation and survival by sponging miR-378a-3p[J]. Cell Death & Disease, 2017, 8(10): e3153. |
| [72] |
Li H, Wei X, Yang J, et al. CircFGFR4 promotes differentiation of myoblasts via binding miR-107 to relieve its inhibition of Wnt3a[J]. Mol Ther Nucleic Acids, 2018, 11: 272-283. DOI:10.1016/j.omtn.2018.02.012 |
| [73] |
Venø MT, Hansen TB, Venø ST, et al. Spatio-temporal regulation of circular RNA expression during porcine embryonic brain development[J]. Genome Biology, 2015, 16(1): 245. DOI:10.1186/s13059-015-0801-3 |
| [74] |
Li C, Li X, Ma Q, et al. Genome-wide analysis of circular RNAs in prenatal and postnatal pituitary glands of sheep[J]. Science Report, 2017, 8(57): 97165-97177. |
| [75] |
Tao H, Xiong Q, Zhang F, et al. Circular RNA profiling reveals chi_circ_0008219 function as microRNA sponges in pre-ovulatory ovarian follicles of goats (Capra hircus)[J]. Genomics, 2017, 10(5): 1-10. |
| [76] |
Zhang C, Wu H, Wang Y, et al. Circular RNA of cattle casein genes are highly expressed in bovine mammary gland[J]. Journal of Dairy Science, 2016, 99(6): 4750-4760. DOI:10.3168/jds.2015-10381 |
| [77] |
Zhu S, Wang YH, Zhao Y, et al. Identification and partial characterization of a novel circular transcript of the Tc2n gene from rat mammary gland[J]. Archives of Biological Sciences, 2016, 68(2): 257-262. DOI:10.2298/ABS151015013Z |
| [78] |
Pei W, Tao L, Zhang LW, et al. Circular RNA profiles in mouse lung tissue induced by radon[J]. Environmental Health & Preventive Medicine, 2017, 22(1): 36. |
| [79] |
Huang JL, Qin MC, Zhou Y, et al. Comprehensive analysis of differentially expressed profiles of Alzheimer's disease associated circular RNAs in an Alzheimer's disease mouse model[J]. Aging, 2018, 10(2): 253-265. DOI:10.18632/aging.v10i2 |
| [80] |
Qiu M, Xia W, Chen R, et al. The circular RNA circPRKCI promotes tumor growth in lung adenocarcinoma[J]. Cancer Research, 2018, 78(11): 2839-2851. DOI:10.1158/0008-5472.CAN-17-2808 |
| [81] |
Yang Q, Du WW, Wu N, et al. A circular RNA promotes tumorigenesis by inducing c-myc nuclear translocation[J]. Cell Death & Differentiation, 2017, 24(9): 1609-1620. |
| [82] |
Li Y, Zheng Q, Bao C, et al. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis[J]. Cell Research, 2015, 25(8): 981-984. DOI:10.1038/cr.2015.82 |
| [83] |
Zeng K, Chen X, Xu M, et al. CircHIPK3 promotes colorectal cancer growth and metastasis by sponging miR-7[J]. Cell Death and Disease, 2018, 9: 417. DOI:10.1038/s41419-018-0454-8 |
| [84] |
Chen G, Shi Y, Liu M, et al. circHIPK3 regulates cell proliferation and migration by sponging miR-124 and regulating AQP3 expression in hepatocellular carcinoma[J]. Cell Death & Disease, 2018, 9(2): 175. |
| [85] |
Jiang MM, Mai ZT, Wan SZ, et al. Microarray profiles reveal that circular RNA hsa_circ_0007385 functions as an oncogene in non-small cell lung cancer tumorigenesis[J]. Journal of Cancer Research & Clinical Oncology, 2018, 144(4): 667-674. |
| [86] |
Wang X, Zhu X, Zhang H, et al. Increased circular RNA hsa_circ_0012673 acts as a sponge of miR-22 to promote lung adenocarcinoma proliferation[J]. Biochem Biophys Res Commun, 2018, 496(4): 1069-1075. DOI:10.1016/j.bbrc.2018.01.126 |
| [87] |
Hsiao KY, Lin YC, Gupta SK, et al. Noncoding effects of circular RNA CCDC66 promote colon cancer growth and metastasis[J]. Cancer Research, 2017, 77(9): 2339-2350. DOI:10.1158/0008-5472.CAN-16-1883 |
| [88] |
Yang C, Yuan W, Xiao Y, et al. Circular RNA circ-ITCH inhibits bladder cancer progression by sponging miR-17/miR-224 and regulating p21, PTEN expression[J]. Molecular Cancer, 2018, 17(1): 19. DOI:10.1186/s12943-018-0771-7 |
| [89] |
Zhong L, Wang Y, Cheng Y, et al. Circular RNA circC3P1 suppresses hepatocellular carcinoma growth and metastasis through miR-4641/PCK1 pathway[J]. Biochem Biophys Res Commun, 2018, 499(4): 1044-1049. DOI:10.1016/j.bbrc.2018.03.221 |
| [90] |
Du WW, Fang L, Yang W, et al. Induction of tumor apoptosis through a circular RNA enhancing Foxo3 activity[J]. Cell Death & Differentiation, 2016, 24(2): 357-370. |
| [91] |
Yao Z, Luo J, Hu K, et al. ZKSCAN1 gene and its related circular RNA (circZKSCAN1) both inhibit hepatocellular carcinoma cell growth, migration, and invasion but through different signaling pathways[J]. Molecular Oncology, 2017, 11(4): 422-437. DOI:10.1002/1878-0261.12045 |
| [92] |
Ouyang H, Chen X, Li Z, et al. Circular RNA circSVIL promotes myoblast proliferation and differentiation by sponging miR-203 in chicken[J]. Frontiers in Genetics, 2018, 9: 172. DOI:10.3389/fgene.2018.00172 |
| [93] |
Zhang X, Yan Y, Lei X, et al. Circular RNA alterations are involved in resistance to avian leukosis virus subgroup-J-induced tumor formation in chickens[J]. Oncotarget, 2017, 8(21): 34961-34970. |
| [94] |
Çlaåžar P, Papavasileiou P, Rajewsky N. CircBase: a database for circular RNAs[J]. RNA, 2014, 20(11): 1666-1670. DOI:10.1261/rna.043687.113 |
| [95] |
Ghosal S, Das S, Sen R, et al. Circ2Traits: a comprehensive database for circular RNA potentially associated with disease and traits[J]. Frontiers in Genetics, 2013, 4(283): 283. |
| [96] |
Liu YC, Li JR, Sun CH, et al. CircNet: a database of circular RNAs derived from transcriptome sequencing data[J]. Nucleic Acids Res, 2016, 44(D1): D209-D215. DOI:10.1093/nar/gkv940 |
| [97] |
Conn SJ, Pillman KA, Toubia J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015, 160(6): 1125-1134. DOI:10.1016/j.cell.2015.02.014 |