2. 中国科学院大学, 北京 100049;
3. 河南师范大学水产学院, 新乡 453007
2. University of Chinese Academy of Sciences, Beijing 100049;
3. College of Fisheries Henan Normal University, Xinxiang 453007
温度是影响鱼类生理、行为及进化过程中最重要的环境因素。作为变温动物, 由于缺乏自身的体温调控系统, 鱼类的体温与环境温度基本一致, 机体内多种生理、生化过程直接受到环境温度的调控, 这使得鱼类对环境温度的依赖性极高。鱼类的低温适应是一个长期进化的过程, 在与低温环境的长期博弈中鱼类也进化形成了多种适应机制以保证其能够在昼夜、季节甚至常年低温的环境下生存[1-3]。鲤(Cyprinus carpio)是一种分布于世界各地的古老经济鱼类, 通过长期的人工驯化, 已形成上百个品种, 具有丰富的遗传多态性[4]。由于其适中的体型、与模式生物斑马鱼(Danio rerio)近缘、广温适应等特点, 鲤也是研究变温动物低温适应的理想实验材料[5]。
转录组测序技术(RNA-seq technology)能够在整体水平上较为全面地挖掘低温响应基因集, 并能够动态监测其转录变化规律。Gracey等[6]利用由13 440个EST探针组成的芯片对鲤响应低温胁迫时多组织的转录变化进行检测, 获得3 400个低温响应基因, 其中多数基因的表达调控具有器官、组织特异性。Liang等[7]通过RNA-seq对具有耐低温性状的黑龙江野鲤多组织的转录活动进行检测, 发现大量低温响应基因, 其中参与蛋白定位及蛋白转运等生物学过程的基因被显著富集, 参与生物节律调控、内质网蛋白加工、内吞作用、胰岛素信号通路及溶酶体等有关通路的基因被显著富集。
能量代谢调控是保证鱼类在长期低温环境下维持基本生理活动的基础。肝脏是脊椎动物能量代谢过程最重要的功能器官, 也是衡量鱼类生理状态时重点监测的指征器官[8]。由于鱼类肝脏(鲤科鱼类中则为肝胰腺)在糖原储备上有一定的缺陷, 肝脏中糖质新生等内源性生糖途径在鱼类维持机体葡萄糖平衡具有重要的作用[9]。然而, 目前尚无针对低温耐受差异鲤品种在低温胁迫条件下肝胰腺代谢活动相关分子机制的研究。因此, 本研究以低温耐受品种松浦镜鲤, 低温敏感品种荷包红鲤为研究对象, 通过RNA-seq技术纵向比较各品种肝胰腺中低温响应基因集, 同时横向比较品种间低温响应差异基因, 并将二者相结合相对全面地进行分析, 有利于鉴定鲤低温耐受相关重要基因及其参与的功能通路, 从而探索鱼类低温耐受的潜在分子调控机制。
1 材料与方法 1.1 材料荷包红鲤20尾(无直接亲缘关系), 松浦镜鲤20尾(无直接亲缘关系)均来自新乡金龙生态养殖基地, 为人工同期授精得到的6月龄幼鱼。
1.2 方法 1.2.1 低温试验处理及样品制备将40尾参试鱼从暂养池(水温24℃)随机分为两组(10尾/品种):常温对照组(24℃)、低温组(4℃)。对于低温组的降温过程为:以4℃/h的降温过程进行, 直到降至实验设定温度。实验期间两组除温度差异外, 饲喂、供氧、水循环及管理一致。在低温驯化后的15 d对各组进行采样, 每组随机取6尾健康个体进行采样。取样时用丁香酚将鲤麻醉后立即解剖取肝胰腺, 液氮暂存。提取各样本总RNA(提取试剂盒为:RNAprep Pure Tissue Kit, TIANGEN), 对提取的总RNA进行纯度和质量检测, 后将每组的总RNA样本进行等含量混合成3个生物学重复(每组中的2个RNA样本等量混为1个样本)。
1.2.2 cDNA文库构建及Illumina测序通过带有OligodT(25)的磁珠富集、纯化mRNA。以mRNA为模板合成cDNA。按照标准的Illumina建库流程对每个测序样本的cDNA构建小片段测序文库。采用2×100 pair-end的模式在Illumina HiSeq2000测序平台进行测序。
1.2.3 转录本重构及基因注释利用比对软件STAR1(2.5.0)将每个样本的clean reads比对到参考基因组上(基因组来源:http://www.carpbase.org/index.php), 使用cuffmerge与原有的基因组注释信息进行比较合并, 除去小于200 bp的转录本, 获得最终的转录本。取每个基因中最长的转录本作为Unigene, 基于Unigene进行基因的功能注释。通过Trinotate软件对Unigene行功能注释。Trinotate软件整合了多个数据库, 分别是蛋白数据库NCBI-nr、SwissProt、KEGG和COG/KOG数据库。比对得到跟Unigene序列相似性最高的蛋白, 从而得到该Unigene的蛋白功能注释信息。
1.2.4 表达丰度估计对重构的转录本进行表达丰度估计。首先使用bowtie 2.0将每个样品的reads分别比对到组装出来的转录本上, 然后使用RSEM计算比对到特定基因或转录本上的reads数目。为了便于不同基因之间进行表达量的比较, 使用FPKM对表达量进行标准化。FPKM(Expected number of Fragments Per Kilobase of transcript sequence per Millions base pairs sequenced)是每百万fragments中来自某一基因每千个碱基长度的fragments数目, 其同时考虑了测序深度和基因长度对fragments计数的影响。
1.2.5 差异表达分析通过Cufflink软件对两两实验处理组之间的比较, 鉴定差异表达的基因。基因差异表达的计算是基于基因表达水平分析中得到的read count数据。使用DESeq程序进行差异分析, 筛选阈值为FDR < 0.05且|log2FoldChange| > 1, 从而获得差异表达矩阵。基于差异表达矩阵, 对差异表达基因进行功能及代谢通路的富集分析(GO和KEGG富集分析)。
1.2.6 差异表达基因的qRT-PCR验证从糖酵解/糖质新生代谢通路(Pathway ID:ko00010)中选取5个编码酶蛋白的差异基因(cycg001314、cy-cg034145、cycg035940、cycg021976和cycg041259)进行荧光定量PCR验证分析, 样品取材与RNA-seq一致。根据相关文献[10]选择鲤β-actin基因为内参, 使用Primer Primers 6.0设计扩增引物, 引物信息见表 1。反应体系如下:上游引物1 μL, 下游引物1 μL, SYBR green 10 μL, cDNA模板1 μL以及ddH2O 7 μL。反应程序设置为:95℃ 30 s; 95℃ 5 s, 60℃ 30 s, 40个循环。相对表达量按照2-ΔΔCT方法计算, 利用R语言基础软件包中的LSD法对样品中各基因表达量变化做统计学分析, 以P < 0.05为具有显著性差异。所有反应设置3次技术重复, 所有样本设置3次生物学重复。
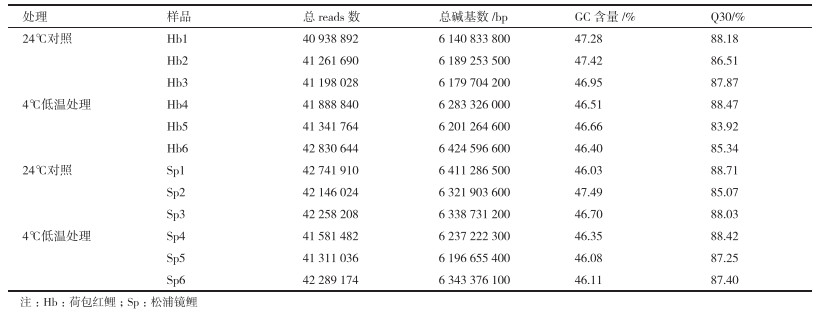
采用Illumina HiSeq2000高通量测序平台对2个鲤品种在低温胁迫15 d后肝胰腺进行转录组测序, 如表 2所示, 各测序样品GC含量在46.03%-47.49%, 较为一致, Q30在85%以上, 说明RNA-seq数据量和质量都较高, 为后续的数据拼接组装提供了可靠的数据源。
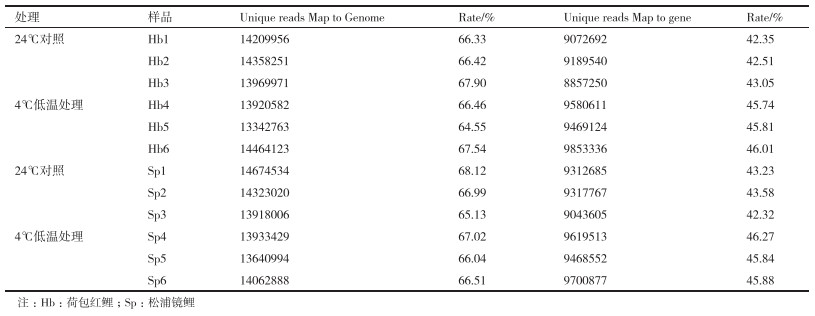
对样品reads在参考基因组的比对效率进行分析(表 3), 各样本reads在参考基因组的比对率在64.55%-67.90%, 且品种间无显著差异; 各样本reads比对到注释基因上的比对率在42.51%-46.27%, 较为一致。对转录组测序所获得的reads通过组装和拼接, 获得转录本总长度达366100425bp(约349.14 Mb), 平均长度为2 260 bp, N50为3 301 bp。对转录本提取Unigene后共获得73 088个Unigene, 其中包括17 088个新基因。
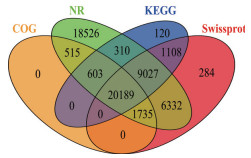
将Unigene序列比对到蛋白数据库NR、SwissProt、KEGG和COG/KOG进行基因注释, 如图 1显示。73 088个Unigene序列在NR数据库中有57 237(占78.31%)个基因被成功注释, 在SwissProt数据库中有38 985(53.34%)个基因得到注释; 在KEGG数据库中有31 357(42.90%)个基因得到注释; 在COG/KOG数据库中有23 042(31.53%)个基因得到注释; 4个数据库一共注释了58 153(79.57%)个Unigene序列, 未能够得到注释的Unigene序列有14 935(20.43%)个, 有20 189(27.62%)个Unigene序列序列被4大数据库同时注释。绝大多数基因可以通过NR数据库得到注释, 只注释到KEGG、COG数据库的基因相对较少, 分别为360和995。多数基因可以同时至少在2个数据库得到注释。
|
| 图 1 Unigene功能注释韦恩统计图 |
对重构的转录本进行表达定量, 筛选FDR < 0.05且至少在两组之间表达差异2倍以上(|log2FoldChange| > 1)的基因后, 共获得由10 521个基因组成差异表达矩阵。根据矩阵中基因的FPKM值进行PCA分析。在PC1-PC2二维坐标系中(图 2), 相同品种、温度处理的样本能够聚在一起, 说明样本重复性良好; 而不同品种、温度处理的样本分布分散, 表明低温胁迫后能够明显改变鲤肝胰腺的整体转录活动, 且低温耐受鲤品种和低温敏感鲤品种在响应低温胁迫时肝胰腺的转录应答模式差异较大。

|
| 图 2 差异表达矩阵PCA分析 Hb:荷包红鲤; Sp:松浦镜鲤 |
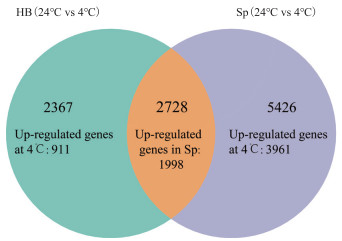
通过温恩分析, 对10 521个差异基因在品种间的分布进行统计。如图 3所示, 与24℃对照组相比, 荷包红鲤中有2 367个特异基因在低温下的表达量发生了明显变化, 其中911个基因表达上调; 松浦镜鲤中共有5 426个特异基因在低温下的表达量发生了明显变化, 其中3 961个基因表达上调。另外, 共有2 728个基因是在松浦镜鲤和荷包红鲤中在低温胁迫后都发生表达变化的基因, 其中, 1 998个基因在松浦镜鲤表达上调, 1 303个基因在荷包红鲤表达上调, 1 017个基因在两个鲤品种中均表现为低温诱导上调。
|
| 图 3 差异表达基因韦恩统计图 Hb:荷包红鲤; Sp:松浦镜鲤 |
基于差异表达矩阵, 对品种内低温胁迫差异表达基因(与各品种24℃对照组比较)及品种间差异表达基因进行功能及代谢通路的富集分析(GO和KEGG富集分析)。
GO分类可分为生物学过程、细胞组分和分子功能3大类条目。分别对荷包红鲤、松浦镜鲤各自的差异表达基因及其共有的差异基因进行GO富集分析。对3大类条目中显著富集基因最多的5个类别进行统计分析, 如图 4所示, 在生物学过程条目中, 基因多富集在转录、转录调控、小分子代谢过程、基于RNA聚合酶Ⅱ型启动子的转录调控等方面; 在细胞组分条目中, 基因多富集在胞核、胞质及胞包膜成分; 在分子功能条目中, 参与ATP结合、锌离子结合、金属离子结合的基因所占比例最高。在富集基因最多的GO分类条目上, 松浦镜鲤与和荷包红鲤基本一致, 但在松浦镜鲤鲤中参与上述生物学过程的基因数目都显著多于荷包红鲤。
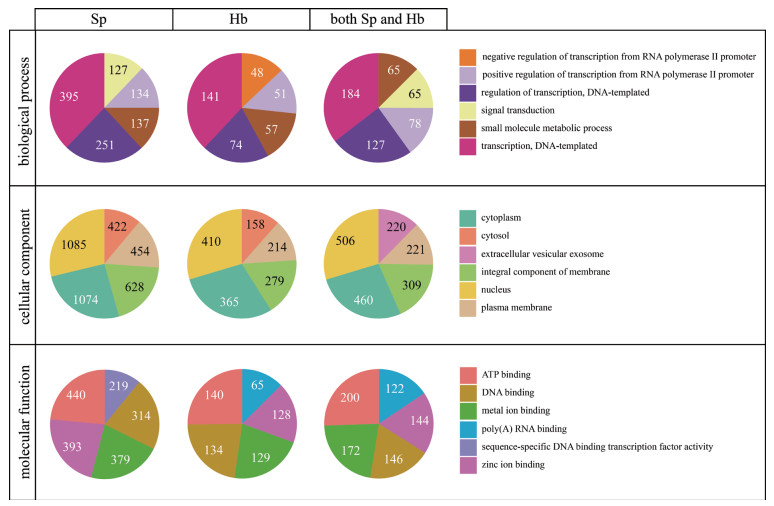
|
| 图 4 GO富集分析 Hb:荷包红鲤; Sp:松浦镜鲤 |
分别对荷包红鲤、松浦镜鲤的差异表达基因在KEGG数据库中进行代谢通路富集分析。对富集基因最多的10个KEGG二级条目进行统计分析, 如图 5所示, 富集在多种信号传导、翻译、内分泌系统等代谢通路中的基因所占比例最大。在松浦镜鲤中, 蛋白质折叠、排序及降解过程、糖酵解/糖质新生过程被显著富集。在糖酵解/糖质新生代谢途径中(Pathway ID:ko00010), 多种编码关键酶或催化亚基基因的表达量发生了显著的变化(图 6), 其中包括乳酸脱氢酶、1, 6-二磷酸果糖激酶、6-磷酸果糖激酶、葡萄糖-6-磷酸酶、己糖激酶、葡萄糖激酶、葡萄糖磷酸变位酶、葡萄糖-6-磷酸异构酶、丙酮酸脱氢酶等多种关键酶。
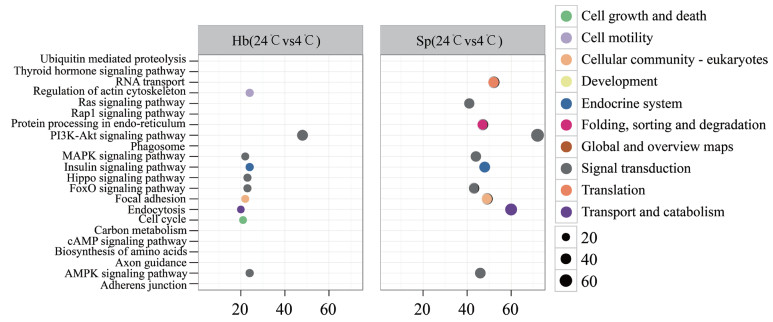
|
| 图 5 KEGG富集分析 Hb:荷包红鲤; Sp:松浦镜鲤 |
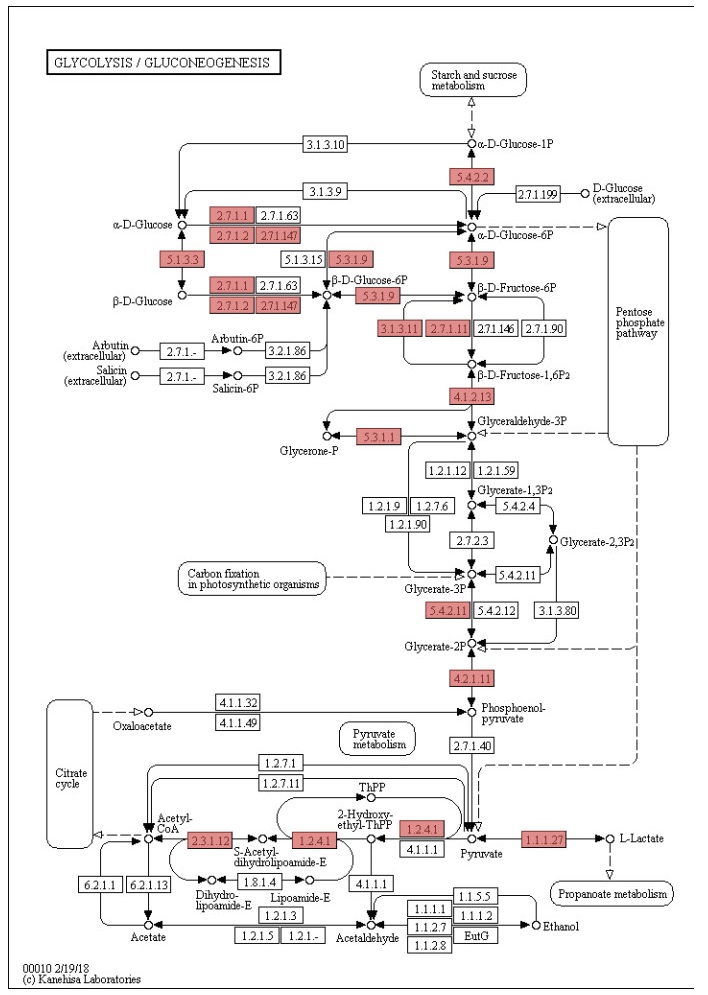
|
| 图 6 糖酵解/糖质新生代谢通路富集结果 |
进一步对表达上调/下调基因进行GO和KEGG富集分析(表 4), 发现在富集基因最多的GO和KEGG条目中, 松浦镜鲤低温诱导表达上调的基因数目显著多于荷包红鲤。其中, 参与转录调控等生物学过程的基因在松浦镜鲤中的上调/下调比显著高于荷包红鲤, 富集在DNA模板介导转录过程的基因, 在荷包红鲤表现为明显的低温表达下调的趋势, 而在松浦镜鲤表现为低温表达上调; 参与细胞组分的基因在两个鲤品种中的上调/下调比值较为接近, 且近似于1:1;而在分子功能条目中, 荷包红鲤多数基因的上调/下调比接近于1:1, 而松浦镜鲤中的表达上调基因数显著多于表达下调基因数; 在细胞凋亡通路中, 荷包红鲤比松浦镜鲤有更多的基因发生低温表达上调和下调。
从糖酵解/糖质新生代谢通路(Pathway ID:ko00010)中选取5个编码葡萄糖-6-磷酸酶、1, 6-二磷酸果糖激酶、磷酸烯醇式丙酮酸激酶、柠檬酸合酶及乳酸脱氢酶等关键酶蛋白的差异基因:g6pc、fbp、pck、cisy和ldhb2(基因ID分别是:cycg001314、cycg034145、cycg035940、cycg021976和cycg041259), 运用qRT-PCR的方法, 对低温胁迫下荷包红鲤及松浦镜鲤幼鱼肝胰腺中的表达模式进行分析。通过qRT-PCR验证(图 7-A-E), 低温胁迫后5个基因在松浦镜鲤中的表达量都显著的高于荷包红鲤, 其表达趋势与RNA-seq定量结果(图 7-F)一致。对两种方法下基因表达变化倍数值的相关性分析, 两种方法所得结果的相关系数达到0.89, 结果高度一致, 进一步验证了转录组测序数据的可靠性。
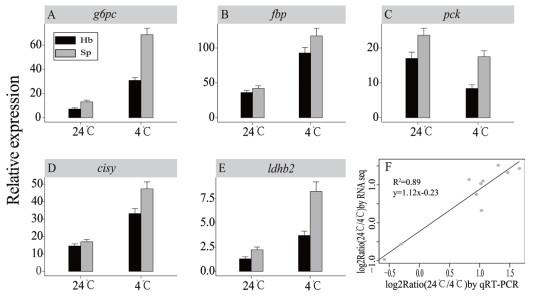
|
| 图 7 qRT-PCR定量结果及相关性验证 Hb:荷包红鲤; Sp:松浦镜鲤 |
鱼类作为变温脊椎动物, 其生长、繁殖等重要的生命活动更易受到环境温度影响。长期低温胁迫对鱼类的摄食活动、应激反应、营养代谢等生理、生化活动会产生显著地影响[11], 同种鱼类由于遗传背景和地理分布的差异, 也会表现出迥然不同的低温适应能力[12]。荷包红鲤的育成地我国南方低纬度温暖地区, 常年自然水温都保持在10℃以上, 而松浦镜鲤主产区在我国东北高纬度的寒冷地区, 冰封期长达6个月之久, 水温稳定维持在0-4℃。本研究通过RNA-seq技术对上述2个低温耐受能力不同的鲤品种在低温胁迫条件下肝胰腺的转录活动进行检测, 发现在转录模式上二者具有显著的差异。通过差异表达分析, 松浦镜鲤在低温胁迫条件下差异表达基因数目显著多于荷包红鲤, 这个可能是松浦镜鲤在长期低温适应过程中进化而来的一种复杂网络调控机制, 但也有可能是由于两个参试品种与参考基因组来源品种[4]亲缘关系不同, reads比对效率差异导致的结果(松浦镜鲤与参考基因组品种具有较近的亲缘关系)。
为了排除reads比对效率对结果的影响, 对两种鲤RNA-seq reads在参考基因组上的比对率进行分析(表 3), 发现两个鲤品种间的基因组比对率及基因比对率并无显著的差异, 故而可以排除品种间序列比对效率差异对差异表达基因挖掘造成的影响。因此推测松浦镜鲤在低温胁迫过程中调动更多的基因参与低温应答可能是保证其在长期低温环境下生存的重要分子调控过程。值得注意的是, Xin等[13]在研究葡萄低温适应的分子调控机制时同样发现在低温胁迫下, 耐低温葡萄品种比非耐低温葡萄品种转录谱的表达变化更为广泛。
相似地结果表明, 在长期的进化过程中, 低温适应的物种会调动更多的功能基因参与抵抗低温带来的不良影响, 而这种特殊的转录变化在不同的物种间都较为普遍。耐低温鲤品种不但比低温敏感鲤品种具有更为广泛的低温转录谱变化, 其差异表达基因的表达上调/下调模式与后者也有显著地差异。例如, 多数富集在DNA模板介导转录过程的基因, 在耐低温的松浦镜鲤中表现为低温表达上调, 而在低温敏感的荷包红鲤中则是表达下调的趋势。这说明在低温胁迫下, 低温耐受品种表现出更为强烈的正向转录调控。
更多参与转录正调控、信号转导、能量代谢、内分泌等生理活动的基因表达激活、上调都有助于耐低温对低温环境的适应; 而低温敏感品种中大量参与细胞凋亡、转录负调控等生理活动的基因表达上调则可能是低温对其细胞造成损害导致的。除此之外, 大量参与细胞细胞膜、细胞质膜及细胞基质等组分的功能基因在低温胁迫下发生转录变化, 而低温敏感品种与低温耐受品种的上调/下调比较为相似, 而以往的研究也证实了低温会改变鱼类细胞膜的流动性和通透性[14], 以上说明低温环境造成的细胞结构组分的改变、重组是普遍存在于鱼类中的。
当环境温度低于适宜的生理温度范围时, 鱼类的摄食行为会减弱, 而食物转化率也会受到抑制[15]。同时, 鱼类为抵抗低温环境而发生的一系列行为、生理生化变化本身也是一个耗能的过程。当外源性能源的获取受到抑制时, 机体需要动员更多的内源性能源物质或潜在产能底物来提供能量应对低温环境。前人的研究发现, 鱼类在响应低温胁迫时, 血糖会显著的升高以应对不良胁迫造成的生理压力[16-17]。由于鱼类对直接产能物质(肝糖原、肌糖原等)的贮备能力有限[9], 糖质新生作用能够利用机体内源产能物质保证机体重要生理活动的能源补给, 在长期的低温适应过程中需要持续葡萄糖供给, 这对于维持鲤机体血糖稳定起关键作用。与Long等[18-19]在斑马鱼(Danio rerio)低温适应的研究结果类似, 本研究发现糖质新生过程的关键酶编码基因g6pc、fbp和pck的表达量在鲤低温适应过程中都显著地升高了, 而其在松浦镜鲤的表达量都显著高于荷包红鲤, 也进一步说明糖质新生途径对鲤低温适应的重要性。当鱼类应对低温等非生物胁迫时耗氧量会增加并且产生乳酸[20], 乳酸脱氢酶将乳酸催化生成丙酮酸, 而后者是糖质新生作用的重要底物。利用乳酸作为产能物质既是一种经济的能源利用策略也能防止机体的乳酸或丙酮酸中毒。Kuo等[16]发现具有耐低温性状的草鱼(Ctenopharyngodon idella)在低温胁迫下乳酸脱氢酶的活性显著高于适温性的虱目鱼(Chanos chanos)。本研究发现在低温胁迫后, 编码乳酸脱氢酶催化亚基的ldhb2在松浦镜鲤中的表达量显著高于荷包红鲤, 也说明乳酸脱氢酶在鱼类的低温适应中起关键作用, 且其活性及mRNA转录水平可以作为评价鱼类低温耐受能力的生化指标。
4 结论本研究采用RNA-Seq技术比较分析了低温敏感鲤品种荷包红鲤与低温耐受鲤品种松浦镜鲤在低温胁迫15 d后的肝胰腺转录组数据, 通过差异表达分析筛选出10 521个差异基因, 其中5 426个基因仅在松浦镜鲤中表达变化显著。低温胁迫会显著改变鲤肝胰腺的转录活动, 编码糖酵解/糖质新生途径的多个关键酶基因在低温耐受品种的表达量显著高于低温敏感品种, 推测这些基因编码蛋白功能有助于其低温适应。通过对低温耐受、低温敏感性状极端鲤品种的比较转录分析, 有利于鉴定鲤低温耐受相关重要基因及其参与的功能通路, 从而探索鱼类低温耐受的潜在分子调控机制。
| [1] |
Vergauwen L, Benoot D, Blust R, et al. Long-term warm or cold acclimation elicits a specific transcriptional response and affects energy metabolism in zebrafish[J]. Comparative Biochemistry & Physiology Part A Molecular & Integrative Physiology, 2010, 157(2): 149-157. |
| [2] |
Tiku PE, Gracey AY, Macartney AI, et al. Cold-induced expression of delta 9-desaturase in carp by transcriptional and posttranslational mechanisms[J]. Science, 1996, 271(5250): 815. DOI:10.1126/science.271.5250.815 |
| [3] |
Zerai DB, Fitzsimmons KM, Collier RJ. Transcriptional response of delta-9-desaturase gene to acute and chronic cold stress in Nile tilapia, Oreochromis niloticus[J]. Journal of the World Aquaculture Society, 2010, 41(5): 800-806. DOI:10.1111/jwas.2010.41.issue-5 |
| [4] |
Xu P, Zhang X, Wang X, et al. Genome sequence and genetic diversity of the common carp, Cyprinus carpio[J]. Nature Genetics, 2014, 46(11): 1212. DOI:10.1038/ng.3098 |
| [5] |
Kolder IC, Sj PD, Tan G, et al. A full-body transcriptome and proteome resource for the European common carp[J]. BMC Genomics, 2016, 17(1): 701. DOI:10.1186/s12864-016-3038-y |
| [6] |
Gracey AY, Fraser EJ, Li WZ, et al. Coping with cold:An integrative, multitissue analysis of the transcriptome of a poikilothermic vertebrate[J]. Proc Natl Acad Sci USA, 2004, 101(48): 16970. DOI:10.1073/pnas.0403627101 |
| [7] |
Liang L, Chang Y, He X, et al. Transcriptome analysis to identify cold-responsive genes in amur carp(Cyprinus carpio haematopterus)[J]. PLoS One, 2015, 10(6): e0130526. DOI:10.1371/journal.pone.0130526 |
| [8] |
Qian B, Xue L. Liver transcriptome sequencing and de novo annotation of the large yellow croaker(Larimichthy crocea)under heat and cold stress[J]. Marine Genomics, 2016, 25: 95-102. DOI:10.1016/j.margen.2015.12.001 |
| [9] |
Moon TW, Foster GD. Chapter 4 tissue carbohydrate metabolism, gluconeogenesis and hormonal and environmental influences[J]. Biochemistry & Molecular Biology of Fishes, 1995, 4(6): 65-100. |
| [10] |
Murakami M, Ohi M, Ishikawa S, et al. Adaptive expression of uncoupling protein 1 in the carp liver and kidney in response to changes in ambient temperature[J]. Comparative Biochemistry & Physiology Part A Molecular & Integrative Physiology, 2015, 185: 142-149. |
| [11] |
于淼, 胡雪松, 李池陶, 等. 松浦镜鲤越冬期的形态、组织结构及生化组成变化[J]. 中国水产科学, 2015, 22(3): 460-468. |
| [12] |
Petricorena ZL, Somero GN. Biochemical adaptations of notothenioid fishes:comparisons between cold temperate South American and New Zealand species and Antarctic species[J]. Comparative Biochemistry & Physiology Part A Molecular & Integrative Physiology, 2007, 147(3): 799-807. |
| [13] |
Xin H, Zhu W, Wang LN, et al. Genome wide transcriptional profile analysis of Vitis amurensis and vitis Vinifera in response to cold stress[J]. PLoS One, 2012, 8(3): 58740. |
| [14] |
赵小立, 徐德主, 张铭.低温胁迫中两种鱼类细胞膜流动性变化的比较[C].华东六省一市生物化学与分子生物学会2003年学术交流会论文摘要集, 2003.
|
| [15] |
Abbink W, Garcia AB, Roques JAC, et al. The effect of temperature and PH on the growth and physiological response of juvenile yellowtail kingfish Seriola lalandi in recirculating aquaculture systems[J]. Aquaculture, 2012, s330-333(1): 130-135. |
| [16] |
Kuo CM, Hsieh SL. Comparisons of physiological and biochemical responses between milkfish(Chanos chanos)and grass carp(Ctenopharyngodon idella)to cold shock[J]. Aquaculture, 2006, 251(2-4): 525-536. DOI:10.1016/j.aquaculture.2005.05.044 |
| [17] |
Qiang J, He J, Yang H, et al. Temperature modulates hepatic carbohydrate metabolic enzyme activity and gene expression in juvenile GIFT tilapia(Oreochromis niloticus)fed a carbohydrate-enriched diet[J]. Journal of Thermal Biology, 2014, 40(1): 25. |
| [18] |
Long Y, Song GL, Yan JJ, et al. Transcriptomic characterization of cold acclimation in larval zebrafish[J]. BMC Genomics, 2013, 14(1): 612. DOI:10.1186/1471-2164-14-612 |
| [19] |
Long Y, Li LC, Li Q, et al. Transcriptomic characterization of temperature stress responses in larval zebrafish[J]. PLoS One, 2012, 7(5): e37209. DOI:10.1371/journal.pone.0037209 |
| [20] |
Blagojevic DP, Grubor-Lajsic GN, Spasic M B. Cold defence responses:the role of oxidative stress[J]. Frontiers in Bioscience, 2011, 3(2): 416. |