杏扁(Armeniaca vulgaris×A.sibirica)是我国特有的杏属栽培品种, 属于苦杏仁甙含量低仁用杏类型。杏仁食用价值大, 不仅含有大量的糖类、蛋白质、脂类等营养成分, 而且有丰富的铜、锌、钾、磷、铁等无机微量元素及10多种维生素, 是滋养补身的上佳果品。据2015年统计, 全国杏扁种植面积超过约333 hm2, 年产甜杏仁约合5.13万t, 出口甜杏仁1.0-1.5万t, 创汇居出口土特产首位[1]。杏扁种植在北方, 属于早花植物, 早春开花时易受到低温倒寒流的冻害, 造成杏果的大量减产, 严重时甚至颗粒无收, 故冻害一直是杏扁的种植和生产的最大威胁[2-4]。自20世纪70年代开始, 我国的研究者对杏扁的抗寒性生理机制进行了大量的研究, 期望选育出抗冻害的优良树种。可是杏扁的抗寒性受到内在生长发育时营养物质含量和外在栽培条件等因素的影响, 仅仅通过生理指标的评判易造成误差, 导致抗寒育种进程缓慢。因此, 要从根本上解决杏扁的抗寒性, 需要从DNA水平上分析杏扁的遗传背景, 构建连锁图谱和抗寒性QTL研究。
自Weeden等[5]提出了“双假测交”(Double pseudo test cross)的理论后, 国内外的研究者对鲜食杏遗传图谱构建的研究取得了一定的进展。Hurtado等[6]利用AFLP、RAPD、RFLP和SSR标记, 以‘Goldrich’בValenciano’杂交的F1代为作图群体, 构建了父母本的遗传连锁图谱。Vilanova等[7]以‘Stark Early Orange’בTyrinthos’的F1代自交获得的76株F2群体为材料, 采用AFLP和SSR标记构建了杏的遗传连锁图谱。国内, 章秋平等[8]利用杏‘串枝红’ב金太阳’的F1为作图群体, 采用SSR和SRAP标记构建了双亲的连锁图谱。然而, 在杏扁遗传连锁图谱构建方面的研究还未见报道。
本研究选用抗寒性差的‘龙王帽’授粉抗寒性强的‘优一’获得的F1代为作图群体, 利用SRAP和SSR标记构建杏扁的遗传连锁图谱, 以期为杏扁的抗寒性QTL定位及分子标记辅助育种奠定基础。
1 材料与方法 1.1 材料以抗寒性强的品种‘优一’为母本, 抗寒性差的品种‘龙王帽’为父本杂交产生的F1代为作图群体。2015年春季去雄杂交, 当年秋季采收获种子, 2016年早春催芽后进行种植。材料种植在张家口市农业科学院的仁用杏资源圃。
1.2 方法 1.2.1 DNA提取和F1杂种的鉴定2017年春, 采摘父母本和2年生F1代植株的嫩叶, 采用本实验室改良的CTAB[9]法提取DNA, 稀释到50 ng/μL后, 贮存于-20℃冰箱备用。为保证作图群体材料的真实性, 选用5对具有父本特异性条带的SRAP引物, 对F1单株进行鉴定。
1.2.2 SRAP与SSR反应体系及扩增程序实验所用的SRAP引物采用Li和Quiros[10]公布的序列, SSR引物采用方闪闪[11]开发的杏扁SSR引物系列。引物由北京鼎国昌盛生物技术有限公司合成。
SRAP-PCR反应体系:Taq酶1.5 U, Mg2+ 2.5 mmol/L, DNA模板30 ng, dNTPs 0.2 mmol/L, 引物0.9 μmol/L, 1×Buffer。PCR反应程序:94℃预变性5 min; 94℃变性1 min, 35℃复性1 min, 72℃延伸1.5 min, 5个循环; 94℃变性1 min, 52℃复性1 min, 72℃延伸1.5 min, 28个循环; 72℃延伸10 min; 4℃保存。扩增产物采用2.0%琼脂糖凝胶电泳, 凝胶成像系统拍照记录。
SSR-PCR反应中各组分浓度为:Mg2+1.5 mmol/L, dNTPs 0.3 mmol/L, Taq酶1.5 U, 引物浓度0.25 μmol/L, DNA模板30 ng, 1×Buffer。PCR反应程序:94℃预变性4 min; 94℃变性45 s, Tm±2℃复性45 s, 72℃延伸90 s, 30个循环; 72℃延伸10 min; 4℃保存。扩增产物采用6%非变性聚丙烯酰胺凝胶(PAGE)电泳, 银染后拍照分析。
1.2.3 分子连锁图谱的构建根据“双假测交”理论, 参照梁小玉等[12]进行SRAP条带的统计, 每个位点用“引物组合+条带大小”命名, 将凝胶图上扩增出的条带记作“1”, 没有条带记作“0”, 模糊不清或者缺失的记作“-”, 然后按照JoinMap 4.0软件CP模式将初始数据转化为数据矩阵。SSR标记按照Join Map 4.0软件中CP模式的要求对原始数据的进行记录并整理, 不清晰或无条带的标记记为“-”。按照孟德尔分离规律, 对F1群体所有条带进行卡方(χ2)检验, 显著水平为0.01, 将偏分离显著的标记剔除。利用Join Map 4.0软件对余下的标记进行遗传连锁分析, 将LOD值设置为3.0-8.0运算, 设置遗传分析的参数最大重组率为r=0.4, 通过Kosambi函数[13]将重组率转换为图距(cM), 分别构建出父本和母本的分子遗传图谱。
2 结果 2.1 多态性引物筛选以父母本的DNA为模板对374对SRAP引物及21对SSR引物进行扩增多态性筛选, SRAP标记选出65对能在父母本之间扩增出位点多样、条带清晰, 并能够重复试验的引物, 引物入选率为17.38%。SSR标记选出17对引物在父母本间表现出丰富的多态性, 引物入选率为81%。
2.2 杂种鉴定自筛出的SRAP引物中选取的5对能扩增出父本特异性条带的引物组合。对122株杂种后代进行鉴定, 以出现父本特异性条带为标准, 从122株子代中鉴定出98株为双亲杂交的真实子代, 在田间对这98株子代个体的表观特征进行复查, 从而更加确定了这98株子代就是‘优一’ב龙王帽’真实的杂交子代。
2.3 SRAP及SSR标记的多态性和偏分离分析SRAP标记扩增结果(图 1):采用65对筛选出多态性良好的引物, 对子代群体进行PCR扩增, 总共检测出标记位点234个, 将这些位点分为3种类型统计:(1)np×nn分离类型, 即父本为杂合基因型np, 母本为纯合基因型nn, 子代孟德尔分离比为1:1, 属于该类型的标记位点为41个, 占多态性标记总数的17.52%。(2)lm×ll分离类型, 即母本为杂合基因型lm, 父本为纯合基因型ll, 子代孟德尔分离比为1:1, 属于该类型的标记位点为96个, 占多态性标记总数的41.03%。(3)hk×hk分离类型, 即双亲均为杂合基因型hk, 子代可能的基因型为h-和kk或hh和k-, 孟德尔分离比为3:1或1:3, 属于该类型的标记位点为97个, 占多态性标记总数的41.45%。平均每对引物组合扩增出3.6条多态性条带。通过卡方(χ2)检验(α=0.01)发现:偏离孟德尔分离比的父本位点有12个, 在所有的父本特异标记中占29.2%;偏离孟德尔分离比的母本位点有23个, 在所有的母本特异标记中占23.9%;偏离孟德尔分离比的父母本共有的位点28个, 在父母本共有的标记中占28.9%。
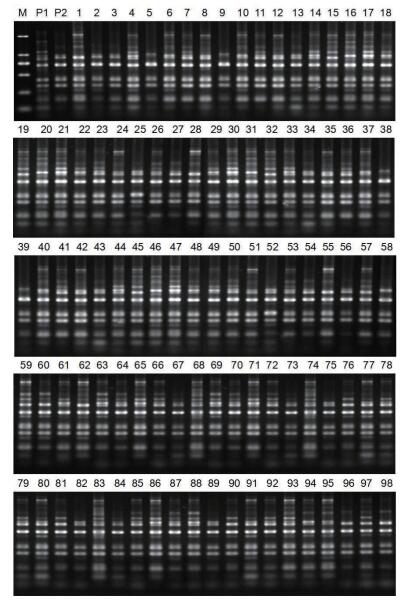
|
| 图 1 SRAP引物M4-E4扩增群体的电泳图 M:Marker; P1:优一; P2:龙王帽; 1-98:F1群体的98个单株 |
SSR标记扩增结果(图 2):利用17对选出的SSR引物对作图群体进行扩增, 由于SSR引物较少, 所以扩增出的结果只有np×nn和lm×ll两种类型。其中属于np×nn分离类型的有7个, 占多态性标记总数的41.18%。属于lm×ll分离类型的有10个, 占多态性标记总数的58.82%。这两种分离类型的孟德尔分离比均为1:1。通过卡方(χ2)检验(α=0.01), 有1个父本杂合位点出现偏离分离现象, 占父本特有标记总数的14.3%;有1母本杂合位点出现偏离分离现象, 占母本杂合位点总数的10%。
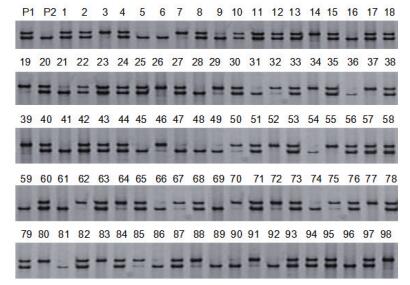
|
| 图 2 SSR引物UDP96-008扩增群体的电泳图 P1:优一; P2:龙王帽; 1-98:F1群体的98个单株 |
本实验共扩增得到234个SRAP标记和17个SSR标记, 利用Join Map 4.0软件中的CP作图模型, 先将48个np×nn分离类型及97个hk×hk分离类型的标记位点进行连锁分析, 构建父本‘龙王帽’的分子连锁图谱, 命名为M1-M8(图 3, 表 1), 父本图谱拥有8个遗传连锁群, 共定位了53个SRAP标记位点以及9个SSR标记位点, 图谱的图距总长度为694.8 cM, 平均图距为11.21 cM, 相邻位点间最小距离为0.6 cM, 平均每个连锁群上有7.75个标记位点, 连锁群平均长度为86.85 cM。

|
| 图 3 父本‘龙王帽’的分子连锁图谱 |
再利用106个lm×ll分离类型及97个hk×hk分离类型的标记位点进行连锁分析, 构建母本‘优一’的分子连锁图谱, 命名为Y1-Y8(图 4, 表 2), 母本图谱同样拥有8个遗传连锁群, 定位了79个SRAP标记位点以及8个SSR标记位点, 图谱的图距总长度为924.8 cM, 平均图距为10.63 cM, 相邻位点间最小距离为0.4 cM, 平均每个连锁群上有10.87个标记位点, 连锁群平均长度为115.6 cM。为了得到标记密度较高的图谱在其中加入部分偏分离标记, 此类位点在遗传图谱中用“*”标出。

|
| 图 4 母本‘优一’的分子连锁图谱 |
亲本的选择和群体的创建是构建遗传连锁图谱的基础。本研究选取的杂交亲本‘龙王帽’和‘优一’虽属种内杂交, 但杏扁属于遗传变异丰富且高度杂合的树种, 导致种内杂交所产生的F1代群体存在较高频率的分离位点。“双假测交”理论为高度杂合的木本植物遗传图谱的创建提供了理论依据, 已被广泛地应用于异花授粉植物F1群体的作图中。与其它作图群体相比, F1群体作图节省时间且操作简单, 同时也避免了自交不亲和的障碍。故本研究基于“双假侧交”策略对F1群体进行作图, 可视为回交群体模型来进行图谱构建[14]。
前人研究表明, 标记的偏分离在作图过程中普遍存在[15, 25], 且在连锁图中的比例一般在6.8%-31.8%之间[16]。本实验对标记偏分离进行分析, 获得的234个多态性标记位点中, 有65个标记属于偏分离标记, 在所有的多态性标记中占27.8%。依据大多数科研学者的共识, 导致遗传位点发生偏分离的主要因素如下:基因组在杂交时发生结构上的重排、缺失、插入和突变[17], 偏分离可能与群体类型有联系[18], 环境因素、非同源重组、基因转化和转座子等因素[19], 叶绿体DNA、线粒体DNA等表现为母性遗传的细胞质DNA[20]。
如何处理偏分离标记, 绝大多数研究者认为可以将偏分离标记加入到图谱构建中[21]。Bradshaw等[22]通过实验证明, 偏分离标记拥有与正常分离的标记同等效果的作图效率, 如果去除偏分离标记可能导致某些连锁图谱中连锁群的基因失去连锁关系。因此, 较认同的作法是将偏分离的标记与正常分离的标记同等分析, 如果能定位到图谱上用“*”标出。本研究中, 同样也有16个偏分离标记被应用到遗传连锁图图谱的构建中, 占作图标记的11%。
一般认为, 遗传图谱中的连锁群上的标记间平均间隔不大于20 cM[23-24]。本研究构建的杏扁双亲2张图谱中, 母本8个连锁群, 标记间平均图距为10.63 cM, 父本也是8个连锁群, 标记间平均图距为11.21 cM, 达到了连锁图谱的基本要求。但本研究中父母本的连锁群上的标记分布不均匀, 这与分析时所采用的标记类型和数量偏少有关。今后我们应该扩大作图群体, 采用更多类型和数量的分子标记加密杏扁的遗传图谱。
4 结论本研究采用筛选出的65对SRAP引物和17对SSR引物对杏扁‘龙王帽’ב优一’的F1代群体进行遗传分析, 在获得了234个SRAP标记和17个SSR标记的基础上成功构建了两亲本“龙王帽”和“优一”的遗传连锁图谱。该结果为后续深入研究杏扁重要农艺性状QTL定位奠定基础。
| [1] |
张加延. 李杏资源研究与利用进展[M]. 北京: 中国林业出版社, 2008.
|
| [2] |
Suranya D. Hormonal control of frost injuries on apricot trees[J]. Acta Horticulturae, 1991, 293(1): 341-344. |
| [3] |
Bassi D, Rizzo M, Foschi S, et al. Breeding apricot in Northern Italy[J]. Acta Horticulturae, 2010, 862: 151-158. |
| [4] |
王飞, 李嘉瑞. 用电导法配合Logistic方程确定杏花期的抗寒性[J]. 西北农业大学学报, 1997, 25(5): 59-63. DOI:10.3321/j.issn:1671-9387.1997.05.012 |
| [5] |
Weeden NF, Hemmat M, Lawson DM, et al. Development and appli-cation of molecular marker linkage maps in woody fruit crops[J]. Euphytic, 1994, 77(1-2): 71-75. DOI:10.1007/BF02551464 |
| [6] |
Hurtado M, Romero C, Vilanova S, et al. Genetic linkage maps of two apricot cultivars(Prunus armeniaca L.)and mapping of PPV(sh-arka)resistance[J]. Theoretical and Applied Genetics, 2002, 105(2-3): 182-191. DOI:10.1007/s00122-002-0936-y |
| [7] |
Vilanova S, Romero C, Abbott AG, et al. An apricot(Prunus armeniaca L.)F2 progeny linkage map based on SSR and AFLP markers, mapping plum pox virus resistance and self-incompatibility traits[J]. Theoretical and Applied Genetics, 2003, 107(2): 239-247. DOI:10.1007/s00122-003-1243-y |
| [8] |
章秋平, 刘威生, 刘宁, 等. 杏杂合位点共显性标记的分离方式及连锁图谱构建[J]. 园艺学报, 2011, 38(10): 1983-1990. |
| [9] |
艾鹏飞, 方闪闪, 甄志军, 等. 仁用杏SSR反应体系的建立与优化[J]. 北方园艺, 2009, 8: 68-70. |
| [10] |
Li G, Quiros CF. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2-3): 455-461. DOI:10.1007/s001220100570 |
| [11] |
方闪闪.仁用杏资源的SSR遗传多样性评价[D].石家庄: 河北科技大学, 2010.
|
| [12] |
梁小玉, 季杨, 白史且, 等. 基于SRAP分子标记构建的菊苣遗传连锁图谱[J]. 草业学报, 2015, 24(5): 153-158. |
| [13] |
Kosambi DD. The geometric method in mathematical statistics[J]. The American Mathematical Monthly, 1944, 51(7): 382-389. DOI:10.1080/00029890.1944.11999118 |
| [14] |
Yu C, Jin MY, Zhang BZ, et al. Genetic linkage map of Anthurium andraeanum based on SRAP molecular markers[J]. Acta Horticulturae Sinica, 2012, 39(6): 1151-1158. |
| [15] |
彭捷, 崔翠菊, 梁广津, 等. 基于SSR标记的海带、裙带菜的遗传构成分析[J]. 生物技术通报, 2018, 34(3): 156-162. |
| [16] |
王晓武, 娄平, 何杭军, 等. 利用芥蓝×青花菜DH群体构建AFLP连锁图谱[J]. 园艺学报, 2005, 32(1): 30-34. DOI:10.3321/j.issn:0513-353X.2005.01.007 |
| [17] |
Nishio S, Terakami S, Matsumoto T, et al. Indentification of QTLs for agronomic traits in the Japanese chestnut(Castanea crenata Sieb.et Zucc.)breeding[J]. The Horticulture Journal, 2018, 87(1): 43-54. DOI:10.2503/hortj.OKD-093 |
| [18] |
Xu SJ, Singh RJ, Hymowitz T. Establishment of a cytogenetic map of soybean:progress and prospective[J]. Soybean Genetics Newsletter, 1997, 24: 121-122. |
| [19] |
Knox MR, Ellis TH. Excess heterozygosity contributes to genetic map expansion in pea recombinant inbred populations[J]. Genetics, 2002, 162(2): 861-873. |
| [20] |
Tulsieram LK, Glaubitz JC, Kiss G, et al. Single tree genetic linkage mapping in conifers using haploid DNA from megagametophytes[J]. Nature Biotechnology, 1992, 10(6): 686-690. DOI:10.1038/nbt0692-686 |
| [21] |
郭印山, 赵玉辉, 刘朝吉, 等. 利用多种分子标记构建龙眼高密度分子遗传图谱[J]. 园艺学报, 2009, 36(5): 655-622. DOI:10.3321/j.issn:0513-353X.2009.05.005 |
| [22] |
Bradshaw Jr BH, Villar M, Watson BD, et al. Molecular genetics of growth and development in Populus.Ⅲ.A genetic linkage map of a hybrid poplar composed of RFLP, STS and RAPD markers[J]. Teoretical and Applied Genetics, 1994, 89(2-3): 167-178. |
| [23] |
于翠, 金茂勇, 张宝珠, 等. 基于SRAP分子标记的安祖花遗传连锁图谱的构建[J]. 园艺学报, 2012, 39(6): 1151-1158. |
| [24] |
唐露, 金梦雅, 黄琳凯, 等. 基于SSR标记的四倍体鸭茅遗传图谱加密[J]. 中国农业科学, 2018, 51(5): 991-998. |
| [25] |
赵能, 原晓龙, 缪福俊, 等. 思茅松转录组SSR分析及标记开发[J]. 生物技术通报, 2017, 33(5): 71-77. |






