2. 北京林业大学材料科学与技术学院,北京 100083
2. College of Materials Science and Technology, Beijing Forestry University, Beijing 100083
PHA(Polyhydroxyalkanoates,聚羟基脂肪酸酯)是一种高值生物聚合物,可作为生物塑料或粘合剂[1],可被分解转化成药物或手性化合物的中间体[2],也可经甲酯化后作为生物柴油[3]。中长链PHA的生产一般采用葡萄糖、有机酸或烷烃等作为碳源和能源,采用脉动给料法和分批给料法等生产工艺发酵生产PHA。该法虽PHA产量高,但是碳源底物增加了生产成本,限制了PHA的大规模生产和应用。利用来源广泛、价格低廉的木质素生物合成PHA是未来的发展趋势,更是当前的研究热点之一。
木质素是木质纤维素的主要成分之一,约占自然界中木质纤维素干重的15%-30%,是仅次于纤维素的可再生生物质资源,也是含量最为丰富的芳香族类聚合物。木质素由3种苯丙烷单体通过C-O、C-C等化学性质稳定的化学键连接构成[4],很难被分解利用,经常作为废弃物燃烧产热,造成了极大的资源浪费和环境污染[5-8]。目前有很多关于木质素高温降解和化学催化降解方面的报道[9-10],但是所获得的产物是混合物,使木质素的高值化利用成为了一个挑战。木质素的生物转化涉及多个步骤,主要包括木质素解聚,芳香族化合物利用和代谢产物形成等过程。每一步都需要多种酶的参与。细菌具有广泛的环境适应性、强大的产物积累能力、相对较短的生长周期、以及较成熟的遗传操作系统等,在木质素生物转化方面具有得天独厚的优势。但是,细菌的木质素降解能力远远低于白腐真菌[11-12],而且系统研究细菌木质素生物转化过程的研究相对较少[13-14]。因此,对木质素降解细菌进行代谢工程改造,使菌株合成有价值的化学品或其他高值化合物,将为木质素生物转化途径的系统性研究和全局调控提供研技术基础和研究思路[15]。
在限制氮源的条件下,P. putida KT2440可利用木质素合成PHA[8, 14]。在细菌中,hsdR基因一般编码限制-修饰系统中Ⅰ型限制酶EcoK或EcoB的一个亚基。宿主的限制-修饰现象能辨别自身的DNA与外源DNA,并能使后者降解掉。为了便于对P. putida KT2440进行遗传操作,前期我们通过同源重组构建了hsdR基因缺失突变株P. putida QSR1。本文以QSR1为出发菌株,采用代谢工程的方法,强化该菌株利用木质素积累PHA的能力,旨在探索提高木质素转化效率的方法,为PHA的高效、低成本生产提供研究思路。
1 材料与方法 1.1 材料 1.1.1 相关菌株、质粒与试剂本实验所用到的质粒和菌株见表 1。用于构建载体的引物由Invitrogen公司合成,DNA Ladder、限制性内切酶、高保真DNA聚合酶Mix、T4 DNA连接酶、PCR纯化和胶回收试剂盒均购自TaKaRa公司,质粒提取试剂盒购自Biomega公司,细菌基因组提取试剂盒购于北京天根生化科技有限公司。玉米芯木质素由山东龙力生物科技有限公司提供。
LB培养基:10 g/L胰蛋白胨,5 g/L酵母提取物,10 g/L NaCl;S.O.C培养基:20 g/L胰蛋白胨,5 g/L酵母提取物,10 mmol/L NaCl,2.5 mmol/L KCl,10 mmol/L MgCl2,10 mmol/L MgSO4;10×M9溶液:6.78 g/L Na2HPO4(15.138 g/L Na2HPO4·12H2O)、3 g/L KH2PO4、0.5 g/L NaCl;微量元素液:CuSO4、MnSO4、FeSO4、ZnSO4各100 μmol/L。
1.2 方法 1.2.1 pK18PHAC1C2质粒的构建首先,利用引物对PHAC1-F/R和PHAC2-F/R分别从P. putida KT2440基因组中扩增phaZ基因的上游phaC1(1 772 bp)和下游phaC2(1 824 bp)DNA片段。接着,先将纯化后的phaC1片段经酶切、连接插入pUC18载体的Hind Ⅲ/Xba Ⅰ克隆位点,获得载体pUCPHAC1;再将phaC2片段插入pUCPHAC1的Kpn Ⅰ/ Xba Ⅰ位点,获得载体pUCPHAC1C2。然后,以pUCPHAC1C2为模板,利用引物对PHAC1-FH/PHAC2-RB扩增phaC1C2 DNA片段,并将该片段插入pK18mobsacB载体的Hind Ⅲ/BamH Ⅰ位点,构建pK18PHAC1C2载体。
1.2.2 pVLTPHAZ质粒的构建以PHAZ-FK/RH为引物,利用高保真DNA聚合酶从KT2440基因组中PCR扩增获得phaZ基因的编码区序列;该基因经纯化、酶切和连接插入pVLT33质粒的Kpn Ⅰ/Hind Ⅲ位点,获得重组载体pVLTPHAZ。
1.2.3 pVLTDYPB质粒的构建以dypB-FK/RH为引物,利用高保真DNA聚合酶从R. jostii RHA1基因组[16]中扩增获得dypB基因。之后,通过酶切和连接将纯化后dypB基因片段分别插入pVLT33的Kpn Ⅰ/Hind Ⅲ位点,获得重组载体pVLTpelBDYPB。
1.2.4 突变株的构建和筛选采用电转化法将重组载体转入E. coli S17-1及P. putida QSRZ6;采用双亲接合法[17]将重组载体pK18PHAC1C2导入P. putida QSR1。利用20%蔗糖筛选同源重组双交换突变株;筛选过程中卡那霉素的使用浓度为50 μg/mL,氨苄青霉素的使用浓度为50 μg/mL;利用相应引物的菌落PCR法验证各步骤的突变株。
1.2.5 过氧化物酶酶活的测定通过ABTS的氧化速率(Ƹ420=36 mmol·L -1·cm-1)来测定DypB的活性。1 mL的反应体系中含有50 mmol/L醋酸钠缓冲液(pH 4.5),0.5 mmol/L H2O2,2.5 mmol/L ABTS以及400 μL粗酶液(上清液)。使用酶标仪测定波长420 nm条件下1分钟内体系中吸光度的增加量。以每分钟氧化1 μmol ABTS所需的酶量定义为1个酶活单位U。
1.2.6 PHA提取和定量以含有2 mL 1 mol/L MgSO4、1 mL 1 mol/L MnSO4、100 μL 1 mol/L CaCl2和1 mL微量元素溶液的1×M9为基础培养基(pH7.0),分别添加10 g/L葡萄糖和10 g/L玉米芯木质素作为碳源和能源积累PHA。挑取平板上活化的单菌落接种到LB液体培养基中,过夜培养;接着将菌液加入到四倍体积的新鲜的LB培养基中,再培养2 h以达到对数生长期;然后按照5%的接种量将其接种到100 mL产PHA培养基中;30℃、150 r/min培养48 h。在培养过程中的0 h,6 h,12 h,24 h,48 h取样测量菌体的生长情况和PHA的积累情况。PHA的提取与定量参照Lu等[15]的方法。
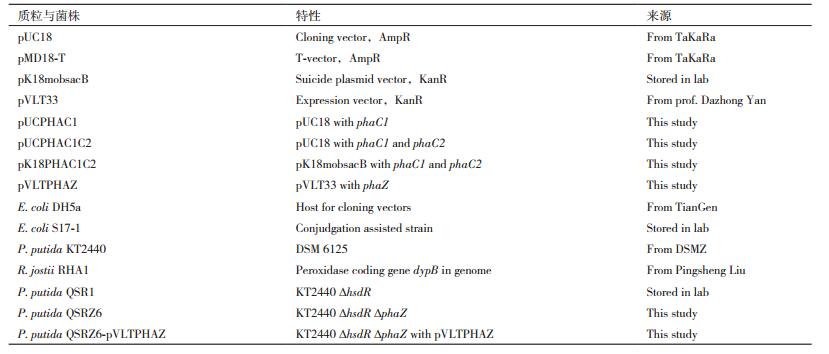
2 结果 2.1 基因突变株的构建 2.1.1 ∆phaZ突变株的构建使用酶切连接的方法将phaZ基因上游phaC1片段和下游phaC2片段进行连接,并插入到pK18mobsacB中,构建phaZ敲除质粒pK18PHAC1C2。然后,通过双亲接合法将pK18PHAC1C2导入hsdR基因缺失突变株P. putida QSR1,再通过抗生素和蔗糖筛选获得phaZ基因失活突变株P. putida QSRZ6(简称QSRZ6),并且利用引物对PHA-F/R(表 2)进行PCR验证。验证引物PHA-F/R分别位于敲除片段上下游同源臂上,若phaZ被成功敲除,验证引物所扩增的条带应为626 bp,若未敲除成功,扩增产物大小为1 426 bp(图 1-A)。
|
| 图 1 各基因突变株的验证 A:phaZ缺失突变株P. putida QSRZ6的验证;B:P. putida QSRZ6-pVLTPHAZ的验证;C:P. putida QSRZ6-pVLTpelBDYPB的验证,M:DL2000 marker |
为了验证phaZ缺失突变株的功能,我们构建了phaZ的功能互补菌株。pVLT33为广宿主表达载体,含有一个lacqI-tac启动子。我们将phaZ基因(852 bp)插入该载体,利用引物对pVLT33-F/R进行PCR扩增验证该质粒正确后(图 1-B,验证中引入了pVLT33上约200 bp序列),将其导入突变株QSRZ6获得P. putida QSRZ6 - pVLTPHAZ(后简称PVLTPHAZ)。
2.1.2 DypB强化表达菌株的构建来源于可降解木质素R. jostii RHA1的DypB蛋白是木质素降解酶,属于过氧化物酶类[18]。DypB属于胞外酶,需要一段前导肽序列以协助其穿过细胞膜。通过SignalP4.1软件分析,发现DypB序列前端不存在前导肽序列。在扩增DypB编码基因时,通过引物在DypB的N端引入了编码软腐欧文氏菌PelB的前导肽序列(序列详见表 2中dypB-F中双下滑线)。我们将pelBdypB基因片段分别插入载体pVLT33中,经过PCR验证(图 1-C),得到重组质粒pVLTpelBDYPB,又利用电转化法将这重组质粒转入phaZ缺失突变株QSRZ6中,得到P. putida QSRZ6B(简称QSRZ6B)。
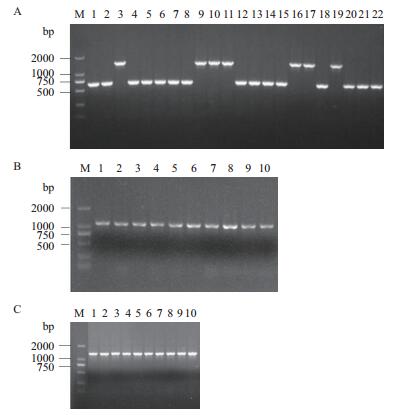
2.2 phaZ基因缺失突变株的PHA积累能力聚羟基脂肪酸酯(PHA)是原核微生物在碳氮比失衡的情况下,作为碳源储藏物的一种聚酯类物质,具有重要的应用价值。PhaZ具有mcl-PHA解聚功能,本研究构建了phaZ基因缺失突变株(QSRZ6)和phaZ的功能互补菌株(PVLTPHAZ)。以P. putida QSR1为对照,研究了phaZ基因和对菌体生长和细胞积累PHA的能力的影响。
以葡萄糖为碳源的培养过程中,QSR1在24 h时进入稳定期;功能互补菌株经历了较长时间的延迟期,较其他两株菌生长缓慢;与QSR1相比,QSRZ6对数生长期延长,48 h时还在继续对数生长,且生长量明显增加,OD600达到0.55,提高了29%(图 2-A)。提取培养48 h的胞内PHA,发现出发菌株QSR1的PHA产量为131 mg/L,由于过量表达PhaZ分解利用了部分PHA,功能互补菌株PVLTPHAZ产量仅为63 mg/L,而phaZ基因敲除突变株QSRZ6的PHA产量为236 mg/L,是出发菌株的1.8倍(图 2-B)。
|
| 图 2 phaZ敲除突变株的生长和PHA的积累 A:以葡萄糖为碳源的生长曲线;B:以葡萄糖为碳源的菌体干重和PHA产率;C:以木质素为碳源的CFU;D:以木质素为碳源的菌体干重和PHA产率 |
在使用木质素为碳源时,由于木质素溶于培养基后使培养基颜色较深,若使用OD600不能很好表征生长量,所以我们选用菌落形成单位来表征菌株生长情况。从图 2-C中可看出,功能互补菌株的菌落形成单位(CFU/mL)明显低于菌株QSR1和敲除突变株QSRZ6,而QSRZ6和QSR1的生长情况基本一致。QSR1、QSRZ6和PVLTPHAZ的PHA产量分别为44 mg/L、124 mg/L和31 mg/L。QSRZ6的PHA产量最高,是出发株的2.8倍(图 2-D),说明敲除phaZ基因对QSR1利用木质纤维素积累PHA有显著的提升。
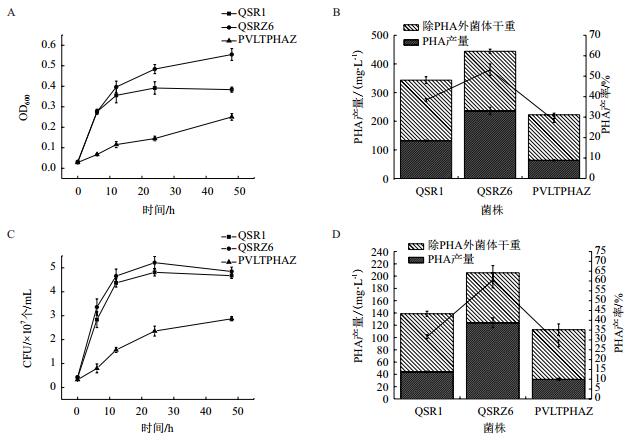
2.3 P. putida QSRZ6B的PHA积累能力在phaZ基因失活的基础上,我们强化表达了R. jostii RHA1的DypB。利用木质素为唯一碳源和能源发酵培养过氧化物酶强化表达菌株时,与转入空载体pVLT33的菌株P. putida QSRZ6-pVLT33相比,在48 h时QSRZ6B的菌落形成单位均提高了133%,菌体干重也提高146%。取24 h和36 h的样品测定过氧化物酶活性,对照菌株自身存在过氧化物酶活性,但酶活在不同时段均处于较低水平;QSRZ6B的酶活分别为0.11 U/mL和0.23 U/mL。发酵48 h,QSRZ6B的PHA积累量为140 mg/L,较QSRZ6和QSR1分别提高了13%和218%(图 3)。上述结果表明,强化表达R. jostii RHA1的DypB提高了菌株降解和利用木质素的水平,使QSRZ6B的生长能力和PHA的积累能力都得到增强。
|
| 图 3 菌株QSRZ6B的特征 A:生长曲线;B:过氧化物酶活性;C:菌体干重和PHA积累量 |
利用代谢工程法改造木质素降解细菌是提高木质素生物转化率的一种有效方法,该方法可以通过表达有效的功能基因和敲除某些基因来定向合成和积累高值化合物。Vardon等[19]在敲除多余代谢分支的同时,又引入了aroY、catA和苯酚代谢通路操纵子,使P. putida KT2440利用木质素类似物积累己二酸,产量达到了13.5 g/L;Lu等[15]在蛋白组学和转录组学等方法的指导下,导入来源于放线菌的过氧化物酶的编码基因dyp2、香草醛脱氢酶基因vanAB以及自身的PHA合成酶基因phaJ4和phaC1,使菌株P. putida A514利用预处理后的木质素纤维素合成PHA的量提高至161 mg/L;以香草酸为碳源时PHA含率达到了73%。
本研究以P. putida KT2440的hsdR缺失突变株为出发菌株,利用同源重组敲除了PHA代谢基因簇中phaZ基因,构建了phaZ缺失菌株P. putida QSRZ6。在P. putida KT2440基因组中,PHA合成相关基因以基因簇(phaC1ZC2- DFI)的形式存在,phaZ基因位于phaC1与phaC2基因之间[20],其编码蛋白PhaZ为胞内mcl-PHA解聚酶[21],在碳源缺乏的条件下,代谢mcl-PHAs[22]。因此,敲除phaZ基因可能有利于PHA的积累。而phaZ基因的上下游基因phaC1和phaC2是PHA合成基因,在利用这两个基因片段为敲除phaZ基因的同源臂的同时,需要避免引入碱基突变。为此,我们在扩增phaC1和phaC2基因时采用了高保真DNA聚合酶,并通过测序验证了QSRZ6中phaC1和phaC2的完整性。在分别以葡萄糖和木质素为碳源培养QSRZ6时,该菌株的生物量明显不同。这主要由于葡萄糖为简单糖类,易被微生物代谢利用,而木质素本身成分复杂,含有较多芳香族类化合物,需要诱导产生代谢酶类,从而降低了木质素的利用。但不论利用葡萄糖还是木质素,QSRZ6均获得了最大的生物量,菌体干重较初始菌株分别提升了29%和48%。在以木质素为碳源时,QSRZ6中PHA的积累量为124 mg/L,提高了1.8倍。Linger[13]等利用APL(木质素含量约32%的玉米芯碱-蒽醌处理液,同时富含单糖和木质素单体)发酵合成PHA的量为252 mg/L。相比之下,我们获得的菌株QSRZ6B的PHA积累量要低44%。原因在于本文使用的木质素为高纯度碱木质素(木质素含量90%以上),仅含有少于5%的不同类型碳水化合物,比APL更为难利用,导致PHA的积累量也相应较低。
进而我们又在QSRZ6菌株中引入来源于R. jostii RHA1的过氧化物酶(DypB)编码基因,构建了菌株QSRZ6B以强化木质素降解能力。在以木质素为碳源的培养基中,该菌的生长量和过氧化物酶活性均得到了不同程度的提高,而且QSRZ6B的PHA的积累量达到140 mg/L,比QSRZ6和QRS1分别提高了13%和218%。Ahmad等[18]开展了相似的研究工作,构建了R. jostii RHA1的dypB的敲除突变株,dypB缺失菌株在降解木质素方面均具有不同程度的下降,而且在大肠杆菌中重组表达的DypB也显示出了对Kraft木质素和麦草原木质素的降解活性,表明DypB在木质素的降解过程中具有一定的作用,与本实验结果一致。
在hsdR基因缺失突变株的基础上,本实验又敲除了phaZ基因,获得了hsdR-phaZ双基因缺失突变菌株P. putida QSRZ6,该突变株可以作为全局调控木质素降解酶系统和PHA合成系统以提高利用木质素积累PHA的优势出发菌株。进而我们利用该菌株强化表达的异源过氧化物酶,所获得的代谢工程菌株QSRZ6B的生长情况和PHA的合成能力都得到了提高,说明采用代谢工程的方法对木质素降解系统和PHA积累途径进行基因修改是一种提高木质素转化和PHA积累量的有效方法。在未来的研究中,我们将对利用木质素积累PHA的整体途径进行深入研究和理性设计,进而将木质素降解、芳香族化合物利用和高值产物的形成与积累结合起来,建立高效的利用木质素积累PHA的细胞工厂,以实现木质素的高效生物转化和PHA的低成本合成。
4 结论本实验以P. putida KT2440的hsdR缺失突变株P. putida QSR1为出发菌株,构建了phaZ基因缺失突变株P. putida QSRZ6,进而在QSRZ6中强化表达了R. jostii RHA1的过氧化物酶DypB,获得了代谢工程菌株QSRZ6B。菌株QSRZ6的生物量和PHA积累量分别增长了48%和182%。QSRZ6B的PHA积累量达到了140 mg/L,相比QSRZ6和QSR1分别提高了13%和218%。
| [1] |
Chen GQ. A microbial polyhydroxyalkanoates (PHA) based bio- and materials industry[J]. Chemical Society Reviews, 2009, 38(8): 2434-2446. DOI:10.1039/b812677c |
| [2] |
Gangoiti J, Santos M, Llama MJ, et al. Production of Chiral (R)-3-Hydroxyoctanoic Acid Monomers, Catalyzed by Pseudomonas fluorescens GK13 Poly (3-Hydroxyoctanoic Acid) Depolymerase[J]. Applied & Environmental Microbiology, 2010, 76(11): 3554-3560. |
| [3] |
Zhang X, Luo R, Wang Z, et al. Application of (R)-3-hydroxyalkanoate methyl esters derived from microbial polyhydroxyalkanoates as novel biofuels[J]. Biomacromolecules, 2009, 10(4): 707-711. DOI:10.1021/bm801424e |
| [4] |
Boerjan W, Ralph J, Baucher M. Lignin biosynthesis[J]. Annual Review of Plant Biology, 2003, 54(1): 519-546. DOI:10.1146/annurev.arplant.54.031902.134938 |
| [5] |
Himmel ME, Ding SY, Johnson DK, et al. Biomass recalcitrance:engineering plants and enzymes for biofuels production[J]. Science, 2007, 315(5813): 804-807. DOI:10.1126/science.1137016 |
| [6] |
Chundawat SP, Beckham GT, Himmel ME, et al. Deconstruction of lignocellulosic biomass to fuels and chemicals[J]. Annual Review of Chemical & Biomolecular Engineering, 2011, 2(2): 121-145. |
| [7] |
Tuck CO, Perez E, Horvath IT, et al. Valorization of biomass:deriving more value from waste[J]. Science, 2012, 337(6095): 695-699. DOI:10.1126/science.1218930 |
| [8] |
Ragauskas AJ, Beckham GT, Biddy MJ, et al. Lignin valorization:improving lignin processing in the biorefinery[J]. Science, 2014, 344(6185): 1246843. DOI:10.1126/science.1246843 |
| [9] |
Kirk TK, Farrell RL. Enzymatic "combustion":the microbial degradation of lignin[J]. Annual review of microbiology, 1987, 41(1): 465-505. DOI:10.1146/annurev.mi.41.100187.002341 |
| [10] |
Zakzeski J, Bruijnincx PCA, Jongerius AL, et al. The catalytic valorization of lignin for the production of renewable chemicals[J]. Chemical Reviews, 2010, 110(6): 3552-3559. DOI:10.1021/cr900354u |
| [11] |
Floudas D, Binder M, Riley R, et al. The paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes[J]. Science, 2012, 336(6089): 1715-1719. DOI:10.1126/science.1221748 |
| [12] |
Dixon RA. Microbiology:Break down the walls[J]. Nature, 2013, 493(7430): 36-37. DOI:10.1038/493036a |
| [13] |
Linger JG, Vardon DR, Guarnieri MT, et al. Lignin valorization through integrated biological funneling and chemical catalysis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(33): 12013-12018. DOI:10.1073/pnas.1410657111 |
| [14] |
Salvachua D, Karp EM, Nimlos CT, et al. Towards lignin consolidated bioprocessing:simultaneous lignin depolymerization and product generation by bacteria[J]. Green Chemistry, 2015, 17(11): 4951-4967. DOI:10.1039/C5GC01165E |
| [15] |
Lin L, Cheng Y, Pu Y, et al. Systems biology-guided biodesign of consolidated lignin conversion[J]. Green Chem, 2016(18): 5536-5547. |
| [16] |
Mcleod MP, Warren RL, Hsiao WWL, et al. The complete genome of Rhodococcus sp. RHA1 provides insights into a catabolic powerhouse[J]. Proceedings of the National Academy of Sciences, 2006, 103(42): 15582-15587. DOI:10.1073/pnas.0607048103 |
| [17] |
Wang JP, Wu LX, Xu F, et al. Metabolic engineering for ethylene production by inserting the ethylene-forming enzyme gene (efe) at the 16S rDNA sites of Pseudomonas putida KT2440[J]. Bioresource Technology, 2010, 101(16): 6404-6409. DOI:10.1016/j.biortech.2010.03.030 |
| [18] |
Ahmad M, Roberts JN, Hardiman EM, et al. Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase[J]. Biochemistry, 2011, 50(23): 5096-5107. DOI:10.1021/bi101892z |
| [19] |
Vardon DR, Franden MA, Johnson CW, et al. Adipic acid production from lignin[J]. Energy & Environmental Science, 2015, 8(2): 617-628. |
| [20] |
Nelson KE, Weinel C, Paulsen IT, et al. Complete genome sequence and comparative analysis of the metabolically versatile Pseudomonas putida KT2440[J]. Environmental Microbiology, 2002, 4(12): 799-808. DOI:10.1046/j.1462-2920.2002.00366.x |
| [21] |
De-Eugenio LI, Garcia P, Luengo JM, et al. Biochemical evidence that phaZ gene encodes a specific intracellular medium chain length polyhydroxyalkanoate depolymerase in Pseudomonas putida KT2442:characterization of a paradigmatic enzyme[J]. J Biol Chem, 2007, 282(7): 4951-4962. DOI:10.1074/jbc.M608119200 |
| [22] |
Laura Isabel DE, Escapa IF, Valle M, et al. The turnover of medium-chain-length polyhydroxyalkanoates in Pseudomonas putida KT2442 and the fundamental role of PhaZ depolymerase for the metabolic balance[J]. Environmental Microbiology, 2010, 12(1): 207-221. DOI:10.1111/emi.2010.12.issue-1 |