2. 中国农业科学院蔬菜花卉研究所,北京 100081
2. The Institute of Vegetables and Flowers in Chinese Academy of Agricultural Sciences, Beijing 100081
百合(Lilium spp.)为我国栽培历史悠久的药、食、赏多用途植物,世界五大鲜切花之一。随着现代分子生物学的不断发展,百合花色[1]、花香[2]、发育[3]及抗性[4]等相关基因的研究都取得了较大进展,为今后百合分子育种奠定了良好基础[5]。
前人对百合基因功能研究中,基因表达分析占据了重要地位[6]。实时荧光定量PCR(Real-time quantitative PCR,qRT-PCR)是一种快速检测基因表达量的方法,因其具有高通量、高灵敏和实时检测等特点,目前被广泛应用于植物基因表达水平研究中[7-8]。尽管qRT-PCR操作简单,但qRT-PCR的准确性却受多方面因素影响,如引物特异性、反应体系和RNA质量等[9-12]。RNA样本中基因组DNA残留对后期基因表达分析的准确性具有很大影响,残留的基因组DNA一方面会影响RNA浓度的精确定量;另一方面,残留的DNA也会影响qRT-PCR扩增效率[13]。目前,常采用设计跨内含子引物的方法来避免基因组DNA残留对供试基因表达结果的影响[14-16]。但是很多物种的基因组测序尚未开展,很难将所有待研究基因的引物都设计为跨内含子引物[17]。因此,为了保证qRT-PCR结果的准确性,获得高质量且无基因组DNA残留的总RNA样本是非常必要的。
目前,市面上RNA提取试剂盒和第一链cDNA合成试剂盒种类繁多。虽然大部分RNA提取试剂盒中都包含有DNA去除步骤,但DNA是否去除干净无法得知。此外,为了避免总RNA样本中有残留的基因组DNA污染,部分第一链cDNA合成试剂盒中也加入了基因组DNA去除步骤,但cDNA质量无法预知。因此,为了检测总RNA和cDNA样本中基因组DNA残留是否去除干净,本研究从前期得到的转录组数据(尚未发表)中筛选了1个高度保守的持家基因TIP41-like,拟针对其内含子序列设计引物,以期可简便快捷的检测百合总RNA以及cDNA样本中有无基因组DNA残留,为后续相关基因表达分析的准确性奠定基础。
1 材料和方法 1.1 材料 1.1.1 供试材料供试材料为岷江百合(Lilium regale)组织培养植株,保存于中国农业科学院蔬菜花卉研究所组培室。随机选取6株生长发育一致的组培苗,分别取其叶片,经液氮速冻后,于-80℃冰箱保存备用。
1.1.2 试剂选择3种不同公司生产的RNA提取试剂盒(分别标记为A、B、C,均含有基因组DNA去除步骤)用于提取岷江百合叶片RNA。采用2种不同公司品牌的第一链cDNA合成试剂盒(分别标记为Ⅰ、Ⅱ;Ⅰ不含有基因组DNA去除步骤,Ⅱ含有基因组DNA去除步骤)用于合成cDNA。
1.2 方法 1.2.1 含内含子的DNA序列扩增从岷江百合转录组数据(尚未公布)中获得了持家基因TIP41-like的一段mRNA序列,利用DNAMAN 5.0软件将其序列与NCBI数据库中所有物种的TIP41-like基因组序列进行比对,推测该岷江百合TIP41-like部分序列对应的基因组DNA序列中含有2个内含子,并使用Primer Premier 5.0软件根据此段序列设计DNA扩增产物中含2个内含子的引物:F:5'-CCGAAAATCAGGGTAGGGTG-3'及R:5'-CGAAGCCAGAAACGGAGAAGA-3'。引物由上海生工生物公司合成。
以岷江百合叶片DNA为模板进行PCR扩增,PCR反应体系20 μL:模版DNA 2 μL,PCR Forward Primer 0.5 μL,PCR Reverse Primer 0.5 μL,2×Taq PCR Magic Mix 10 μL,ddH2O 7 μL。
PCR反应程序为:94℃预变性3 min;94℃变性30秒,58℃退火30 s,72℃延伸1 min,循环35次;72℃延伸5 min。反应结束后,取6 μL产物用2%琼脂糖凝胶电泳并观察拍照。
1.2.2 基因组DNA残留检测引物设计将含内含子序列的DNA扩增产物送至上海生工生物公司测序,利用DNAMAN 5.0软件对所得到的序列进行核酸拼接;将拼接好的序列与TIP41-like转录组mRNA序列进行比对,寻找出内含子序列。根据所获得的TIP41-like内含子序列,利用Primer Premier 5.0软件设计基因组DNA残留检测引物,引物设计时应保证引物其中任意一端序列全部落在内含子序列上,引物设计完成后由上海生工生物公司合成。以岷江百合叶片DNA为模板进行PCR扩增,反应体系参照1.2.1。反应结束后,取6 μL产物用2%琼脂糖凝胶电泳并观察拍照。
1.2.3 RNA样本中基因组DNA残留检测通过1.2%琼脂糖凝胶电泳对三种不同RNA提取试剂盒提取的岷江百合叶片的总RNA样本进行检测,利用Quawell Q3000超微量紫外分光光度计检测总RNA的质量和浓度。
以3种不同RNA试剂盒提取的岷江百合叶片总RNA为模板,使用基因组DNA残留检测引物进行PCR扩增,PCR反应体系20 μL:模版RNA 2 μL,PCR Forward Primer 0.5 μL,PCR Reverse Primer 0.5 μL,2×Taq PCR Magic Mix 10 μL,ddH2O 7 μL。
PCR反应程序为:94℃预变性3 min;94℃变性30 s,58℃退火30 s,72℃延伸1 min,循环35次;72℃延伸5 min。反应结束后,取6 μL产物用2%琼脂糖凝胶电泳,并观察拍照。
1.2.4 cDNA中基因组DNA残留检测以两种不同公司品牌的第一链cDNA合成试剂盒对3种RNA提取试剂盒提取的18个RNA样本分别进行第一链cDNA合成。以获得的cDNA为模板,分别使用基因组DNA残留检测引物进行PCR扩增,反应体系参照1.3.3。反应结束后,取6 μL产物用2%琼脂糖凝胶电泳并观察拍照。
2 结果 2.1 含内含子的DNA序列扩增及序列比对以DNA为模板,使用扩增产物中含内含子的引物(F/R)进行PCR扩增,获得了条带单一的目的片段(图 1)。将此DNA扩增产物送上海生工生物公司测序,得到了片段大小为367 bp的碱基序列。利用DNAMAN 5.0软件将测序获得的DNA产物片段与转录组mRNA序列进行比对发现,此DNA扩增产物共有2段内含子序列(图 2)。
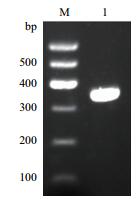
|
| 图 1 岷江百合TIP41-like含内含子DNA片段扩增结果 M:Marker Ⅰ;1:TIP41-like含内含子DNA片段 |

|
| 图 2 TIP41-like含内含子DNA片段和对应的mRNA片段序列比对结果 红色方框:基因组DNA检测引物设计位置 |
根据2.1比对结果,设计了一对基因组DNA残留检测引物:LDRG-F:5'-CTCTTTTATGACGAGGT-TGGTA-3'及LDRG-R:5'-CAGGGAAATCGGTCAGT-GTT-3'。其中LDRG-R引物全部落在内含子序列上(图 2)。
岷江百合叶片DNA为模板,对设计的基因组DNA残留检测引物(LDRG-F/LDRG-R)进行PCR扩增验证,获得了条带单一与预期产物长度(182 bp)结果相一致的扩增片段(图 3)。
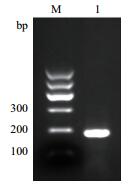
|
| 图 3 岷江百合基因组DNA残留检测片段扩增结果 M:Marker Ⅰ;1:基因组DNA残留检测片段 |
三种RNA提取试剂盒提取的总RNA经1.2%琼脂糖凝胶电泳检测,结果(图 4)显示,28S和18S rRNA条带清晰,无杂带和弥散现象。经Quawell Q3000检测,OD260/280在1.8-2.1范围内,表明提取的RNA完整性好,无蛋白质污染。
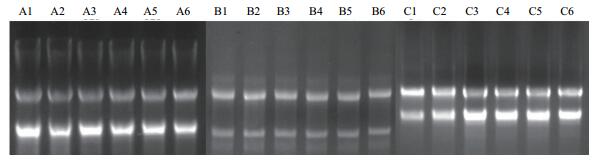
|
| 图 4 三种不同RNA提取试剂盒提取的岷江百合叶片总RNA电泳检测结果 A、B、C分别代表 3种不同的RNA提取试剂盒;1-6分别代表不同单株的叶片样本 |
使用基因组DNA残留检测引物(LDRG-F/LDRG-R)对提取的总RNA进行检测,结果(图 5)显示,使用试剂盒A和B提取的12个总RNA样本中均能扩增出条带,且条带清晰,表明总RNA样本中含有较高浓度的DNA残留。使用试剂盒C提取的总RNA样本中未能扩增出条带,表明此总RNA样本中无基因组DNA残留。
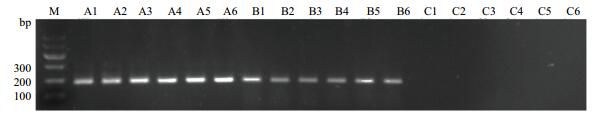
|
| 图 5 总RNA样本中基因组DNA残留检测片段扩增结果 M:Marker Ⅰ;A、B、C分别代表 3种不同的RNA提取试剂盒;1-6分别代表不同单株的叶片样本 |
利用第一链cDNA合成试剂盒Ⅰ、Ⅱ对三种RNA提取试剂盒提取的总RNA样本分别进行反转录,使用基因组DNA残留检测引物(LDRG-F/LDRG-R)对获得的cDNA样本进行检测,结果(图 6)表明:两种cDNA合成试剂盒合成的cDNA样本中A1-A6和B1-B6均能扩增出目的片段,C1-C6无法扩增出目的条带,表明两种cDNA合成试剂盒均无法去除RNA样本中残留的基因组DNA。
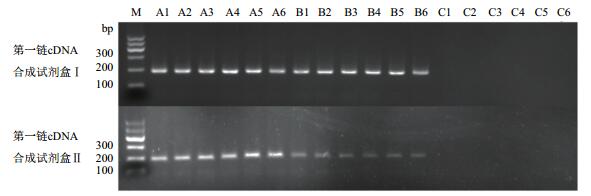
|
| 图 6 cDNA样本中基因组DNA残留检测片段扩增结果 M:Marker Ⅰ;Ⅰ、Ⅱ分别代表两种不同的cDNA合成试剂盒;A、B、C分别代表 3种不同的RNA提取试剂盒;1-6分别代表不同单株的叶片样本 |
RNA质量是影响qRT-PCR准确性的关键因素之一[18-21],本课题组在前期qRT-PCR实验中发现,待测基因的相对表达量不稳定,推测总RNA样本可能存在基因组DNA残留,因此,建立一种快速高效地检测总RNA样本中基因组DNA残留的方法具有重要的实践意义。本实验在前期得到的转录组数据中挖掘了一个持家基因TIP41-like,根据其部分内含子序列设计了一对基因组DNA残留检测引物,用于检测总RNA和cDNA样本中的基因组DNA残留。
基因组DNA残留是抑制后续实验中反转录和实时荧光定量PCR反应的重要因素[22-23]。目前RNA提取过程中去除基因组DNA最常用的方法就是加入DNase Ⅰ消化[24]。本研究采用了3种不同公司生产的RNA提取试剂盒对岷江百合组培苗叶片进行了总RNA提取,通过RNA凝胶电泳和纯度检测无法得知总RNA样本中基因组DNA是否去除干净(图 4)。但使用基因组DNA残留检测引物(LDRG-F/LDRG-R)对总RNA样本进行扩增后发现,尽管3种试剂盒提取过程中均含有DNA去除步骤,但是RNA质量差异较大,总RNA提取试剂盒A、B提取的RNA样本均存在严重的基因组DNA污染,而试剂盒C提取的总RNA样本无基因组DNA残留(图 5)。
此外,部分试剂公司为了避免总RNA样本中残留基因组DNA,第一链cDNA合成试剂盒中增加了基因组DNA去除步骤。在本试验中,尽管第一链cDNA合成试剂盒Ⅰ中含有基因组DNA去除步骤,但还是无法去除总RNA样本残留的基因组DNA(图 6)。
目前,很多研究都是将获得的总RNA和cDNA样本直接用于后续试验[25-26],尽管市面上大多数RNA提取试剂盒和部分第一链cDNA合成试剂盒中都加入了基因组DNA去除步骤,但获得的总RNA和cDNA样本质量无法预知,因此,为了保证qRT-PCR实验的准确性,必须对提取的总RNA及cDNA样本进行基因组DNA残留检测,这一点应该引起从事类似科学研究者的重视。本研究依据持家基因TIP41-like部分内含子序列设计的基因组DNA残留检测引物,可快速检测岷江百合总RNA以及cDNA样本中有无基因组DNA残留,以确保后续待测基因表达分析的准确性,并为其他百合材料以及其他物种相关研究提供参考。
4 结论依据转录组中持家基因TIP41-like的部分内含子序列设计了1对基因组DNA残留检测引物:LDRG-F:5'-CTCTTTTATGACGAGGTTGGTA-3'及LDRG-R:5'-CAGGGAAATCGGTCAGTGTT-3',能够快速检测总RNA样本中的基因组DNA残留。
| [1] |
Yamagishi M, Yoshida Y, Nakayama M. The transcription factor LhMYB12 deter mines anthocyanin pigmentation in the tepals of Asiatic hybrid lilies(Lilium spp.)and regulates pigment quantity[J]. Molecular Breeding, 2012, 30(2): 913-925. DOI:10.1007/s11032-011-9675-6 |
| [2] |
Du F, Fan JM, Wang T, et al. Identification of differentially expressed genes in flower, leaf and bulb scale of Lilium oriental hybrid 'Sorbonne' and putative control network for scent genes[J]. BMC Genomics, 2017, 18(1): 899. DOI:10.1186/s12864-017-4303-4 |
| [3] |
Yang PP, Xu LF, Xu H, et al. Histological and transcriptomic analysis during bulbil formation in Lilium lancifolium[J]. Frontiers in Plant Science, 2017, 8: 1508. DOI:10.3389/fpls.2017.01508 |
| [4] |
Howlader J, Park JI, Robin AHK, et al. Identification, characterization and expression profiling of stress-related genes in Easter Lily(Lilium formolongi)[J]. Genes, 2017, 8(7): 172. DOI:10.3390/genes8070172 |
| [5] |
黄洁, 刘晓华, 管洁, 等. 百合分子育种研究进展[J]. 园艺学报, 2012, 39(9): 1793-1808. |
| [6] |
Zhang J, Gai MZ, Xue BY, et al. The use of miRNAs as reference genes for miRNA expression normalization during Lilium, somatic embryogenesis by real-time reverse transcription PCR analysis[J]. Plant Cell, Tissue and Organ Culture, 2016, 129(1): 105-118. |
| [7] |
Huggett J, Dheda K, Bustin S, et al. Real-time RT-PCR normalisation; strategies and considerations[J]. Genes and Immunity, 2005, 6(4): 279-284. DOI:10.1038/sj.gene.6364190 |
| [8] |
Lekshmy S, Jha SK. Selection of reference genes suitable for qRT-PCR expression profiling of biotic stress, nutrient deficiency and plant hormone responsive genes in bread wheat[J]. Indian Journal of Plant Physiology, 2017, 22(1): 101-106. DOI:10.1007/s40502-017-0282-3 |
| [9] |
Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR[J]. Nature Protocols, 2006, 1(3): 1559-1582. DOI:10.1038/nprot.2006.236 |
| [10] |
苏晓娟, 樊保国, 袁丽钗, 等. 实时荧光定量PCR分析中毛果杨内参基因的筛选和验证[J]. 植物学报, 2013, 48(5): 507-518. |
| [11] |
Li XY, Cheng JY, Zhang J, et al. Validation of reference genes for accurate normalization of gene expression in Lilium davidii var. unicolor for real time quantitative PCR[J]. PLoS One, 2015, 10(10): e0141323. DOI:10.1371/journal.pone.0141323 |
| [12] |
姜琼, 王幼宁, 王利祥, 等. 盐胁迫下大豆根组织定量PCR分析中内参基因的选择[J]. 植物学报, 2015, 50(6): 754-764. |
| [13] |
陈旭, 齐凤坤, 康立功, 等. 实时荧光定量PCR技术研究进展及其应用[J]. 东北农业大学学报, 2010, 21(8): 148-155. DOI:10.3969/j.issn.1005-9369.2010.08.029 |
| [14] |
Kosir R, Acimovic J, Golicnik M, et al. Determination of reference genes for circadian studies in different tissues and mouse strains[J]. BMC Mol Biol, 2010, 11: 60. DOI:10.1186/1471-2199-11-60 |
| [15] |
Eildermann K, Aeckerle N, Debowski K, et al. Developmental expression of the pluripotency factor sal-like protein 4 in the monkey, human and mouse testis:restriction to premeiotic germ cells[J]. Cells Tissues Organs, 2012, 196(3): 206-220. DOI:10.1159/000335031 |
| [16] |
Carvalho LC, Coito JL, Colaço S, et al. Heat stress in grapevine:the pros and cons of acclimation[J]. Plant Cell and Environment, 2015, 38(4): 777-789. DOI:10.1111/pce.2015.38.issue-4 |
| [17] |
Xu LF, Xu H, Cao YW, et al. Validation of reference genes for quantitative real-time PCR during bicolor tepal development in asiatic hybrid lilies(Lilium spp.)[J]. Frontiers in Plant Science, 2017, 8: 669. DOI:10.3389/fpls.2017.00669 |
| [18] |
Fleige S, Pfaffl MW. RNA integrity and the effect on the real-time qRT-PCR performance[J]. Molecular Aspects of Medicine, 2006, 27(2-3): 126-139. DOI:10.1016/j.mam.2005.12.003 |
| [19] |
任锐, 戴鹏辉, 曹福祥, 等. 珙桐CAC基因的克隆及其作为内参基因的评价[J]. 生物技术通报, 2017, 33(4): 119-129. |
| [20] |
杨丹, 李清, 王贵禧, 等. 平欧杂种榛实时荧光定量PCR内参基因的筛选与体系建立[J]. 中国农业科学, 2017, 50(12): 2399-2410. DOI:10.3864/j.issn.0578-1752.2017.12.020 |
| [21] |
张佳佳, 张力维, 李勇鹏, 等. 不同非生物胁迫下香樟实时定量PCR内参基因的选择[J]. 生物技术通报, 2016, 32(10): 205-211. |
| [22] |
Wilson IG. Inhibition and facilitation of nucleic acid amplification[J]. Appl Environ Microbiol, 1997, 63(10): 3741-3751. |
| [23] |
刘岳霖.大鼠死后脑、心肌和肾组织β-actin mRNA的降解与早期死亡时间的关系[D].上海: 复旦大学, 2011. http://www.cnki.com.cn/Article/CJFDTotal-FYXZ201101003.htm
|
| [24] |
李小燕, 黄欢, 王珏, 等. 不同来源的脱氧核糖核酸酶在基因表达量分析中应用效果的比较研究[J]. 现代生物医学进展, 2013, 13(35): 6838-6841. |
| [25] |
Zheng TC, Chen ZL, Ju YQ, et al. Reference gene selection for qRT-PCR analysis of flower development in Lagerstroemia indica and L. speciosa[J]. PLoS One, 2018, 13(3): e0195004. DOI:10.1371/journal.pone.0195004 |
| [26] |
Liu X, Guan HR, Song M, et al. Reference gene selection for qRT-PCR assays in Stellera chamaejasme subjected to abiotic stresses and hormone treatments based on transcriptome datasets[J]. Peer J, 2018, 6: e4535. DOI:10.7717/peerj.4535 |




