2. 华东师范大学物理与材料科学学院,上海 200241
2. College of Physics and Materials Science, East China Normal University, Shanghai 200241
如今,荧光蛋白已经被广泛用于生命科学的研究中,而作为生物探针是它的一项重要应用。通过基因编码的荧光蛋白探针可以实现对生物小分子的特异性检测,以及实时监测生命过程,从而为病理研究、疾病早期发现提供一种强有力的工具。相比于化学染料探针,基因编码的荧光蛋白探针生物兼容性更好,细胞毒性更小,同时在细胞内的定位也更加准确,一般不会损坏细胞,可以更好的减少人为因素的干扰。环化荧光蛋白探针是近些年来发展比较迅速的一类生物探针,独特的构造方式使其更容易感受外界环境的变化,从而在生物检测方面表现出优异的性能,引起了越来越多科学家的注意,现已发展成为一类重要的荧光蛋白探针。但受制于生物活体内的复杂性及目前检测方法的局限性,还有环化荧光蛋白自身的原因,如亮度低、易受pH值影响、会被光漂白等原因,导致能够真正用于科学研究的环化荧光蛋白探针并不多。本文主要从环化荧光蛋白发展,现阶段的检测方法,以及目前所存在的问题这些方面进行综述,旨在为环化荧光蛋白探针的进一步发展及应用提供参考和帮助。
1 环化荧光蛋白探针发展状况 1.1 环化荧光蛋白的设计理念对于绿色荧光蛋白而言,其由11条螺旋组成的桶和1条穿过桶中心的螺旋构成,发色团则为处于桶内部65-67位的3个氨基酸残基[1-2]。通常,由于单独的发色团会与周围的溶剂相互作用,以及自身的振动和转动,导致非辐射跃迁过程太强,所以是不发光的。而当处于桶内,由于限制了振动与转动,减少了发色团的非辐射跃迁而增强了辐射跃迁过程,从而产生荧光。对于其他颜色的荧光蛋白,其组成结构与绿色荧光蛋白基本类似。而荧光蛋白生物探针对物质检测的原理是探测物与荧光蛋白相互作用影响了发色团的光物理性质,从而根据光强或者其他信息的变化来实现对检测物浓度或者其他信息的标定。
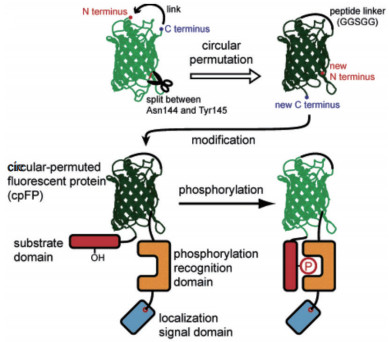
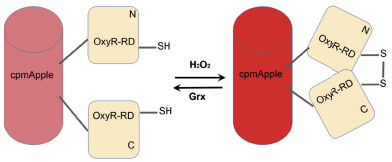
对于环化荧光蛋白探针而言,目前设计的基本原理是在原始荧光蛋白的合适位点打开肽链,并在产生的初始N端和C端之间接上合适的肽链,就可以在发色团的附近产生新的N端和C端。然后在新的N端和C端接上合适的、对某种探测物特异性响应的结合域蛋白[4-5],并通过多次筛选,从而得到所需的环化荧光蛋白探针(图 1)。当结合域蛋白和探测物结合后会改变结合域蛋白的构象结构,从而影响荧光蛋白发色团所处的微环境,进而改变其发光特性,利用其光物理性质的变化对探测物的浓度等信息进行监测。例如,基于环化荧光蛋白的HyPer系列氧化还原类探针,依赖的原理就是H2O2进入结合域蛋白的疏水腔从而改变疏水结构,在残基间形成二硫键使得结合域蛋白的构象急剧变化,影响了荧光蛋白发色团所处的微环境,导致光物理性质发生改变(图 2),而后根据光谱的变化实现测量[6-9]。因为二硫键在一定条件下对氧化还原状态比较敏感,所以Fan等[10]在2015年的报道中提出直接将二硫键插入到环化荧光蛋白的N端和C端之间,以此来检测氧化还原状态的改变。可见,环化荧光蛋白探针并不一定需要具备结合域蛋白,重要的是在新产生N端和C端之间提供一个可以感受探测物的位点,这表明环化荧光蛋白的结合区域也可以由化学小分子或者其他物质来充当。
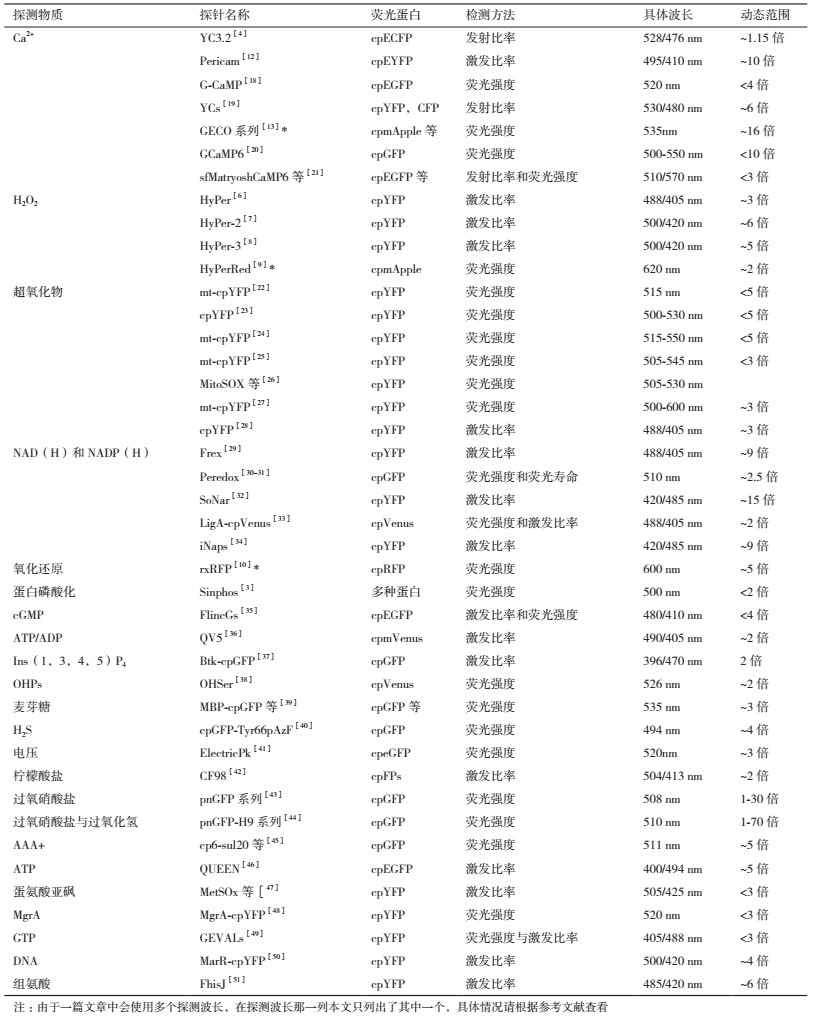
1.2 环化荧光蛋白生物探针的发展自从1997年,Miyawaki和Tsien等[11]在Nature杂志上报道首个基于绿色荧光蛋白的Ca2+探针后,该课题组便不断尝试使用其他方法构建新的Ca2+探针。1999年,Tsien和Baird等[4]使用环化青色荧光蛋白取代供体的原始青色荧光蛋白,试图提高探针的动态响应范围。尽管最后在加入饱和的Ca2+后强度比率变化仅为15%,但作者认为这个结果并不能否定环化荧光蛋白在生物探针方向的潜力,也为环化荧光蛋白的设计打开了大门。在此启发下,Miyawaki课题组[12]在2001年报道了基于环化荧光蛋白开发的第一种实用Ca2+探针,即Pericam。Pericam融合了携钙素(CaM)和目标肽M13作为Ca2+响应区域,在结合Ca2+之后,利用激发光谱中的峰值强度比例变化情况来检测Ca2+浓度。在他们的工作中得到的一系类变体均具有较大的动态响应范围,并成功地实现了细胞内的Ca2+的检测。之后,一系列的Ca2+环化荧光蛋白探针被开发出来,并被用于多个方面[13-17]。表 1列出了这些年来出现的大部分的基于环化荧光蛋白设计的生物探针。
可以看出,近些年来基于环化荧光蛋白设计的生物探针越来越多。主要的使用范围还是用于对细胞内代谢产物的研究、细胞内氧化还原状态的监测以及药物筛选等领域,对体外物质的检测比较少。而且,值得注意的是,对某一类物质特异性响应的环化荧光蛋白探针的发展也是一个不断改进的过程。例如,第一代Hyper由环化黄色荧光蛋白和来自大肠杆菌中对H2O2敏感的结合域蛋白OxyR-RD组成,显示出对H2O2亚微摩尔的亲和力,但是动态范围只有3倍左右[6]。为解决第一代Hyper动态范围小的问题,Hyper-2被研发出来,其动态范围达到6倍[7]。但是Hyper-2是强二聚体,导致其对氧化还原动力学响应较慢,无法实现实时监测。在此基础之上,又开发了第三代Hyper-3[8],它对H2O2具有更快的响应速率,而且动态变化范围和Hyper-2类似。从上面表 1中可以看出来,目前环化荧光蛋白探针的动态响应范围还不是很大,普遍在10倍以内,有的甚至变换不到2倍,如2016年在Science上报道的对NAD+检测的环化荧光蛋白探针[33]。总的来说,动态范围大、响应快速的环化荧光蛋白探针是一个重要的发展方向。
2 环化荧光蛋白生物探针的检测方法目前环化荧光蛋白探针对物质检测时所使用的方法主要还是基于荧光强度和荧光比率这两种。对于使用荧光强度方法的环化荧光蛋白探针,其稳态荧光光谱一般只有单一的激发峰和发射峰。如对过氧硝酸盐检测的pnGFP[43],通过探测加入探测物之后单个荧光通道的荧光强度变化,从而实现对物质的定量测量。而使用荧光比率方法的环化荧光蛋白探针,其稳态荧光光谱一般具有两个激发峰,或者两个发射峰,可进一步分为激发比率型探针和发射比率型探针。如YCs系列环化荧光蛋白探针就是基于发射比率的方法对Ca2+进行检测,FlincGs探针则是基于激发比率来检测cGMP[19, 35]。无论激发比率型还是发射比率型环化荧光蛋白探针,都是通过测量两个波长处的荧光强度比值变化情况来实现对探测物的定量检测。由于两个波长处的荧光强度来自同一蛋白,所以二者的比值跟环化荧光蛋白生物探针的浓度无关。最理想的情况是两个波长处荧光强度变化情况相反,从而荧光比值能够更灵敏的检测探测物的变化。总而言之,荧光比率的方法可以不受环化荧光蛋白在细胞内表达的影响,具备了可以直接检测探测物绝对浓度的能力,同时又具有大的灵敏度。
从上面介绍中我们可以得知,能使用荧光比率方法的探针一般来说都可以使用荧光强度的方法,但是对于只具有单一激发峰和发射峰的环化荧光蛋白探针一般只能使用荧光强度的方法。如果针对某一物质为了实现比率型方法测量而重新设计一种全新的探针,势必需要大量的工作。科学家们为了解决这个问题,通过在原有环化荧光蛋白探针上接上另一种不同颜色的荧光蛋白来实现比率测量的目的。由于两种发光蛋白都是已知的,这样一来也就减少了工作量。例如,Peredox是一种检测还原型辅酶和其氧化物浓度比值的探针(NADH/NAD+),它只有单一的激发峰和发射峰[30]。Hung和Yellen等[30]通过在Peredox上连接红色的荧光蛋白mCherry,来形成一种新的探针Peredox-mCherry。在使用过程中利用mCherry的荧光强度来标定环化荧光蛋白探针在细胞内表达的量,从而消除荧光强度型探针易受细胞内表达量影响的缺点,对NADH/NAD+比率的测量则还是利用新探针的内部的Peredox。即使用mCherry荧光强度对Peredox的荧光强度幅值进行修正,从而实现了比率测量的目的。但是作为一个有多个发色团的环化荧光蛋白,Peredox-mCherry的分子量很大,容易引起蛋白聚集等效应,影响探针的使用。而且,该探针中的绿色荧光蛋白和红色荧光蛋白的成熟和光漂白过程不同步,限制了该探针的性能。不过由于减少了工作量,这样的方法也为环化荧光蛋白的设计提供了新的思路。
3 时间分辨荧光在环化荧光蛋白探针中的应用 3.1 时间分辨荧光方法的优势虽然相对于电化学等方法,荧光强度和荧光比率的检测方式可以避免对细胞产生损害。但是这两种方法都属于稳态荧光方法的范畴,本质上还是基于荧光强度的测量来实现检测目的。这就带来了一个问题,因为测量荧光强度的仪器设备在不同厂商制造时所使用的标准并不一样,无论是稳态荧光光谱仪、酶标仪,还是共聚焦显微成像系统都有这个问题,所以在用不同的设备对细胞内的物质进行定量测量时所得到的结果严格来说并不能直接进行比较。时间分辨荧光的方法为此提供一个解决途径,它通过测量荧光寿命的变化来标定分析物的浓度。一般来说,环化荧光蛋白的荧光寿命会随着探测物的加入而发生改变,通过测量荧光寿命的变化,从而可以得出探测物浓度等信息。相对于稳态荧光方法,时间分辨荧光方法由于提供了一个通用的标准——“时间”,这就允许了在不同设备上测得的数据可以直接比较。同时荧光寿命反映的是环化荧光蛋白生物探针从激发态返回基态的过程,在合理的浓度下,与荧光蛋白的数量无关。所以环化荧光蛋白在细胞内表达的数量并不影响其荧光寿命,具备了比率型方法不受发色团浓度影响的优势。同时有些荧光物质由于光谱相似,使用稳态的方法难以区分。如还原型辅酶NADH和NADPH,由于它们的光谱非常相似,如何利用光学的方法将它们区分曾一度困扰着科学家[52]。除了发展对它们各自特异性响应的荧光探针外,2014年,Blacker等[53]使用时间分辨荧光技术,结合数学模型很好的解决了这个问题。这显示出时间分辨荧光方法在物质检测领域具有独特的优势,未来在环化荧光蛋白探针中也必将发挥更大的作用。
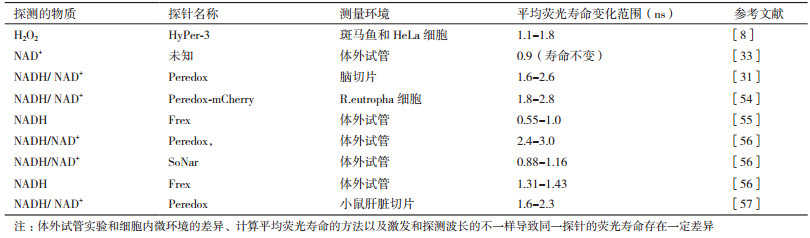
3.2 荧光寿命成像在环化荧光蛋白探针中的应用相比于荧光显微镜,基于荧光强度对生物组织进行成像,也可以在此基础上,基于时间分辨荧光的方法,利用荧光寿命大小对生物组织成像。通过不断的激发和扫描样品,再经过分析得到每个像素点处的荧光寿命大小,进而观察生物组织不同位置的荧光寿命变化情况,以此来监测探测物。即将体外一维的寿命信息,通过成像技术转变为二维的图像。通过我们的文献调研,表 2列出了目前已知的环化荧光蛋白探针在荧光寿命方面研究的结果。
从表 2中可以看出目前荧光寿命成像(Fluorescence lifetime imaging microscopy,FLIM)技术在环化荧光蛋白探针上的应用刚刚起步,近些年才开始逐渐使用。而且大多数被用于还原型辅酶NAD(H)方面[31, 33, 54-57],应用的还不是十分广泛,但已表明时间分辨荧光方法的优势。除了仪器比较昂贵以外,限制FLIM技术应用的主要原因,在于环化荧光蛋白探针的平均荧光寿命在探测物浓度改变的时候变化并不明显,通常只有不到1 ns的荧光寿命变化。甚至有的环化荧光蛋白探针荧光寿命几乎不变化,如2016年报道的对NAD+检测的环化荧光蛋白[33]。如此小的平均荧光寿命变化限制了探针对探测物响应的动态范围,自然限制了这种方法在该类探针上的使用。如果要解决这个问题,一方面是要通过不断的突变和筛选,得到荧光寿命变化更明显的环化荧光蛋白探针;另一方面要不断的研究环化荧光蛋白的发光机理,探究影响环化荧光蛋白荧光寿命变化的因素,从根本上解决目前环化荧光蛋白平均荧光寿命变化小的问题,为以后的环化荧光蛋白的设计提供指导。
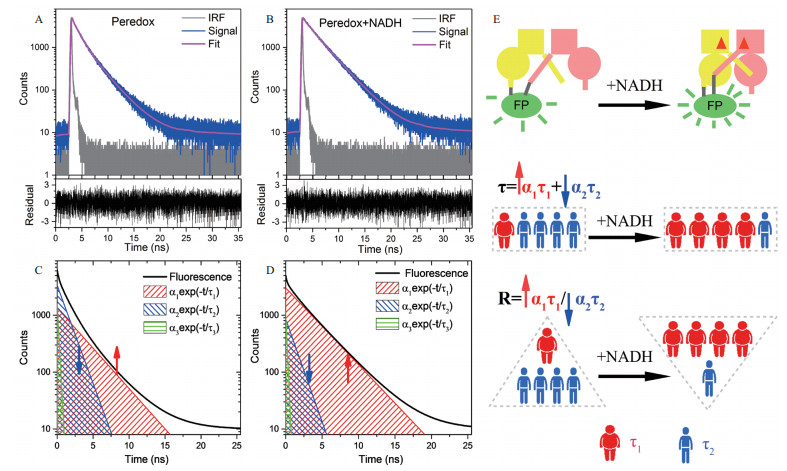
在现有的条件下,也可以通过探索新的方法达到增大变化动态范围的目的。我们知道一般环化荧光蛋白探针的荧光衰减并不是按照单一指数衰减,而是有多个衰减成分。平均荧光寿命只是各组分寿命综合作用的表现,并不代表每一组份寿命的变化情况,这也表明可以进一步挖掘荧光寿命的信息进行物质检测。2017年,Chang等[56]利用部分荧光强度比值的方法实现了对探测物浓度的表征,极大的扩大了动态范围。其原理如图 3所示,通过对Peredox环化荧光蛋白探针在加入NADH前后的时间分辨荧光衰减曲线进行拟合(图 3-A,3-B),拆分出τ1、τ2和τ3这3个荧光寿命组成成分以及它们各自所占的比重α1、α2和α3。在图 3-C,3-D中分别用绿蓝红这3种颜色来表示τ1、τ2和τ3,其中不同的直线斜率代表了不同的寿命大小,而荧光衰减曲线下不同颜色所占的面积则表示了不同组分对整体荧光强度所占的贡献,它的大小跟部分强度相关。可以看出,加入NADH后部分强度α1τ1增大,而α2τ2减小,利用它们比值α1τ1/α2τ2的变化来实现对探测物浓度的定量测量(图 3-E)。这种方法可以极大的扩展动态范围,即使像Frex这种平均荧光寿命变化动态范围变化只有1.09倍左右的环化荧光蛋白探针,在此方法的处理下,也可以得到了4倍多的动态范围[56]。而且对于Peredox这种只有单一激发峰和发射峰的环化荧光蛋白探针,利用此方法同时实现了比率测量,无需在其上面接上新的荧光蛋白,这是在稳态荧光方法中目前还实现不了的。随着环化荧光蛋白探针的研究越来越深入,时间分辨荧光这种方法会发挥出越来越重要的作用。
4 现阶段的问题无论是使用哪种方法对物质进行检测,前提条件还是环化荧光蛋白探针的性质要优良。值得注意的是,目前大多数的环化荧光蛋白荧光探针都会或多或少受pH值的影响。主要的原因是pH值会影响处于质子型和离子型状态的环化荧光蛋白比例,从而干扰对探测物浓度的测量。现在消除pH影响可以通过努力筛选出一种对pH值不敏感的荧光蛋白探针,这需要极大的工作量,如Peredox经过大量的定点突变消除了探针的pH敏感性[30]。更普遍的方法是利用额外的荧光蛋白对pH的影响进行校准和修正,如SoNar和FHisJ[32, 51]。而且目前大多数的环化荧光蛋白探针都是基于黄色或者绿色荧光蛋白设计的(表 1),这些短波长的荧光在生物组织内部的散射性太大,导致成像深度不深,而且对细胞的光毒性也比较大。而环化红色荧光蛋白探针由于突变的困难,目前还没有被广泛的开发出来。相比于化学染料探针,目前的环化荧光蛋白探针的动态范围还比较小,这也是普遍存在的一个问题。同时,现阶段的使用的探测方法还比较单一,时间分辨荧光的还没有被普遍使用。同时值得注意的是,抛开环化荧光蛋白探针自身性质的因素,FLIM这种基于时间分辨荧光的技术也有其自身的缺点。因为FLIM得到的是一个二维图像,其图像上的每个像素点都代表着一条时间分辨荧光衰减曲线。在我们的实验中,每一幅图有6万多个像素点,这也就是6万多条时间分辨荧光衰减曲线。而且,每条曲线如果要得到准确的拟合,就要保证在每个像素点有足够多的光子数被记录。同时为了保证单光子探测的效果,光子计数率不能太大。这些原因会导致为了得到一副准确的荧光寿命图像需要测量大量的时间。一般发光强度比较弱的环化荧光蛋白并不适合使用FLIM技术,因为会导致细胞长时间处于在光照下,从而产生光漂白以及损害细胞活性等问题。同时,开发出一个新的荧光探针的工作量仍然很大,科学家们在过去十几年间也对设计环化荧光蛋白的方法进行不断的研究[21, 58-59]。例如,2017年,Wongso等[59]报道了一种基于环化荧光蛋白设计生物探针的新方法,并把这类探针称为Flashbody。这类探针采用抗体作为结合域蛋白,通过优化荧光蛋白与抗体的连接方式产生最佳的效果。相比于从天然蛋白质中筛选结合域蛋白这种费时费力的方法,使用抗体充当结合域蛋白则会使探针的开发简单得多,在某些方面会极大的减少工作量,也会减少开发的盲目性。Ast等[21]也报道了一种基于环化荧光蛋白设计生物探针的通用开发平台。它采用俄罗斯套娃的模式,将两个荧光蛋白进行嵌套,然后将其插入在结合域蛋白的合适位置。其中一个荧光蛋白用来修正非探测物因素对荧光探针的影响,另一个蛋白则用来对探测物进行响应,利用二者的荧光强度比值来实现对探测物的准确测量。可以看出,目前的环化荧光蛋白的一大研究方向就是尽量减少开发的工作量,使开发流程能够简单化,同时又要保证荧光探针能实现对探测物的准确测量。
5 结语本文回顾了这些年来环化荧光蛋白探针的发展状况,比较了几种探测方法的优劣,以及环化荧光蛋白目前存在的问题。总体上说,环化荧光蛋白探针正在越来越多的被科学家注意到,更多的环化荧光蛋白探针也被愈加广泛的被应用在生命科学领域去解决不同的问题。虽然目前还存在着诸多问题,但是随着对环化荧光蛋白研究的不断深入,以及设计方法不断的完善,动态范围更大、特异性更好、响应速度更快、亮度更强、抗漂白性更好、pH值耐受性更强、发光波长更长的环化荧光蛋白生物探针也必定会被陆续被开发出来。在探测的方法上,也会随着对这类蛋白机理研究的更加深入,不断的发展新的探测和分析方法。同时随着学科之间融合的更加密切,环化荧光蛋白也必将会在生命科学领域发挥越来越重要的作用。
| [1] |
Ormo M, Cubitt AB, Kallio K, et al. Crystal structure of the Aequorea victoria green fluorescent protein[J]. Science, 1996, 273(5280): 1392-1395. DOI:10.1126/science.273.5280.1392 |
| [2] |
Yang F, Moss LG, Phillips GN. The molecular structure of green fluorescent protein[J]. Nature Biotechnology, 1996, 14(10): 1246-1251. DOI:10.1038/nbt1096-1246 |
| [3] |
Kawai Y, Sato M, Umezawa Y. Single color fluorescent indicators of protein phosphorylation for multicolor imaging of intracellular signal flow dynamics[J]. Analytical Chemistry, 2004, 76(20): 6144-6149. DOI:10.1021/ac040037s |
| [4] |
Baird GS, Zacharias DA, Tsien RY. Circular permutation and receptor insertion within green fluorescent proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(20): 11241-11246. DOI:10.1073/pnas.96.20.11241 |
| [5] |
Topell S, Hennecke J, Glockshuber R. Circularly permuted variants of the green fluorescent protein[J]. Febs Letters, 1999, 457(2): 283-289. DOI:10.1016/S0014-5793(99)01044-3 |
| [6] |
Belousov VV, Fradkov AF, Lukyanov KA, et al. Genetically encoded fluorescent indicator for intracellular hydrogen peroxide[J]. Nature Methods, 2006, 3(4): 281-286. DOI:10.1038/nmeth866 |
| [7] |
Markvicheva KN, Bilan DS, Mishina NM, et al. A genetically encoded sensor for H2O2 with expanded dynamic range[J]. Bioorganic & Medicinal Chemistry, 2011, 19(3): 1079-1084. |
| [8] |
Bilan DS, Pase L, Joosen L, et al. HyPer-3:A genetically encoded H2O2 probe with improved performance for ratiometric and fluorescence lifetime imaging[J]. ACS Chemical Biology, 2013, 8(3): 535-542. DOI:10.1021/cb300625g |
| [9] |
Ermakova YG, Bilan DS, Matlashov ME, et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide[J]. Nature Communications, 2014, 5: 5222-5231. DOI:10.1038/ncomms6222 |
| [10] |
Fan Y, Chen Z, Ai H. Monitoring redox dynamics in living cells with a redox-sensitive red fluorescent protein[J]. Analytical Chemistry, 2015, 87(5): 2802-2810. DOI:10.1021/ac5041988 |
| [11] |
Miyawaki A, Llopis J, Heim R, et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin[J]. Nature, 1997, 388(6645): 882-887. DOI:10.1038/42264 |
| [12] |
Nagai T, Sawano A, Park ES, et al. Circularly permuted green fluorescent proteins engineered to sense Ca2+[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(6): 3197-3202. DOI:10.1073/pnas.051636098 |
| [13] |
Zhao Y, Araki S, Wu J, et al. An expanded palette of genetically encoded Ca2+ indicators[J]. Science, 2011, 333(6051): 1888-1891. DOI:10.1126/science.1208592 |
| [14] |
Helassa N, Podor B, Fine A, et al. Design and mechanistic insight into ultrafast calcium indicators for monitoring intracellular calcium dynamics[J]. Scientific Reports, 2016, 6: 38276-38288. DOI:10.1038/srep38276 |
| [15] |
Shen Y, Dana H, Abdelfattah AS, et al. A genetically encoded Ca2+ indicator based on circularly permutated sea anemone red fluorescent protein eqFP578[J]. Bmc Biology, 2018, 16: 9. DOI:10.1186/s12915-018-0480-0 |
| [16] |
Akerboom J, Calderon NC, Tian L, et al. Genetically encoded calcium indicators for multi-color neural activity imaging and combination with optogenetics[J]. Frontiers in Molecular Neuroscience, 2013, 6: 2. |
| [17] |
Carlson HJ, Campbell RE. Circular permutated red fluorescent proteins and calcium ion indicators based on mCherry[J]. Protein Engineering Design & Selection, 2013, 26(12): 763-772. |
| [18] |
Nakai J, Ohkura M, Imoto K. A high signal-to-noise Ca2+ probe composed of a single green fluorescent protein[J]. Nature Biotechnology, 2001, 19(2): 137-141. |
| [19] |
Nagai T, Yamada S, Tominaga T, et al. Expanded dynamic range of fluorescent indicators for Ca2+ by circularly permuted yellow fluorescent proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(29): 10554-10559. DOI:10.1073/pnas.0400417101 |
| [20] |
Chen TW, Wardill TJ, Sun Y, et al. Ultrasensitive fluorescent proteins for imaging neuronal activity[J]. Nature, 2013, 499(7458): 295-302. DOI:10.1038/nature12354 |
| [21] |
Ast C, Foret J, Oltrogge LM, et al. Ratiometric Matryoshka biosensors from a nested cassette of green-and orange-emitting fluorescent proteins[J]. Nature Communications, 2017, 8: 431-443. DOI:10.1038/s41467-017-00400-2 |
| [22] |
Wang W, Fang H, Groom L, et al. Superoxide flashes in single mitochondria[J]. Cell, 2008, 134(2): 279-290. DOI:10.1016/j.cell.2008.06.017 |
| [23] |
Schwarzlander M, Logan DC, Fricker MD, et al. The circularly permuted yellow fluorescent protein cpYFP that has been used as a superoxide probe is highly responsive to pH but not superoxide in mitochondria:implications for the existence of superoxide 'flashes'[J]. Biochemical Journal, 2011, 437(3): 381-387. DOI:10.1042/BJ20110883 |
| [24] |
Cao Y, Zhang X, Shang W, et al. Proinflammatory cytokines stimulate mitochondrial superoxide flashes in articular chondrocytes in vitro and in situ[J]. PLoS One, 2013, 8(6): e66444. DOI:10.1371/journal.pone.0066444 |
| [25] |
Wei-Lapierre L, Gong G, Gerstner BJ, et al. Respective contribution of mitochondrial superoxide and pH to mitochondria-targeted circularly permuted yellow fluorescent protein (mt-cpYFP) flash activity[J]. J Biol Chem, 2013, 288(15): 10567-10577. DOI:10.1074/jbc.M113.455709 |
| [26] |
Zhang X, Huang Z, Hou T, et al. Superoxide constitutes a major signal of mitochondrial superoxide flash[J]. Life Sciences, 2013, 93(4): 178-186. DOI:10.1016/j.lfs.2013.06.012 |
| [27] |
Wang W, Zhang H, Cheng H. Mitochondrial flashes:From indicator characterization to in vivo imaging[J]. Methods, 2016, 109: 12-20. |
| [28] |
Schwarzlander M, Wagner S, Ermakova YG, et al. The 'mitoflash' probe cpYFP does not respond to superoxide[J]. Nature, 2014, 514(7523): E12-14. DOI:10.1038/nature13858 |
| [29] |
Zhao Y, Jin J, Hu Q, et al. Genetically encoded fluorescent sensors for intracellular NADH detection[J]. Cell Metabolism, 2011, 14(4): 555-566. DOI:10.1016/j.cmet.2011.09.004 |
| [30] |
Hung YP, Albeck JG, Tantama M, et al. Imaging cytosolic NADH-NAD+ redox state with a genetically encoded fluorescent biosensor[J]. Cell Metabolism, 2011, 14(4): 545-554. DOI:10.1016/j.cmet.2011.08.012 |
| [31] |
Mongeon R, Venkatachalam V, Yellen G. Cytosolic NADH-NAD+ redox visualized in brain slices by two-photon fluorescence lifetime biosensor imaging[J]. Antioxidants & Redox Signaling, 2016, 25(10): 553-563. |
| [32] |
Zhao Y, Hu Q, Cheng F, et al. SoNar, a highly responsive NAD+/NADH sensor, allows high-throughput metabolic screening of anti-tumor agents[J]. Cell Metabolism, 2015, 21(5): 777-789. DOI:10.1016/j.cmet.2015.04.009 |
| [33] |
Cambronne XA, Stewart ML, Kim D, et al. Biosensor reveals multiple sources for mitochondrial NAD+[J]. Science, 2016, 352(6292): 1474-1477. DOI:10.1126/science.aad5168 |
| [34] |
Tao R, Zhao Y, Chu H, et al. Genetically encoded fluorescent sensors reveal dynamic regulation of NADPH metabolism[J]. Nature Methods, 2017, 14(7): 720-728. DOI:10.1038/nmeth.4306 |
| [35] |
Nausch LW, Lecloux J, Bonev AD, et al. Differential patterning of cGMP in vascular smooth muscle cells revealed by single GFP-linked biosensors[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(1): 365-370. DOI:10.1073/pnas.0710387105 |
| [36] |
Berg J, Hung YP, Yellen G. A genetically encoded fluorescent reporter of ATP:ADP ratio[J]. Nature Methods, 2009, 6(2): 161-166. DOI:10.1038/nmeth.1288 |
| [37] |
Sakaguchi R, Endoh T, Yamamoto S, et al. A single circularly permuted GFP sensor for inositol-1, 3, 4, 5-tetrakisphosphate based on a split pH domain[J]. Bioorganic & Medicinal Chemistry, 2009, 17(20): 7381-7386. |
| [38] |
Zhao BS, Liang Y, Song Y, et al. A highly selective fluorescent probe for visualization of organic hydroperoxides in living cells[J]. Journal of the American Chemical Society, 2010, 132(48): 17065-17067. DOI:10.1021/ja1071114 |
| [39] |
Marvin JS, Schreiter ER, Echevarria IM, et al. A genetically encoded, high-signal-to-noise maltose sensor[J]. Proteins, 2011, 79(11): 3025-3036. DOI:10.1002/prot.23118 |
| [40] |
Chen S, Chen Z, Ren W, et al. Reaction-based genetically encoded fluorescent hydrogen sulfide Sensors[J]. Journal of the American Chemical Society, 2012, 134(23): 9589-9592. DOI:10.1021/ja303261d |
| [41] |
Barnett L, Platisa J, Popovic M, et al. A fluorescent, genetically-encoded voltage probe capable of resolving action potentials[J]. PLoS One, 2012, 7(9): e43454. DOI:10.1371/journal.pone.0043454 |
| [42] |
Honda Y, Kirimura K. Generation of circularly permuted fluorescent-protein-based indicators for in vitro and in vivo detection of citrate[J]. PLoS One, 2013, 8(5): e64597. DOI:10.1371/journal.pone.0064597 |
| [43] |
Chen Z, Ren W, Wright QE, et al. Genetically encoded fluorescent probe for the selective detection of peroxynitrite[J]. Journal of the American Chemical Society, 2013, 135(40): 14940-14943. DOI:10.1021/ja408011q |
| [44] |
Chen Z, Tian Z, Kallio K, et al. The NB interaction through a water bridge:understanding the chemoselectivity of a fluorescent protein based probe for peroxynitrite[J]. Journal of the American Chemical Society, 2016, 138(14): 4900-4907. DOI:10.1021/jacs.6b01285 |
| [45] |
Wohlever ML, Nager AR, Baker TA, et al. Engineering fluorescent protein substrates for the AAA+ Lon protease[J]. Protein Engineering Design and Selection, 2013, 26(4): 299-305. DOI:10.1093/protein/gzs105 |
| [46] |
Yaginuma H, Kawai S, Tabata KV, et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging[J]. Scientific Reports, 2014, 4: 6522-6529. |
| [47] |
Tarrago L, Peterfi Z, Lee BC, et al. Monitoring methionine sulfoxide with stereospecific mechanism-based fluorescent sensors[J]. Nature Chemical Biology, 2015, 11(5): 332-338. DOI:10.1038/nchembio.1787 |
| [48] |
Wang Y, Zhang H, Zhang Q, et al. Genetically encoded fluorescence screening probe for MgrA, a global regulator in Staphylococcus aureus[J]. RSC Advances, 2015, 5(106): 87216-87220. DOI:10.1039/C5RA11455A |
| [49] |
Bianchi-smiraglia A, Rana MS, Foley CE, et al. Internally ratiometric fluorescent sensors for evaluation of intracellular GTP levels and distribution[J]. Nature Methods, 2017, 14(10): 1003-1009. DOI:10.1038/nmeth.4404 |
| [50] |
Zhu R, Hao Z, Lou H, et al. Structural and mechanistic study of the cysteine oxidation-mediated induction of the Escherichia coli MarR regulator[J]. Tetrahedron, 2017, 73(26): 3714-3719. DOI:10.1016/j.tet.2017.05.039 |
| [51] |
Hu H, Gu Y, Xu L, et al. A genetically encoded toolkit for tracking live-cell histidine dynamics in space and time[J]. Scientific Reports, 2017, 7: 43479-43488. DOI:10.1038/srep43479 |
| [52] |
Mayevsky A, Rogatsky GG. Mitochondrial function in vivo evaluated by NADH fluorescence:from animal models to human studies[J]. American Journal of Physiology-Cell Physiology, 2007, 292(2): C615-C640. DOI:10.1152/ajpcell.00249.2006 |
| [53] |
Blacker TS, Mann ZF, Gale JE, et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM[J]. Nature Communications, 2014, 5: 3936-3945. DOI:10.1038/ncomms4936 |
| [54] |
Tejwani V, Schmitt FJ, Wilkening S, et al. Investigation of the NADH/NAD+ ratio in Ralstonia eutropha using the fluorescence reporter protein Peredox[J]. Biochimica et Biophysica Acta, 2017, 1858(1): 86-94. DOI:10.1016/j.bbabio.2016.11.001 |
| [55] |
Wilkening S, Schmitt FJ, Horch M, et al. Characterization of Frex as an NADH sensor for in vivo applications in the presence of NAD+ and at various pH values[J]. Photosynthesis Research, 2017, 133(1-3): 305-315. DOI:10.1007/s11120-017-0348-0 |
| [56] |
Chang M, Li L, Hu H, et al. Using fractional intensities of time-resolved fluorescence to sensitively quantify NADH/NAD+ with genetically encoded fluorescent biosensors[J]. Scientific Reports, 2017, 7: 4209-4218. DOI:10.1038/s41598-017-04051-7 |
| [57] |
Masia R, Mccarty WJ, Lahmann C, et al. Live cell imaging of cytosolic NADH/NAD+ ratio in hepatocytes and liver slices[J]. American Journal of Physiology-Gastrointestinal and Liver Physiology, 2018, 314(1): G97-G108. DOI:10.1152/ajpgi.00093.2017 |
| [58] |
Pandey N, Kuypers BE, Nassif B, et al. Tolerance of a knotted near-Infrared fluorescent protein to random circular permutation[J]. Biochemistry, 2016, 55(27): 3763-3773. DOI:10.1021/acs.biochem.6b00258 |
| [59] |
Wongso D, Dong J, Ueda H, et al. Flashbody:A next generation fluobody with fluorescence intensity enhanced by antigen binding[J]. Analytical Chemistry, 2017, 89(12): 6719-6725. DOI:10.1021/acs.analchem.7b00959 |