2. 北京大数据精准医疗高精尖创新中心 北京航空航天大学,北京 100191
2. Beijing Advanced Innovation Center for Big Data-Based Precision Medicine, Beihang University, Beijing 100191
高通量测序技术的快速发展促进现代基因组学研究的蓬勃发展,然而全基因组测序因成本昂贵,分析过程复杂,使科研人员倍感压力[1],目标序列捕获技术的出现,在很大程度上缓解了上述的问题。目标序列捕获技术是通过特异探针与基因组目标区域杂交,有选择性的富集基因组中的特定片段。近年发展起来的捕获技术,如杂交捕获技术,其样本在杂交前被随机打断成500 bp左右的片段,寡核苷酸探针可能与一些含有部分目标区段的非靶标序列杂交,导致捕获的特异性较差。多重PCR捕获技术是在一个反应体系中同时扩增多个目的片段,扩增子越多,不均一性也越高,PCR扩增的效率使其应用受到一定的限制。分子倒置探针(Molecular Inversion Probe,MIP)技术是最新发展起来的一种分子捕获技术,因其特异性强、重复性好、操作简单、费用低廉且对DNA完整度要求不高等特点,使其一经出现便备受青睐,被广泛应用于分子生物学检测的各个领域。本文主要对MIP技术的原理、实验流程、发展过程、技术特点及其应用进行了综述,旨在使相关领域学者对MIP技术进行全面的了解,为研究者进行目标序列捕获研究时提供更多的参考和选择。
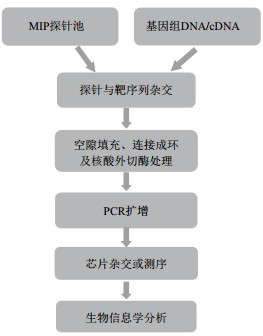
1 MIP技术原理及实验流程MIP技术是指线性的单链DNA序列探针与靶序列基因组杂交形成环状结构,用来捕获目标序列的分子生物学技术。
MIP技术的探针设计颇为新颖,是一段较长的单链DNA序列,两端含有大约20-40 nt长度的特异序列,该序列能与靶标序列互补形成含有一个核苷酸至几百个核苷酸缺口的环状结构。以4种游离的核苷酸(dATP、dTTP、dGTP和dCTP)为原料,在DNA聚合酶作用下以靶标序列为模板填补缺口;DNA连接酶催化探针两端的3',5'-磷酸二酯键,形成完整的环化探针;核酸外切酶降解反应体系中未参与反应的探针和基因组DNA序列,只保留环形探针;以滚环或者线性方式扩增的PCR产物可用于芯片杂交或者高通量测序,获取的实验数据可用于多种生物信息学分析。MIP技术的具体实验流程,见图 1。
|
| 图 1 MIP技术实验流程图 |
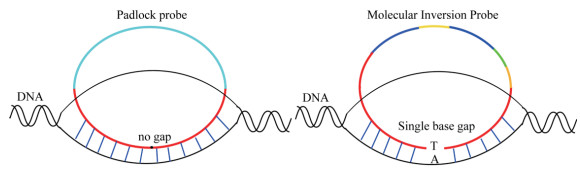
MIP技术是基于锁式探针技术发展而来的一项新型分子捕获技术。锁式探针是一条两端含特异性检测序列的DNA单链,探针的5'端和3'端与靶标序列毗邻互补,DNA连接酶可将线性的锁式探针连接成环,如果探针两端之间有一个碱基不能与模板互补配对,或者有一个碱基的空隙,则无法形成环状结构(图 2左),因此锁式探针技术只能检测已知的DNA序列,用于相似DNA序列的分析[2]。Hardenbol等[3]于2003年首次报道根据锁式探针原理开发出MIP技术,用于单核苷酸多态性(Single nucleotide polymorphism,SNP)分型分析:当线性单链DNA探针与基因组靶标杂交时,在靶标序列的SNP位置有一个碱基的空隙,空隙填充的核苷酸与靶标序列SNP位点的核苷酸互补配对(图 2右)。这种改进的探针较锁式探针具有更多优点,如对于同一SNP位点而言,MIP技术仅需设计一条探针即可,而锁式探针则需设计多条探针。
|
| 图 2 锁式探针与分子倒置探针的比较 |
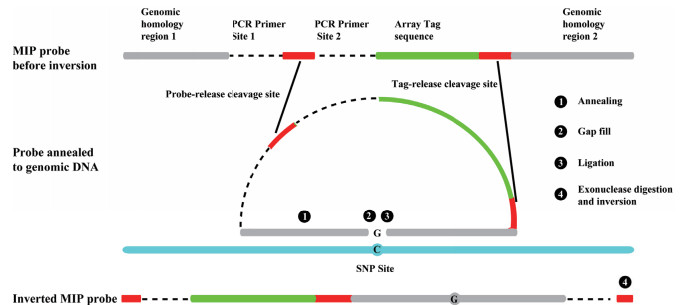
最初,MIP技术仅用于SNP分型,此时的探针共由7个结构元件组成:探针的5'和3'端各有一段与目的基因组序列互补的特异序列(Genomic homology region 1/2)、两段通用引物识别序列(PCR primer Site 1/2)、与芯片特异性结合的标签序列(Array tag sequence)及两段酶切序列(Probe/Tag-release cleavage site),总长度约120 nt[4]。探针与基因组DNA混合杂交形成环状结构,在一系列酶促反应下形成倒置的探针(图 3),线性扩增的产物与芯片杂交,通过杂交信号强度进行SNP分型。Wang等[5]利用此技术检测细胞中完整基因组DNA及FFPE样本DNA的拷贝数变异(Copy number variation,CNV),发现MIP技术也适用于FFPE样本的检测。
|
| 图 3 与芯片杂交结合的MIP技术原理 |
MIP技术与高通量测序技术的结合实现了更长基因组区域的捕获。2007年,Akhras等[6]对探针进行改进,改进后的探针由5个结构元件组成,包括两端与靶标序列结合的片段,两段通用引物配对序列及一段限制性酶切位点,探针3'端延伸到5'端,能形成大约100 nt的空隙填充长度,他们将此探针称为“连接倒置探针(Connector inversion probe,CIPer)”。
运用MIP技术捕获外显子测序主要存在两个困难,即捕获效率低和杂合等位基因捕获不均一。为了克服这些困难,2009年,Turner等[7]对MIP技术进行改进以适用于外显子大规模的捕获。他们延长了探针与靶标序列结合配对长度及空隙填充的时间,增加了探针及连接酶的浓度,同时将线性PCR扩增产物混合后用于构建标准鸟枪法测序文库[8]。该探针长度为70 nt,包括两端特异的杂交序列及骨架序列[9]。改进后的MIP技术能同时扩增并测序50 000个外显子。
2013年,Hiatt等[10]将分子倒置探针技术与单分子标记技术相结合,使MIP技术能借助高通量测序检测低频率变异;改进后的方法被称为单分子分子倒置探针技术(Single molecule Molecular Inversion Probes,smMIP)。单分子标记能去除偏差和PCR扩增带来的冗余序列,保留原始样本中真实的信息,能检测到MAF≤1%的低频率变异。该探针主要由3部分构成(图 4):5'-3'端与靶标序列互补的片段(Targeting arm)、骨架序列(Backbone)和单分子标记序列(Molecular tag)。此时使用的PCR扩增正向引物序列包括:测序引物序列和Illumina接头序列1,反向引物包括:测序引物序列、条形码序列和Illumina接头序列2。因此运用此技术扩增的产物无需再构建高通量测序文库,可直接进行上机测序,简化步骤节约时间。
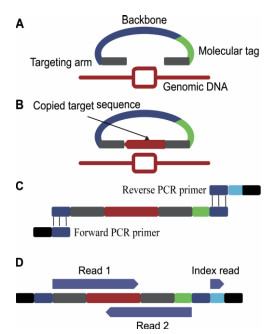
|
| 图 4 与测序技术结合的smMIP流程图 A:smMIP探针; B:空隙填充; C:PCR扩增; D:高通量测序 |
为了降低MIP技术检测的费用,2012年,O’Roak等[11]采用新的MIP设计算法改进工作流程、优化捕获步骤及条件使其适用于高通量检测,开发出了在大样本中实现超低花费的多重靶向候选基因重测序新探针,使得每个样本的单个基因检测费用少于1美元。除此之外,所需DNA样品量少,灵敏度和阳性预测值分别可达到99%和98%,显著提高了捕获的效率。
2014年中国研究者将MIP技术与操作简单、费用更低的琼脂糖凝胶电泳结合,用于区分乙型肝炎病毒菌株单一位点的SNP分型,MIP探针与目的序列杂交后,探针的3'端获得一个与靶标序列SNP位点互补的碱基空隙,反应产物分为4等份,每一等份加入一种游离的核苷酸填充空隙,随后PCR扩增产物进行琼脂糖凝胶电泳,通过泳道条带对应加入的核苷酸种类即可判断SNP分型[12](图 5)。
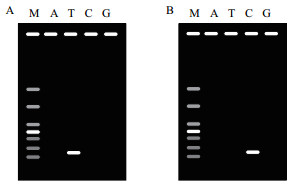
|
|
图 5 MIP技术与琼脂糖凝胶电泳结合检测SNP分型模式图
A图为野生型;B图为突变株;M标示DNA标准带(1 000 bp) A标示dATP;T标示dTTP;C标示dCTP;G标示dGTP |
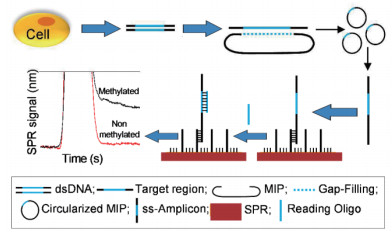
2014年,Carrascosa等[13]将MIP技术与等离子体共振技术(Surface plasmon resonance technology,SPR)结合,用于检测DNA甲基化区域。具体改进的地方包括:探针与经过亚硫酸氢盐处理过的基因组DNA和未处理的基因组DNA杂交形成环形探针后,通过不对称PCR进行扩增产生单链寡核苷酸DNA序列,与固定在SPR上的生物传感芯片杂交,Reading Oligo只与含有甲基化序列的片段结合而使SPR信号强度增强,从而将含有甲基化的片段与非甲基化片段区分开。具体流程如图 6。
3 MIP技术的特点分子倒置探针与线性探针相比,能够指数级减少由于线性引物所引起的交叉反应及二聚体。此外,MIP技术还具有如下主要特点。
3.1 特异性强通过探针与基因组序列杂交互补来捕获靶标序列,减少了非特异性序列的影响。DNA聚合酶、连接酶和核酸外切酶等一系列酶促反应处理后,降解反应体系中的线性探针及基因组DNA序列,有效减少其对PCR扩增的影响,降低背景对结果的干扰[5]。此外,DNA聚合酶校正3'端的错配能力较5'端高,MIP探针3'端的单碱基延伸及后续的连接反应确保能更好的区分出单个碱基突变。这些巧妙的设计显著增强了MIP技术的特异性。而其他传统捕获技术如杂交捕获因探针可与非靶标序列结合,而造成特异性较差。
3.2 对DNA样品完整度要求不严格探针两端与靶标序列结合的长度仅需40 bp左右,因此适用于部分降解的DNA样本。而高通量测序需要靶向富集和深度覆盖,才能获得福尔马林石蜡包埋样本(Formalin-fixed paraffin-embedded,FFPE)的拷贝数信息。正因如此,Affymetrix公司基于MIP技术针对900个癌基因优化设计探针,研发出适用于FFPE样本的OncoScanTM FFPE Assay kit和OncoScanTM CNV FFPE Assay Kit,该试剂盒在一次试验中可同时检测出拷贝数变异、杂合性丢失等信息。
3.3 操作简单、时间短MIP技术检测中用到的试剂、仪器都非常常见,许多实验室均能满足其操作要求。同时,在PCR扩增过程中,除使用线性扩增外,还可以采用滚环扩增,且PCR扩增产物无需专门构建文库便可以直接上机测序,这不仅简化操作步骤还大大缩短操作时间。例如,罗氏NimbleGen基于此技术最新推出的针对肿瘤和遗传病研究的HEAT-Seq目标片段富集系列产品如HEAT-SEQ oncology panel,具有操作流程简单省时的特点,且不乏高效可信的变异检测能力。从样本DNA提取到完成测序文库构建耗时不足8 h,其中手工操作时间不到2 h。
3.4 所需DNA样品少,花费低基于MIP技术捕获的目标序列经PCR扩增后可以直接上机测序,因此只需少量的DNA样品即可完成操作(≥200 ng)。而PCR捕获技术、杂交捕获技术需要的样品量要达到µg级。此外,尽管MIP技术总体费用较高,但与其他捕获技术相比,背景对目的片段的富集影响小,使得其通量更高,进而将每个样本的单个基因检测花费降低到不足1美元[10]。
3.5 重复性高使用MIP捕获技术结合芯片杂交对同一个体SNP进行分型时,重复性可以达到99.9%[3]。
3.6 MIP技术不足MIP技术在捕获目的序列的过程中,捕获的目的序列不能过长,否则空间效应可能会影响探针与靶标序列的成功杂交,对实验结果产生影响。因此适用于小规模DNA片段的研究(≤5 Mb)。此外,对于一些位点如高GC含量区目前还无法设计探针,因此捕获效率较差。
4 MIP技术在疾病研究上的应用人类基因组计划和国际人类基因组单倍型图计划(HapMap)的完成以及生物芯片研发和高通量测序技术的发展,极大地促进了人们对疾病的认识与理解,为疾病的预防、诊断及治疗提供了新的思路。值得注意的是,研究人员利用MIP技术参与HapMap项目的实施,用于7号染色体上成千上万个SNP的分型,其中大于11 000个非同义SNP被归为HapMap项目的一部分[14]。MIP技术作为一种新型的分子捕获技术,因其操作简单、费用低等优势在研究人类疾病方面具有其独特的价值。
4.1 在遗传疾病方面的应用孤独症谱系障碍(Autism spectrum disorder,ASD)近年呈现逐渐攀升的趋势,严重影响人类正常生活。2012年,O'Roak等[11]利用MIP技术对2000多位受到不同类型ASD影响的患者进行了多基因分析发现,6种频发突变可能是造成1%偶发性ASD的病因。2016年,中南大学夏昆教授课题组利用MIP技术在1 543例中国人群ASD患者中完成了针对189个前期研究所提示的孤独症风险基因的靶向捕获测序,并在29个孤独症相关的风险基因中发现大约4%的患者都携带与孤独症相关的de novo(DN)突变,从而揭示了我国孤独症患者潜在的病因[15],这项研究不仅填补了我国孤独症研究领域的空白,同时在国际上也属于领先水平。
脊髓性肌萎缩(Spinal muscular atophy,SMA)是常染色体隐性遗传病,也是最常见的致死性神经肌肉疾病之一。台湾研究者设计不同长度的MIP探针用于识别相同的核苷酸变异,采用不连续滚环进行扩增,利用短柱毛细管电泳检测不同长度PCR产物的基因型,通过检测SMN1基因和SMN2基因的剂量来进行SMA的诊断[16]。
4.2 在癌症方面的研究在癌症研究领域,MIP技术最先应用于结肠直肠癌的研究。Ji等[17]分别对200多个癌基因及染色体臂18q设计MIP探针,分析结肠直肠癌细胞系和原发性结肠直肠癌基因拷贝数变异及外显子突变,结果发现原发性肿瘤有几类不同的结肠直肠癌,并且每类都有特定基因及18q缺失,这一发现具有重要的临床意义,18q丢失可以作为Ⅱ期结直肠癌辅助治疗的潜在指标。Zhang等[18]利用MIP技术检测140例≤35岁患家族或非家族结肠直肠癌患者的7个易感基因,结果表明16例患者中6个基因发生变异,5例患者APC基因发生致病突变,3例患者MLH1基因发生致病突变,以及2例患者MSH2基因发生致病突变,此外3例患者检测到新的突变。以上数据说明利用MIP技术诊断孟德尔式结肠直肠癌综合症是可靠的。
基质金属蛋白酶-7(MMP-7)是一类具有广泛底物的分泌蛋白水解酶,能够降解所有的细胞外基质蛋白,许多研究已经表明其表达与各种肿瘤的侵袭、转移及存活有关[19-21]。Beeghly-Todiel等[22]利用MIP技术结合芯片杂交对MMP-7基因中11个SNP进行基因分型,评估MMP-7中SNP与乳腺癌存活之间的关系,结果发现MMP-7基因多态性是中国女性乳腺癌患者生存的重要决定因素。拷贝数变异一直是了解乳腺癌潜在分子机制的有效工具,Thompson等[23]使用高密度的MIP探针检测971例乳腺癌Ⅰ期/Ⅱ期患者染色体拷贝数增加或缺失情况,鉴定出了12个新的拷贝数变异作为预后标记。随后,研究者继续利用MIP技术对乳腺癌患者的不同染色体的拷贝数进行检测,为乳腺癌的诊断、治疗及预后策略提供了非常重要的参考价值[24-27]。乳腺癌易感基因主要有BRCA1和BRCA2这两种,这两种基因发生突变患乳腺癌的风险高达75%和65%。因此,对BRCA1/2基因的检测显得尤为重要。Neveling等[28]利用smMIP技术分析BRCA1和BRCA2基因编码区拷贝数变异,并开发出一种基于smMIP技术的自动化操作流程。
卵巢癌是女性最常见的恶性肿瘤之一,死亡率居妇科恶性肿瘤之首,MIP技术有助于提高对卵巢癌肿瘤发生过程和疾病进展阶段的了解。2008年,Brown等[29]采用MIP技术对33例卵巢高级别浆液性癌患者中11q13区的扩增子进行研究发现,扩增子的核心区域位于4个致癌基因的6 Mb区域内,在卵巢癌早期4个癌基因扩增与浆液性肿瘤密切相关。研究者利用同样的技术对抑制受体酪氨酸激酶信号传导的Sprouty蛋白基因座进行拷贝数分析发现,在卵巢高级别浆液性癌患中Sprouty 2的拷贝数缺失失,过表达Sprouty将增加表皮生长因子(EGF)诱导的E-钙黏连蛋白的表达,抑制EGF诱导的细胞侵袭[30]。Weren等[31]利用smMIP技术对BRCA1和BRCA2基因进行捕获测序,获得的结果为卵巢癌的治疗策略提供重要的参考价值。
肾细胞癌简称肾癌,男性发病率高于女性。为了确定与透明细胞乳头状肾细胞癌有潜在关系的遗传变异并评估MIP技术在检测肾癌FFPE样本的可行性,Alexiev等[32]利用MIP技术对两种透明细胞乳头状肾细胞癌进行SNP分型分析,结果表明基于MIP技术的SNP芯片可以很好的检测杂合性缺失及中性拷贝杂合性缺失,并且MIP技术对FFPE样本的检测效果良好,商业化OncoScan FFPE Assay kit(Affymetrix,Santa Clara,CA)能够快速、经济地利用肿瘤样本中的少量DNA检测出全基因组拷贝数变异、杂合性丢失及体细胞突变。透明细胞肾细胞癌也被认为是一种代谢性癌症,大约80%患癌都是由VHL基因失活突变或者表观遗传沉默导致的代谢异常引起的[33-34]。Bitter等[35]采用新型smMIP技术检测编码代谢酶的基因活性及其突变情况,研究结果显示smMIP技术可以提供有关代谢途径的相关信息,检测出调节代谢的靶基因活性,并且费用较低操作流程简单。
研究表明许多肿瘤发生的早期阶段抑癌基因甲基化增加,因此可通过DNA甲基化谱进行癌症的诊断及预后分析。肝癌是临床上常见的消化系统恶性肿瘤之一,早期阶段治疗比晚期阶段治疗具有更好的预后效果。2017年,Xu等[36]利用MIP技术捕获亚硫酸氢盐处理过的肝癌组织DNA及正常血液ctDNA,通过深度测序检测DNA甲基化水平,发现肝癌组织的DNA甲基化与血浆中的ctDNA甲基化具有强相关性,之后通过多种统计方法开发出了可用甲基化标记进行诊断和预后预测的模型。
此外,MIP技术还用于其他肿瘤的研究,如黑素瘤、Burkitt淋巴瘤、尤因肉瘤、白血病、滤泡树突状细胞肉瘤[37]、胆管癌[38]和胃癌[39]等,为这些肿瘤的发生、发展、诊断、治疗及预后研究作出了重要的贡献。
4.3 在其他疾病方面的研究乙型肝炎病毒(Hepatitis B virus,HBV)是一类严重危害人类身体健康的病原微生物,其耐药基因突变检测对指导临床抗病毒药物治疗具有重要意义。唱凯等[12]利用MIP技术对病毒HBV耐药基因突变位点进行检测,结果发现结合琼脂糖凝胶电泳的MIP技术检测结果与测序结果一致,并成功建立了检测HBV耐药基因单碱基突变的MIP技术,为以后检测HBV耐药基因单碱基突变提供重要的参考价值。近年来,糖尿病的患者越来越多,并且患病的人群中青少年呈大幅度增加,因此对于糖尿病的研究引起很多学者的关注。Smyth等[40]利用MIP技术结合芯片杂交分析1型糖尿病(T1D)易感区域,结果在染色体2q24.3上发现了一个新的T1D易感区域,此区域的一个基因KCNH7可能参与胰岛素的分泌过程。此外,研究发现人类的许多疾病是由RNA错剪接造成的,MIP技术同样可用来检测可变剪接位点,Bachmann-Gagescu等[41]通过该技术捕获了Joubert综合征相关的28个基因及编码纤毛发生所需的中心体蛋白KIAA0586基因的全外显子,并通过Illumina HiSeq测序,发现KIAA0586基因第23号外显子最后一对碱基发生同义突变,此突变使得该处出现隐蔽剪接位点,导致移码突变使蛋白翻译提前终止。
5 结语目标序列捕获是目前基因组学研究的一个热点,与全基因组测序相比,不仅降低了高通量测序的费用而且结果分析更加容易。MIP技术作为一种新的目标序列捕获技术,自2003年首次报道至今,已被各领域的学者或专家不断的改进和优化,捕获的片段长度从一个核苷酸增加到几百个核苷酸,检测范围也从SNP分型扩大到拷贝数变异、杂合性丢失、DNA甲基化及可变剪接等。MIP技术不仅在疾病研究方面应用广泛,在其他方面例如植物病原菌检测等领域也发挥重要的作用。尽管MIP技术的探针制备费用较昂贵,但核酸外切酶消化未反应的线性探针,减少了背景对实验结果的影响,其通量往往很高,同时能捕获成千上万个基因,对于大样本检测而言,费用较低。与其他目标序列捕获技术相比,MIP技术不仅所需要的DNA含量少、对样本的完整度要求不高,而且实验操作简单、时间也短。
纵然MIP技术经历了不断的发展和改进,但其仍然存在一些不足。研究发现,探针的捕获效率不超过90%,捕获的目的片段较短,甚至一些位点无法设计出探针及MIP技术结果分析存在欠缺。因此,具有较高捕获效率的探针和相应的分析软件还需要进一步设计和开发。另外,由于每个探针的工作效率不同,MIP文库的构建仍需优化。考虑到该技术在分子生物学的广泛应用,若在今后的研究中能够很好地克服以上的不足,该方法将为疾病诊断、分子病理学研究及疾病治疗等方面提供强有力的手段。
| [1] |
黄建锋, 肖华胜. 目标序列捕获技术及其应用[J]. 生物产业技术, 2011(4): 64-69. DOI:10.3969/j.issn.1674-0319.2011.04.006 |
| [2] |
Nilsson M, Malmgren H, Samiotaki M, et al. Padlock probes:circularizing oligonucleotides for localized DNA detection[J]. Science,, 1994, 265(5181): 2085. DOI:10.1126/science.7522346 |
| [3] |
Hardenbol P, Banér J, Jain M, et al. Multiplexed genotyping with sequence-tagged molecular inversion probes[J]. Nature Biotechnology, 2003, 21(6): 673-678. DOI:10.1038/nbt821 |
| [4] |
Ji H, Welch K. Molecular inversion probe assay for allelic quantitation[M]. Microarray Analysis of the Physical Genome: Methods and Protocols, 2009: 67-87.
|
| [5] |
Wang Y, Moorhead M, Karlin-Neumann G, et al. Analysis of molecular inversion probe performance for allele copy number determination[J]. Genome Biology, 2007, 8(11): R246. DOI:10.1186/gb-2007-8-11-r246 |
| [6] |
Akhras MS, Unemo M, Thiyagarajan S, et al. Connector inversion probe technology:a powerful one-primer multiplex DNA amplification system for numerous scientific applications[J]. PLoS One, 2007, 2(9): e915. DOI:10.1371/journal.pone.0000915 |
| [7] |
Turner EH, Lee C, Ng SB, et al. Massively parallel exon capture and library-free resequencing across 16 genomes[J]. Nature Methods, 2009, 6(5): 315. DOI:10.1038/nmeth.f.248 |
| [8] |
Bentley DR, Balasubramanian S, Swerdlow HP, et al. Accurate whole human genome sequencing using reversible terminator chemistry[J]. Nature, 2008, 456(7218): 53. DOI:10.1038/nature07517 |
| [9] |
Porreca GJ, Zhang K, Li JB, et al. Multiplex amplification of large sets of human exons[J]. Nature Methods, 2007, 4(11): 931. DOI:10.1038/nmeth1110 |
| [10] |
Hiatt JB, Pritchard CC, Salipante SJ, et al. Single molecule molecular inversion probes for targeted, high-accuracy detection of low-frequency variation[J]. Genome Research, 2013, 23(5): 843-854. DOI:10.1101/gr.147686.112 |
| [11] |
O'Roak BJ, Vives L, Fu W, et al. Multiplex targeted sequencing identifies recurrently mutated genes in autism spectrum disorders[J]. Science, 2012, 338(6114): 1619-1622. DOI:10.1126/science.1227764 |
| [12] |
唱凯, 贾双荣, 潘锋, 等. 基于分子倒置探针的乙型肝炎病毒耐药基因单碱基突变检测技术的建立[J]. 中华检验医学杂志, 2014(5): 337-341. DOI:10.3760/cma.j.issn.1009-9158.2014.05.006 |
| [13] |
Carrascosa LG, Sina AAI, Palanisamy R, et al. Molecular inversion probe-based SPR biosensing for specific, label-free and real-time detection of regional DNA methylation[J]. Chemical Communications, 2014, 50(27): 3585-3588. DOI:10.1039/C3CC49607D |
| [14] |
Wang Y, Cottman ME, Schiffman JD. Molecular inversion probes:a novel microarray technology and its application in cancer research[J]. Cancer Genetics, 2012, 205(7): 341-355. |
| [15] |
Wang T, Guo H, Xiong B, et al. De novo genic mutations among a Chinese autism spectrum disorder cohort[J]. Nature Communications, 2016, 7: 13316. DOI:10.1038/ncomms13316 |
| [16] |
Kou HS, Wang CC. Molecular inversion probes equipped with discontinuous rolling cycle amplification for targeting nucleotide variants:Determining SMN1 and SMN2 genes in diagnosis of spinal muscular atrophy[J]. Analytica Chimica Acta, 2017, 977: 65-73. DOI:10.1016/j.aca.2017.04.037 |
| [17] |
Ji H, Kumm J, Zhang M, et al. Molecular inversion probe analysis of gene copy alterations reveals distinct categories of colorectal carcinoma[J]. Cancer Research, 2006, 66(16): 7910-7919. DOI:10.1158/0008-5472.CAN-06-0595 |
| [18] |
Zhang J, Wang X, De Voer RM, et al. A molecular inversion probe-based next-generation sequencing panel to detect germline mutations in Chinese early-onset colorectal cancer patients[J]. Oncotarget, 2017, 8(15): 24533. |
| [19] |
Almendro V, Ametller E, García-Recio S, et al. The role of MMP7 and its cross-talk with the FAS/FASL system during the acquisition of chemoresistance to oxaliplatin[J]. PLoS One, 2009, 4(3): e4728. DOI:10.1371/journal.pone.0004728 |
| [20] |
Yokoyama Y, Grünebach F, Schmidt SM, et al. Matrilysin (MMP-7) is a novel broadly expressed tumor antigen recognized by antigen-specific T cells[J]. Clinical Cancer Research, 2008, 14(17): 5503-5511. DOI:10.1158/1078-0432.CCR-07-4041 |
| [21] |
Huo N, Ichikawa Y, Kamiyama M, et al. MMP-7 (matrilysin) accelerated growth of human umbilical vein endothelial cells[J]. Cancer Letters, 2002, 177(1): 95-100. DOI:10.1016/S0304-3835(01)00772-8 |
| [22] |
Beeghly-Fadiel A, Shu X, Long J, et al. Genetic polymorphisms in the MMP-7 gene and breast cancer survival[J]. International Journal of Cancer, 2009, 124(1): 208-214. DOI:10.1002/ijc.v124:1 |
| [23] |
Thompson PA, Brewster AM, Kim-Anh D, et al. Selective genomic copy number imbalances and probability of recurrence in early-stage breast cancer[J]. PLoS One, 2011, 6(8): e23543. DOI:10.1371/journal.pone.0023543 |
| [24] |
Brewster AM, Thompson P, Sahin AA, et al. Copy number imbalances between screen and symptom-detected breast cancers and impact on disease-free survival[J]. Cancer Prevention Research, 2011, 4(10): 1609. DOI:10.1158/1940-6207.CAPR-10-0361 |
| [25] |
Johnson CE, Gorringe KL, Thompson ER, et al. Identification of copy number alterations associated with the progression of DCIS to invasive ductal carcinoma[J]. Breast Cancer Research and Treatment, 2012, 133(3): 889-898. DOI:10.1007/s10549-011-1835-1 |
| [26] |
Huw LY, O'brien C, Pandita A, et al. Acquired PIK3CA amplification causes resistance to selective phosphoinositide 3-kinase inhibitors in breast cancer[J]. Oncogenesis, 2013, 2(12): e83. DOI:10.1038/oncsis.2013.46 |
| [27] |
Esserman LJ, Berry DA, Cheang MCU, et al. Chemotherapy response and recurrence-free survival in neoadjuvant breast cancer depends on biomarker profiles:results from the I-SPY 1 TRIAL (CALGB 150007/150012;ACRIN 6657)[J]. Breast Cancer Research and Treatment, 2012, 132(3): 1049-1062. DOI:10.1007/s10549-011-1895-2 |
| [28] |
Neveling K, Mensenkamp AR, Derks R, et al. BRCA testing by single-molecule molecular inversion probes[J]. Clinical Chemistry, 2017, 63(2): 503-512. DOI:10.1373/clinchem.2016.263897 |
| [29] |
Brown LA, Kalloger SE, Miller MA, et al. Amplification of 11q13 in ovarian carcinoma[J]. Genes, Chromosomes and Cancer, 2008, 47(6): 481-489. DOI:10.1002/gcc.v47:6 |
| [30] |
So WK, Cheng JC, Fan Q, et al. Loss of Sprouty2 in human high-grade serous ovarian carcinomas promotes EGF-induced E-cadherin down-regulation and cell invasion[J]. Febs Letters, 2015, 589(3): 302-309. DOI:10.1016/j.febslet.2014.12.012 |
| [31] |
Weren RDA, Mensenkamp AR, Simons M, et al. Novel BRCA1 and BRCA2 tumor test as basis for treatment decisions and referral for genetic counselling of patients with ovarian carcinomas[J]. Human Mutation, 2017, 38(2): 226-235. DOI:10.1002/humu.2017.38.issue-2 |
| [32] |
Alexiev BA, Zou YS. Clear cell papillary renal cell carcinoma:a chromosomal microarray analysis of two cases using a novel Molecular Inversion Probe (MIP) technology[J]. Pathology-Research and Practice, 2014, 210(12): 1049-1053. DOI:10.1016/j.prp.2014.10.001 |
| [33] |
Kim WY, Kaelin WG. Role of VHL gene mutation in human cancer[J]. Journal of Clinical Oncology, 2004, 22(24): 4991-5004. DOI:10.1200/JCO.2004.05.061 |
| [34] |
Hakimi AA, Reznik E, Lee CH, et al. An integrated metabolic atlas of clear cell renal cell carcinoma[J]. Cancer Cell, 2016, 29(1): 104-116. DOI:10.1016/j.ccell.2015.12.004 |
| [35] |
Bitter T, Water C, Heuvel C, et al. Profiling of the metabolic transcriptome via single molecule molecular inversion probes[J]. Scientific Reports, 2017, 7(1): 11402. DOI:10.1038/s41598-017-11035-0 |
| [36] |
Xu R, Wei W, Krawczyk M, et al. Circulating tumour DNA methylation markers for diagnosis and prognosis of hepatocellular carcinoma[J]. Nature Materials, 2017, 16(11): 1155. DOI:10.1038/nmat4997 |
| [37] |
Andersen EF, Paxton CN, O'Malley DP, et al. Genomic analysis of follicular dendritic cell sarcoma by molecular inversion probe array reveals tumor suppressor-driven biology[J]. Modern Pathology, 2017, 30(9): 1321. DOI:10.1038/modpathol.2017.34 |
| [38] |
Arnold A, Bahra M, Lenze D, et al. Genome wide DNA copy number analysis in cholangiocarcinoma using high resolution molecular inversion probe single nucleotide polymorphism assay[J]. Experimental and Molecular Pathology, 2015, 99(2): 344-353. DOI:10.1016/j.yexmp.2015.08.003 |
| [39] |
Weren RDA, van der Post RS, Vogelaar IP, et al. Role of germline aberrations affecting CTNNA1, MAP3K6 and MYD88 in gastric cancer susceptibility[J]. Journal of Medical Genetics, 2018: jmedgenet-2017-104962. https: //jmg.bmj.com/content/jmedgenet/55/10/669.full.pdf
|
| [40] |
Smyth DJ, Cooper JD, Bailey R, et al. A genome-wide association study of nonsynonymous SNPs identifies a type 1 diabetes locus in the interferon-induced helicase (IFIH1) region[J]. Nature Genetics, 2006, 38(6): 617-619. DOI:10.1038/ng1800 |
| [41] |
Bachmann-Gagescu R, Phelps IG, Dempsey JC, et al. KIAA0586 is mutated in Joubert syndrome[J]. Human Mutation, 2015, 36(9): 831-835. DOI:10.1002/humu.2015.36.issue-9 |