线粒体是细胞进行有氧呼吸产生ATP的场所,是细胞的“动力车间”,是一种半自主复制细胞器。粟酒裂殖酵母线粒体基因组能够编码25种tRNA、8种mRNA、2种rRNA及1个rnpB[1]。线粒体与人类健康有密切的联系,线粒体受损会引发诸如癌症,糖尿病,Leigh综合征,帕金森,婴儿猝死综合征等一系列疾病[2-4],大约有30%-40%的线粒体疾病是由于线粒体呼吸链酶缺陷[5-7]造成的,因此有关线粒体的研究越来越受到人们的关注。
线粒体复合体Ⅴ(ATP合酶)分为F1(α3β3γδε)和F0组分。粟酒裂殖酵母数据库Pombase预测Atp11(SPAC3A12.12)是F1-ATP酶的伴侣蛋白,参与ATP合酶的组装。粟酒裂殖酵母中Atp11具有两个同源蛋白,分别是芽殖酵母中的ATP11和人中的Atp11p。芽殖酵母中ATP11参与F1的组装蛋白,其中ATP11的缺失在非发酵型培养基中出现生长呼吸受损,这是由于线粒体F1组装缺陷导致的[8]。并且免疫共沉淀和酵母双杂交实验证明ATP11与F1中β亚基的结合位点在Gly114和Leu318核苷酸结构域之间[9]。Δatp11突变体中除了ATPase的含量减少,还降低了细胞色素氧化酶和辅酶QH2-细胞色素c还原酶的浓度[10]。在人中Atp11p是管家蛋白,参与F1-ATPase的组装并结合β亚基位点[11]。研究表明Atp11p保护胰岛素B链免于在体外聚集,因此充当分子伴侣[12]。ATPAF1是儿童患有哮喘病的易感基因,同时患有阿尔茨海默病的患者样品中ATP合酶的量相对于正常群体中明显减少[13]。因此,ATP11与线粒体功能有着密不可分关系。
芽殖酵母中ATP11的功能已经研究的比较透彻,但粟酒裂殖酵母中Atp11的功能还没有被报道。因此,本研究旨在探究粟酒裂殖酵母中Atp11参与线粒体功能的机制,为进一步研究粟酒裂殖酵母线粒体蛋白的功能提供一定的理论依据。
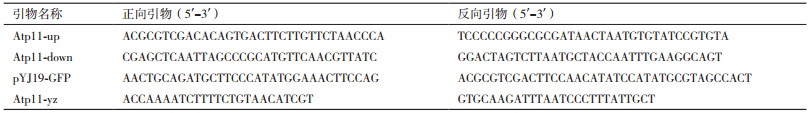
1 材料与方法 1.1 材料 1.1.1 菌株与质粒粟酒裂殖酵母单倍体菌株yHL6381;大肠杆菌E.coli Top10;质粒pFA6a-hphMX6,pYJ19(图 1)为本实验室保存。
|
| 图 1 pFA6a-hphMX6及pYJ19图谱 |
YES培养基:0.5%酵母粉,3%葡萄糖,4种微量元素(adenine、histidine、uracil和leucine),固体培养基添加2%的琼脂粉;YES+3%甘油或YES+6%甘油:将YES培养基中的葡萄糖替换成甘油;YES+hph(潮霉素):YES固体培养基添加潮霉素至终浓度为100 mg/mL。
基础培养基EMM-leucine(100 mL):potassium hydrogen phthalate 0.3 g,Na2HPO4·12H2O 0.555 g,NH4Cl 0.5 g,1 000 × vitamin 1 mL,10 000 × mineral stock 0.01 mL,50 × salt stock 2 mL,Agar Powder 2 g,glucose 2 g;3种微量元素:adenine、histidine和uracil各22.5 mg。其中glucose分开灭菌以防止碳化。
1.1.3 主要试剂PrimeSTAR高保真聚合酶等PCR反应试剂、限制性内切酶、连接酶均购自TaKaRa公司;PCR引物由上海英骏生物有限公司合成;DNA Marker购自南京百思凯;DNA纯化试剂盒以及割胶回收试剂盒购自博巧生物科技公司;SDS、HEPES、Sorbitol、Tris、30%丙烯酰胺购自上海生工。
1.1.4 主要仪器PCR仪;台式高速离心机;Odyssey双色红外激光成像系统;电泳槽。
1.2 方法 1.2.1 引物设计利用DNAMAN软件设计引物,见表 1。
粟酒裂殖酵母atp11基因序列来源于酵母基因组数据库(S.pombe_GeneDB,http://www.pombase.org/);粟酒裂殖酵母Atp11及其人的同源蛋白Atp11p的序列来源于NCBI数据库(http://www.ncbi.nlm.nih.gov/);芽殖酵母中同源蛋白ATP11的序列来源于SGD数据库(http://www.yeastgenome.org/);利用NCBI Protein BLAST进行蛋白序列同源性比对分析;用MitoProt Ⅱ(http://ihg.gsf.de/ihg/mitoprot.html)分析线粒体信号肽。
1.2.3 载体构建及鉴定atp11-del-hphMX6重组质粒的构建:以野生型菌株yHL6381基因组为模板,以Atp11-up及Atp11-down为引物分别扩增得到atp11的基因5'端上游同源臂(528 bp)和3'端下游同源臂(533 bp),分别将其构建在载体pFA6a-hphMX6(图 1-A)的Sal Ⅰ/Sma Ⅰ和Sac Ⅰ/Spe Ⅰ位点,并进行双酶切验证。
Atp11-GFP重组质粒的构建:以野生型菌株yHL6381基因组为模板和pYJ19-GFP为引物通过PCR扩增得到atp11基因目的片段,将其构建到载体pYJ19(图 1-B)的Pst Ⅰ/Sal Ⅰ位点上,从而实现在C端添加GFP标签,并进一步进行酶切验证。
1.2.4 酵母醋酸锂转化[14]及菌株鉴定以atp11-del-hphMX6重组质粒为模板,Atp11-up的正向引物和Atp11-down的反向引物为引物,扩增目的片段,通过醋酸锂转化导入yHL6381菌株中,并在YES+hph(潮霉素)抗性平板上筛选;重组质粒Atp11-GFP经Nru Ⅰ线性化,获得目的片段,醋酸锂转化导入yHL6381菌株中,涂布于EMM-leucine平板上;目的片段通过同源重组的方法整合在酵母基因组上。菌株鉴定:以yHL6381基因组和挑取的醋酸锂转化子基因组为模板,再以Atp11-yz为引物进行PCR验证;Atp11-GFP菌株通过荧光显微镜观察进行验证。
1.2.5 Δatp11突变体表型研究挑取yHL6381菌株和Δatp11突变体于YES液体培养基中培养至对数期,将细胞依次按10倍梯度稀释,分别点至YES,YES+3%甘油,YES+6%甘油平板上,30℃恒温培养5 d,拍照。
1.2.6 GFP荧光定位实验接种Atp11-GFP菌株于EMM-leucine液体培养基中培养,收菌。用1×PBS洗涤两次之后,在30℃与10 μmol/L MitoTracker染料孵育1-2 min;制片,观察。
1.2.7 Western blotting提取yHL6381和Δ atp11菌株的线粒体[15],通过SDS-PAGE聚丙烯凝胶电泳分离蛋白质,转膜。用抗体检测:一抗:anti-Cob1(1/1 000),anti-Cox1(1/2 000),anti-Cox2(1/300),anti-Cox3(1/300),anti-Atp6(1/2 000),anti-Hsp60(1/2 000,作为内参)。二抗:IRDye800CW羊抗兔抗体。Odyssey双色红外激光成像系统检测结果。
2 结果 2.1 重组质粒验证与菌株鉴定以野生型基因组yHL6381为模板扩增atp11基因的上游同源臂和下游同源臂大小分别为528 bp和533 bp(图 2-A);重组质粒atp11-del-hphMX6和Atp11-GFP构建成功(图 2-B-D)。

|
| 图 2 重组质粒的构建及酶切验证 M:DNA Marker分子量标准(100 bp-8 kb);A:1-4号泳道为atp11上游同源臂PCR(528 bp)扩增结果,5-8号泳道为atp11下游同源臂PCR(533 bp)扩增结果;B:1号泳道为sal Ⅰ和sma Ⅰ对atp11-del-hphMX6上游同源臂双酶切验证;C:1和2号泳道为Sac Ⅰ和Spe Ⅰ对atp11-del-hphMX6下游同源臂双酶切验证;D:1号泳道是Pst Ⅰ和Sal Ⅰ对重组质粒Atp11-GFP双酶切验证 |
同源重组的结果为在裂殖酵母基因组上抗性筛选标记hph潮霉素基因(1651 bp)替代了atp11(861 bp)基因,PCR验证结果(图 3-A)显示Δatp11突变体构建成功。另外,Atp11-GFP菌株在荧光显微镜下观察到绿色荧光,说明Atp11-GFP菌株构建成功(图 3-B)。

|
| 图 3 敲除菌Δatp11和Atp11-GFP的验证 A:1号泳道为以yHL6381基因组为模板扩增得到的目的片段(2016 bp);2号泳道为以敲除菌基因组扩增得到的目的片段(2784 bp);B:通过荧光显微镜观察鉴定菌株Atp11-GFP |
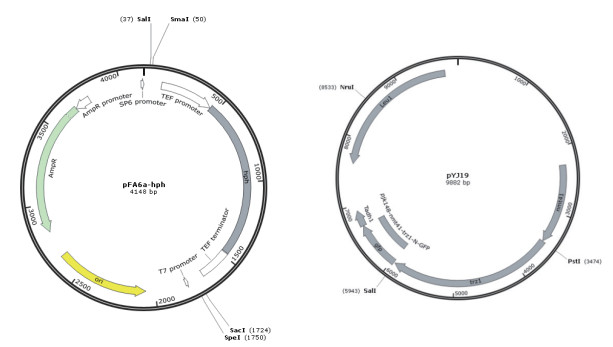
为了研究Atp11的生物学功能,通过构建Δatp11突变体,在以甘油为唯一碳源的非发酵型培养基中来培养yHL6381和Δatp11菌株。结果(图 4)表明,一方面,在以葡萄糖为碳源的YES培养基上,yHL6381和Δ atp11突变体生长状态一致,此时,它们可以利用葡萄糖进行无氧呼吸,不依赖于线粒体。另一方面,在以甘油为碳源的非发酵型培养基上,相比于野生型菌株,Δatp11敲除菌的生长受到严重的抑制。这是由于在非发酵型培养基上,细胞只能进行有氧呼吸,依赖于线粒体产生ATP。因此Atp11对于线粒体功能是必不可少的。
|
| 图 4 Δatp11在非发酵培养基上的生长情况 |
atp11基因缺失之后影响了线粒体功能,暗示Atp11蛋白很有可能进入线粒体行驶功能。生物信息学分析结果显示,Atp11的N端有一段由31个氨基酸残基组成的线粒体定位序列(MLPIWKLPVNH-LLCHSFKSIPRT),可信度高达90.61%。
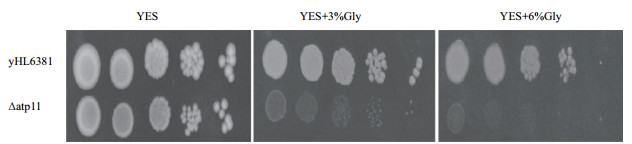
利用线粒体染料来确定Atp11的定位,线粒体marker MitoTracker Red是一种用于染活细胞线粒体的染料,在荧光显微镜Red通道下发出红色荧光。Atp11-GFP能够表达绿色荧光蛋白,在GFP通道下发绿色荧光。如图 5所示,同一视野中绿色荧光和红色荧光重叠后发出橙色荧光,这就证实Atp11确实定位在线粒体,结合前期表型实验,再次证实Atp11蛋白与线粒体功能密切相关。
|
| 图 5 Atp11-GFP的荧光显微镜观察 MitoTracker:红色荧光;GFP:绿色荧光蛋白;Merge:Red和GFP共定位 |
通过Western blotting技术检测atp11缺失之后对线粒体基因组编码的蛋白稳态期水平的影响。结果(图 6)显示Δatp11突变体中,Cob1、Cox1、Cox2、Cox3和Atp6蛋白水平相对于野生型菌株显著降低,Δatp11突变体中几乎没有mt-DNA编码蛋白质的残留。Cox1、Cox2和Cox3参与组成呼吸链复合体Ⅳ,Atp6参与组成复合体Ⅴ。线粒体呼吸链复合体组装受损,导致Δatp11突变体不能在非发酵型培养基上正常的进行氧化磷酸化,从而表现出呼吸缺陷的生长状态。因此,Atp11对线粒体呼吸链复合体亚基的组装是必需的,对线粒体呼吸链功能的发挥起重要作用。

|
| 图 6 Western blotting检测Δatp11菌中线粒体基因组编码的蛋白质表达量 |
线粒体呼吸链包含多种线粒体呼吸链酶,如NADH脱氢酶、琥珀酸氧化还原酶、细胞色素c还原酶、细胞色素c氧化酶和ATP合成酶。Cob1由细胞色素c还原酶编码,Cox1、Cox2和Cox3由细胞色素c氧化酶编码,而Atp6则由ATP合成酶编码。线粒体呼吸链酶经过一系列的氧化还原过程最终形成ATP,为肌体提供能量。如果线粒体呼吸链酶合成受损,将会引起线粒体功能缺陷,从而引发线粒体相关疾病。
已知芽殖酵母中敲除ATP11会在非发酵型培养基中出现生长呼吸受损现象。通过蛋白序列比对发现芽殖酵母中的ATP11蛋白与裂殖酵母中的Atp11同源性约为38%。因此,本研究在粟酒裂殖酵母中构建Δatp11菌株,发现Δatp11在非发酵培养中表现出呼吸链受损生长;进一步研究得出Atp11定位于线粒体中,从而推测Atp11可能与线粒体基因组编码蛋白有关。通过Western blotting实验证明了Atp11会影响Cob1、Cox1、Cox2、Cox3和Atp6蛋白稳态水平。虽然在Δatp11菌中蛋白的稳态水平显著降低了,但Atp11是影响这些蛋白的转录、合成还是蛋白的稳定,有待进一步研究。
另外,芽殖酵母ATP11主要序列分为4个结构域:括线粒体前导序列(残基1-39),N端结构域(NTD,残基40-111),功能结构域(残基112-183)和C末端结构域(残基184-318)。Atp11功能结构域位于苯丙氨酸-120和天冬氨酸-174序列之间,并且这段功能结构域对是维持线粒体内蛋白质的稳定性是非常重要的[16]。而裂殖酵母中的Atp11p含有一个ATP超家族结构域,这个结构域是否对线粒体中蛋白质的稳定性起作用目前还不清楚。接下来可以通过设计相关的实验来研究Atp11p在线粒体中发挥的作用。
4 结论粟酒裂殖酵母atp11基因大小为861 bp,共编码286氨基酸残基。本文主要研究芽殖酵母ATP11的同源蛋白Atp11。敲除atp11构建突变体菌株,发现Δatp11在非发酵培养基上表现出呼吸缺陷型生长,说明Δatp11菌是线粒体功能缺陷菌。通过荧光定位实验结果得出Atp11定位于线粒体中,这进一步说明Atp11是在线粒体中发挥作用。最后,Western blotting实验证明了Atp11的缺失会导致复合体Ⅲ的Cob1、复合体Ⅳ的部分亚基Cox1,Cox2,Cox3、复合物Ⅴ中的Atp6的蛋白表达水平显著降低。复合体亚基组装受阻导致线粒体呼吸功能受损而引发在甘油培养基上不能正常生长。
| [1] |
Kuhl I, Dujeancourt L, Gaisne M, et al. A genome wide study in fission yeast reveals nine PPR proteins that regulate mitochondrial gene expression[J]. Nucleic Acids Research, 2011, 39(18): 8029-8041. DOI:10.1093/nar/gkr511 |
| [2] |
Debray FG, Lambert M, Allard. P, et al. Low citrulline in Leigh disease:still a biomarker of maternally inherited Leigh syndrome[J]. Child Neurol, 2010, 25(8): 1000-1002. DOI:10.1177/0883073809351983 |
| [3] |
Jorstad ØK, Ødegaard EM, Heimdal KR, et al. Leber hereditary optic neuropathy caused by a mitochondrial DNA 10663T > C Point mutation and its response to idebenone treatment[J]. Journal of Neuroradiology, 2018, 38(1): 129-131. |
| [4] |
Larsen SB, Hanss Z, Krüger R. The genetic architecture of mitochondrial dysfunction in Parkinson's disease[J]. Cell Tissue Research, 2018, 373(1): 21-37. DOI:10.1007/s00441-017-2768-8 |
| [5] |
Hatakeyama H, Goto YI. Respiratory chain complex disorganization impairs mitochondrial and cellular integrity:Phenotypic variation in cytochrome c oxidase deficiency[J]. American Journal of Pathology, 2017, 187(1): 110-121. DOI:10.1016/j.ajpath.2016.09.003 |
| [6] |
Mayr JA, Haack TB, Freisinger P, et al. Spectrum of combined respiratory chain defects[J]. Journal of inherited metabolic disease, 2015, 38(4): 629-640. DOI:10.1007/s10545-015-9831-y |
| [7] |
Roloff GA, Henry MF. Mam33 promotes cytochrome c oxidase subunit Ⅰ translation in Saccharomyces cerevisiae mitochondria[J]. J Mol Biol Cell, 2015, 26(16): 2885-2894. DOI:10.1091/mbc.e15-04-0222 |
| [8] |
Ackerman SH, Martin J, Tzagoloff. A. Characterization of ATP11 and detection of the encoded protein in mitochondria of Saccharomyces cerevisiae[J]. J Biol Chem, 1992, 267(11): 7386-7394. |
| [9] |
Wang ZG, Ackerman SH. The assembly factor Atp11p binds to the beta-subunit of the mitochondrial F(1)-ATPase[J]. J Biol Chem, 2000, 275(8): 5767-5772. DOI:10.1074/jbc.275.8.5767 |
| [10] |
Ackerman SH, Tzagoloff A. Identification of two nuclear genes(ATP11, ATP12)required for assembly of the yeast F1-ATPase[J]. Proc Natl Acad Sci USA, 1990, 87(13): 4986-4990. DOI:10.1073/pnas.87.13.4986 |
| [11] |
Wang ZG, White PS, Ackerman SH. Atp11p and Atp12p are asse-mbly factors for the F(1)-ATPase in human mitochondria[J]. J Biol Chem, 2001, 276(33): 30773-30778. DOI:10.1074/jbc.M104133200 |
| [12] |
Hinton A, Zuiderweg ER, Ackerman SH. A purified subfragment of yeast Atp11p retains full molecular chaperone activity[J]. J Biol Chem, 2003, 278(36): 34110-34113. DOI:10.1074/jbc.M305353200 |
| [13] |
Schauberger EM, Ewart SL, Arshad SH, et al. Identification of ATPAF1 as a novel candidate gene for asthma in children[J]. J Allergy Clini Immunol, 2011, 128(4): 753-760. DOI:10.1016/j.jaci.2011.04.058 |
| [14] |
Gietz RD, Schiestl RH. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J]. Nat Protocols, 2007, 2(1): 31-4. DOI:10.1038/nprot.2007.13 |
| [15] |
Maheshwari KK, Marzuki S. Defective assembly of the mitochondrial ribosomes in yeast cells grown in the presence of mitochondrial protein synthesis inhibitors[J]. Biochim Biophys Acta, 1985, 824(4): 273-283. DOI:10.1016/0167-4781(85)90033-8 |
| [16] |
Wang ZG, Ackerman SH. Identification of functional domains in Atp11p Protein required for assembly of the mitochondrial F1-ATPase in yeast[J]. J Biol Chem, 1996, 271(9): 4887-4894. DOI:10.1074/jbc.271.9.4887 |