随着有机磷农药长久和广泛的使用,不仅污染了土壤和水源,破坏自然界的生态系统平衡,同时大量的残留在瓜果蔬菜及动物性食品上的有机磷农药对人体产生很多不良的影响[1]。有机磷降解酶是一种高效,低毒,无残留的生物绿色催化剂,是消除农药残留最有潜力的新方法[2]。
由于甲基对硫磷水解酶可水解最常见有机磷农药之一,甲基对硫磷,这使该酶成为生物降解领域的主要研究对象。本实验所关注的甲基对硫磷水解酶MPH源于Pseudomonas sp.WBC-3[3]。通过对MPH晶体结构的解析发现[4],MPH是一种金属酶,在催化活性中心具有含有两个二价金属离子,并通过活性中心内部的关键残基His147、His149、Asp151、His152、His234、His302和Asp255催化底物的水解。但是MPH水解效率较低,如何提高该酶的催化效率是本项研究的核心问题。
目前对酶的改造策略多种多样,例如,定向进化[5-7]、计算机辅助设计[8-10]、酶的全新设计[11-13]等,但是酶活性中心均为改造的关键区域。酶的活性中心有底物结合和提供反应基团的关键残基。在引入新的催化活性或改变酶的专一性方面,靠近活性中心的位置突变所产生的影响明显高于远离活性中心的位置[14]。活性中心附近的很多位点都是高保守性的。蛋白质的高保守序列是在中性进化的过程中极少积累突变的序列[15],而且高保守序列往往是蛋白质的功能区域[16-18],一旦高保守序列发生突变极有可能导致蛋白质的功能丧失。而低保守的序列经过高频率的突变和正向的选择很有可能出现有利的突变[15],因此选择低保守序列进行高通量筛选更能提高定向进化的效率。本实验利用酶的分子结构信息,计算距离活性中心较近的位点;并结合每个残基的生物信息学进化保守性[19],进一步限定突变区域的范围,提高了正向突变株的筛选效率。
1 材料与方法 1.1 材料 1.1.1 菌株及质粒大肠杆菌(Escherichia coli)BL21以及pET-28a质粒购于Novagen公司。含有甲基对硫磷水解酶基因的重组质粒pET-28a-mpd由本实验室保藏。
1.1.2 主要试剂甲基对硫磷购自AccuStandard公司。PCR所用的酶购自TaKaRa的HS PrimeSTAR。测定蛋白质浓度的试剂为碧云天公司生产的BCA蛋白浓度测定试剂盒。
1.2 方法 1.2.1 文库的构建与筛选定点饱和突变以pET-28a-mpd为模板,利用软件Primer 5.0设计简并引物进行定点饱和突变,简并引物如表 1。
PCR体系:5× GXL loading buffter 10 μL;dNTP 4 μL;上下游引物各1 μL;模板(10 ng/ μL)0.5 μL;GXL polymerase 1 μL;加入无菌水至50 μL。PCR条件为:95℃ 3 min;98℃ 10 s;55℃ 15 s;68℃ 1 kb/min;68℃ 10 min。PCR产物经过胶回收后,使用Dpn Ⅰ酶消除模板。之后采用T4 Polynucleotide Kinase试剂盒进行磷酸化,加入T4连接酶使重组载体自连。转化至E. coli BL21感受态细胞中,筛选阳性克隆并测序,突变率在75%以上即可使用。
据公式Pi=100[1-exp(-T×Fi)][20],Pi是某一突变点i突变体的覆盖率,T是需要挑选的克隆子,Fi是某一突变点i在文库中出现的频率。在引物中NNK,Fi=1/4×1/4×1/2=1/32,当覆盖率达到94%时需要挑88个阳性克隆子。
挑取阳性转化子接入每孔含有400 μL TB液体培养基的96深孔培养板,同时预留两个孔作为对照,一孔接入野生酶重组菌株,一孔不接入任何菌株。37℃,800 r/min培养至OD600=0.8时,每孔再加入4 μL的异丙基-β-D-硫代半乳糖苷(IPTG),25℃诱导12 h。发酵结束后,每孔加入10 mg/mL的溶菌酶37℃处理2 h,冻融一次。4 800 r/min,10 min离心。吸取2 μL上清液至96微孔板中,其中每孔加入190 μL Tris-HCl缓冲液(含250 μmol/L的甲基对硫磷),37℃反应5 min。测定吸光值A405,筛选酶活力较高的菌株并保藏。对初筛的菌株进行进一步的复筛,摇瓶诱导表达、纯化、测定其比酶活力。
1.2.2 MPH及其突变体表达纯化从保藏管中转接50 μL的突变体菌株至含有20 mL LB培养基的摇瓶中,37℃、200 r/min培养8 h。按1%的接种量接种至含有100 mL TB培养基的500 mL摇瓶中,37℃、200 r/min振荡培养至OD600=0.8。按1%的添加量加入IPTG,25℃、200 r/min诱导表达24 h。离心获得菌体,破壁。使用镍柱亲和纯化,分别用60 μmol/L和75 μmol/L浓度的咪唑进行洗杂,再用100 μmol/L浓度的咪唑冲洗获得目的蛋白。
1.2.3 酶活力测定酶活力单位定义:在一定条件下,酶催化甲基对硫磷(MP)每分钟释放出1 μmol无机磷所需要的酶量为一个酶活力单位(U)[21]。
酶活测定方法:在96微孔板中依次加入20 μL酶液,180 μL含有125 μmol/L MP的Tris-HCl(20 mmol/L、pH8.0)缓冲液,立即将96微孔板放入多功能酶标仪中进行测量。MPH水解甲基对硫磷的产物对硝基苯酚,在波长405 nm有最大的吸收值,检测波长设为405 nm,温度设置37℃,每隔15 s读取一次数据。选取酶的一级反应的阶段,计算酶活。
1.2.4 动力学参数的测定取20 μL样品至96微孔板,之后吸取180 μL含不同MP浓度的Tris-HCl缓冲液[22](20 μmol/L、pH8.0),混合溶液中底物浓度范围为30 μmol/L、50 μmol/L、75 μmol/L、100 μmol/L和125 μmol/L。在37℃,测定酶的反应过程。选定酶反应的一级阶段绘制双倒数曲线,通过米氏方程求得动力学参数。
1.2.5 最适反应pH和最适反应温度的测定在pH5.0-12.0范围内测到酶的最适pH值,不同pH值对应的缓冲液(20 μmol/L)为:pH5.0-6.0乙酸钠-HCl缓冲液、pH6.0-9.0 Tris-HCl缓冲液、pH9.0-12.0甘氨酸-NaOH缓冲液。测到酶的最适反应温度时,不同温度分别为30、35、40、45、50、55、60和65℃,其他条件均与酶活测到方法一致。
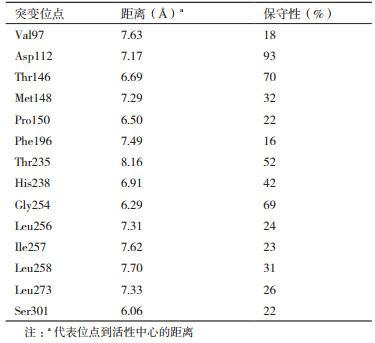
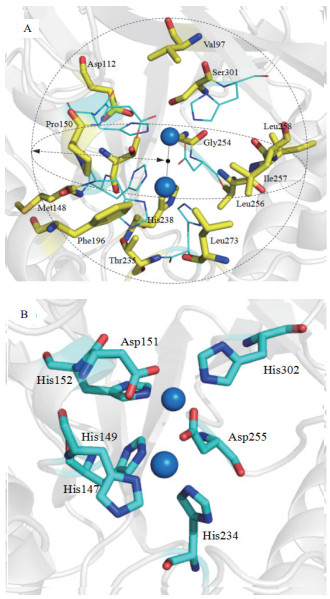
2 结果 2.1 突变位点的确定利用MPH的结构信息(PDB:1P9E),催化活性中心具有含有两个二价金属离子,并通过活性中心内部的关键残基His147、His149、Asp151、His152、His234、His302、Asp255催化底物的水解(图 1-B)。首先根据活性中心两个金属离子的坐标,计算出两个金属离子中心点的坐标,中心点的坐标为1.899、40.1325、25.138。
|
| 图 1 甲基对硫磷水解酶的三维结构图(1P9E) A:距离活性中心的金属离子中点8Å内残基结构图;B:甲基对硫磷水解酶的活性中心示意图 |
利用公式: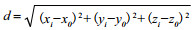
通过软件EVcoupling计算出在进化中每个残基的保守性分数[20],选取保守性低于40%的氨基酸残基作为定点饱和突变的位点:Val97、Met148、Pro150、Phe196、Leu256、Ile257、Leu258、Leu273和Ser301等9个位点(表 2)。
以pET-28a-mpd为模板,采用引物(表 1)进行扩增。PCR扩增片段为整个重组质粒,mpd片段长度996 bp,pET-28a片段长度5 369 bp,PCR扩增目的片段长度约6.3 kb。图 2中片段大小与理论值一致,并通过测序验证正确。
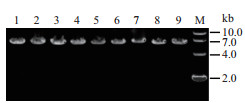
|
| 图 2 全质粒定点突变PCR产物电泳图 M:10.0kb Marker 1-9:分别是Val97、Met148、Pro150、Phe196、Leu256、Ile257、Leu258、Leu273、Ser301位点 |
根据公式,当覆盖率达到94%时需要挑88个阳性克隆子。从构建的突变体文库中随机挑选10个克隆进行测序,其突变率为70%以上则可使用。为了确保其碱基覆盖率,每个突变位点挑取150株克隆子进行初筛。经过第一轮的初筛,从1 500株中得到38株正向突变(图 3)。
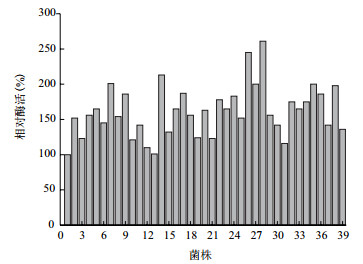
|
| 图 3 MPH突变体文库的初筛 |
为了进一步准确测定MPH突变体的催化效率,需要获得比较纯的酶蛋白。通过IPTG诱导、超声波破碎、离心、镍柱纯化得到MPH及其突变体较纯的目的蛋白。MPH理论分子量为32.3 kD,本实验纯化所得目的蛋白在29.0 kD和44.3 kD之间有一条清晰的条带(图 4)。通过与对照比较,确定纯化获得的蛋白为目的蛋白。
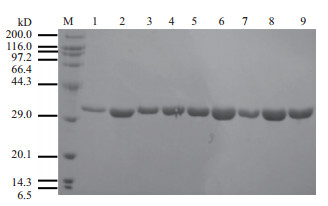
|
| 图 4 纯化后MPH及其突变体的SDS-PAGE图谱 M:Marker;1:野生型MPH;2-9:突变体 |
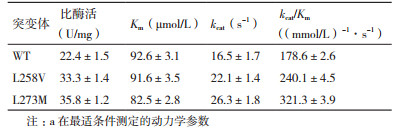
经过复筛,从初筛得到的38株中筛选到了2株比酶活力明显提高,分别是L258V和L273M。L258V、L273M的比酶活力分别是19.8 U/mg和21.1 U/mg,而野生型MPH的比酶活力为14.13 U/mg。L258V、L273M的比酶活力分别较野生型分别提高了1.4和1.5倍(图 5)。

|
| 图 5 MPH突变体文库的复筛 |
与野生型MPH相比,突变体的最适反应pH发生了一定的变化(图 6-A)。当pH < 10.0时,野生型MPH的酶活一直呈上升趋势。到达最高值之后开始缓慢下降,pH8.0-12.0时,酶活均较高,说明该酶在碱性条件具有较高酶活性。突变后,突变体L258V的最适反应pH由野生酶的10.0升高至11.0,突变体呈现了更偏碱的趋势。但突变体L273M的最适反应pH由野生酶的10.0降低至9.0,说明该突变促使酶在酸性条件下具有更高的酶活。
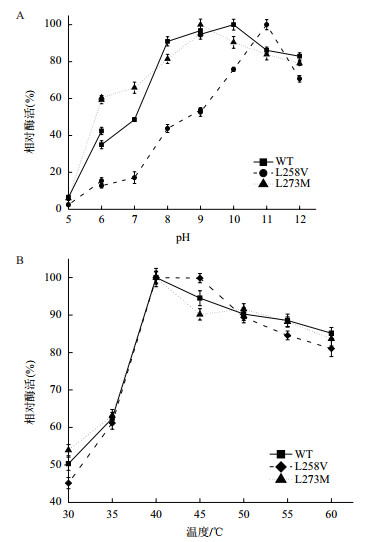
|
| 图 6 L258V突变和L273M突变对MPH的最适反应pH(A)和最适反应温度(B)的影响 |
如图 6-B所示,L258V和L273M突变对MPH的最适反应温度没有影响。野生型MPH的最适反应温度为40℃,当反应温度为30-40℃时,野生型MPH的酶活呈快速上升趋势,但当温度高于45℃时,酶活开始下降。突变后,突变体L258V和L273M的最适反应温度均为40℃,与野生酶相比较没有变化。
2.3.2 L258V突变和L273M突变对MPH的动力学参数的影响测定了相同蛋白浓度下野生型MPH及两个突变体以甲基对硫磷为底物的反映过程曲线(图 7)。与野生型相比,突变体L258V和L273M的初始反应速率有了显著的提高,分别提高了48.9%和58.1%。
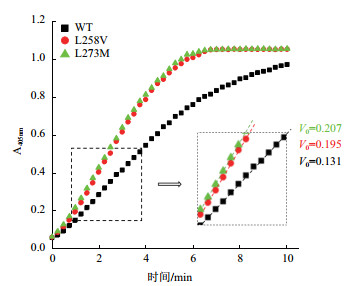
|
| 图 7 野生型MPH及其突变体反应过程曲线 |
MPH突变体L258V和L273M的催化效率相比野生型有明显的提高。野生型MPH的kcat/Km值是105.3(mmol/L)-1·s-1,L258V和L273M的kcat/Km值分别是134.1(mmol/L)-1·s-1和166.5(mmol/L)-1·s-1,这两个突变体的催化效率kcat/Km值分别提高了27.3%和58.1%(表 3,表 4)。
随着分子技术的发展,按照人类的需要对酶进行分子改造成为大家关注的热点。Arnold[23]在20世纪90年代年初期提出了定向进化的概念,定向进化技术由于简单易行,是目前最广泛应用的酶改造方法。但此方法需要构建庞大的突变体文库进行筛选,且只有极少数的突变体是有益的突变,因此其工作效率较低。理性设计需要对酶的晶体结构和催化机理有深入的了解,对改造前期工作要求很高。虽然理性设计和非理性设计(定向进化)都可以有效的改变酶的性能,但是两者结合起来改变酶的特性将代表着未来的发展方向[24]。
本研究通过基于甲基对硫磷水解酶的分子结构及其生物信息学等多维度特征的定向进化,筛选得到两株催化效率明显提高的突变体L258V和L273M。由于甲基对硫磷水解酶的活性中心处于疏水环境中,周围被7个疏水残基包围。包括:Leu65、Leu67、Phe119、Trp179、Phe196、Leu258和Leu273。Dong[4]使用低温保护剂甘油分子与MPH建模,发现甘油分子与金属活性中心周围的疏水残基构成疏水接触,说明这些疏水残基可能对底物构成空间位阻或与底物形成氢键,在底物与酶结合时限定底物取向,进而影响酶与底物结合的方式以及反应方式。L258V和L273M是构成活性中心口袋的一部分且距离酶的活性中心较近(图 1-A),因此推测其突变对催化活性中心区域产生轻微的影响,进而影响酶与底物的反应,促使其催化效率提高。
随着蛋白质测序技术不断发展,在数据库中已积累了大量蛋白质同源序列的大数据。基于分子进化理论,利用生物信息学手段分析同源序列,限定酶的改造位点,缩小筛选范围,是酶工程的新思路。同时增加了我们对于酶的关键位点的进一步理解,对以后酶的工程改造和对数据库中的新的蛋白序列的功能预测提供一些参考。
4 结论通过计算活性中心附近的位点并进行同源序列比对,选取距离活性中心8 Å以内的9个低保守性位点进行定向进化。经过两轮筛选,从1500株突变体中获得2株催化效率明显提高的突变体L258V和L273M。
| [1] |
俞发荣, 李登楼. 有机磷农药对人类健康的影响及农药残留检测方法研究进展[J]. 生态科学, 2015, 34(3): 197-203. |
| [2] |
虞云龙, 盛国英, 傅家谟. 一株农药降解菌的分离与鉴定[J]. 华南理工大学学报, 1996(S1): 192-196. |
| [3] |
陈亚丽, 张先恩, 刘虹, 等. 甲基对硫磷降解菌Pseudomonas sp. WBC-3的筛选及其降解性能的研究[J]. 微生物学报, 2002, 42(4): 490-497. DOI:10.3321/j.issn:0001-6209.2002.04.017 |
| [4] |
Dong YJ, Bartlam M, Sun L, et al. Crystal structure of methyl parathion hydrolase from Pseudomonas sp. WBC-3[J]. Journal of Molecular Biology, 2005, 353(3): 655-663. DOI:10.1016/j.jmb.2005.08.057 |
| [5] |
Carr R, Alexeeva M, Dr AE, et al. Directed evolution of an amine oxidase possessing both broad substrate specificity and high enantioselectivity[J]. Angewandte Chemie, 2003, 42(39): 4807-4810. DOI:10.1002/(ISSN)1521-3773 |
| [6] |
Kim JH, Choi GS, Kim SB, et al. Enhanced thermostability and tolerance of high substrate concentration of an esterase by directed evolution[J]. Journal of Molecular Catalysis B Enzymatic, 2004, 27(4): 169-175. |
| [7] |
Böttcher D, Bornscheuer UT. Protein engineering of microbial enzymes[J]. Current Opinion in Microbiology, 2010, 13(3): 274-282. DOI:10.1016/j.mib.2010.01.010 |
| [8] |
Dwyer MA, Looger LL, Hellinga HW. Computational design of a biologically active enzyme[J]. Science, 2004, 304(5679): 1967-1971. DOI:10.1126/science.1098432 |
| [9] |
Khoury GA, Fazelinia HChin JW. Computational design of candida boidinii xylose reductase for altered cofactor specificity[J]. Protein Science, 2009, 18(10): 2125-2138. DOI:10.1002/pro.v18:10 |
| [10] |
Chen CY, Georgiev I, Anderson AC, et al. Computational structure-based redesign of enzyme activity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(10): 3764-3769. DOI:10.1073/pnas.0900266106 |
| [11] |
Röthlisberger D, Khersonsky O, Wollacott AM, et al. Kemp elimination catalysts by computational enzyme design[J]. Nature, 2008, 453(7192): 190-195. DOI:10.1038/nature06879 |
| [12] |
Jiang L, Althoff EA, Clemente FR, et al. De novo computational design of retro-aldol enzymes[J]. Science, 2008, 319(5868): 1387-1391. DOI:10.1126/science.1152692 |
| [13] |
Siegel JB, Zanghellini A, Lovick HM, et al. Computational design of an enzyme catalyst for a stereoselective bimolecular diels-alder reaction[J]. Science, 2010, 329(5989): 309-313. DOI:10.1126/science.1190239 |
| [14] |
Toscano MD, Woycechowsky KJ, Hilvert D. Minimalist active-site redesign:teaching old enzymes new tricks[J]. Angewandte Chemie International Edition, 2007, 46(18): 3212-3236. DOI:10.1002/(ISSN)1521-3773 |
| [15] |
Ponting CP. Biological function in the twilight zone of sequence conservation[J]. Bmc Biology, 2017, 15(1): 1-9. DOI:10.1186/s12915-016-0343-5 |
| [16] |
Glaser F, Pupko T, Paz I, et al. Consurf:identification of functional regions in proteins by surface-mapping of kphylogenetic information[J]. Bioinformatics, 2003, 19(1): 163-164. |
| [17] |
Wilkins A, Bachman B, Erdin S, et al. The use of evolutionary patterns in protein annotation[J]. Current Opinion in Structural Biology, 2012, 22(3): 316-325. DOI:10.1016/j.sbi.2012.05.001 |
| [18] |
Sankararaman S, Kolaczkowski B, Sjölander K. INTREPID:a web server for prediction of functionally important residues by evolutionary analysis[J]. Nucleic Acids Research, 2009, 37(Web Server issue): 390-395. |
| [19] |
Hopf TA, Schärfe CPI, Rodrigues JPGLM, et al. Sequence co-evolution gives 3D contacts and structures of protein complexes[J]. Elife, 2014, 3(6): 380-389. |
| [20] |
Engqvist MK, Mcisaac RS, Dollinger P, et al. Directed evolution of Gloeobacter violaceus rhodopsin spectral properties[J]. Journal of Molecular Biology, 2015, 427(1): 205-220. DOI:10.1016/j.jmb.2014.06.015 |
| [21] |
Wu NF, Deng MJ, Shi XY, et al. Isolation, purification and characterization of a new organphosphorus hydrolase OPHC2[J]. Chinese Science Bulletin, 2004, 49(3): 268-272. |
| [22] |
Xie J, Zhao Y, Zhang H, et al. Improving methyl parathion hydrolase to enhance its chlorpyrifos-hydrolysing efficiency[J]. Letters in Applied Microbiology, 2013, 58(1): 53-59. |
| [23] |
Chen K Q, Arnold F H. Enzyme engineering for nonaqueous solvents:random mutagenesis to enhance activity of subtilisin E in polar organic media[J]. Biotechnology, 1991, 9(11): 1073-1077. DOI:10.1038/nbt1191-1073 |
| [24] |
张秀艳, 何国庆, 陈启和. 理性和非理性蛋白质设计策略在酶工程中应用[J]. 科技通报, 2007, 23(2): 191-97. DOI:10.3969/j.issn.1001-7119.2007.02.008 |