2. 中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193;
3. 河北农林科学院植物保护研究所,保定 071000
2. State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193;
3. Institute of Plant Protection, Hebei Academy of Agricultural and Forestry Sciences, Baoding 071000
作为目前研究最多、应用最广的生防微生物,苏云金芽胞杆菌(Bacillus thuringiensis,Bt)广泛分布于土壤、水体、尘埃、病死昆虫、植被等基质中,其最大的特点是在形成芽胞的同时生成伴胞晶体,这种伴胞晶体由不同的杀虫晶体蛋白组成(Insecticidal crystal proteins,ICPs),包括Cry和Cyt蛋白[1],对多种昆虫具有特异的高效杀灭活性,如鳞翅目、鞘翅目、双翅目、半翅目,及线虫、螨类[2-7]等,并且对非靶标天敌安全、对人畜无害、不污染环境,因此Bt广泛用于防治农、林、医、仓贮等领域的害虫[8]。
目前国际上Bt商品化产品约有数百种,其中用于防治鳞翅目害虫的Bt产品主要源自库斯塔克亚种(B. thuringiensis subsp.kurstaki,血清型H3a,3b,3c,简称Btk)菌株,如HD1菌株(Bt杀虫剂Dipel的生产菌株)。该菌株已经在全世界应用几十年,并且是目前用于评估鳞翅目害虫特异Bt杀虫剂毒力效价的标准参考菌株[9]。在国家有关项目的大力支持下,我国研究机构也分离出一系列对鳞翅目高毒力的Bt菌株。例如,河北农林科学院植物保护研究所分离到的鲇泽亚种(B. thuringiensis subsp.aizawai,血清型7,简称Bta)菌株G03,同样对多种鳞翅目害虫有效,中国农业科学院植物保护研究所在此基础上构建出了同时对鳞翅目和鞘翅目(叶甲科)害虫均具有高毒性的工程菌株G033A[10-11],并且目前已经获得了农药登记证(PD20171726),商标为“禁卫军”。
为了进一步分析G03等鳞翅目害虫高效菌株的特点,本研究对G03菌株进行了基因组测序,并与已公布序列的Bt生产菌株HD1以及Btk模式菌株HD73进行比较基因组的研究,相关研究结果对G03应用以及今后Bt菌株遗传改良均具有重要的指导意义。此外,为了分析不同杀虫活性特性Bt菌株之间的进化差异,本研究选择了两株蛴螬有效的菌株(Bt185、HBF18),以及对鳞翅目害虫有效但是杀虫活性相对较差的菌株HD12进行了基因组进化分析。
1 材料与方法 1.1 供试菌株G03菌株是实验室保存菌株。从NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载菌株HD1、HD73,Bt185、HBF18以及HD12基因组数据用于后续分析。
1.2 方法 1.2.1 基因组制备与测序按照张彦蕊等方法提取[12]G03菌株的基因组DNA并检测基因组DNA的质量,合格后送交公司测序。利用TruSeq DNA无PCR文库制备试剂盒构建测序文库,并交由华大基因公司利用Illumina HiSeq 2500测序平台测序。
1.2.2 测序数据的组装、注释对测序获得的原始Reads进行质量控制,删除低质量的Reads,利用IDBA_UD软件包[13]对剩余高质量的pair-end clean reads数据进行基因组序列组装。并利用NCBI原核生物基因组注释流程PGAP(Prokaryotic Genome Annotation Pipeline,https://www.ncbi.nlm.nih.gov/genome/annotation_prok/)对组装基因组数据进行基因预测与注释。
利用泛基因组分析PGAP软件包[14]进行基因家族聚类。利用维恩图在线绘制网站(http://bioinfogp.cnb.csic.es/tools/venny/)进行菌株间共享基因及特有基因的统计分析。利用Blast软件包[15]、TBtools软件[16]进行Go功能富集。并利用Blast软件包[15]进行杀虫基因及功能基因的注释。利用MEGAX软件包[17]和CVTree软件包[18]分别进行蛋白质系统发生分析和全基因组系统发生分析。
2 结果 2.1 基因组测序、组装与注释利用IDBA_UD软件包对G03菌株进行基因组组装。结果显示,基因组大小为6.38 M,组装contig数量为395,GC含量为34.71%,Contig N50为45.68 kb,Contig L50为41。相关组装结果已经提交到NCBI GenBank数据库,基因组登录号为NHNQ00000000.1。进一步利用NCBI原核生物基因组注释流程PGAP对基因组进行了注释,结果显示G03菌株包含6916个基因,其中蛋白质编码基因(Coding sequence,CDS)6 848个。
2.2 CVTree系统进化分析CVTree(Composition vector tree,简称CVTree)是郝柏林研究组于2003年提出的一种全新的用于推测物种之间亲缘关系与分类的研究方法[18]。这种方法基于全基因组,不需要进行序列比对,根据全基因组序列中不同核苷酸碱基或氨基酸残基短串的出现频率来构建系统发育树。迄今为止,CVTree已经广泛用于分析多种不同物种及数据,包括古生菌[19-20]、原核生物[18, 21]、真菌[22]、病毒[23]、叶绿体序列[24]、tRNA序列[25]、癌症序列[26]等。
我们利用CVTree对包括G03、HD1、HD73菌株在内的7株Bt菌株的全基因组构建组分矢量并作出Neighbor-joining系统发生树(氨基酸短串K值取6)。结果(图 1)发现,Btk菌株HD1和HD73聚在同一个分支,菌株G03与Btk菌株HD1和HD73所在分支相聚较近,属于同一大分支。而同样对鳞翅目害虫有效的菌株HD12则相距较远。此外,对鞘翅目害虫有效的菌株Bt185与HBF18与G03所在分支也有较大的距离。
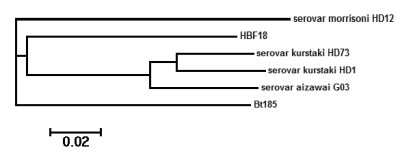
|
| 图 1 菌株CVTree系统进化分析树图 |
我们对CVTree分析中亲缘关系较近的Bta菌株G03以及Btk菌株HD1、HD73这3株Bt菌株进行了比较基因组的分析,首先利用PGAP软件包对其进行基因家族聚类,并利用维恩图在线绘制网站(http://bioinfogp.cnb.csic.es/tools/venny/)对这3株菌株的共享基因及特有基因家族进行绘图统计分析,如图 2所示。PGAP分析共鉴定出了6 860个基因家族,其中核心基因家族共有4 720个(全部菌株均含有的基因家族),占全部基因家族的68.8%。而G03、HD1和HD73菌株分别有540、433和327个特有基因(仅在一株菌株中存在的基因)。G03与HD1有5122基因家族是共有的,这大于HD1与HD73共有基因家族(5101)。
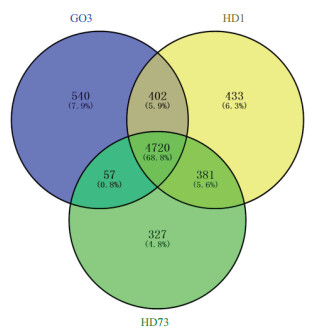
|
| 图 2 3株Bt菌株共享及特有基因的维恩图分析 |
我们利用Blast程序包分别将G03和HD1菌株的全部基因、共有基因以及特有基因序列与uniprot数据库(https://www.uniprot.org/)进行序列比对,并利用TBtools软件进行Go功能注释以及level2水平上的功能解析。图 3统计了菌株G03基因组Go功能注释在level 2水平上的统计情况,红色是菌株G03基因组特有基因的统计结果,蓝色部分是G03和HD1菌株都有的基因的统计结果。
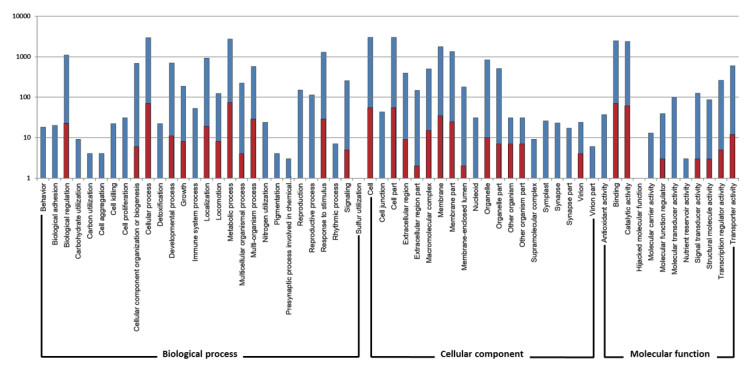
|
| 图 3 G03菌株Go功能注释 蓝色部分代表G03与HD1两者共有的基因的Go功能富集;红色部分是G03特有基因的Go功能富集 |
结果分析发现生物学途径(biological process)相关功能部分,G03特有基因在cellular process和metabolic process两个方面数量最多;在细胞学组件(cellular component)相关功能部分,G03特有基因在cell和cell part两个方面数量最多;在分子功能(molecular function)方面,G03特有基因在binding和catalytic activity两个方面数量最多。其中生物学途径与分子功能相关的Go富集反映了G03在生物学水平与分子水平上的特点。
cellular process是指在细胞水平上发生的过程相关功能富集。例如,细胞对环境的应答过程,在该方面的功能有较多Go富集显示菌株G03在环境适应等细胞过程方面有较多的特点。metabolic process是指生物体转化化学物质的化学反应和途径相关功能富集,包括合成代谢和分解代谢,这方面较多Go富集说明G03菌株在物质分解利用或化合物合成方面有较特别的能力。
Binding和catalytic activity分别指结合与催化方面的功能富集,这两个功能是紧密相关的,G03特有基因在这两个方面有较多的富集,显示其在分子识别催化方面有特别的功能。
2.5 G03菌株杀虫基因分析我们对Bta及Btk菌株包含的杀虫基因种类以及数量做了比较分析,HD73菌株仅有cry1Ac一个杀虫基因,菌株G03与HD1均含有cry1Aa、cry1Ac、cry1Ia、cry2Ab、vip3Aa这5个高毒力杀虫基因[3, 27-29]。已有研究表明Cry1A、Cry1I、Cry2A、Vip3A这几类杀虫蛋白具有不同的受体,并且杀虫蛋白之间还有协同增效的作用,这可能是G03与HD1具有高活性的原因。此外,与HD1相比,G03菌株还含有cry1Ca、cry1Da、cry9Ea基因。
2.6 G03、HD1菌株特有基因分析利用基因家族分析的方法,对菌株G03与HD1进行比较分析,结果显示G03有597个特有基因家族,而HD1有814个特有基因家族。其中,G03菌株特有的基因家族中有232个可以注释到功能,而HD1菌株特有的基因家族中有404个可以注释到功能。进一步,对这些注释到功能的基因家族进行分析,发现噬菌体与转座酶相关的基因家族最为丰富。HD1菌株特有的基因家族中有34个基因家族与噬菌体相关,而G03菌株特有的基因家族中只有10个与噬菌体有关。
此外,特有基因家族中,HD1菌株转座酶基因也比较多,有26个基因家族属于转座酶,而G03菌株特有的基因家族中只有8个。转座酶是转座子执行转座功能的酶,通常由转座子编码。本研究进一步比较了两个菌株含有转座酶的数量与多样性。结果显示,G03菌株仅仅含有33个转座酶或片段,而HD1多达216个转座酶或片段。这些转座酶按照序列一致性(按照序列一致性50%设定阈值)分为58个家族,其中16个基因家族是两个菌株共有的。这些基因家族中,最大的基因家族含有45个转座酶基因,其中有4个来源于菌株G03,41个来源于菌株HD1。该家族转座酶含有TNP1 DDE结构域,是IS4、IS421、IS5377、IS427、IS402、IS1355以及IS5等多种插入序列(Insert sequence,IS)的转座酶。进一步用UPGMA对这些含有TNP1 DDE结构域的转座酶进行系统进化分析(去除了4个转座酶片段),结果(图 4)显示这些转座酶分为3个类群,其中类群1是两个菌株共有的;类群2有6个基因,是HD1特有的;类群3只有1个基因,是G03特有的。
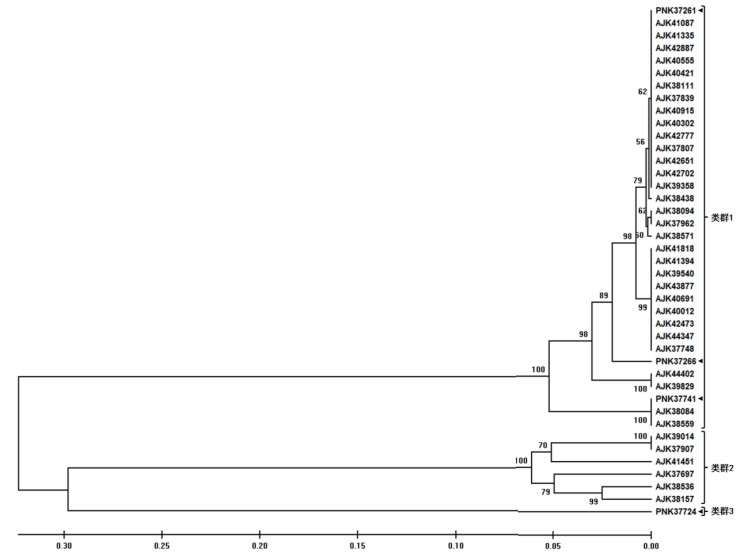
|
| 图 4 含有TNP1 DDE结构域转座酶进化关系分析。箭头标出的是G03转座酶 |
噬菌体与插入序列、转座子是微生物获得外源基因的重要途径,同时也是微生物基因组变异的重要因素。上述分析结果表明G03含有较少的噬菌体与插入序列、转座子相关的基因,显示菌株G03与HD1相比,应该具有较低的变异概率。
3 讨论目前市场上常用的Bt生产菌株种类相对较少,主要以Btk菌株HD1为主,相关研究报道较多。Bta菌株G03是我国科学家自主分离并且已经用于害虫防控的菌株,已通过基因工程改造拓展了其杀虫谱,在鳞翅目和鞘翅目(叶甲科)害虫防治中发挥重要作用,然而该菌株遗传学、基因组相关研究鲜见报道。本研究利用Illumina测序技术进行了菌株G03基因组测序,并进行了基因功能注释。在此基础上与NCBI数据库中已经公开的Btk生产菌株HD1、Btk模式菌株HD73、两株蛴螬有效的菌株(Bt185、HBF18)以及含有多种杀虫基因类型的菌株HD12进行了比较基因组研究,旨在从基因组角度分析G03等鳞翅目害虫高效菌株的特点、比较与Btk菌株的异同,为进一步对Bt菌株进行改良、推进我国自主分离研发的Bt菌株及其杀虫剂产品的应用奠定基础。
本研究首次利用基因组测序技术,从基因组水平上全面的分析了G03杀虫相关的基因。与PCR杀虫基因鉴定技术相比,基因组测序技术避免了PCR鉴定技术由于引物设计不全面、PCR扩增条件不合适等原因,导致基因鉴定的误差。通过基因组分析发现,菌株G03含有8个杀虫基因(cry1Aa、cry1Ac、cry1Ca、cry1Da、cry1Ia、cry2Ab、cry9Ea、vip3Aa),其中cry1Ca、cry1Da是菌株HD1不具备的。目前,表达Cry1A、Cry2A和Vip3A等蛋白的转基因的抗虫作物已经在多个国家商业化种植(International Service for the Acquisition of Agri-biotech Applications,www.isaaa.org/),使用表达与上述蛋白无交互抗性的Bt杀虫剂在害虫抗性治理方面具有重要的意义。利用小菜蛾抗Cry1Ac种群研究表明,Cry1Ca与Cry1Ac蛋白没有交互抗性[30],因此,菌株G03比HD1在害虫的Bt杀虫剂抗性风险控制方面更具优势。
Bt菌株中与杀虫活性相关的免疫抑制因子A(Immune Inhibitor A,InhA)、双效菌素ZwA(Zwittermicin A,ZwA)、酰基高丝氨酸内酯水解酶(AHLs Hydrolase,aiiA)以及几丁质酶(Chitinase)对Bt菌株杀虫活性及环境适应性都有重要意义。InhA是Bt分泌的一种金属蛋白酶,能够降解昆虫产生的抗菌肽从而逃避宿主的免疫系统[31-32]。aiiA可以水解细菌的群体感应相关的信号分子酰基高丝氨酸内酯(Acylated Homoserine Lactones,AHLs),对多种细菌有抑制作用,因此有助于提高Bt在昆虫肠道内竞争优势[33-34]。Chitinase是一种可溶性的胞外蛋白质类杀虫活性物质,可以帮助Bt菌株降解昆虫肠道围食膜中的几丁质成分,使其能通过穿孔的肠道进入血腔引起昆虫败血症,对Bt杀虫蛋白具有增效作用[35-36]。前期研究发现,菌株G03与HD1都含inhA、aiiA、chitinase基因以及合成ZwA的基因簇,并且aiiA、chitinase基因进行系统发生分析发现鳞翅目害虫高效的菌株aiiA、chitinase基因序列比较接近,说明G03菌株在感染寄主、适应环境方面与HD1类似[37]。
此外,本研究还发现与HD1相比,G03含有较少的噬菌体与转座子基因,说明G03基因组应该具有更好的遗传稳定性。作为生产菌株,遗传稳定性对产品发酵生产尤为重要,基因组比较分析结果显示G03菌株在该方面更有优势。
综上所述,本研究对生产菌株G03进行的基因组测序、注释与比较基因组分析,不仅可以从杀虫基因角度揭示生产菌株高活性的原因,还可以为进一步菌株改良提供基因组数据支撑。
| [1] |
Höfte H, Whiteley HR. Insecticidal crystal proteins of Bacillus thuringiensis[J]. Microbiological Reviews, 1989, 53(2): 242-255. |
| [2] |
Xue J, Liang G, Crickmore N, et al. Cloning and characterization of a novel Cry1A toxin from Bacillus thuringiensis with high toxicity to the Asian corn borer and other lepidopteran insects[J]. Fems Microbiology Letters, 2008, 280(1): 95-101. DOI:10.1111/fml.2008.280.issue-1 |
| [3] |
李长友, 张杰, 宋福平, 等. 苏云金芽孢杆菌B-Pr-88菌株中cry2Ab4基因的表达和杀虫活性研究[J]. 生物工程学报, 2007, 23(4): 634-638. DOI:10.3321/j.issn:1000-3061.2007.04.014 |
| [4] |
Bi Y, Zhang Y, Shu C, et al. Genomic sequencing identifies novel Bacillus thuringiensis Vip1/Vip2 binary and Cry8 toxins that have high toxicity to Scarabaeoidea larvae[J]. Appl Microbiol Biotechnol, 2015, 99(2): 753-760. DOI:10.1007/s00253-014-5966-2 |
| [5] |
张学雯, 束长龙, 陆宴辉, 等. 对绿盲蝽具有杀虫活性Bt菌株的筛选及Cry15Aa蛋白活性的研究[J]. 植物保护, 2016, 42(3): 56-62. DOI:10.3969/j.issn.0529-1542.2016.03.009 |
| [6] |
Jr UJ, Hu Y, Miller MM, et al. Bacillus thuringiensis-derived Cry5B has potent anthelmintic activity against Ascaris suum[J]. PLoS Negl Trop Dis, 2013, 7(6): e2263. DOI:10.1371/journal.pntd.0002263 |
| [7] |
Erban T, Nesvorna M, Erbanova M, et al. Bacillus thuringiensis var. tenebrionis control of synanthropic mites(Acari:Acaridida)under laboratory conditions[J]. Experimental and Applied Acarology, 2009, 49(4): 339-346. DOI:10.1007/s10493-009-9265-z |
| [8] |
Bravo A, Likitvivatanavong S, Gill SS, et al. Bacillus thuringiensis:A story of a successful bioinsecticide[J]. Insect Biochemistry and Molecular Biology, 2011, 41(7): 423-431. DOI:10.1016/j.ibmb.2011.02.006 |
| [9] |
Beegle CC, Couch TL, Alls RT, et al. Standardization of HD-1-S-1980:U. S. standard for assay of lepidopterous-active Bacillus thuringiensis[J]. Bulletin of the Esa, 1986, 32(1): 44-45. |
| [10] |
王广君, 张杰, 宋福平, 等.苏云金芽孢杆菌工程菌G033A及其制备方法[P].中国发明专利. CN 1318584 C. 2007.
|
| [11] |
Wang G, Zhang J, Song F, et al. Engineered Bacillus thuringiensis GO33A with broad insecticidal activity against lepidopteran and coleopteran pests[J]. Appl Microbiol Biotechnol, 2006, 72(5): 924-930. DOI:10.1007/s00253-006-0390-x |
| [12] |
张彦蕊, 束长龙, 宋福平, 等. 一种简单、快速的苏云金芽胞杆菌基因组DNA提取方法[J]. 生物技术通报, 2012(11): 197-201. |
| [13] |
Peng Y, Leung HCM, Yiu SM, et al. IDBA-UD:a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [14] |
Zhao Y, Wu J, Yang J, et al. PGAP:pan-genomes analysis pipeline[J]. Bioinformatics, 2012, 28(3): 416-8. DOI:10.1093/bioinformatics/btr655 |
| [15] |
Lo bo. Basic Local Alignment Search Tool(BLAST)[J]. Journal of Molecular Biology, 2012, 215(3): 403-410. |
| [16] |
Chen C, Xia R, Chen H, et al. TBtools, a Toolkit for biologists integrating various HTS-data handling tools with a user-friendly interface[J]. bioRxiv, 2018. |
| [17] |
Kumar S, Stecher G, Tamura K. MEGA7:Molecular Evolutionary Genetics Analysis Version 7. 0 for Bigger Datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870. DOI:10.1093/molbev/msw054 |
| [18] |
Qi J, Wang B, Hao BL. Whole proteome prokaryote phylogeny without sequence alignment:a K-string composition approach[J]. Journal of Molecular Evolution, 2004, 58(1): 1-11. |
| [19] |
Sun JD, Xu Z, Hao BL. Whole-genome based Archaea phylogeny and taxonomy:A composition vector approach[J]. Science Bulletin, 2010, 55(22): 2323-2328. DOI:10.1007/s11434-010-3008-8 |
| [20] |
Zuo G, Zhao X, Hao B. Phylogeny and Taxonomy of Archaea:A comparison of the whole-genome-based CVTree approach with 16S rRNA sequence analysis[J]. Life, 2015, 5(1): 949-968. DOI:10.3390/life5010949 |
| [21] |
Li Q, Xu Z, Hao B. Composition vector approach to whole-genome-based prokaryotic phylogeny:success and foundations[J]. Journal of Biotechnology, 2010, 149(3): 115-119. DOI:10.1016/j.jbiotec.2009.12.015 |
| [22] |
Wang H, Xu Z, Gao L, et al. A fungal phylogeny based on 82 complete genomes using the composition vector method[J]. BMC Evolutionary Biology, 2009, 9(1): 195. DOI:10.1186/1471-2148-9-195 |
| [23] |
Gao L, Qi J. Whole genome molecular phylogeny of large dsDNA viruses using composition vector method[J]. BMC Evolutionary Biology, 2007, 7(1): 1-7. DOI:10.1186/1471-2148-7-1 |
| [24] |
Chu KH, Qi J, Yu ZG, et al. Origin and phylogeny of chloroplasts revealed by a simple correlation analysis of complete genomes[J]. Molecular Biology and Evolution, 2004, 21(1): 200-206. |
| [25] |
毛源泽, 韦芳萍, 王俊美, 等. 利用cvTree方法构建及分析tRNA序列的亲缘关系进化网络[J]. 基因组学与应用生物学, 2011, 30(4): 371-378. DOI:10.3969/j.issn.1674-568X.2011.04.018 |
| [26] |
沈路明, 韦芳萍. 基于cvtree方法和复杂网络理论的癌症进化树分析[J]. 基因组学与应用生物学, 2014, 33(2): 405-412. |
| [27] |
Schnepf HE, Wong HC, Whiteley H R. The amino acid sequence of a crystal protein from Bacillus thuringiensis deduced from the DNA base sequence[J]. J Biol Chem, 1985, 260(10): 6264. |
| [28] |
Carlson CR, Kolstø AB. A complete physical map of a Bacillus thuringiensis chromosome[J]. Journal of Bacteriology, 1993, 175(4): 1053-1060. DOI:10.1128/jb.175.4.1053-1060.1993 |
| [29] |
Chakroun M, Ferré J. In vivo and in vitro binding of Vip3Aa to Spodoptera frugiperda midgut and characterization of binding sites by(125)Ⅰ radiolabeling[J]. Appl Environ Microbiol, 2014, 80(20): 6258-65. DOI:10.1128/AEM.01521-14 |
| [30] |
Zhu X, Lei Y, Yang Y, et al. Construction and characterisation of near-isogenic Plutella xylostella(Lepidoptera:Plutellidae)strains resistant to Cry1Ac toxin[J]. Pest Management Science, 2015, 71(2): 225. DOI:10.1002/ps.2015.71.issue-2 |
| [31] |
Fedhila S, Nel P, Lereclus D. The InhA2 metalloprotease of Bacillus thuringiensis strain 407 is required for pathogenicity in insects infected via the oral route[J]. Journal of Bacteriology, 2002, 184(12): 3296. DOI:10.1128/JB.184.12.3296-3304.2002 |
| [32] |
Dalhammar G. Characterization of inhibitor A, a protease from Bacillus thuringiensis which degrades attacins and cecropins, two classes of antibacterial proteins in insects[J]. Febs Journal, 1984, 139(2): 247-252. |
| [33] |
Zhang L, Murphy PJ, Kerr A, et al. Agrobacterium conjugation and gene regulation by N-acyl-L-homoserine lactones[J]. Nature, 1993, 362(6419): 446-8. DOI:10.1038/362446a0 |
| [34] |
Molina L, Rezzonico F, Défago G, et al. Autoinduction in Erwinia amylovora:evidence of an acyl-homoserine lactone signal in the fire blight pathogen[J]. Journal of Bacteriology, 2005, 187(9): 3206. DOI:10.1128/JB.187.9.3206-3213.2005 |
| [35] |
Thamthiankul S, Moar WJ, Miller ME, et al. Improving the insecticidal activity of Bacillus thuringiensis subsp. aizawai against Spodoptera exigua by chromosomal expression of a chitinase gene[J]. Appl Microbiol Biotechnol, 2004, 65(2): 183-192. |
| [36] |
Ding X, Luo Z, Xia L, et al. Improving the insecticidal activity by expression of a recombinant cry1Ac gene with chitinase-encoding gene in Acrystalliferous Bacillus thuringiensis[J]. Current Microbiology, 2008, 56(5): 442-446. DOI:10.1007/s00284-008-9112-1 |
| [37] |
黄颖, 王奎, 束长龙, 等. 基于比较基因组学分析苏云金芽胞杆菌防治蛴螬的相关功能基因[J]. 中国生物防治学报, 2018, 34(5): 793-800. |




