2. 东北农业大学 国家教育部大豆生物学重点实验室,哈尔滨 150030
2. Soybean Research Institute, Key Laboratory of Soybean Biology in Chinese Ministry of Education, Northeast Agricultural University, Harbin 150030
纤维素是自然界中分布最广且含量最多的碳水化合物,同时又是数量最大的可再生资源。目前,利用可再生生物质资源生产替代燃料越来越受到人们的重视。但是,纤维素的结构复杂[1],纤维素酶对天然纤维素降解效率比较低,导致工业化成本很高,从而无法实现大规模的生产。所以,高的水解效率通常需要对底物进行预处理,一般包括物理、化学、物理化学和生物四种预处理的方式。物理和化学方法通常会造成一定的环境污染,同时产生的一些副产物还会抑制酶的水解活性[2]。而生物预处理是一种绿色环保的方法,通过对纤维素晶体结构的疏松和破坏,可以使木质纤维素更容易的被纤维素酶水解[3]。研究发现,一些非水解破坏活性的蛋白能够促进纤维素酶的水解[4]。这些蛋白与纤维素酶存在协同作用,包括植物膨胀素[5],来源于细菌[6]、真菌和线虫类的类膨胀素蛋白[7-8],以及一些辅助蛋白如裂解多糖单加氧酶(Lytic polysaccharide mon-ooxygenases,LPMO)和Cerato platanin(CP)[9-10]。
膨胀素(expansin)是一类扩展蛋白,能够破坏并松散植物的细胞壁。膨胀素通过破坏纤维素微纤维间或纤维素和其他细胞壁多糖间的氢键来发挥作用,但对报道的膨胀素一般对它们没有水解活性[11],因此可以提高纤维素酶对纤维素类底物的水解[12]。膨胀素的分类是基于其系统发育关系,根据文献报道,有4种膨胀素家族在植物中已经被鉴定[13],分别是α-膨胀素(EXPA)、β-膨胀素(EXPB)、类α-膨胀素(EXLA)、类β-膨胀素(EXLB)。通过实验证明,EXPA和EXPB能够使细胞壁松弛[14],类似膨胀素的蛋白可能在其他的生物体中发挥作用。有研究发现,类膨胀素在病原微生物在植物根的定殖[15]及真菌中降解木质纤维素等[16]中发挥作用。非植物的膨胀素蛋白大多属于类膨胀素家族X(Expansin-like X,EXLX),这些蛋白大多参与细胞壁的延伸过程。目前为止,来源于真菌的里氏木霉(Trichoderma reesei),曲霉属烟曲霉(Aspergillus fumigatus)和烟管菌属烟管菌(Bjerkandera adusta)的TrSwo1,AfSwo1和BaLoosenin,以及细菌河氏菌属(Hahella chejuensis)的HcEXLX2,细菌枯草芽孢杆菌(Bacillus subtilis)的BsEXLX1和线虫球异皮线虫属金线虫(Globodera rostochiensis)的GrEXPB1都能够通过破坏纤维素结构来增强纤维素酶的活性[7, 17-20]。另外,膨胀素参与细胞壁松弛相关的其他发育过程,如果实软化[21]、花瓣的生长发育[22]、种子的发芽[23]、谷物的生长(在早期的生长过程中,膨胀素主要在谷粒皮中被发现,随后在胚乳也能发现,说明膨胀素可能是取决于谷粒最终重量的原因)[24]等过程。
传统膨胀素的结构包括两个结构域D1和D2。D1结构域以极性残基为主,并且与糖苷水解酶GH45酶有较低的相似性,其对于细胞壁的活性是非常重要的。D2结构域是一种非催化模块,作用类似于碳水化合物结合模块(Carbohydrate-binding module,CBM),有助于其结合纤维素。D2结构域具有扁平的芳香族氨基酸残基,可以与糖环的疏水中心相互作用[25]。
真菌来源的swollenin通常包括3个结构域:N端的信号肽和一个典型的真菌类型的CBM区,一个结构与GH45相似的同源区以及一个与膨胀素类似的区域。植物膨胀素通常不大于250个氨基酸残基,而真菌中的swollenin是这种尺寸的2倍。该大小的差异进一步支持细菌和真菌膨胀素相关蛋白的进化通过水平基因转移和独立结构域融合事件发生的假设[26]。此外,swollenin显示出与植物扩展蛋白不同的独特作用模式,但其类似于内切葡聚糖酶和纤维二糖水解酶的作用,但其不适合任何标准的纤维素酶类[27]。
在本研究中,我们从嗜热真菌T. leycettanus JCM12802中克隆得到了一个新的类膨胀素基因Tlexlx1,并构建了CBM融合基因CBM-Tlexlx1,类膨胀素基因及其融合基因在毕赤酵母中实现了表达,并对其基本性质进行研究,为在生物工程等高新技术领域中解决现代纤维素利用率低,难降解等的问题上提供了新的思路,以及对于非植物来源的类膨胀素EXLX家族的基因功能的鉴定提供新的素材。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒嗜热真菌T. leycettanus JCM12802购自日本微生物菌种保藏中心(Japan collection of microorganisms,JCM);毕赤酵母Pichia pastoris GS115和质粒pPIC9购自Thermo Fisher(原Invitrogen);克隆菌株Escherichia coli Trans-T1和质粒pEASY-T3购自北京全式金公司。
1.1.2 培养基LB液体及固体培养基、YPD培养基的配制见《分子克隆实验指南》(J.萨姆布鲁克D.W.拉塞尔著),MD固体培养基、MM固体培养基、BMGY培养基、BMMY培养基按照Invitrogen公司发布的毕赤酵母表达系统操作手册配方配制。
1.1.3 试剂真菌DNA提取试剂盒、质粒提取试剂盒及DNA胶回收试剂盒购自OMEGA公司;真菌RNA提取试剂盒购自Promega公司;反转录试剂盒TOYOBO购自Tokyo公司;蛋白定量试剂盒购自Bio-Rad公司;限制性内切酶购自TaKaRa公司;T4 DNA连接酶购买自New England Biolab公司;FastPfu DNA聚合酶购自北京全式金公司,蛋白Marker够买于北京GeneStar生物公司。商品纤维素酶购自Sigma公司。其它试剂均为国产分析纯。
1.2 方法 1.2.1 类膨胀素基因Tlexlx1的克隆及融合基因CBM-Tlexlx1的构建嗜热真菌T. leycettanus JCM12802的全基因组序列已由上海美吉公司完成测序。经过对注释序列进行分析,Tlexlx1是一个典型的类膨胀素基因。通过美国国立生物技术信息中心NCBI(National Center for Biotechnology Information)BLAST模块(http://blast.ncbi.nlm.nih.gov/Blast/)对该基因的新颖性进行评估,利用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)预测其信号肽序列。利用Vector NTI Advance 10.0软件分析Tlexlx1基因序列及编码蛋白理论蛋白分子量大小、等电点。利用ClustalW软件和ESPript 3.0(http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行多序列比对分析。
为了获得类膨胀素基因Tlexlx1的cDNA序列,将T. leycettanus JCM12802接种于PDB培养基中,30℃、200 r/min摇床震荡培养3 d后,以2%接种量转接于以复合培养基中,30℃、200 r/min摇床震荡培养3 d。用干净滤纸过滤菌液得到菌体后,置于液氮中用研钵充分研磨,提取总RNA,用反转录试剂盒TOYOBO(Tokyo,Japan)合成cDNA单链。并以cDNA为模板,用特异性引物Tlexlx1-F(5'-CGGAATTCGGGCCTCTTGTTGGACGTCAGGA-3',下划线为Eco RⅠ酶切位点)和Tlexlx1-R(5'-TTGCGGCCGCCTAAAAGTTTGAAGACGCAGTCGTGGT-3',下划线为NotI的酶切位点)进行PCR扩增,扩增后将得到符合目的片段大小的PCR产物连接至pEASY-T3载体上,并转化Trans-T1菌株,阳性克隆进行测序验证。
将同样来源的膨胀素基因的CBM区编码序列Tlswo-CBM用overlap PCR的方法融合到Tlexlx1的N端,构建了融合基因CBM-Tlexlx1。第一轮PCR使用引物CBMF(CGGAATTCCAGAGCAGCTGTGCAGGGACC,下划线为EcoR Ⅰ酶切位点)和Tlexlx1-CBM-R1(TCCAACAAGAGGCCCCATGGTTCCGGATTTGCACATGGAC)扩增得到CBM区编码序列,使用引物Tlexlx1-CBM-F1(5'-AAATCCGGAACCATGGGGCCTCTTGTTGGACGTCAG-3')和Tlexlx1-R扩增等到Tlexlx1编码序列。第二轮PCR将第一轮PCR产物混合后作为模板,使用引物CBMF和Tlexlx1-R扩增等到CBM和Tlexlx1的融合编码基因CBM-Tlexlx1。
1.2.2 毕赤酵母表达重组质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1的构建用限制性内切酶EcoRⅠ和NotⅠ对测序正确的Tlexlx1基因和CBM融合基因CBM-Tlexlx1以及酵母表达载体pPIC9进行双酶切,将正确的目的片段与载体回收,用T4DNA连接酶室温下连接2 h,转入E. coli TransⅠ-T1感受态细胞中,37℃孵育30-60 min,涂于具有氨苄抗性的LB平板上,37℃过夜培养,挑取单菌落并进行阳性克隆的筛选。经测序正确后得到重组质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1。
1.2.3 重组质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1在毕赤酵母中的表达与纯化将重组质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1通过Bgl Ⅱ单酶切进行线性化,线性化产物通过电击转化到GS115感受态细胞中,涂至MD平板上,30℃培养2-3 d待菌落长出。然后,从MD板上挑取96个单克隆子到带有1-96编号MD平板,培养1-2 d。按编号挑取MD平板上正常生长转化子接种于装有BMGY培养基的15 mL离心管中,30℃、220 r/min摇床培养2 d后,离心去上清,更换为含有0.5%甲醇的BMMY培养基诱导培养2 d,然后离心取上清用于酶活性检测。筛选出酶活相对较高,蛋白量相对较大的转化子进行摇瓶的放大培养。酶活高的转化子接入YPD培养基,30℃培养2 d,然后按1%接种量接种于400 mL BMGY培养基30℃、260 r/min摇床中培养2 d,离心去上清,用200 mL BMMY诱导培养基继续培养2 d(中间补加甲醇一次)。将酶液在12 000 r/min下离心10 min,收集上清液进行酶活的测定及蛋白电泳(SDS-PAGE)分析。
用10 kD的膜包对上清的粗酶液进行浓缩同时去除小分子等杂质并更换缓冲液。浓缩后的酶液用Tris-HCl缓冲液(pH 8.0)过夜透析(3 kD透析袋)进行脱盐处理。经过处理后的低盐浓度的酶液,使用HiTrap Q XL阴离子柱进行纯化,用1 mol/L的NaCl进行梯度洗脱,收集洗脱峰并进行酶活力测定。SDS-PAGE电泳检测并分析其纯度,纯化后的酶液进行酶的性质分析。并且对表达的蛋白进行质谱鉴定,样品由中国农业科学院蜜蜂所测定。
1.2.4 酶活测定方法本实验采取DNS法测定类膨胀素TlEXLX1的活性。底物:大麦葡聚糖、地衣多糖或CMC-Na(900 μL),缓冲液:柠檬酸-磷酸二氢钠(终浓度为0.1 mol/L),稀释酶液100 μL,终止剂DNS(1.5 mL,煮沸5 min),OD540下读取吸光值。
酶活定义:最适反应条件下,每分钟分解底物生成1 μmoL葡萄糖所需的酶量定义为一个酶活力单位U。
1.2.5 蛋白定量用蛋白定量试剂盒对TlEXLX1和CBM-TlEXLX1进行蛋白定量。将不同浓度的蛋白标品(BSA)和待测样品TlEXLX1、CBM-TlEXLX1、加入在酶标条中,然后加入200 µL考马斯亮蓝显色剂。室温反应8 min后,在波长(OD595)处读取吸光值。根据所测得的吸光值计算出标准曲线,根据标准曲线公式计算出样品的蛋白质含量(mg/mL)。
1.2.6 类膨胀素TlEXLX1和CBM-TlEXLX1基本酶学性质的测定 1.2.6.1 最适pH的测定将纯化后TlEXLX1和CBM-TlEXLX1,加入到不同pH(pH 2.2-12.0)100 mmol/L的缓冲液(pH 2.2-8:柠檬酸-磷酸氢二钠;pH 8-9:Tris-HCl;pH10-12:Gly-NaOH)的底物中,在60℃下反应10 min,测定其最适pH。每个反应3个平行及一个空白对照,根据测定结果绘制最适pH曲线。
1.2.6.2 最适温度的测定用pH 6.0的缓冲液把酶液稀释合适的倍数,在不同温度(30-70℃)下反应10 min测定其最适温度,每个反应3个平行及一个空白对照,根据测定结果绘制最适温度曲线。
1.2.6.3 类膨胀素TlEXLX1和CBM-TlEXLX1比活力及底物特异性的测定选取地衣多糖(1%),大麦葡聚糖(1%)、昆布多糖(1%)、CMC-Na(2%)、微晶纤维素(1% Avicel)、葡甘露聚糖(1%)、角豆胶(1%)、木聚糖(1%)和瓜尔豆胶(1%)作为底物,pH 6.0的柠檬酸-磷酸氢二钠(终浓度100 mmol/L)缓冲液配置。通过DNS法测定TlEXLX1和CBM-TlEXLX1的比活力。
比活力单位定义为,每毫克酶蛋白所具有的酶活力单位数。(1%为质量体积比)。
1.2.7 TlEXLX1及CBM-TlEXLX1与纤维素酶的协同实验采用两步法进行TlEXLX1与纤维素酶的协同实验。以10 mg/mL的微晶纤维素(Avicel)作为底物,pH 6.0的柠檬酸-磷酸氢二钠(终浓度50 mmol/L)缓冲液,体系1 mL。过程:取TlEXLX1 0 µg、1 µg、5 µg、10 µg、20 µg和30 µg分别用缓冲液稀释到100 µL,加入用900 µL 10 mg/mL的微晶纤维素(Avicel)底物中进行预处理,放入金属浴(金属浴的作用是能够使微晶纤维素反应充分)37℃下振荡24 h(600 r/min),取出样品90℃下灭活10 min,离心(12 000 r/min),用缓冲液洗3-4遍初去残留TlEXLX1或CBM-TlEXLX1,分别加入100 µL(0.3 U)的商品纤维素酶,buffer补齐到1 mL,0、30、60和90 min分别取样120 µL测定酶活。用DNS法进行测定释放还原糖的量。
用10 mg/mL的微晶纤维素(Avicel)作为底物,pH5.0的柠檬酸-磷酸氢二钠(终浓度50 mmol/L)缓冲液,体系1 mL。过程:加入100 µL的TlEXLX1及CBM-TlEXLX1(10 µg)对底物(900 µL)进行预处理,放入金属浴37℃下振荡24 h(600 r/min),取出90℃下灭活10 min,离心(12 000 r/min),用缓冲液洗3-4遍去除TlEXLX1和CBM-TlEXLX1。随后,加入100 µL(0.3 U和0.6 U)的商品纤维素酶,buffer补齐到1 mL,每30 min取样120 µL测定酶活。用DNS法进行测定酶活力。
按照上述方法,测定(1 U,2 U)的商品纤维酶与类膨胀素TlEXLX1(10 µg)时的协同作用。过程同上。上述实验均用金属浴与完成。
1.2.8 重组酶TlEXLX1和CBM-TlEXLX1处理微晶纤维素的扫描电镜实验用TlEXLX1和CBM-TlEXLX1各300 µg加入到微晶纤维素(5 mg/mLAvicel)和滤纸(5 mg/mL)的底物中用pH 6的柠檬酸-磷酸氢二钠缓冲液(终浓度50 mmol/L)将体系补齐到1 mL,37℃下轻微震荡处理24 h,然后烘干被处理过的底物。分别用Buffer和BSA代替膨胀素作为对照实验。
扫描电镜观察实验前,需对样品进行处理,在样品上粘上少量的导电胶,用棉签粘取少量干燥的固体样品微晶纤维素和滤纸后涂贴在导电胶上,然后去除多余未粘在导电胶上的粉末,进行喷金处理30 min后,进行观察。
2 结果 2.1 TlEXLX1基因的克隆及序列分析T. leycettanus JCM12802类膨胀素基因Tlexlx1基因全长为1 196 bp,含有1个内含子(83 bp)。cDNA全长为1 113 bp,编码370个氨基酸和一个终止密码子。SignalP预测N-端前22个氨基酸为信号肽。用NetNGlyc 1.0 Server对TlEXLX1进行N-糖基化预测,没有N-糖基化位点,融合蛋白CBM-TlEXLX1有两个N-糖基化位点。Tlexlx1编码蛋白TlEXLX1的理论蛋白分子量大小为35.4 kD,等电点为4.3,而CBM-Tlexlx1的理论分子量为51.5 kD,等电点为4.3。
根据BlastP比对结果显示Tlexlx1编码蛋白与Penicilliopsis zonata CBS 506.65来源的假想蛋白hypothetical protein ASPZODRAFT_140583具有最高的序列相似性为81%,与Penicillium digitatum Pd1来源的Expansin-like protein 1序列相似性为56%。基因Tlexlx1提交至GenBank数据库中,序列注册号为MH036891。
2.2 表达载体pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1的构建与重组蛋白的表达与纯化分析将不含有自身信号肽的Tlexlx1和CBM-Tlexlx1c-DNA序列,以正确的阅读框克隆到质粒pPIC9上信号肽编码序列α因子后,得到重组表达质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1,测序结果表明构建成功。
对重组表达质粒pPIC9/Tlexlx1和pPIC9/CBM-Tlexlx1转化毕赤酵母的克隆子通过酶活力测定及蛋白电泳检测进行筛选,成功选到了阳性克隆子。将阳性克隆子通过摇瓶水平发酵,获得发酵上清液。通过浓缩和纯化得到并收集得到了纯化的蛋白。纯化后的蛋白进行蛋白电泳检测,根据蛋白电泳结果(图 1)可知,重组蛋白TlEXLX1(泳道1)表观分子量为75 kD,比理论分子量大(TlEXLX1理论分子量为35.4 kD)。融合蛋白CBM-TlEXLX1(泳道2)在75 kD和180 kD处均可发现明显条带,均比理论分子量大(融合蛋白CBM-TlEXLX1的理论分子量为51.5 kD)。
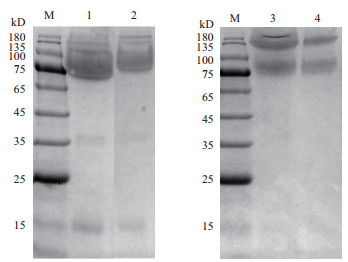
|
| 图 1 重组蛋白TlEXLX1和融合蛋白CBM-TlEXLX1的SDS-PAGE图 M:蛋白分子量标准;1,2:原酶和纯化后的TlEXLX1;3,4:原酶和纯化后的CBM-TlEXLX1 |
质谱鉴定结果显示,重组蛋白测定结果能够与理论蛋白序列匹配,覆盖上的肽段为:GPSGNSIK和NKDGTSAYWFSMQVVNANEPVASLEVSTDGGSTWQSTTRTYYNYFEKQSGFGTDTVDVRITSTSGATITVK)。
分子量75 kD(180 kD)融合蛋白质谱鉴定结果与理论蛋白序列也都能匹配,覆盖上的肽段分别为:SGTMGPLVGR,GPSGNSIK和NKDGTSAYWFSMQVVNANEPVASLEVSTDGGSTWQSTTRTYYNYFEKQSGFGTDTVDVRITSTSGATITVKNVSCQSESTTTASSNF(SGTMGPLVGR,GPSGNSIK和NKDGTSAYWFSMQVVNANEPVASLEVSTDGGSTWQSTTRTYYNYFEKQSGFGTDTVDVRITSTSGATITVK)。
在分子量75 kD和180 kD融合蛋白的质谱鉴定结果中发现,野生型N-端的部分序列和CBM(TlSWO1)末端的部分序列连接成一条肽段(SGTMGPLVGR,下划线部分属于CBM序列,无下划线属于野生型序列)。质谱结果说明,该蛋白是正确表达的TlEXLX1类膨胀素蛋白,同时也确定融合蛋白构建成功。
通过N-糖基化预测,结果可知重组蛋白没有N-糖基化位点,而融合蛋白存在两个N-糖基化位点。而通过对重组蛋白和融合蛋白进行O-糖基化预测,结果发现存在氧糖基化位点。所以,推测蛋白分子量变大很大程度上受到氧糖基化的影响。
2.3 类膨胀素TlEXLX1和CBM-TlEXLX1酶学性质测定 2.3.1 类膨胀素TlEXLX1和CBM-TlEXLX1最适温度和最适pH重组蛋白TlEXLX1和融合蛋白CBM-TlEXLX1的最适温度均为60℃(图 2-A),最适pH均为6.0(图 2-B)。重组酶TlEXLX1在50Ü和70℃下均有90%以上的酶活,而突变体CBM-TlEXLX1在在50℃和70℃下都保持着80%以上的酶活。在pH在4.0-7.0条件下,均能保持85%以上的酶活性。
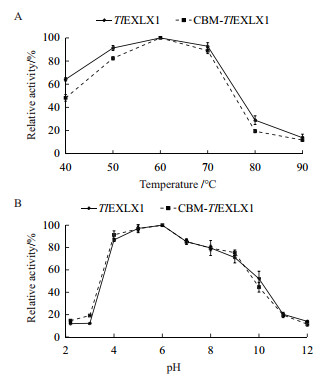
|
| 图 2 温度和pH值对两种蛋白催化活性的影响 |
实验结果(图 3-A)可以看出,在所选择TlEXLX1的浓度范围内,协同效果没有明显差异,10 µg,20 µg的效果相对较好,在90 min时,分别产生了1.26和1.17 mmol/L的还原糖。
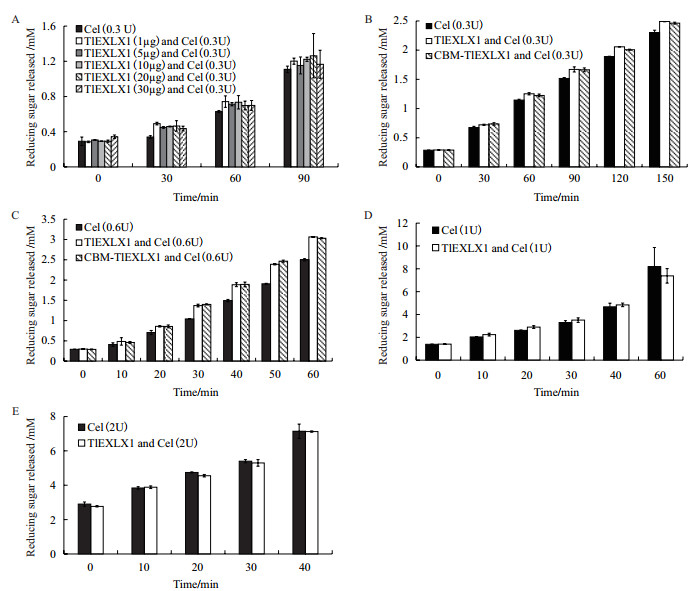
|
| 图 3 TlEXLX1和CBM-TlEXLX1与商品纤维素酶协同效果 A:不同时间点,用不同量的TlEXLX1处理Avicel后与0.3 U的商品纤维素酶的协同;B:不同时间点,用10 μg的TlEXLX1和CBM-TlEXLX1处理Avicel后与0.3 U的商品纤维素酶的协同;C:不同时间点,用10 μg的TlEXLX1和CBM-TlEXLX1处理Avicel后与0.6 U的商品纤维素酶的协同;D:不同时间点,用10 μg的TlEXLX1处理Avicel后与1 U的商品纤维素酶的协同;E:不同时间点,用10 μg的TlEXLX1处理Avicel后与2 U的商品纤维素酶的协同 |
接下来选择10 µg的TlEXLX1和CBM-TlEXLX1与0.3 U,0.6 U的商品纤维素酶协同。结果加入0.6 U的纤维素酶比加入0.3 U的协同效果明显。在经过TlEXLX1和CBM-TlEXLX1处理后,加入纤维素酶量为0.3 U时,在150 min时分别产生2.49 mmol/L和2.46 mmol/L的还原糖,略高于没经过膨胀素处理的直接添加0.3 U纤维素酶所产生的2.29 mmol/L的还原糖。而经过TlEXLX1和CBM-TlEXLX1处理后,加入纤维素酶量为0.6 U时,在60 min时分别产生3.06 mmol/L和3.03 mmol/L的还原糖,高于对照直接添加0.6 U纤维素酶产生的2.49 mmol/L的还原糖(图 3-B,C)。
而选择10 µg的TlEXLX1与1 U,2 U的商品纤维素酶协同,由实验结果可知,加入1 U,2 U的商品纤维素酶在1 h内几乎都没有产生协同效果。在60 min时,1 U纤维素酶产生了7.38 mmol/L还原糖,略低于空白产生的8.18 mmol/L还原糖。在反应40 min后,加入2 U的纤维素酶产生7.12 mmol/L几乎等于空白产生的7.14 mmol/L还原糖(图 3-D,E)。
2.3.3 TlEXLX1和CBM -TlEXLX1底物特异性及比活力测定TlEXLX1和CBM-TlEXLX1对地衣多糖有最高的水解活力,比活分别为7.2 U/mg和17.2 U/mg。其次是大麦葡聚糖和昆布多糖,TlEXLX1的比活力为4.4 U/mg、1.6 U/mg,CBM-TlEXLX1对大麦葡聚糖和昆布多糖的比活力为9.4 U/mg、3.7 U/mg。TlEXLX1和CBM-TlEXLX1对CMC-Na、微晶纤维素、葡甘露聚糖等有微弱的活性。突变体相对于野生型对以上3种底物的比活分别提高了2.4倍、2.1倍和2.3倍。可见,CBM区能有效提高类膨胀素对底物的水解能力。
2.4 TlEXLX1和CBM-TlEXLX1扫描电镜实验从扫描电镜的结果可以看到,未经处理的微晶纤维素和滤纸表面很光滑并且很致密,结构性很强。经过重组蛋白TlEXLX1和融合蛋白CBM-TlEXLX1处理过的微晶纤维素(图 4-A-D)和滤纸(图 4-E-H),其表面明显被破坏,变的粗糙,致密的纤维素结构出现断裂,有明显被腐蚀的痕迹。说明了TlEXLX1和CBM-TlEXLX1对滤纸和微晶纤维素致密的结构造成了一定的破坏作用,从而推测更有利于与纤维酶的协同。
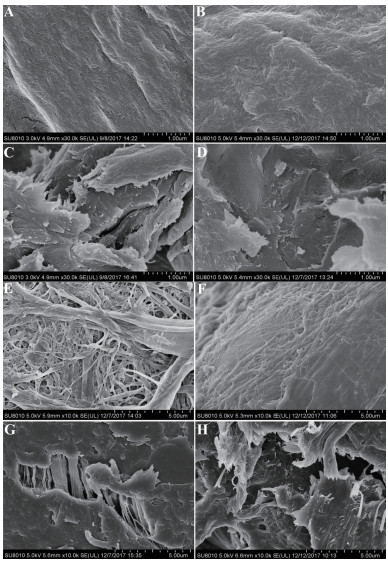
|
| 图 4 重组蛋白TlEXLX1和融合蛋白CBM-TlEXLX1的电镜扫描图 A:底物为Avicel,缓冲液空白对照扫描电镜图;B:BSA处理Avicel后的对照扫描电镜图;C:重组蛋白TlEXLX1处理Avicel后的扫描电镜图;D:融合蛋白CBM-TlEXLX1处理Avicel后的扫描电镜图;E:底物为滤纸,缓冲液空白对照扫描电镜图;F:BSA处理滤纸后的对照扫描电镜图;G:重组蛋白TlEXLX1处理滤纸后的扫描电镜图;H:融合蛋白CBM-TlEXLX1处理滤纸后的扫描电镜图。以上实验条件:缓冲液为柠檬酸-磷酸氢二钠,pH为6.0,反应温度为37℃,反应时间为24 h |
真菌来源的类膨胀素主要是swollenin,细菌来源的有EXLX和swollenin[28]。来源于不同家族膨胀素蛋白的氨基酸序列有着明显不同,相似性只有20%-40%[29]。本实验从嗜热真菌克隆到的类膨胀素基因Tlexlx1与P. digitatum Pd1来源的Expansin-like protein 1相似性仅为56%,而与有晶体结构报道的膨胀蛋白(PDB编号4JVC)只有30%的最高序列相似性。到目前为止,关于来源于真菌类膨胀素EXLX家族基因的相关报道还很少见,因此可以作为研究EXLX类膨胀素的新颖性材料。通过多序列比对,发现该基因具有典型膨胀素的结构特点[13-14],但是其N端有一段未知区域,这与大多数真菌来源的类膨胀素有着明显不同。Swollenin的N端存在一个非常保守的CBM区,CBM区通常有利于swollenin蛋白更好的结合到纤维素底物上[16],而在Tlexlx1基因中有一段未知结构和功能区域而缺少类似CBM区。该段未知区域的功能我们将在今后的工作中继续研究。我们将swollenin的N端CBM区与Tlexlx1的N端融合发现,融合蛋白CBM-TlEXLX1相对于野生型TlEXLX1对葡聚糖酶等底物的水解活力提高了2-2.4倍。因此,可以推断CBM的加入能更好的的结合底物并充分发挥水解功能。
在已报道的4类膨胀素EXPA、EXPB、EXLA和EXLB中,多数EXPA和EXPB家族的成员在体外都具有膨胀素活性,EXLA和EXLB虽然具有典型的膨胀素的结构(包含两个结构域),其成员还没有发现有类似膨胀素的活性[30]。本实验从嗜热真菌T. leycettanus JCM12802成功克隆到了一个类膨胀素基因Tlexlx1,成功在毕赤酵母中进行了表达,纯化的重组酶具有水解葡聚糖底物的活力,及微弱的滤纸和微晶纤维素的水解活力。在大多数报道的膨胀素文献中,均没有发现水解活性。例如,来源于Trichoderma harzianum(ThSwo)[31],Trichoderma reesei(TrSwo1),Bacillus subtilis(BsEXLX1)[28]以及来源于白腐真菌裂褶菌属(ScExlx1)[32]。
类膨胀素TlEXLX1和商品纤维素酶混合反应表现出一定的协同作用。通过两步法用TlEXLX1对微晶纤维素先进行处理,然后再用纤维素酶处理,发现能够提高纤维酶水解作用,也证明存在协同作用。根据报道,两步法能够克服酶对纤维素样本的竞争,并且通过洗脱被膨胀素处理的底物,能够得更多利于纤维素酶结合的位点。类膨胀素蛋白能够与纤维素进行可逆的结合,因此两步法不仅能够更有利于酶与底物的结合,同时还能释放更多的结合位点[33]。经过预处理后再用纤维素酶水解比膨胀素和纤维素酶同时反应水解效应增强[6]。扫描电镜的实验也证实了类膨胀素TlEXLX1对微晶纤维素和滤纸有一定松散和破坏作用。之前的文献也报道了类膨胀素swollenin也具有类似的作用效果[31]。
报道称植物膨胀素在异源系统中以活性的形式表达是非常困难的[24],本研究通过异源表达,获得了真菌来源的与植物膨胀素类似作用的蛋白。同时TlEXLX1还具有水解活性,为膨胀素的研究开辟了新的途径,也为EXLX支架蛋白的分子工程开辟了道路,以阐明蛋白活性的结构需求。TlEXLX1还能够与纤维素酶协同配合将木质纤维素有效转化成可发酵的糖,提高其水解效率,具有重要的工业应用前景。今后我们将从蛋白结构、分子进化、作用机制等都方面对其进行深入的研究。
4 结论本实验从嗜热真菌T. leycettanus JCM12802中成功克隆得到一个类膨胀素基因Tlexlx1,并成功构建融合基因CBM-Tlexlx1。TlEXLX1和CBM-TlEXLX1成功在毕赤酵母中表达,并对其进行性质测定。
TlEXLX1和CBM-TlEXLX1对地衣多糖有最高的水解活力,比活分别为7.2 U/ mg和17.2 U/mg,对CMC-Na和微晶纤维素均有微弱的水解活性,以地衣多糖为底物的最适pH和最适温度均为6.0和60℃。类膨胀素与商品纤维素酶有一定的协同作用,使用10 µg类膨胀素TlEXLX1和0.6 U商品纤维素酶处理微晶纤维素,协同效果最好。
通过扫描电镜观察,TlEXLX1和CBM-TlEXLX1对微晶纤维素和滤纸的表面有破坏作用。综上,该真菌来源的类膨胀素TlEXLX1(expansin)有很好的新颖性并在协同降解纤维素上有潜在的应用价值。
| [1] |
Zhang L, Ruan D, Zhou J. Structure and properties of regenerated cellulose films prepared from cotton linters in NaOH/Urea aqueous solution[J]. Industrial & Engineering Chemistry Research, 2016, 40(25): 5923-5928. |
| [2] |
Meng X, Ragauskas AJ. Recent advances in understanding the role of cellulose accessibility in enzymatic hydrolysis of lignocellulosic substrates[J]. Current Opinion in Biotechnology, 2014, 27(6): 150-158. |
| [3] |
Singh P, Suman A, Tiwari P, et al. Biological pretreatment of sugarcane trash for its conversion to fermentable sugars[J]. World Journal of Microbiology & Biotechnology, 2008, 24(5): 667-673. |
| [4] |
Suwannarangsee S, Bunterngsook B, Arnthong J, et al. Optimisation of synergistic biomass-degrading enzyme systems for efficient rice straw hydrolysis using an experimental mixture design[J]. Bioresource Technology, 2012, 119(7): 252-261. |
| [5] |
Baker JO, King MR, Adney WS, et al. Investigation of the cell-wall loosening protein expansin as a possible additive in the enzymatic saccharification of lignocellulosic biomass[J]. Applied Biochemistry & Biotechnology, 2000, 84-86(1-9): 217. |
| [6] |
Kang K, Wang S, Lai G, et al. Characterization of a novel swollenin from Penicillium oxalicumin facilitating enzymatic saccharification of cellulose[J]. Bmc Biotechnology, 2013, 13(1): 1-9. DOI:10.1186/1472-6750-13-1 |
| [7] |
Quiroz-Castañeda RE, Martínez-Anaya C, Cuervo-Soto LI, et al. Loosenin, a novel protein with cellulose-disrupting activity from Bjerkandera adusta[J]. Microbial Cell Factories, 2011, 10(1): 1-9. DOI:10.1186/1475-2859-10-1 |
| [8] |
Qin L, Kudla U, Roze EH, et al. Plant degradation:a nematode expansin acting on plants[J]. Nature, 2004, 427(6969): 30. DOI:10.1038/427030a |
| [9] |
Beeson WT, Phillips CM, Cate JH, et al. Oxidative cleavage of cellulose by fungal copper-dependent polysaccharide monooxygenases[J]. Journal of the American Chemical Society, 2012, 134(2): 890. DOI:10.1021/ja210657t |
| [10] |
Baccelli I, Luti S, Bernardi R, et al. Cerato-platanin shows expansin-like activity on cellulosic materials[J]. Applied Microbiology & Biotechnology, 2014, 98(1): 175-184. |
| [11] |
Mcqueen-Mason S, Cosgrove DJ. Disruption of hydrogen bonding between plant cell wall polymers by proteins that induce wall extension[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(14): 6574-6578. DOI:10.1073/pnas.91.14.6574 |
| [12] |
Andberg M, Penttilä M, Saloheimo M. Swollenin from Trichoderma reesei exhibits hydrolytic activity against cellulosic substrates with features of both endoglucanases and cellobiohydrolases[J]. Bioresource Technology, 2015, 181: 105-113. DOI:10.1016/j.biortech.2015.01.024 |
| [13] |
Kende H, Bradford K, Brummell D, et al. Nomenclature for members of the expansin superfamily of genes and proteins[J]. Plant Molecular Biology, 2004, 55(3): 311-314. DOI:10.1007/s11103-004-0158-6 |
| [14] |
Cho HT, Kende H. Expression of expansin genes is correlated with growth in deepwater rice[J]. Plant Cell, 1997, 9(9): 1661. DOI:10.1105/tpc.9.9.1661 |
| [15] |
Georgelis N, Nikolaidis N, Cosgrove DJ. Biochemical analysis of expansin-like proteins from microbes[J]. Carbohydrate Polymers, 2014, 100(2): 17-23. |
| [16] |
Saloheimo M, Paloheimo M, Hakola S, et al. Swollenin, a Trichoderma reesei protein with sequence similarity to the plant expansins, exhibits disruption activity on cellulosic materials[J]. Febs Journal, 2010, 269(17): 4202-4211. |
| [17] |
Wang M, Cai J, Huang L, et al. High-level expression and efficient purification of bioactive swollenin in Aspergillus oryzae[J]. Applied Biochemistry & Biotechnology, 2010, 162(7): 2027-2036. |
| [18] |
Chen XA, Ishida N, Todaka N, et al. Promotion of efficient Saccharification of crystalline cellulose by Aspergillus fumigatus Swo1[J]. Applied & Environmental Microbiology, 2010, 76(8): 2556. |
| [19] |
Lee HJ, Lee S, Ko HJ, et al. An expansin-like protein from Hahella chejuensis, binds cellulose and enhances cellulase activity[J]. Molecules & Cells, 2010, 29(4): 379-385. |
| [20] |
Kudla U, Qin L, Milac A, et al. Origin, distribution and 3D-modeling of Gr-EXPB1, an expansin from the potato cyst nematode Globodera rostochiensis[J]. Febs Letters, 2005, 579(11): 2451. DOI:10.1016/j.febslet.2005.03.047 |
| [21] |
Hubbard NL, Pharr DM, Huber SC. Sucrose phosphate synthase and other sucrose metabolizing enzymes in fruits of various species[J]. Physiologia Plantarum, 1991, 82(2): 191-196. DOI:10.1111/ppl.1991.82.issue-2 |
| [22] |
Harada T, Torii Y, Morita S, et al. Cloning, characterization, and expression of xyloglucan endotransglucosylase/hydrolase and expansin genes associated with petal growth and development during carnation flower opening[J]. Journal of Experimental Botany, 2011, 62(2): 815-823. DOI:10.1093/jxb/erq319 |
| [23] |
Magneschi L, Kudahettige RL, Alpi A, et al. Expansin gene expression and anoxic coleoptile elongation in rice cultivars[J]. Journal of Plant Physiology, 2009, 166(14): 1576-1580. DOI:10.1016/j.jplph.2009.03.008 |
| [24] |
Lizana XC, Riegel R, Gomez LD, et al. Expansins expression is associated with grain size dynamics in wheat(Triticum aestivum L.)[J]. Journal of Experimental Botany, 2010, 61(4): 1147-1157. DOI:10.1093/jxb/erp380 |
| [25] |
Georgelis N, Tabuchi A, Nikolaidis N, et al. Structure-function analysis of the bacterial expansin EXLX1[J]. J Biol Chem, 2011, 286(19): 16814-1623. DOI:10.1074/jbc.M111.225037 |
| [26] |
Nikolaidis N, Doran N, Cosgrove DJ. Plant expansins in bacteria and fungi:evolution by horizontal gene transfer and independent domain fusion[J]. Molecular Biology & Evolution, 2014, 31(2): 376-386. |
| [27] |
Andberg M, Penttilä M, Saloheimo M. Swollenin from Trichoderma reesei exhibits hydrolytic activity against cellulosic substrates with features of both endoglucanases and cellobiohydrolases[J]. Bioresour Technol, 2015, 181: 105-113. DOI:10.1016/j.biortech.2015.01.024 |
| [28] |
Wang W, Liu C, Ma Y, et al. Improved production of two expansin-like proteins in Pichia pastoris, and investigation of their functional properties[J]. Biochemical Engineering Journal, 2014, 84(3): 16-27. |
| [29] |
任明玄, 李凤兰, 胡宝忠. 膨胀素的基因家族及功能[J]. 安徽农业科学, 2011, 39(20): 11991-11992. DOI:10.3969/j.issn.0517-6611.2011.20.003 |
| [30] |
金漫, 王轲, 晏月明. 植物细胞壁松弛蛋白——膨胀素[J]. 生物技术通报, 2010(8): 7-11. |
| [31] |
Santos CA, Ferreirafilho J A, O'Donovan A, et al. Production of a recombinant swollenin from Trichoderma harzianum in Escherichia coli and its potential synergistic role in biomass degradation[J]. Microbial Cell Factories, 2017, 16(1): 83. DOI:10.1186/s12934-017-0697-6 |
| [32] |
Chen XA, Ishida N, Todaka N, et al. Promotion of efficient Saccharification of crystalline cellulose by Aspergillus fumigatus Swo1[J]. Applied & Environmental Microbiology, 2010, 76(8): 2556. |
| [33] |
Liu X, Liu C, Ma Y, et al. Heterologous expression and functional characterization of a novel cellulose-disruptive protein LeEXP2 from Lycopersicum esculentum[J]. Journal of Biotechnology, 2014, 186: 148-155. DOI:10.1016/j.jbiotec.2014.07.013 |




