鱼类含有很多促进人类健康的化合物,如优质蛋白质,维生素(A、D、E和B12),硒和碘以及对心脏有益的多不饱和脂肪酸,尤其是二十碳五烯酸和二十二碳六烯酸对心律失常,癌症和高血压患者非常有益,并且在维持神经功能和预防某些精神疾病方面发挥重要作用[1]。其中,生活在水温常年低于20℃的额尔齐斯河流域的新疆冷水鱼类,因其肉质鲜美和独特的口感广受消费者青睐。因此,野生冷水鱼类已经不能满足人们日益增长的消费需求,人们开始对冷水鱼进行人工养殖。然而,为了预防和治疗在渔业养殖中发生的疾病,抗生素被广泛应用于渔业产品。研究显示发展中国家90%的水产养殖业存在抗生素使用监督不完善,这导致抗生素污染水体,此外,水产养殖企业通常位于河口,来自人类和动物粪便中的抗生素耐药菌株便也会污染水体[2-3]。水中抗生素残留的选择性压力会诱导抗生素耐药菌的形成,因此,水生环境中的抗生素残留是一个潜在的危害。水中生活的鱼类是较容易受到生存水体污染的渔业产品,如栖息于水中的耐药细菌等,而食源性耐药细菌可以通过食物链在人类肠道定殖,这使得渔业产品中抗生素耐药性的问题很令人关注。
Enterococcus是一种能够耐受许多极端环境[4],且自然界广泛分布的细菌,如植物、动物(包括人类)胃肠道、土壤、海水、淡水及其他废水等[5-7]。不仅如此,肠球菌通过质粒、染色体或携带抗性基因的转座子,能够快速高效的获得对几乎所有临床用抗生素的耐药性[8]。如临床开始使用氯霉素,红霉素和四环素后,肠球菌很快就出现了对它们的耐药性,在某些案例中甚至达到了阻止了它们在临床上基于经验主义治疗的使用。美国环境保护署将肠球菌作为水体污染程度的指示菌[9],因此,肠球菌是一种较为理想的用来评估渔业产品抗生素耐药性的指示菌。本研究以E. faecium为代表性菌株对新疆阿勒泰的冷水鱼进行抗生素耐药及毒力因子的检测,评估其食品安全性,以期为我国水产养殖业的抗生素耐药性扩散问题的评估提供理论参考依据。
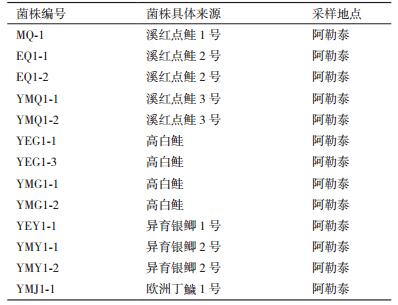
1 材料与方法 1.1 材料 1.1.1 样品及菌株39条新疆冷水鱼类购买于阿勒泰水产品零售商店(全部为当天捕获),包括1条细鳞鲑;1条高白鲑;3条溪红点鲑;2条欧洲丁鱥;1条大麻哈鱼;1条长颌白鲑;1条东方欧鳊;5条贝尔加雅罗鱼;2条赤眼鳟;1条黑斑狗鱼;1条白斑狗鱼;2条河鲈;3条梭鲈;2条异育银鲫;3条江鳕;1条红尾鱼;1条哲罗鲑;3条鲫鱼和3条草鱼。标记后于4℃车载冰箱内冷藏,12 h内运回实验室。药敏实验用质控菌株Enterococcus faecium(CGMCC 1.2136)购自中国普通微生物菌种保藏管理中心。
1.1.2 培养基改良MRS培养基主要成分为葡萄糖20 g、蛋白胨10 g、牛肉膏10 g、酵母浸提物5 g、柠檬酸二胺2 g、磷酸氢二胺2 g、乙酸钠5 g、MgSO4.7H2O 0.58 g、MnSO4.4H2O 0.25 g、吐温-80 1 mL、蒸馏水1 000 mL(调pH 6.2-6.4);肠球菌琼脂(胆汁七叶苷叠氮钠琼脂)(BAA)培养基购自于青岛高科园海博生物技术有限公司。
1.1.3 主要试剂和仪器12种药敏纸片购自于英国OXOID;PCR反应相关试剂购自于上海生工生物工程有限公司;扩增引物均购自于上海捷瑞生物工程有限公司;DNA分子量maker购于北京全式金生物技术有限公司。高速冷冻离心机为Fresco21型(德国Thermo公司);PCR仪为德国Biometra公司Tprofessional;凝胶成像系统为法国Vilber多功能成像系统;水平电泳仪为美国Bio-Rad公司PowerPac Universal。
1.2 方法 1.2.1 肠球菌分离纯化75%(V/V)酒精擦拭整个冷水鱼体,超净工作台中解刨冷水鱼,移出整个肠道;75%(V/V)酒精擦拭整个肠道的外表面,灭菌解剖剪剖开肠道,收集肠道内溶物和粘液。称取1 g内溶物和粘液与99 mL无菌生理盐水混匀,后取0.1 mL匀浆进行梯度稀释后,涂布在MRS,M17,BAA 3种固体培养基上,置于16℃恒温培养箱中,培养4 d后,连续转接划线培养3次后得到单菌落。
1.2.2 菌株DNA的提取采用urea-SDS-NaOH方法提取菌株DNA[10],保存至-20℃即可进行PCR扩增。
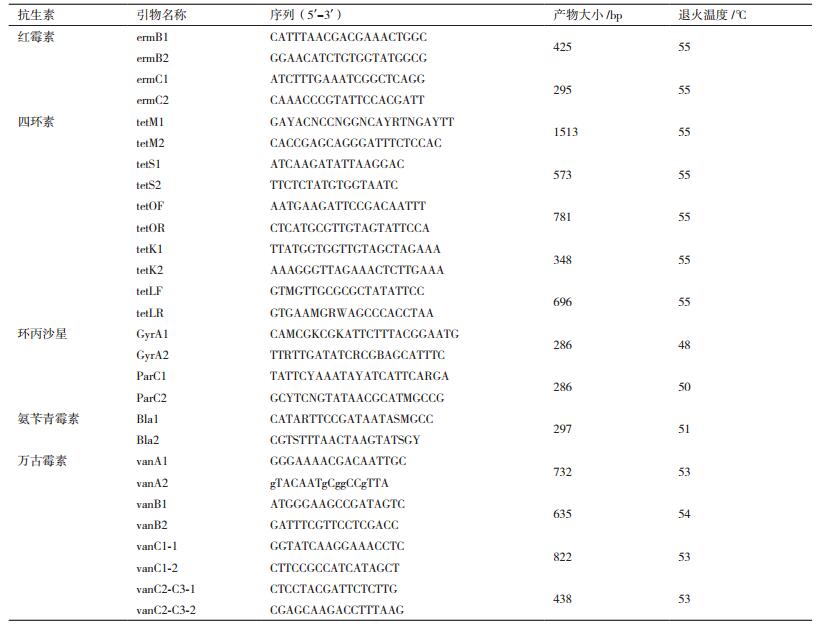
1.2.3 屎肠球菌种、属鉴定将革兰氏染色阳性,接触酶试验阴性且镜检下细胞形态为球状的细菌用Enterococcus sp引物扩增[11],确定其为Enterococci属,以模式菌E. faecium CGMCC 1.2136为阳性对照,再采用E. faecium的种引物扩增[10],确定到具体种(表 1)。
采用通用引物27F/1492R进行PCR扩增,扩增体系(25 μL):2×EsTaq Master Mix 12.5 μL,10 μmol/L 27F 0.5 μL,10 μmol/L 1492R 0.5 μL,100 ng/μL DNA模板1 μL,ddH2O 10.5 μL。PCR反应程序:94℃ 5 min;94℃ 30 s,55℃ 30 s,72℃ 1 min,35次循环;72℃ 10 min。取3 μL PCR扩增产物于1.2%琼脂糖凝胶进行目的片段的电泳检测。PCR产物由上海生工生物科技有限公司进行测序,测序所得16S rDNA序列提交到GenBank数据库,用BLAST进行相关序列的搜索,与GenBank数据库中现有的近缘菌株序列比较,序列的同源性大于等于99%,则可认为菌株为该种。
1.2.5 药敏性测定利用K-B纸片法进行药敏性测定,药敏纸片:氨苄青霉素(AMP,10 μg)、青霉素(P,10 IU)、庆大霉素(CN,120 μg)、链霉素(S,300 μg)、万古霉素(VA,30 μg)、环丙沙星(CIP,5 μg)、四环素(TE,30 μg)、红霉素(E,15 μg)、呋喃妥因(F,300 μg)、利奈唑胺(LZD,30 μg)、氯霉素(C,30 μg)、奎奴普丁(QD,15 μg)。结果参照CLSI标准将供试菌判读为敏感、中介和耐药3种。
1.2.6PCR检测红霉素、四环素、环丙沙星、氨苄青霉素和万古霉素耐药基因[11-14]耐药基因引物序列及退火温度如表 2所示,进行PCR扩增。反应总体系25 μL,2×EsTaq Master Mix 12.5 μL,10 μmol/L引物各0.5 μL,100 ng/μL DNA模板1 μL,ddH2O 10.5 μL。PCR反应程序:94℃ 5 min;94℃ 1 min,合适温度1 min,72℃ 2 min,30个循环;72℃ 10 min。
毒力基因引物序列[15]及相应退火温度见表 3,进行PCR扩增。反应总体系25 μL,2×EsTaq Master Mix 12.5 μL,10 μmol/L引物各0.5 μL,100 ng/μL DNA模板1 μL,ddH2O 10.5 μL。PCR反应程序:95℃ 5 min;94℃ 30 s,合适温度1 min,7℃ 1 min,30个循环;72℃ 10 min。
经镜检、革兰氏染色、接触酶试验及种属特异性引物PCR扩增,从39条冷水鱼的肠道中分离到13株屎肠球菌(表 4)。16S rDNA基因测序结果提交到GenBank数据库,经过比对确定为屎肠球菌。13株屎肠球菌中有5株菌来自3条溪红点鲑,4株菌来自1条高白鲑,3株菌来自2条异育银鲫,最后1株来自1条欧洲丁鱥;其余冷水鱼肠道未分离到屎肠球菌。
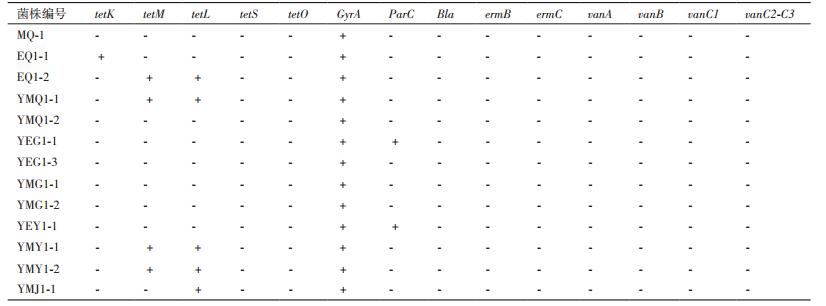
13株屎肠球菌对12种抗生素的敏感性如表 5所示。13株屎肠球菌都对氨苄青霉素、青霉素G、庆大霉素、万古霉素、四环素、红霉素、呋喃妥因、利奈唑胺和奎奴普丁/达福普敏感。除屎肠球菌YEY1-1和YMY1-1外,其余11株屎肠球菌均对环丙沙星中介(即中度敏感)。此外,屎肠球菌MQ-1和EQ1-1对链霉素中介(即中度敏感)。
四环素、环丙沙星、氨苄青霉素、红霉素和万古霉素耐药基因在屎肠球菌中的分布如表 6所示。屎肠球菌都不携带氨苄青霉素耐药基因Bla、红霉素耐药基因ermB、ermC及万古霉素耐药基因vanA、vanB、vanC1和vanC2-C3。所有屎肠球菌都携带环丙沙星耐药基因gyrA,甚至有2株屎肠球菌同时携带另一个环丙沙星耐药基因parC。在5个四环素耐药基因中,屎肠球菌都对tetS和tetO的PCR结果呈阴性,此外仅有1株屎肠球菌携带tetK基因,4株屎肠球菌同时携带tetM和tetL基因,还有1株屎肠球菌仅携带tetL基因。
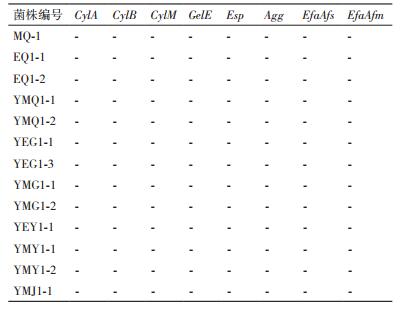
13株屎肠球菌对8种毒力因子的PCR扩增结果(表 7)显示,全部屎肠球菌不携带任何一种毒力因子。
抗生素是一类主要用于治疗各种细菌感染或致病微生物感染类疾病的药物,它具有成本低、使用方便、疗效显著等特点,因而被广泛使用。中国作为世界第一的水产养殖国家,每年的养殖产量为世界养殖总产量的70%左右[16]。这与其养殖模式有很大关系,尤其是密集型渔业养殖模式,但这种养殖模式为致病菌迅速生长提供了很好的契机,导致了水产养殖中细菌性疾病的发病率极高[16-17]。因此,抗生素在水产养殖业中被广泛的应用来控制这些疾病,使得渔业产品存在抗生素残留的问题[17]。抗生素残留会诱导耐药菌株的产生,因此许多关于养殖鱼类的研究都报道了耐药菌株[18-19],不仅如此,临床常用抗生素的耐药性也在养殖鱼类中有报道。如Broughton和Walker对广东100条淡水养殖鱼的研究,发现分离的菌株全部对红霉素和氨苄青霉素耐药[20],然而本研究的冷水鱼肠道屎肠球菌对所测试的抗生素均未表现出耐药性。研究表明水产品(鱼、虾等)中的抗生素含量与其生活习性,食物链等级和产品特征有关,因此本研究中13株屎肠球菌的敏感性很可能与冷水鱼生活的水层、底栖生物有关,但其具体原因有待进一步深入研究[21]。
应用在水产养殖中的抗生素种类主要有喹诺酮类、四环素类、大环内酯类、氨基糖苷类等[16],因此,本研究检测了环丙沙星、四环素、红霉素、万古霉素及氨苄青霉素的耐药基因。其中喹诺酮类抗生素(如环丙沙星,诺氟沙星等)是水产养殖业中使用率很高的抗生素,因此其在水产养殖业中的检出率很高[22]。Liu等[17]对234份中国渔业产品抗生素残留的研究进行统计分析后,发现渔业产品对6个种类的32种不同抗生素呈阳性,其中喹诺酮类抗生素是最常检出的抗生素,分析抗生素具体种类发现,最少有10例研究对环丙沙星呈阳性。然而本研究中的屎肠球菌对环丙沙星并未表现出耐药性,该结果很可能与我国政府在2016年开始对食用动物使用部分诺酮类药物进行管理和控制,其使用量在水产养殖业的减少有关[23]。尽管没有检测到耐药性,但屎肠球菌均携带环丙沙星耐药基因,这一现象应该引起人们的关注,因此,加强抗生素在水产养殖业中的合理及规范化使用很有必要。
本研究肠球菌的抗生素耐药表型及常见抗生素耐药基因的检测结果表明冷水鱼的安全性较高,不仅如此,13株菌不携带所检测的任何一种毒力因子,而研究发现肠球菌要引起感染,必须携带毒力因子帮助其定殖宿主组织并逃避宿主免疫防御机制[24],这又进一步说明了冷水鱼的安全性,因此冷水鱼的消费对消费者身体健康并不存在安全隐患。
4 结论对13株阿勒泰冷水鱼肠道屎肠球菌进行了耐药性、常用抗生素的耐药基因、毒力基因的分析。13株屎肠球菌不携带任何毒力基因,且并未表现出对测试抗生素的耐药性。此外,常用抗生素耐药基因的整体检出率很低。综上所述,认为新疆阿勒泰冷水鱼食品安全性很高,对消费者的身体健康不具有潜在危害。
| [1] |
Dib AL, Agabou A, Chahed A, et al. Isolation, molecular characterization and antimicrobial resistance of enterobacteriaceae isolated from fish and seafood[J]. Food Control, 2018, 88: 54-60. DOI:10.1016/j.foodcont.2018.01.005 |
| [2] |
Boss R, Overesch G, Baumgartner A. Antimicrobial resistance of Escherichia coli, Enterococci, Pseudomonas aeruginosa, and Staphylococcus aureus from raw fish and seafood imported into Switzerland[J]. J Food Prot, 2016, 79(7): 1240-1246. DOI:10.4315/0362-028X.JFP-15-463 |
| [3] |
Harwood VJ, Delahoya NC, Ulrich RM, et al. Molecular confirmation of Enterococcus faecalis and E-faecium from clinical, faecal and environmental sources[J]. Letters in Applied Microbiology, 2004, 38(6): 476-482. DOI:10.1111/lam.2004.38.issue-6 |
| [4] |
Moreno MRF, Sarantinopoulos P, Tsakalidou E, et al. The role and application of enterococci in food and health[J]. International Journal of Food Microbiology, 2006, 106(1): 1-24. DOI:10.1016/j.ijfoodmicro.2005.06.026 |
| [5] |
Boehm AB, Grant SB, Kim JH, et al. Decadal and shorter period variability of surf zone water quality at Huntington Beach, California[J]. Environmental Science & Technology, 2002, 36(18): 3885-3892. |
| [6] |
Svec P, Vancanneyt M, Devriese LA, et al. Enterococcus aquimarinus sp nov., isolated from sea water[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55: 2183-2187. DOI:10.1099/ijs.0.63722-0 |
| [7] |
Byappanahalli MN, Nevers MB, Korajkic A, et al. Enterococci in the environment[J]. Microbiology and Molecular Biology Reviews, 2012, 76(4): 685-706. DOI:10.1128/MMBR.00023-12 |
| [8] |
Chajęcka-Wierzchowska W, Zadernowska A, Łaniewska-Trokenheim Ł. Virulence factors, antimicrobial resistance and biofilm formation in Enterococcus spp. isolated from retail shrimps[J]. LWT-Food Science and Technology, 2016, 69: 117-122. DOI:10.1016/j.lwt.2016.01.034 |
| [9] |
Albertson OE. Changes in the biochemical oxygen demand procedure in the 21st edition of Standard Methods for the Examination of Water and Wastewater[J]. Water Environment Research, 2007, 79(4): 453-455. DOI:10.2175/106143006X116083 |
| [10] |
Singh AK, Ramesh A. Evaluation of a facile method of template DNA preparation for PCR-based detection and typing of lactic acid bacteria[J]. Food Microbiology, 2009, 26(5): 504-513. DOI:10.1016/j.fm.2009.03.006 |
| [11] |
Ortigosa M, Irigoyen A, Urdin M, et al. Sources of enterococci in Idiazábal-type cheese[J]. International Journal of Food Microbiology, 2008, 125(2): 146-152. DOI:10.1016/j.ijfoodmicro.2008.03.035 |
| [12] |
Guo H, Pan L, Li L, et al. Characterization of antibiotic resistance genes from Lactobacillus isolated from traditional dairy products[J]. Journal of Food Science, 2017, 82(3): 724-730. |
| [13] |
Gevers D, Danielsen M, Huys G, et al. Molecular characterization of tet(M)genes in Lactobacillus isolates from different types of fermented dry sausage[J]. Appl Environ Microbiol, 2003, 69(2): 1270-1275. DOI:10.1128/AEM.69.2.1270-1275.2003 |
| [14] |
Comunian R, Daga E, Dupré I, et al. Susceptibility to tetracycline and erythromycin of Lactobacillus paracasei strains isolated from traditional Italian fermented foods[J]. International Journal of Food Microbiology, 2010, 138(1-2): 151-156. DOI:10.1016/j.ijfoodmicro.2009.11.018 |
| [15] |
Eaton TJ, Gasson MJ. Molecular screening of Enterococcus virulence determinants and potential for genetic exchange between food and medical isolates[J]. Appl Environ Microbiol, 2001, 67(4): 1628-1635. DOI:10.1128/AEM.67.4.1628-1635.2001 |
| [16] |
张骞月, 赵婉婉, 吴伟. 水产养殖环境中抗生素抗性基因污染及其研究进展[J]. 中国农业科技导报, 2015, 17(6): 125-134. |
| [17] |
Liu X, Steele JC, Meng XZ. Usage, residue, and human health risk of antibiotics in Chinese aquaculture:A review[J]. Environ Pollut, 2017, 223: 161-169. DOI:10.1016/j.envpol.2017.01.003 |
| [18] |
Conti GO, Copat C, Wang Z, et al. Determination of illegal antimicrobials in aquaculture feed and fish:An ELISA study[J]. Food Control, 2015, 50: 937-941. DOI:10.1016/j.foodcont.2014.10.050 |
| [19] |
Novais C, Campos J, Freitas AR, et al. Water supply and feed as sources of antimicrobial-resistant Enterococcus spp. in aquacultures of rainbow trout(Oncorhyncus mykiss), Portugal[J]. Science of The Total Environment, 2018, 625: 1102-1112. DOI:10.1016/j.scitotenv.2017.12.265 |
| [20] |
Broughton EI, Walker DG. Walker, Prevalence of antibiotic-resistant Salmonella in fish in Guangdong, China[J]. Foodborne Pathogens and Disease, 2009, 6(4): 519-521. DOI:10.1089/fpd.2008.0196 |
| [21] |
Liu X, Lu S, Guo W, et al. Antibiotics in the aquatic environments:A review of lakes, China[J]. Science of The Total Environment, 2018, 627: 1195-1208. DOI:10.1016/j.scitotenv.2018.01.271 |
| [22] |
Rico A, Phu TM, Satapornvanit K, et al. Use of veterinary medicines, feed additives and probiotics in four major internationally traded aquaculture species farmed in Asia[J]. Aquaculture, 2013, 412(2): 231-243. |
| [23] |
中华人民共和国农业部兽医局.中华人民共和国农业部公告第2292号[Z]. 2015-09-07.
|
| [24] |
Hammad AM, Shimamoto T, Shimamoto T. Genetic characterization of antibiotic resistance and virulence factors in Enterococcus spp. from Japanese retail ready-to-eat raw fish[J]. Food Microbiology, 2014, 38: 62-66. DOI:10.1016/j.fm.2013.08.010 |