溶菌酶广泛存在于动物、植物和微生物中,是一种能够水解致病菌中黏性多糖的碱性酶,被认为是体内免疫保护系统的必要组成成分;也是甲壳动物非特异性免疫系统的重要组分,在其免疫防御中发挥重要作用[1-2]。溶菌酶通过破坏细胞壁中的N-乙酰胞壁酸与N-乙酰氨基葡萄糖之间的β-1,4糖苷键而使细胞壁破裂,进而致死细菌。溶菌酶主要分为6种类型:C型(Chicken-type)溶菌酶、G型(Goose-type)溶菌酶、无脊椎动物型溶菌酶、植物溶菌酶、真菌和细菌溶菌酶、T4噬菌体溶菌酶[3]。其中C型溶菌酶已经在昆虫、爬行动物、鸟类和哺乳动物中被发现[4-5],此外,在甲壳动物中也发现有C型溶菌酶[6-7]。
目前,就水产生物而言,在三疣梭子蟹(Portunus trituberculatus)[8]、中国明对虾(Fenneropenaeus chinensis)[9]和凡纳滨对虾(Litopenaeus vannamei)[10]等无脊椎动物中已发现存在C型溶菌酶基因;同时,在斑点叉尾鮰(Ictalurus punctatus)[11]、虹鳟(Oncorhynchus mykiss)[12]、点带石斑鱼(Epinephelus coioides)和赤点石斑鱼(Epinephelus akaara)[13-14]等硬骨鱼中也发现了C型溶菌酶基因。迄今为止,关于水产生物来源的C型溶菌酶在原核细胞中表达的研究报道较多,例如,日本对虾(Penaeus japonicus)[15]、仿刺参(Apostichopus japonicus)[16]、赤点石斑鱼[14]和斑点叉尾鮰[17]的C型溶菌酶已经在大肠杆菌中得到表达;张辉等[8]报道了三疣梭子蟹C型溶菌酶在大肠杆菌中的表达,且表达产物对溶藻弧菌(Vibrio alginolyticus)、哈维氏弧菌(Vibrio harveyi)和鳗弧菌(Vibrio anguillarum)均有一定的抑菌作用。
然而,原核表达系统存在众所周知的缺点,如对表达产物不能进行翻译后加工修饰,以致表达的目的蛋白会形成不溶性的包涵体,需要经过复杂的复性才能恢复构象和活性。与原核表达系统相比,巴斯德毕赤酵母(Pichia pastoris)作为真核表达的宿主,其有助于表达产物空间构象的形成,进而获得具有生物学活性的重组蛋白,并可实现目的蛋白的细胞外分泌表达以方便表达产物的后续处理,是目前公认的外源蛋白表达的良好宿主[18]。
近几年来,我们先后开展了对于尼罗罗非鱼(Oreochromis niloticus)[19]、大西洋庸鲽(Hippoglossus hippoglossus L.)[20]、斑点叉尾鮰[21-24]和斑马鱼(Danio rerio)[25]等数种鱼类中不同抗菌肽的重组DNA表达研究;另一方面,以甲壳动物三疣梭子蟹为研究对象,对其PtCrustin 2和PtCrustin 3抗菌肽实现了在毕赤酵母中的重组DNA表达[26-27]。在上述研究的基础上,最近我们聚焦于鱼类来源溶菌酶的真核表达研究,已经对鲤鱼(Cyprinus carpio)g型溶菌酶基因进行了cDNA克隆,并通过毕赤酵母表达系统获得了重组g型溶菌酶[28]。此外,来自斑点叉尾鮰的C型溶菌酶基因也被克隆,并实现了其在毕赤酵母中的表达,重组C型溶菌酶显示了对于枯草牙孢杆菌(Bacillus subtilis)的抑菌活性[29]。
三疣梭子蟹(Portunus trituberculatus)广泛分布于日本沿海、韩国和中国等国家,是一种重要的商业蟹。目前,随着养殖规模的不断扩大以及养殖集约化程度的不断提高,由细菌、真菌和病毒等引起的各种病害给三疣梭子蟹的养殖带来巨大的损失,由此造成抗生素的滥用也埋下了食品安全的隐患。因此,寻求一种替代或部分替代抗生素的天然免疫因子成为从源头解决这一问题的关键。据此,本研究以三疣梭子蟹C型溶菌酶基因作为重组DNA表达的目标,以期通过毕赤酵母表达系统获得具有良好抑菌活性的三疣梭子蟹重组C型溶菌酶,为开发用于三疣梭子蟹安全养殖的天然抑菌剂提供技术途径。为了实现上述目标,首先通过RT-PCR从其鳃中分离编码C型溶菌酶的cDNA片段,以pPICZαA为表达载体,构建重组表达载体;电转至毕赤酵母X-33后,通过博来霉素筛选阳性转化子;通过甲醇诱导表达重组蛋白,并采用固化金属离子亲和层析(IMAC)和MALDI-TOF/TOF分别对表达产物进行纯化和鉴定,最终通过平板涂布法和琼脂糖扩散法检验三疣梭子蟹重组C型溶菌酶的生物学活性。
1 材料与方法 1.1 材料 1.1.1 质粒和菌株克隆载体pMD-19T simple购自TaKaRa公司(日本);毕赤酵母X-33和表达载体pPICZαA购自Invitrogen公司(美国);大肠杆菌DH5α购自天根生物科技有限公司(北京);用于抑菌活性测定的铜绿假单胞菌(Pseudomonas aeruginosa)、枯草芽胞杆菌、副溶血性弧菌(Vibrio parahaemolyticus)和沙门氏菌(Salmonella lignieres)均为本实验室保藏。
1.1.2 主要试剂限制性内切酶(Xho Ⅰ、Xba Ⅰ、Sac Ⅰ)和T4 DNA连接酶、TaqDNA聚合酶均购自TaKaRa公司;胰蛋白胨、酵母粉购自OXOID公司(英国);YNB(无机氮原)购自BIOSHARP公司(美国);DNA分子量标准、DNA割胶回收试剂盒、质粒提取试剂盒、酵母DNA提取试剂盒等购自天根生物科技有限公司;蛋白质分子量标准购自中科瑞泰生物科技有限公司(北京);Western Blot分析试剂购自康为世纪生物科技有限公司(北京)。
1.1.3 仪器实验所用仪器及型号如下:恒温培养箱(XMT-A7000)、恒温摇床(ZHWY-200H),高速冷冻离心机(CT14RD)、恒温孵育器(Eppendorf,Thermostat plus)、PCR仪(BIO-RAD,PTC-200)、聚丙烯酰胺凝胶电泳仪(BIO-RAD,Power Pac Basic)、电转仪(BIO-RAD,Gene Pulser Xcell)、蛋白质纯化仪(BIO-RAD,Profinia Protein Purification System)、赛多利斯切向回流超滤器(VIVAFLOW200)、酶标仪(BioTek SLFPTAD)。引物合成和DNA测序由上海生物工程有限公司完成。
1.2 方法 1.2.1 目的基因的扩增参考三疣梭子蟹C型溶菌酶的全长cDNA序列(GenBank登录号:FJ589729),设计用于PCR的引物(表 1);以实验室原有的三疣梭子蟹鳃的第一股cDNA为模板[26],通过3次PCR(图 1),扩增含Xho Ⅰ、Xba Ⅰ酶切位点和6×His标签的三疣梭子蟹C型溶菌酶成熟肽基因“ptlyC”。第一次PCR的反应体系和条件:10×Taq Plus Buffer 1 μL、dNTP(2.5 mmol/L)0.8 μL、F1和R1(10 mol/L)各0.2 μL、模板0.3 μL、Taq DNA聚合酶(2.5 U/μL)0.1 μL、dd H2O 7.4 μL;94℃3 min、94℃40 s、57℃ 30 s、72℃1 min、72℃ 5 min,30个循环。第二次PCR:以第一次PCR产物为模板、F2和R2为引物,反应体系和条件同上,除了退火温度改为54.5℃。第三次PCR:以第二次PCR产物为模板、F3和R2为引物,反应体系和条件同第二次PCR。将第3次PCR产物“ptlyC”割胶纯化后与质粒pMD-19T连接后,转入大肠杆菌DH5α感受态细胞中,37℃培养过夜,通过菌落PCR挑取阳性克隆,由上海生工生物工程有限公司测序。
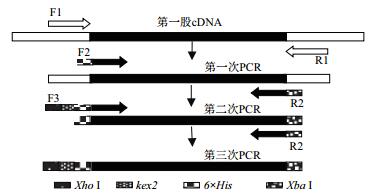
|
| 图 1 PCR扩增目的基因的策略图 |
使用Xho Ⅰ和Xba Ⅰ对重组克隆质粒“pMD-19T-ptlyC”进行双酶切,获得目的片段“ptlyC”;在T4DNA连接酶作用下,将其与被同样酶处理过的表达载体pPICZαA连接(16℃,30 min),转入大肠杆菌感受态细胞DH5α中,37℃培养过夜。通过双酶切和DNA测序验证重组表达载体pPICZαA-ptlyC是否构建成功。
1.2.3 pPICZαA-ptlyC的转化及阳性转化子的筛选重组表达载体“pPICZαA-ptlyC”经Sac Ⅰ酶切后,与毕赤酵母X-33感受态细胞混合(1:8,V/V),转入预冷的0.2 cm电转杯中,冰浴5 min,1.5 kV、25 μF、200 Ω,电击5 ms,立即加入预冷的1 mol/L山梨醇;30℃孵育2 h后,离心将菌体涂布于含100 μg/mL博来霉素的YPD平板(1%酵母提取物、2%胰蛋白胨、2%琼脂粉和2%葡萄糖)上,29℃培养3 d;挑取部分菌落分别接种在含500 μg/mL博来霉素的YPD平板上,筛选高拷贝酵母转化子。同时,将挑取的酵母细胞转接至MM平板(YNB13.4 g/L、甲醇5 mL/L、生物素0.4 mg/L、琼脂15 g/L)上以筛选甲醇利用快速型酵母转化子。对筛选到的酵母转化子提取基因组DNA,采用通用引物5'AOX1和3'AOX1(表 1)进行PCR鉴定。
1.2.4 目的蛋白的诱导表达挑取一个阳性转化子接种于5 mL YPD培养基中扩大培养24 h,取500 μL转接至50 mL BMGY培养基(酵母提取物0.5 g、胰蛋白胨1 g、34% YNB 5 mL、10%甘油5 mL、0.02%生物素100 μL、1 mol/L磷酸钾缓冲液5 mL)培养至OD600为2-6,离心收集菌体,重悬于50 mL BMMY培养基(酵母提取物0.5 g、胰蛋白胨1 g、34%YNB 5 mL、1%甲醇500 μL、0.02%生物素100 μL、1 mol/L磷酸钾缓冲液5 mL)中,每隔24 h补加甲醇至终浓度1%。培养条件:pH6.0、28℃、250 r/min、72 h。每隔12 h收集培养液用于Tricine-SDS-PAGE分析(5%浓缩胶、12%分离胶、0.1 mol/L Tricine)。
1.2.5 表达产物的纯化及Western blot分析挑取1个良好的酵母转化子,对其进行1 000 mL的扩大培养,表达条件同上,培养液经离心(11 000 r/min,15 min)后,收集上清过0.22 μm滤膜,通过切向回流超滤器对其进行超滤浓缩;浓缩液上蛋白质纯化仪,根据仪器使用说明书进行蛋白质纯化,采用BCA法测定蛋白质浓度。纯化产物经Tricine-SDS-PAGE分析后,将凝胶电转至PVDF膜上(100 V,恒压1 h);用TBST漂洗后加入封闭液封闭1 h;再用TBST漂洗后加入抗His标签鼠单克隆抗体,室温孵育2 h;再用TBST漂洗后加入辣根过氧化酶标记山羊抗小鼠IgG,室温孵育1 h;最后用HRP-DAB显色试剂盒显色。
1.2.6 目的蛋白的抑菌活性测定将处于对数生长期的铜绿假单胞菌、枯草芽孢杆菌、副溶血性弧菌和沙门氏菌分别与营养琼脂混合(1:1 000,V/V)倒平板,凝固后打直径为6 mm的孔,加入100 μL纯化的重组ptlyC,37℃培养12 h,观察有无抑菌圈;同时,以100 μL洗脱缓冲液作为阴性对照。另一方面,取100 μL含重组ptlyC的培养液上清与1 μL对数生长期的枯草芽孢杆菌混合,37℃振荡培养2 h后,取30 μL涂布于YPD平板上,37℃培养12 h,观察抑菌效果。同时,以100 μL含空质粒的酵母转化子的培养液上清作为阴性对照。
2 结果 2.1 基于PCR扩增的ptlyC基因及其cDNA序列第一次PCR以三疣梭子蟹鳃的第一股cDNA为模板、F1和R1为引物,获得645 bp的编码三疣梭子蟹C型溶菌酶的全长cDNA(图 2泳道1)。第2次PCR以该全长cDNA为模板、F2和R2为引物,获得652 bp的5'端含6×His标签、3'端含终止密码子TAG和Xba Ⅰ位点的片段(图 2泳道2)。第3次PCR以该片段为模板、F3和R2为引物,扩增到657 bp的5'端含Xho Ⅰ位点和Kex2信号肽酶切位点、3'端含终止密码子TAG和Xba Ⅰ位点的编码三疣梭子蟹C型溶菌酶成熟肽的目的片段“ptlyC”(图 2泳道3)。
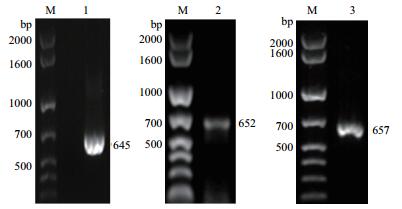
|
| 图 2 三次PCR产物的琼脂糖凝胶电泳 M:DNA分子量标准;1:第1次PCR产物;2:第2次PCR产物;3:第3次PCR产物 |
对目的片段的测序结果(图 3-A)显示,除在5'和3’端添加的相关位点和标签外,ptlyC编码了由205个氨基酸残基组成的三疣梭子蟹C型溶菌酶成熟肽;8个保守的半胱氨酸残基和活性位点的2个残基(谷氨酸和天冬氨酸)位于成熟肽中。
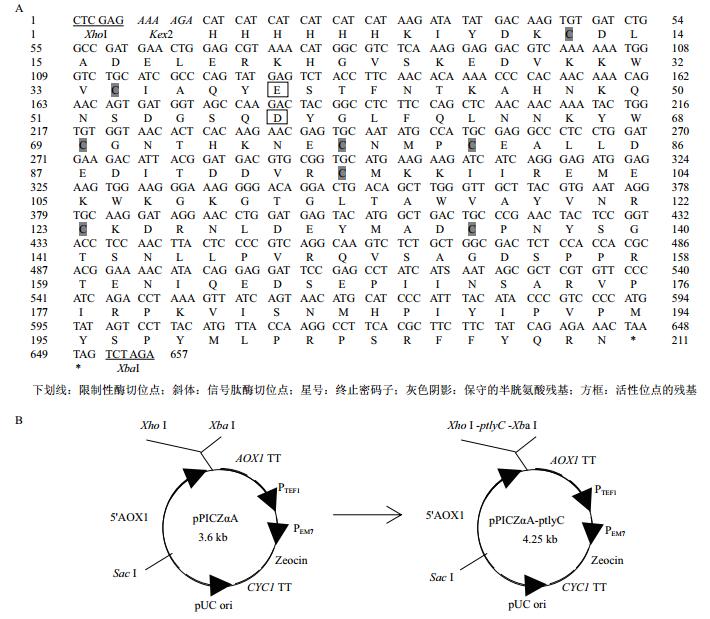
|
| 图 3 目的基因ptlyC的核苷酸及其推断的氨基酸序列(A)和重组表达载体的构建(B) |
重组表达载体pPICZαA-ptlyC的构建如图 3-B所示。采用Xho Ⅰ和Xba Ⅰ对其进行双酶切后,琼脂糖凝胶电泳结果显示,在接近理论分子量657 bp处有一个明显的条带(图 4);此外,DNA测序结果也证明ptlyC与pPICZαA正确连接。
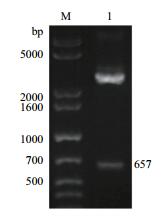
|
| 图 4 重组表达载体pPICZαA-ptlyC的双酶切验证 M:DNA分子量标准;1:Xho Ⅰ和Xba Ⅰ双酶切产物 |
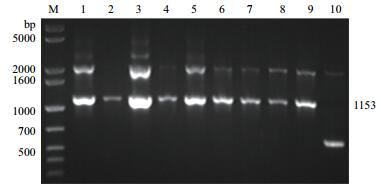
上述pPICZαA-ptlyC电转至毕赤酵母感受态细胞,通过含500 μg/mL博来霉素的YPD和MM平板筛选,得到9株长势良好的酵母转化子;以它们的基因组DNA为模板,采用载体上醇氧化酶AOX1基因的通用引物5'AOX1和3' AOX1进行PCR扩增,结果(图 5)显示:9株酵母菌均在1 153 bp和近2 000 bp处有明显条带,前者为预期的理论分子量位置,证明目的基因ptlyC被成功嵌合进pPICZαA载体;后者为毕赤酵母X-33基因组中也存在醇氧化酶AOX1基因的该两个引物结合位点而扩增的条带。但含pPICZαA的酵母转化子(阴性对照)则未见此条带。
|
| 图 5 高拷贝酵母转化子的PCR鉴定 M:DNA分子量标准;1-9:含pPICZαA-ptlyC的酵母转化子;10:含pPICZαA的酵母转化子 |
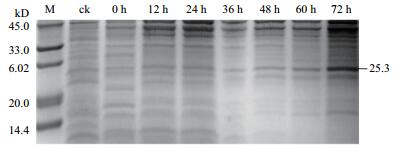
挑取筛选到的高拷贝酵母转化子(图 5,泳道9)于50 mL BMMY培养基中进行诱导表达,结果如图 6 Tricine-SDS-PAGE分析所示,随着表达时间的延长,培养液上清所显示的在理论分子量25.3 kD处的条带逐渐加深,至72 h时最为明显;而含pPICZαA空载体的酵母转化子的培养液上清中未发现该分子量的条带。此结果初步证明重组ptlyC在毕赤酵母X-33中得到成功表达。
|
| 图 6 培养液上清的Tricine-SDS-PAGE分析 M:蛋白质分子量标准;ck:含pPICZαA的酵母转化子的培养液上清 |
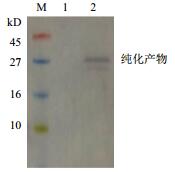
进一步对上述菌株扩大培养后的培养液上清进行IMAC法纯化,得到的纯化产物经Western blot分析显示(图 7),在近27 kD处有明显条带,证明该纯化产物与抗His的鼠单克隆抗体成功杂交;经BCA法测定,纯化产物的浓度为0.21 mg/mL。由于在转膜过程中蛋白质条带可能会发生偏移,以致条带的位置稍偏高。而阴性对照则未见任何条带。Western blot的分析结果进一步证明了重组ptlyC在毕赤酵母X-33中得到成功表达。
|
| 图 7 纯化产物的Western blot分析 M:蛋白分子量标准;1:含pPICZαA的酵母转化子的培养液上清;2:纯化产物 |
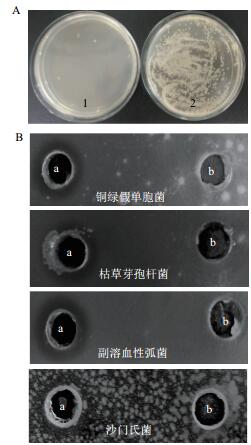
通过菌落计数法考察含重组ptlyC的培养液上清对枯草芽孢杆菌的抑菌活性,结果如图 8-A所示,浓缩30倍的培养液上清(总蛋白质浓度1.9 mg/mL)显示了对枯草芽孢杆菌的明显的抑菌活性。
|
| 图 8 含重组ptlyC的培养液上清(A)和纯化的ptlyC(B)的抑菌活性 1:含重组ptlyC的培养液上清;2:不含重组ptlyC的培养液上清;a:纯化的ptlyC;b:洗脱缓冲液 |
另一方面,通过琼脂糖凝胶扩散法考察了纯化后的重组ptlyC的抑菌活性,结果如图 8-B所示,分别涂有枯草芽孢杆菌、铜绿假单胞菌和副溶血性弧菌的平板上的孔经纯化后的重组ptlyC处理后,均显示了明显的抑菌圈;涂有沙门氏菌的平板虽然菌落分布密集,但亦可辨明有依稀可见的抑菌圈;而用洗脱缓冲液处理的孔则无明显的抑菌圈,由此证明重组ptlyC具有良好的抑制革兰氏阳性和阴性细菌的活性。
3 讨论本研究通过RT-PCR获得编码三疣梭子蟹C型溶菌酶成熟肽的cDNA片段“ptlyC”,并成功构建重组表达载体pPICZαA-ptlyC,电转入毕赤酵母X-33后,在诱导表达阶段,分别使用了以YNB(无机氮)作为氮源的BMM培养基(结果未显示)和有机氮(酵母粉和蛋白胨)作为氮源的BMMY培养基,结果显示基于BMMY培养基培养的菌体的长势及重组蛋白的表达情况更好,因此在后续的扩大培养中使用BMMY培养基进行诱导表达。然而,根据纯化产物的蛋白质浓度估算得到的重组ptlyC的表达量低于1 mg/L,导致如此低的表达量的原因可能与本研究使用的天然cDNA有关,经JAVA Codon Adaption Tool(http://www.jcat.de/Start.jsp)检测,“ptlyC”基因中有80个属于低频密码子(相对适应性低于0.1)。李兵等[30]报道了经密码子优化后在毕赤酵母中表达的重组体黑曲霉阿魏酸酯酶活性较天然cDNA表达的重组蛋白提高了近6倍;Inouye等[31]的研究表明根据人类密码子偏爱性对萤火虫荧光素酶进行密码子优化后,显著提高了其在哺乳动物细胞中的表达水平。因此,进一步的研究将聚焦于对“ptlyC”基因进行密码子优化后的重组DNA表达。
为方便纯化和Western blot分析,本研究设计的重组蛋白在其N端含6×His标签,但无论含重组ptlyC的培养液上清还是纯化的重组ptlyC均显示了抑菌活性,说明N端的6×His标签并不影响重组蛋白的生物学活性;此结果与舒正玉[32]报道的黑曲霉F044脂肪酶基因在大肠杆菌BL21(DE3)和毕赤酵母GS115中表达的结果是一致的,他同样设计了N端含6×His标签的重组蛋白,经大肠杆菌和毕赤酵母表达的重组F044脂肪酶均具有较高的酶活力。值得注意的是,纯化的重组ptlyC除了显示对革兰氏阳性的枯草芽孢杆菌有抑菌活性之外,还对革兰氏阴性的铜绿假单胞菌、副溶血性弧菌和沙门氏菌具有较明显的抑菌活性;张辉等[8]报道的通过原核表达系统获得的重组三疣梭子蟹C型溶菌酶则具有抑制革兰氏阴性的溶藻弧菌、哈维氏弧菌和鳗弧菌的活性。而冯亚东等[29]报道的通过毕赤酵母表达系统获得的重组斑点叉尾鮰C型溶菌酶仅显示对于枯草芽孢杆菌的抑菌活性。三疣梭子蟹与鱼类在C型溶菌酶抑菌谱方面的差异可能源于两者来源的C型溶菌酶在结构上的差异,这种差异与两者分属不同的物种有关,前者为甲壳类动物,而后者属于硬骨鱼类,它们在免疫机制方面存在的客观差异可能导致作为免疫因子的C型溶菌酶在结构和功能上的差异。该方面的研究工作还有待于进一步深入。本研究建立的毕赤酵母表达系统为进一步开发甲壳动物来源的天然抗菌剂奠定了重要的研究基础。
4 结论本研究建立了基于毕赤酵母表达系统的三疣梭子蟹C型溶菌酶的制备方法,其最适的表达条件为:在BMMY培养基中,28℃、250 r/min、pH6.0、1%甲醇,培养72 h。经Tricine-SDS-PAGE分析,重组ptlyC的分子量接近预期的理论分子量(25.3 kD)。抑菌实验证明,重组三疣梭子蟹C型溶菌酶具有抑制革兰氏阳性的枯草芽孢杆菌和革兰氏阴性的铜绿假单胞菌、副溶血性弧菌和沙门氏菌的活性。
| [1] |
Won JL, Paul TB. Isolation and characterization of the lysozyme-encoding gene from the Silkworm bombyx mori[J]. Gene, 1995, 161(2): 199-203. DOI:10.1016/0378-1119(95)00199-G |
| [2] |
郑清梅, 吴锐全, 叶星. 水生动物溶菌酶的研究进展[J]. 上海水产大学学报, 2006, 15(14): 483-487. |
| [3] |
Prager E, Wilson A, Amheim N, et al. Widespread distribution of lysozyme in egg white of birds[J]. J Biol Chem, 1974, 294(22): 7295-7297. |
| [4] |
Menendez-Arias L, Gavilanes JG, Rodriguez R. Amino acid sequence around the cysteine residues of pigeon egg-white lysozyme:comparative study with other type c lysozymes[J]. Comparative Biochemistry and Physiology B, 1985, 82(4): 639-642. DOI:10.1016/0305-0491(85)90500-0 |
| [5] |
McKenzie HA, Shaw DC. The amino acid sequence of equine milk lysozyme[J]. Biochemistry International, 1985, 10(1): 23-31. |
| [6] |
Mai WJ, Hu CQ. Molecular cloning, characterization, expression and antibacterial analysis of a lysozyme homologue from Fenneropenaeus merguiensis[J]. Mol Biol Rep, 2009, 36(6): 1587-1595. DOI:10.1007/s11033-008-9355-8 |
| [7] |
Gao FY, Ye X, Bai JJ, et al. Cloning and expression characterization of lysozyme gene in two freshwater prawns[J]. Acta Hydrobiologica sinica, 2005, 29(6): 615-620. |
| [8] |
张辉, 潘鲁青, 岳峰. 三疣梭子蟹C-型溶菌酶基因的原核表达与活性检测[J]. 中国海洋大学学报:自然科学版, 2013, 43(7): 23-27. |
| [9] |
卜兴江, 杜欣军, 周文杰, 等. 中国明对虾溶菌酶基因克隆、重组表达与性质分析[J]. 生物工程学报, 2008(5): 723-732. DOI:10.3321/j.issn:1000-3061.2008.05.003 |
| [10] |
Nilsenl W, Overo K, Sandsdalen E, et al. Protein purification and gene isolation of chlamysin, a cold-active lysozyme-like with antibacterial activity[J]. FEBS Lett, 1999, 3: 153-158. |
| [11] |
Wang RJ, Feng JB, Li C, et al. Four lysozymes(one c-type and three g-type)in catfish are drastically but differentially induced after bacterial infection[J]. Fish and Shellfish Immunology, 2013, 35(1): 136-145. DOI:10.1016/j.fsi.2013.04.014 |
| [12] |
Dautigny A, Prager EM, Pham-Dinh D, et al. cDNA and amino acid sequences of rainbow trout(Oncorhynchus mykiss)lysozymes and their implications for the evolution of lysozyme and lactalbumin[J]. J Mol Evol, 1991, 32(2): 187-198. DOI:10.1007/BF02515392 |
| [13] |
Wei SN, Huang YH, Cai J, et al. Molecular cloning and characterization of c-type lysozyme gene in orange-spotted grouper, Epinephelus coioides[J]. Fish and Shellfish Immunology, 2012, 33(2): 186-196. DOI:10.1016/j.fsi.2012.03.027 |
| [14] |
Mai W, et al. Cloning and immune characterization of the c-type lysozyme gene in red-spotted grouper, Epinephelus akaara[J]. Fish and Shellfish Immunology, 2014, 36(1): 305-314. DOI:10.1016/j.fsi.2013.11.002 |
| [15] |
杨杰, 丛丽娜, 夏涛, 等. 日本对虾c型溶菌酶的高效重组表达及产物分析[J]. 生物技术通报, 2012(6): 99-105. |
| [16] |
Tian Y, Liang XW, Chang YQ, et al. Expression of c-type lysozyme gene in sea cucumber(Apostichopus japonicus)is highly regulated and time dependent after salt stress[J]. Comparative Biochemistry and Physiology B, 2015, 180: 68-78. |
| [17] |
Pridgeon JW, et al. Chicken-type lysozyme in channel catfish:Expression analysis, lysozyme activity, and efficacy as immunostimulant against Aeromonas hydrophila infection[J]. Fish and Shellfish Immunology, 2013, 35(3): 680-688. DOI:10.1016/j.fsi.2013.05.018 |
| [18] |
Daly R, Hearn MT. Expression of heterologous proteins in Pichia pastoris:A useful experimental tool in protein engineering and production[J]. J Mol Recognit, 2005, 18(3): 119-138. |
| [19] |
李文婧, 陶妍, 赵冬梅, 等. 基于SOE-PCR的两种鱼类hepcidin基因串联及其在毕赤酵母中的表达[J]. 生物工程学报, 2015, 31(5): 682-691. |
| [20] |
李文婧, 陶妍. 大西洋庸鲽(Hippoglossus hippoglossus L.)hipposin抗菌肽在毕赤酵母中的表达及其纯化[J]. 生物学杂志, 2016, 33(3): 15-19. DOI:10.3969/j.issn.2095-1736.2016.03.015 |
| [21] |
宋长丰, 陶妍, 赵冬梅, 等. 斑点叉尾鮰铁调素成熟肽在毕赤酵母中的表达及其抑菌活[J]. 农业生物技术学报, 2015, 23(3): 308-387. |
| [22] |
宋长丰, 李雯, 陶妍. 斑点叉尾鮰LEAP-2和NK-lysin抗菌肽在毕赤酵母中的串联表达[J]. 生物技术, 2015, 25(6): 524-528. |
| [23] |
孙立人, 陶妍, 高北. 斑点叉尾鮰LEAP2成熟肽在毕赤酵母中的表达及其抑菌活性[J]. 上海海洋大学学报, 2017, 26(1): 23-30. |
| [24] |
吴慧, 陶妍. 斑点叉尾鮰NK-lysin成熟肽在毕赤酵母中的表达[J]. 淡水渔业, 2017, 47(1): 78-83. DOI:10.3969/j.issn.1000-6907.2017.01.013 |
| [25] |
Tao Y, Song CF, Li W. Expression of the zebrafish β-defensin 3 mature peptide in Pichia pastoris and its purification and antibacterial activity[J]. Applied Biochemistry and Microbiology, 2017, 53(6): 661-668. DOI:10.1134/S0003683817060126 |
| [26] |
王艳慧, 陶妍. 三疣梭子蟹PtCrustin2成熟肽在毕赤酵母中的表达[J]. 微生物学杂志, 2016, 36(5): 26-31. |
| [27] |
卢希阳, 陶妍. 三疣梭子蟹PtCrustin3成熟肽在毕赤酵母中的重组DNA表达[J]. 山东农业大学学报:自然科学版, 2017, 48(2): 303-309. |
| [28] |
李雯, 陶妍. 鲤鱼g型溶菌酶基因的cDNA克隆及其在毕赤酵母中的表达[J]. 福建农林大学学报:自然科学版, 2017, 46(1): 81-88. |
| [29] |
冯亚东, 陶妍, 李雯, 等. 斑点叉尾鮰C型溶菌酶在毕赤酵母中的表达及其抑菌活性[J]. 生物技术通报, 2017, 33(7): 195-202. |
| [30] |
李兵, 蔡国林, 朱德伟, 等. 黑曲霉阿魏酸酯酶基因密码子优化及在毕赤酵母中的高效表达[J]. 微生物学通报, 2017, 44(5): 1065-1073. |
| [31] |
Inouye S, Sahara-Miura Y, Sato JI, et al. Codon optimization of genes for efficient protein expression in mammalian cells by selection of only preferred human codons[J]. Protein Expression and Purification, 2015, 109: 47-54. DOI:10.1016/j.pep.2015.02.002 |
| [32] |
舒正玉.黑曲霉脂肪酶的酶学性质、基因克隆与表达及结构预测[D].武汉: 华中科技大学, 2007. http://cdmd.cnki.com.cn/Article/CDMD-10487-2009033679.htm
|





