我国水稻种植以籼稻为主,粳稻种植面积较小[1]。但随着生活水平的提高,市场对粳米的需求量日益增长,需要培育出更多优秀品种来满足需求[2-3]。把优异遗传资源应用到育种中,保持粳稻品种的多样性是培育优秀新品种的关键[4]。张立娜等[5]研究表明云南的粳稻地方品种遗传多样性最为丰富,也育成了大量的粳稻系列品种,其中具有优异特性的品种、品系等作为亲本材料常用于育种中。因此,通过对各单位创新的粳稻选育品种的遗传多样性研究,阐明云南不同来源粳稻品种的亲缘关系和遗传结构,了解粳稻品种的生态类型及地理分布,能有效的指导粳稻杂交育种中亲本材料选配,加强育种家间资源共享和种质资源交流,拓宽育种的遗传基础,推动云南粳稻育种发展。
分子标记技术已广泛应用于作物种质资源的遗传多样性研究。其中应用最多的是简单重复序列(Simple sequence repeat,SSR)标记,其具有重复性好、多态性高、数量丰富、呈共显性且广泛分布于基因组等优点[6-7]。本研究精选云南粳稻品种163份,利用SSR分子标记技术研究其遗传多样性,并阐明其遗传结构和亲缘关系,以期为粳稻优质高产育种提供科学支撑。
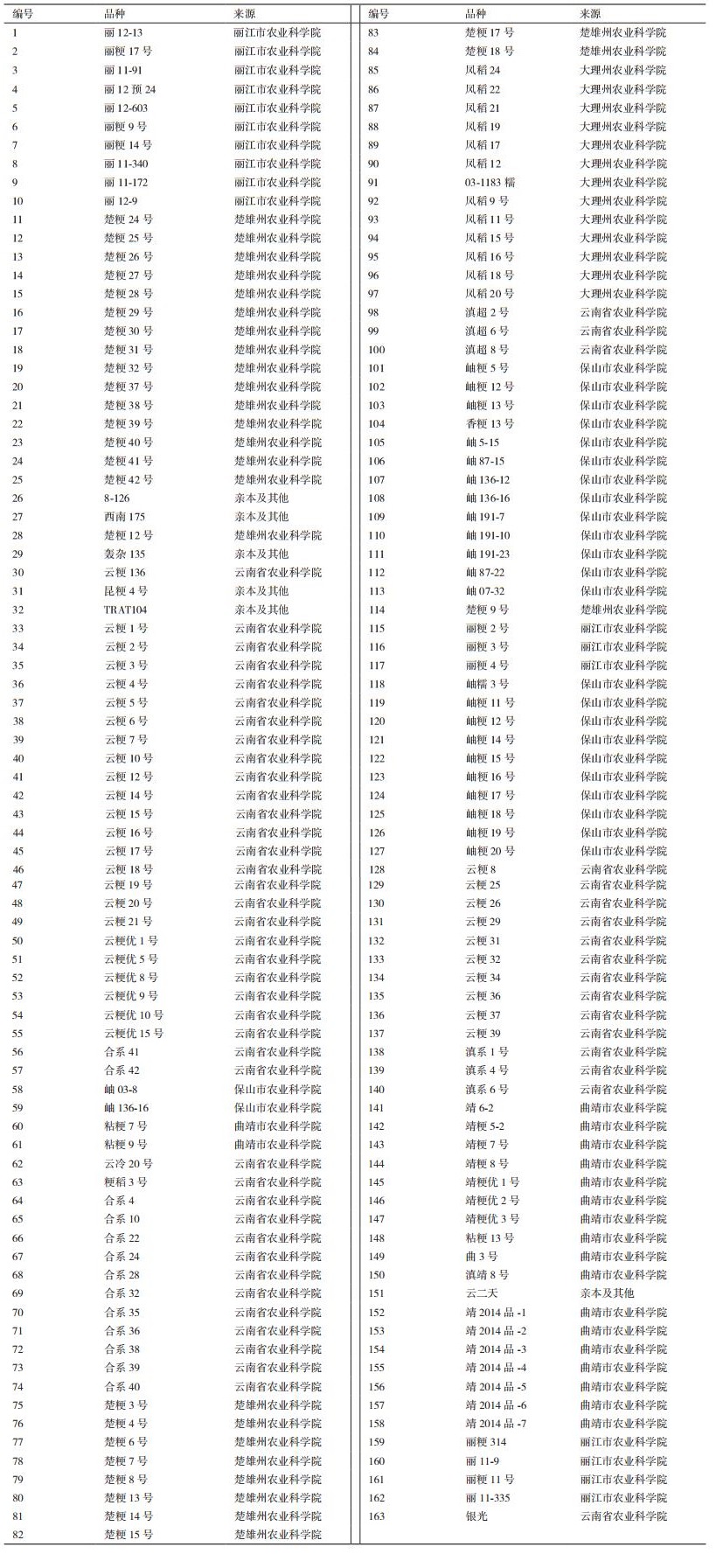
1 材料与方法 1.1 材料选取云南省7个育种单位的163份粳稻品种、品系等作为实验材料(表 1)。包括丽江市农业科学院17份,楚雄州农业科学院27份,大理州农业科学院13份,保山市农业科学院25份,曲靖市农业科学院19份,云南省农业科学院56份,其他亲本材料的6份。
参照农业行业标准《水稻品种鉴定技术规程SSR标记法》(NY/T 1433-2014)的方法,试验材料经催芽播种于秧床上,薄膜覆盖进行育苗。当幼苗长到四叶一心时,混合取20个单株的叶片,用改良的CTAB法提取DNA。
1.2.2 SSR标记检测依据Temnykh等[8]公布的水稻SSR遗传标记连锁图,经过初筛选,在每条染色体上均匀选择1-3对,共30对SSR荧光标记引物用于遗传多样性检测。PCR总反应体系为10 µL,采用2×mix混合液,10 µL的反应体积包括5 mL 2×mix混合液、0.3 mL 10 mmol/L正向引物、0.3 mL 10 mmol/L反向引物,1 mL样品DNA,3.4 mL超纯水。扩增程序为94℃预变性5 min;94℃变性45 s,55℃退火45 s,72℃延伸1 min,循环30次;72℃下延伸10 min,4℃保存。PCR产物在毛细管荧光电泳系统AB 3730XL DNA分析仪上检测。
1.2.3 统计分析用Powermarker Version3.25[9]软件计算群体的等位基因数、基因多样性指数和多态信息含量(PIC),并按非加权配对法(UPGMA)采用Nei’s遗传距离进行聚类。用MEGA Version 3.1[10]软件对生成的聚类图进行编辑修饰。用STRUCTURE Version2.2软件完成参试材料的遗传结构分析[5]。参照Evanno等[11]的K值变化下△K的折线图模型(△K=m(|L(K+1)-2L(k)+L(K-1)|)/s[L(k)],其中m代表K值的平均值,s表示K值的标准差),K值预设为1-10,估计最佳群体组群数K,每个K值都独立运算10次。
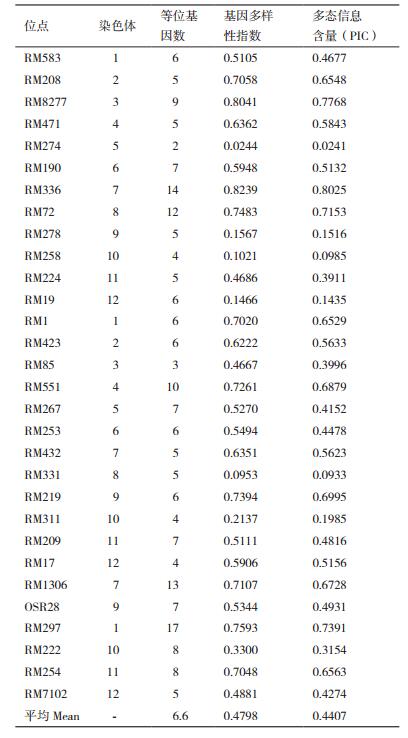
2 结果 2.1 SSR标记多态性分析用30对SSR引物,对163份粳稻品种进行遗传多样性分析,其结果见表 2。30个多态性位点共检测到等位基因207个,每个位点等位基因变幅为2-17,平均等位基因数为6.6。基因多样性指数变异范围为0.024 4-0.823 9,平均为0.479 8;多态信息含量(PIC)的变异范围为0.024 1-0.802 5,平均为0.440 7。其中,RM297、RM336、RM1306、RM72、RM551的等位基因数较多,依次为17、14、13、12、10。等位基因数最少的为RM274,有2个等位基因。基因多样性指数较高的引物依次为RM336、RM8277、RM297、RM72、RM219、RM551、RM1306、RM254、RM1,分别是0.823 9、0.804 1、0.759 3、0.748 3、0.739 4、0.726 1、0.710 7、0.704 8和0.702 0。结果表明,不同的粳稻品种存在着丰富的SSR多态性,SSR标记适合应用于粳稻选育品种的遗传多样性分析及品种鉴定。
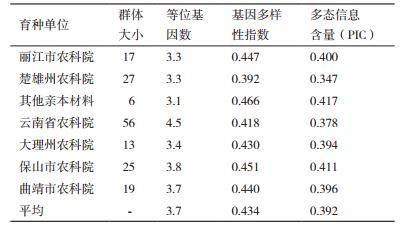
7个育种单位选育粳稻品种的遗传多样性参数分析结果见表 3。7个育种单位粳稻品种的等位基因数变异在3.1-4.5,基因多样性指数变异范围为0.392-0.466,其他分类中的基因多样性水平最高。PIC变异范围为0.347-0.417,其他分类中的PIC值最大,为0.417。等位变异数最多的是云南省农业科学院选育的品种,但遗传多样性并不与群体大小完全正相关,群体最小的其他分类中的基因多样性指数和PIC均表现为最高。此外,保山市农科院和丽江市农科院的基因多样性指数和PIC稍高于其他单位。
来自不同育种单位的163份粳稻品种系统聚类分析结果见图 1。利用30对SSR标记将163份粳稻材料分为4大类群。第Ⅰ类群为来自国际水稻所的一个粳稻亲本TRAT104,第Ⅱ、Ⅲ类群主要包括了丽江市农业科学院选育的9个品种,云南省农业科学院的4个及两个亲本。第Ⅳ大类群包含了其他单位选育的品种,其中又分为不同的小类群。从每个小类群品种的组成可见相同单位的材料虽然并未完全聚类于同一小类群,而是分散于不同小类群中,但大部分品种遗传相似度较高。表明不同育种单位间的育种材料有交叉但交叉不多。
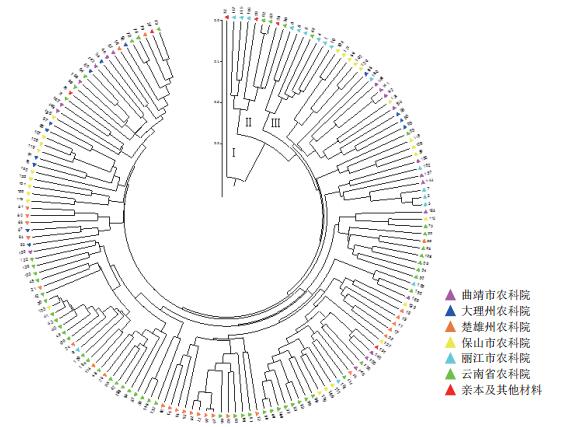
|
| 图 1 粳稻品种基于Nei’s遗传距离的系统聚类图 |
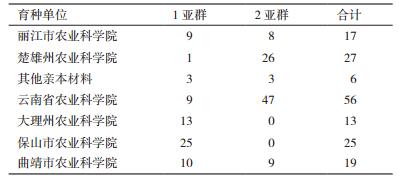
采用基于数学模型的聚类方法分析了163份粳稻品种的群体遗传结构。图 2显示K值变化下△K值的折线图。当K=2时出现拐点,图 3显示K=2时参试材料类群的遗传结构和群体组成。图中所示纵坐标数值表示每份材料划分到相应类群的几率,横坐标表示个体所在群体,163个品种划分为两个类群。某材料属于某类群的概率 > 0.6,被归入此类群,各亚群材料来源列于表 3。在163份材料中,有70份材料被分到红色部分表示的第1亚群,其余93份被分到绿色部分表示的第2亚群。
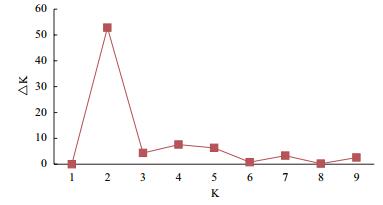
|
| 图 2 每个K值所对应的△ K |

|
| 图 3 163个粳稻品种的群体结构 |
表 4中来自大理州农业科学院和保山市农业科学院的品种全部分在第1亚群中,楚雄州农业科学院和云南省农业科学院的品种主要集中在第2亚群中。丽江市农业科学院、曲靖市农业科学院的品种平均分布在两个亚群中。大理州农业科学院、保山市农业科学院与其他院所的资源遗传差异较大,它们之间的杂交有利于拓宽后代材料的遗传基础。
赵庆勇[12]、束爱萍[4]、杨文毅[13]、张立娜[5]等分别对粳稻育成品种和地方品种进行了遗传多样性分析,筛选出了相应的适合用于粳稻品种遗传多样性检测的标记。本研究中,通过等位基因数与基因多样性指数和PIC值的比较,一个引物上的等位基因数与基因多样性指数和多态性信息值不成直接的线性关系。在遗传多样性分析及品种鉴别中,引物的选择需要综合考虑等位基因的数量及多态性信息值,还要考虑位点与农艺性状的连锁情况,保证所用的分子标记可以有效的评价种质资源。
3.2 云南省粳稻品种的遗传多样性云南特殊的区域条件和众多少数民族聚居,丰富的生态地理和人文习惯及多样栽培等环境孕育了云南粳稻地方品种的遗传多样性[14]。张丽娜等[5]对云南省粳稻地方品种的分析表明,等位基因数、基因多样性指数、多态信息指数分别为4.9、0.622、0.569。在本研究中,育成粳稻品种的等位基因数、基因多样性指数、多态信息指数分别为3.7、0.434、0.392,均低于地方品种。可见,目前育成的粳稻品种在选育过程中丢失了一部分等位基因,云南丰富的种质资源还未充分开发利用。
结合云南省水稻种植区划,分为5个稻作区,分别为滇中一季粳稻、籼稻区,滇南单、双季籼稻区,南部边缘水、陆稻区,滇东北高原粳稻区,滇西北高寒粳稻区。根据杨忠义等[14]的研究,云南粳稻资源在全省各地均有分布,但以滇西南和滇南地区较为丰富多样,从气候类型上看多样性中心主要集中在亚热带地区。但是近年来云南粳稻品种的选育和推广种植主要分布在滇中一季粳稻、籼稻区、滇东北高原粳稻区,滇西北高寒粳稻区,对分布于亚热带地区的热带粳等种质资源开发利用较少,造成了粳稻选育品种遗传多样性远低于地方品种。今后在粳稻育种中应重视热带粳等地方品种的有效利用。
3.3 不同育种单位选育品种的亲缘关系束爱萍等[4]认为在水稻育种中被选育的基因型首先取决于亲本材料的遗传基础,而亲本材料往往根据当地的生态气候环境条件和育种目标而被选择。由此可能导致选育品种间较高的遗传相似性。魏兴华等[15]认为,籼粳稻品种尤其是粳稻的聚类结果显示出较强的季节型和地域特性。张立娜[5]等研究表明,粳稻地方品种间的亲缘关系与地域性存在一定的相关性。
本研究聚类结果显示,相同单位的材料虽然并未完全聚类于同一小类群,而是分散于不同小类群中,但大部分品种遗传相似度较高。说明各育种单位在品种选育时所采用的亲本材料遗传基础较狭窄。育种单位之间有种质资源的交流但交流不多。遗传结构分析表明,处于滇中一季粳稻、籼稻区的楚雄州农业科学院及云南省农业科学院(昆明)选育的大部分品种聚在了第2亚群中。而同样处于滇中一季粳稻、籼稻区的来自大理州农业科学院和保山市农业科学院的品种全部分在第1亚群中,这可能由于这两个育种单位所处的地区具有相同的气候类型,较之楚雄及昆明具有更加丰富的气候类型,以致大理州农业科学院,保山市农业科学院的品种多样性也较高。位于滇西北高寒粳稻区和滇东北高原粳稻区的丽江市农业科学院、曲靖市农业科学院的品种平均分布在两个亚群中。可见,粳稻品种间的亲缘关系与地域性存在一定的相关性。
4 结论各育种单位拥有的粳稻种质资源较单一,加上在环境压力和人为选择导致同一单位选育的品种遗传相似度较高,品种遗传多样性不够丰富,云南省丰富的粳稻资源还没有被完全挖掘利用。因而还需要在资源的高效利用、优异资源评价、资源共享和交流等方面开展工作。不同的粳稻品种存在着丰富的SSR多态性,SSR标记适合应用于粳稻选育品种的遗传多样性分析及品种鉴定。
| [1] |
黄山, 何虎, 张卫星, 等. 不同粳稻品种在江西不同生态区的农学表现[J]. 江西农业大学学报, 2013, 35(1): 25-32. |
| [2] |
张洪程, 许轲, 张军, 等. 双季晚粳生产力及相关生态生理特征[J]. 作物学报, 2014, 40: 283-300. |
| [3] |
王远征, 王晓菁, 李源, 等. 北方粳稻产量与品质性状及其相互关系分析[J]. 作物学报, 2015, 41(6): 910-918. |
| [4] |
束爱萍, 张媛媛, 曹桂兰, 等. 中国不同省份粳稻选育品种的遗传相似性[J]. 中国农业科学, 2009, 42(10): 3381-3387. DOI:10.3864/j.issn.0578-1752.2009.10.001 |
| [5] |
张立娜, 曹桂兰, 韩龙植. 利用SSR标记揭示中国粳稻地方品种遗传多样性[J]. 中国农业科学, 2012, 45(3): 405-413. |
| [6] |
刘承晨, 赵富伟, 等. 云南哈尼梯田当前栽培水稻遗传多样性及群体结构分析[J]. 中国水稻科学, 2015, 29(1): 28-34. |
| [7] |
涂敏, 王云月, 卢宝荣, 等. 云南省不同地理位置水稻品种遗传多样性分析热[J]. 热带作物学报, 2011, 32(6): 998-1003. |
| [8] |
Temnykh S, DeClerck G, Lukashova A, et al. Computational and experimental analysis of microsatellites in rice(Oryza sativa L.):frequency, length variation, transposon associations, and genetic marker potential[J]. Genome Research, 2001, 11: 1441-1452. DOI:10.1101/gr.184001 |
| [9] |
Fukunaga K, Hill J, et al. Genetic diversity and population structure of teosinte[J]. Genetics, 2005, 169: 2241-2254. DOI:10.1534/genetics.104.031393 |
| [10] |
Kumar S, Tamura K, Nei M. MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignmen[J]. Brief Bioinform, 2004, 5(2): 150-163. DOI:10.1093/bib/5.2.150 |
| [11] |
Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Molecular Ecology, 2005, 14: 2611-2620. DOI:10.1111/mec.2005.14.issue-8 |
| [12] |
赵庆勇, 张亚东, 朱镇, 等. 30个粳稻品种SSR标记遗传多样性分析[J]. 植物遗传资源学报, 2010, 11(2): 218-223. |
| [13] |
杨文毅, 等. 不同地理来源水稻品种的SSR分子标记遗传相似性分析[J]. 中国农学通报, 2011, 27(12): 24-30. |
| [14] |
杨忠义, 曹永生, 苏艳, 等. 中国地方粳稻资源特征特性评价及多样性分布[J]. 西南农业学报, 2007, 20(5): 875-882. |
| [15] |
魏兴华, 袁筱萍, 余汉勇, 等. 我国常规稻主栽品种的遗传变异分析[J]. 中国水稻科学, 2009, 23(3): 237-244. |