2. 眉山职业技术学院,眉山 620000
2. Meishan Vocational and Technical College, Meishan 620000
猕猴桃因其品质鲜嫩,营养丰富,被誉为“水果之王”。中国是猕猴桃属植物的起源中心,栽培面积最大,主要分布于陕西、四川、河南、贵州等省,其中,四川主栽品种“红阳”,因其独特的优良红色条纹性状和优异的风味成为新一代的主流品种[1]。
然而,该品种易感猕猴桃溃疡病,病菌为丁香假单胞菌猕猴桃致病变种(Pseudomonas syringae pv. actinidiae,PSA),该菌适应性广,致病力强,来势凶猛,发病严重时,整株枯死,几年间造成果园毁灭[2-4]。该病害于1984年日本静冈县首次发现,随后在韩国、意大利、法国、葡萄牙、西班牙、新西兰和智利等多个国家陆续报道[5-8]。目前中国陕西[9]和四川等多个省份的猕猴桃产区有溃疡病爆发,四川苍溪、雅安、都江堰等多个市县的猕猴桃产区均有大规模溃疡病发生,部分果园完全毁园,对猕猴桃产业的可持续发展及经济损失造成严重影响。近些年,随着猕猴桃溃疡病感病品种的苗木繁育和调运不规范,防治困难等因素的影响,使得猕猴桃溃疡病的发生、传播和蔓延面积进一步扩大,现已成为猕猴桃的毁灭性病害,是生产上亟待解决的重大问题之一。
2010年后,随着溃疡病给猕猴桃产业带来的严重损失,国内外专家学者对猕猴桃溃疡病的病原菌进行了研究和分类。目前,将全球的PSA分成4类[10-12]:PSA1包括日本和1992年在意大利引起的猕猴桃溃疡病;PSA2只存在于韩国,其他国家尚未发现这一类群;PSA3广布于全球,包括中国、智力、新西兰、意大利(2008-2009年发生的猕猴桃溃疡病等)和韩国[13];PSA4主要分布于新西兰和澳大利亚。在各类群中,自从2008年意大利爆发了致病性极强的PSA3类群以来,该病害开始对猕猴桃产业的发展造成重大影响,才逐渐受到人们的关注,PSA3已成为制约全球猕猴桃产业发展的重要致病类群。因此,田间带菌果树的早期检测,是防治猕猴桃溃疡病的关键;对调运苗木的准确诊断,是防治其蔓延的关键。建立一套高效、快速、方便、准确的检测方法,进而有利于实现该病害的早期诊断和及时防治。
目前,主要通过PCR扩增特异基因序列进行PSA的分子检测。2017年,Cimmino等[14]报道制备了一种PSA胞外多糖的多克隆抗体,建立了一种猕猴桃溃疡病菌的血清学检测。本研究则根据2013年McCann等[11]对猕猴桃溃疡病菌不同类群基因组的分析,尤其是效应子在PSA各类群中的差异比较,指出PSA3类群的特异效应子包括Hopz5、NRPS、hopH1、hopAM-2和hopAA1-2。此外,根据NCBI数据库(http://ncbi.nlm.nih.gov/)和T3SE数据库(http://pseudomonas-syringae.org/)提供的信息,表明仅Hopz5效应子不存在于其他细菌中。因此,利用该效应子进行分子检测,以及制备的抗体行血清学检测都将表现高度的特异性及准确性。目前,鉴于Ciarroni等[15]报道中国猕猴桃溃疡病菌为PSA3类群,并且前期我们对四川各地猕猴桃溃疡病菌类群的鉴定,也证实其为该类群。因此,本研究选择PSA3特异效应子Hopz5制备的多克隆抗体,通过对抗体特性分析,旨为提供一种用于PSA3血清学检测用试剂。
1 材料与方法 1.1 材料 1.1.1 供试材料猕猴桃品种为红阳;猕猴桃溃疡病菌分离自苍溪猕猴桃发病果园,并于实验室保存备用;田间各检测用样品材料取自都江堰。
1.1.2 供试试剂及仪器Hopz5多克隆抗体由四川农业大学植物病理实验室提供;限制性内切酶Xho Ⅰ、BamH Ⅰ,Primestar HS polymerase购自宝生物工程(大连)有限公司;质粒小提试剂盒购自Tiangen公司;预染蛋白marker,HRP(辣根过氧化物酶),卡那霉素购自北京全式金公司。TMB显色液购自天根公司。碱性磷酸酶购自中杉金桥公司。NBT/BCIP试剂盒购自康为世纪公司。引物由苏州金唯智公司合成。UVP凝胶成像仪为美国BIO-RAD,DNA浓度检测仪为美国Thermo公司的NANODROP200。
1.2 方法 1.2.1 不同类型样品的制备PSA在LB固体培养基上进行画线,放于25℃恒温培养箱培养,将菌落用无菌水洗脱,制备为1×108 CFU/mL的PSA菌悬液。感病组织总蛋白提取过程:液氮充分研磨材料,按照1 g:2 mL,加入蛋白提取缓冲液(50 mmol/L Tris.HCl(PH 6.8),4% SDS,6%β-巯基乙醇,4 mol/L尿素,10%甘油)。100℃温浴10 min,然后12 000×g离心10 min,取上清即为总蛋白。组织浸泡液制备过程:将带菌组织切成小碎块,0.005 g样品碎片中加入无菌水20 μL,浸泡10 s,吸取浸泡液备用。以制备的PSA菌悬液,感病组织提取的总蛋白和组织浸泡液这3种样品为材料,用于ELISA检测。
1.2.2 ELISA检测ELISA的操作步骤参照以前报道的方法略有改动[16]。取待测样品,平均每孔加入样品10 µL、包被液90 µL,置于37℃充分吸附2 h;倒出固定液,平均各孔加入220 µL PBST,洗板3次后每孔加入100 µL封闭液,置于37℃温育2 h;倒出封闭液,用PBST洗板3次后每孔加入100 µL用PBST稀释成1:2 000的一抗(PAb-Hopz5),37℃孵育2 h;倒出一抗,用PBST洗板3次后每孔加入100 µL用PBS将辣根酶结合的山羊抗小鼠的IgG(二抗)(1:2 000),37℃培养90 min;倒出二抗,用PBST洗板3次后每孔加入100 µL底物缓冲液,于空白对照开始显色时,每孔加入2 mol/L H2SO4 100 µL终止反应,待蓝色转变为黄色,用酶标仪测定450 nm下的OD值。
1.2.3 不同侵染时期的蛋白表达检测取两年生的猕猴桃枝干约20 cm,先用无菌水冲洗,再用1%次氯酸钠浸泡10 min进行表面消毒,然后用无菌水浸泡漂洗3次,每次10 min。PSA于25℃、180 r/min震荡培养至对数生长期(16-18 h),收集菌体细胞(5 000 r/min,10 min),并重悬浮在1×PBS缓冲液中,血球计数板计数法调节菌悬液浓度至1×108 CFU/mL。于接种后3、6、12和24 d取材,提取总蛋白,取10 µL用于ELISA检测。
1.2.4 Hopz5抗体的特异性检测将丁香假单胞菌番茄致病变种、枯草芽孢杆菌、姜瘟菌、巨大芽孢杆菌、恶臭假单胞菌、荧光假单胞菌、大黄欧文氏菌培养24 h后用包被液制成浓度为1×108 CFU/mL的菌悬液,取50 µL用于ELISA检测。
1.2.5 Hopz5抗体的ELISA灵敏度检测将PSA制备为1×108 CFU/mL的菌悬液,用无菌水梯度稀释为107、106、105、104、103、103、102、101、100 CFU/mL和无菌水做对照,每个稀释梯度取10 µL用于ELISA检测;10 µL用于PCR检测。
1.2.6 Hopz5抗体的田间样品检测从都江堰猕猴桃溃疡病发病果园,随机取30个样品进行检测,每个样品分成两份,一份提取DNA,用于PCR检测,所用引物为PSAF1:5' -TTTTGCTTTGC ACACCCGATTTT-3' 和PSAR2:5' -CACGCACCCTTCAATCAGGATG-3' [17],扩增大小280 bp;Hopz5-F:5' -TCACTCCTAGACTGGAATAC-3' 和Hopz5-R:5' -GGCTATCATGAAGGCTGTCA-3' ,扩增大小550 bp。另一份提取植物总蛋白,用于ELISA检测。
2 结果 2.1 Hopz5抗体的Western-blot检测将PSA3特异效应子Hopz5多克隆抗体首先通过Western-blot进行检测,其检出的信号条带为38.98 kD,与预期大小一致,且检测条带单一(图 1),进而将Hopz5抗体进行后续特性分析。

|
| 图 1 PAbs-Hopz5的Western blot检测 |
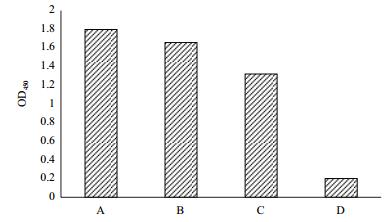
Hopz5是猕猴桃溃疡病菌的一个特异效应子,是病原细菌在侵染寄主过程中的重要致病因子之一。Hopz5多克隆抗体能否用于检测培养的PSA,有待验证。因此,将培养24 h的PSA菌液,以感病猕猴桃组织的总蛋白和感病组织浸泡液为参照,ELISA检测结果(图 2)表明3种样品均能显色,其中菌悬液、蛋白样品和组织浸泡液的OD450值分别为1.74、1.66和1.32,P/N值均大于2,结果为阳性,表明Hopz5抗体对培养基上的PSA同带菌猕猴桃组织一样,可进行检测,具有较好的应用范围。
|
| 图 2 不同类型的PSA样品检测 A:PSA菌悬液;B:发病组织蛋白;C:发病组织浸泡液 |
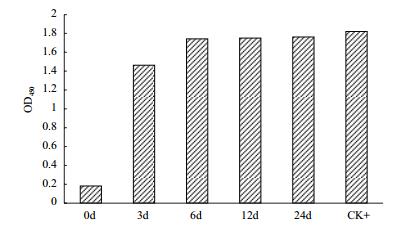
为了明确Hopz5抗体检测是否受PSA不同侵染时期的影响,我们将人工接种PSA的枝干,于接种后3、6、12和24 d,提取总蛋白,进行ELISA检测,OD450值分别为1.46、1.74、1.75和1.76,P/N值均大于2,结果如图 3,表明该抗体可以稳定检测PSA不同侵染时期的材料。
|
| 图 3 不同接种时期的PSA检测 |
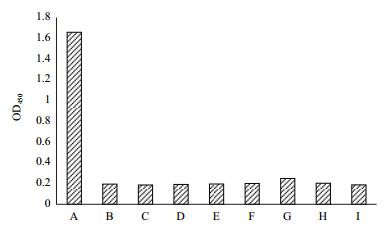
将PSA和其他7种供试细菌进行特异性分析,PSA菌悬液发生明显反应,OD450值为1.71,P/N值大于2。与供试菌枯草芽孢杆菌、姜瘟菌、丁香假单胞菌番茄致病变种、巨大芽孢杆菌、恶臭假单胞菌、荧光假单胞菌和大黄欧文氏菌无交叉反应,OD450值均约0.2,结果如图 4,表明Hopz5抗体对PSA的检测特异性较好。
|
| 图 4 PAb-Hopz5的特异性检测 A:猕猴桃溃疡病菌;B:枯草芽孢杆菌;C:姜瘟菌;D:丁香假单胞菌番茄致病变种;E:巨大芽孢杆菌;F:恶臭假单胞菌;G:荧光假单胞菌;H:大黄欧文氏菌;I:无菌水 |
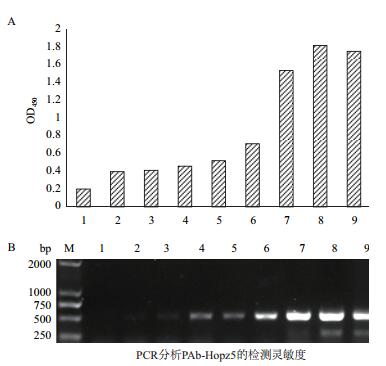
为了明确Hopz5多克隆抗体检测PSA的有效灵敏度,采用ELISA和PCR两种方法对比分析。将PSA菌悬液稀释成3×100-3×108 CFU/mL 9个浓度梯度,每个浓度梯度分别用于PCR和ELISA检测。结果显示(图 5),Hopz5抗体用于ELISA检测的最低菌悬液浓度为3× 105 CFU/mL,受灵敏度限制,利用该抗体可进行带菌苗木和田间样品的诊断。然而,PCR检测PSA菌悬液的最低浓度为3×10 CFU/mL,具有较高的检测灵敏度。
|
| 图 5 ELISA分析PAb-Hopz5的检测灵敏度 1-9:3×100-2.2×108 CFU/mL |
为了明确Hopz5多克隆抗体的检测准确性,我们以田间猕猴桃溃疡病发病样品为材料,结合PCR结果对比分析。将采集的田间样品一份提取DNA,用于PCR检测;另一份提取总蛋白,用于ELISA检测。ELISA检测样品编号为3-19的OD450值大于1.5,P/N值大于2(图 6),均为阳性结果,与PCR检测一致。然而,即使PCR检测菌悬液的灵敏度较高,但对于大部分田间样品材料,PCR扩增的目标条带并不够清晰,推测由于猕猴桃多糖多酚类物质含量高,影响核酸提取质量,进而对PCR扩增产物造成影响。然而,田间样品的ELISA检测结果明显,其可信度高,说明Hopz5可用于ELSIA的田间样品诊断。
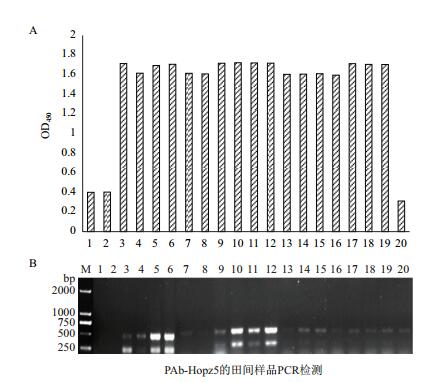
|
| 图 6 PAb-Hopz5的田间样品检测 1-19:田间样品;20:健康样品 |
近些年,猕猴桃溃疡病对猕猴桃产业具有毁灭性的破环,造成了重大的经济损失。因此,对病原菌的检测为限制其进一步扩展和及时防治提供依据。
目前,分子检测是猕猴桃溃疡病菌的主要方式。在全基因组未测序之前,2002年Koh等[18]通过SRAP分析,筛选了一段能够扩增PSA的序列,建立了猕猴桃溃疡病菌的分子检测方式。2010年Rees-George等[17]根据PSA的16S-23SrDNA的ITS区域,设计了一对引物,但该引物不能将P. syringae pv. theae区分。2013年Balestra等[19]建立的PSA双重PCR检测体系,其中利用HopZ3序列设计了一对引物,能够特异检测PSA,并将P. syringae pv. theae区分。虽然,血清学检测具有操作方便的特点,针对PSA却发展较慢,直到2017年,Cimmino等[14]制备了一种PSA胞外多糖的多克隆抗体,才建立了猕猴桃溃疡病菌的ELISA检测。本研究则基于PSA的主要致病类群PSA3,选择特异性效应子Hopz5,制备了相应的多克隆抗体,建立了一种针对我国主要类群的血清学检测方法。
效应子、胞外多糖、胞外酶和毒素等通常为病原菌的致病因子。效应子作为病原细菌的重要致病因子之一,能够通过Ⅲ型分泌系统进入寄主植物中。例如,假单胞菌属的hopW1效应子能够与感病寄主的机动蛋白结合,破坏寄主正常生长[20]。然而,Hopz5效应子多克隆抗体能否广泛用于PSA的检测仍有待验证,毕竟目前鲜有利用效应子进行血清学检测的文献报道。因此,本研究在抗体特性分析中,首先利用培养基上培养的PSA3和不同类型的材料进行检测发现,Hopz5多克隆抗体均能检测PSA3,但发病枝干浸泡液的检测效果较人工培养的菌液和提取的总蛋白效果差,表明选用猕猴桃组织的蛋白进行检测是较好的一种样品类型。此外,通过人工接种方式,对不同接种时期的样品进行检测,均能检出PSA3,说明该抗体的检测不受侵染时期的影响。通过特异性比较,Hopz5多克隆抗体对供试菌无交叉反应。在灵敏度分析中,ELISA检测PSA3菌悬液的最低浓度为3×105 CFU/mL,与PCR的检测灵敏度相比,差异较大,PCR能够检测PSA3的最低浓度为3×10 CFU/mL。然而,田间带菌组织的检测发现,ELISA对样品的检测效果优于PCR。推测原因在于,猕猴桃组织富含多糖多酚类物质,对核酸提取质量影响较大,进而影响PCR的检测结果。Hopz5抗体对田间样品的检测表明其结果较准确,可信度较高。
4 结论本实验通过对Hopz5抗体的特性分析和田间样品检测结果表明,我国由丁香假单胞菌猕猴桃致病变种PSA3类群引起的猕猴桃溃疡病可以利用其效应子Hopz5制备的多克隆抗体进行血清学检测,该抗体可运用于室内培养的菌液和田间样品的检测,可作为其他血清学检测的试剂,具有较广的应用范围。
| [1] |
吴世权, 何仕松, 闫书贵. 红阳猕猴桃[M]. 北京: 中国农业科学出版社, 2014, 1-2.
|
| [2] |
Renzi M, Copini P, Taddei AR, et al. Bacterial canker on kiwifruit in Italy:Anatomical changes in the wood and in the primary infection sites[J]. Phytopathology, 2012, 102: 827-840. DOI:10.1094/PHYTO-02-12-0019-R |
| [3] |
Bull CT, De Boer SH, Denny TP, et al. Comprehensive listof names of plant pathogenic bacteria, 1980-2007[J]. Jouralof Plant Pathology, 2010, 92: 551-592. |
| [4] |
Gardan L, Shafik H, Belouin S, et al. DNA relatedness among the pathovars of Pseudomonas syringae and description of Pseudomonas tremae sp. nov. and Pseudomonas cannabina sp. nov.(ex Sutic and Dowson 1959)[J]. International Journal of Systematic Bacteriology, 1999, 49 Pt 2(2): 469. |
| [5] |
Serizawa S, Ichikawa T, Takikawa Y, et al. Occurrence of bacterial canker of kiwifruit in Japan:description of symptoms, isolation of the pathogen and screening of bactericides[J]. Annals of the Phytopathological Society of Japan, 1989, 55: 427-436. DOI:10.3186/jjphytopath.55.427 |
| [6] |
Scortichini M. Occurrence of Pseudomonas syringae pv. actinidiae on kiwifruit in Italy[J]. Plant Pathology, 1994, 43: 1035-1038. DOI:10.1111/ppa.1994.43.issue-6 |
| [7] |
Abelleira A, López MM, Peñalver J, et al. First report of bacterial canker of kiwifruit caused by Pseudomonas syringae pv. actinidiae in Spain[J]. Plant Disease, 2011, 95(12): 1583-1583. |
| [8] |
Everett KR, Taylor RK, Romberg MK, et al. First report of Pseudomonas syringae, pv. actinidiae, causing kiwifruit bacterial canker in New Zealand[J]. Australasian Plant Disease Notes, 2011, 6(1): 67-71. DOI:10.1007/s13314-011-0023-9 |
| [9] |
梁英梅, 张星耀. 陕西省猕猴桃枝干溃疡病病原菌鉴定[J]. 西北林学院学报, 2000, 15(1): 37-39. |
| [10] |
Chapman JR, Taylor RK, Weir BS, et al. Phylogenetic relationships among global populations of Pseudomonas syringae pv. actinidiae[J]. Phytopathology, 2012, 102(11): 1034-1044. DOI:10.1094/PHYTO-03-12-0064-R |
| [11] |
McCann HC, Rikkerink EH, Bertels F, et al. Genomic analysis of the Kiwifruit pathogen Pseudomonas syringae pv. actinidiae provides insight into the origins of an emergent plant disease[J]. PLoS Pathoglogy, 2013, 9(7): e1003503. DOI:10.1371/journal.ppat.1003503 |
| [12] |
Ferrante P, Scortichini M. Redefining the global popμlations of Pseudomonas syringae pv. actinidiae based on pathogenic, molecular and phenotypic characteristics[J]. Plant Pathology, 2015(64): 51-62. |
| [13] |
Kim GH, Kim KH, Son KI, et al. Outbreak and spread of bacterial canker of kiwifruit caused by Pseudomonas syringae pv. actinidiae biovar 3 in Korea[J]. Plant Pathol Journal, 2016, 32(6): 545-551. DOI:10.5423/PPJ.OA.05.2016.0122 |
| [14] |
Cimmino A, Iannaccone M, Petriccione M, et al. An ELISA method to identify the phytotoxic Pseudomonas syringae pv. actinidiae exopolysaccharides:A tool for rapid immunochemical detection of kiwifruit bacterial canker[J]. Phytochemistry Letters, 2017, 19: 136-140. DOI:10.1016/j.phytol.2016.12.027 |
| [15] |
Ciarroni S, Gallipoli L, Taratufolo MC. Development of a multiple loci variable of tandem repeats analysis(MLVA)to unravel the intra-pathovar structure of Pseudomonas syringae pv. actinidiae populations worldwide[J]. PLoS One, 2015, 10(8): 2018-2025. |
| [16] |
Li N, Chen Z, Liu Y, et al. Development of monoclonal antibodies an serological assays specific for Barley yellow dwarf virus GAV strain[J]. Virology Journal, 2015, 12: 189-191. DOI:10.1186/s12985-015-0418-x |
| [17] |
Rees-George J, Vanneste JL, Cornish DA, et al. Detection of Pseudomonas syringae pv. actinidiae using polymerase chain reaction(PCR)primers based on the 16S-23S rDNA intertranscribed spacer region and comparison with PCR primers based on other gene regions[J]. Plant Pathology, 2010, 59: 453-464. DOI:10.1111/(ISSN)1365-3059 |
| [18] |
Koh YJ, Nou IS. DNA markers for identification of Pseudomonas syringae pv. actinidiae[J]. Molecules and Cells, 2002, 13: 309-312. |
| [19] |
Balestra GM, Taratufolo MC, Vinatzer BA, et al. A mμltiplex PCR assay for detection of Pseudomonas syringae pv. actinidiae and differentiation of popμlations with different geographic origin[J]. Plant Diease, 2013, 97(4): 472-478. DOI:10.1094/PDIS-06-12-0590-RE |
| [20] |
Kang Y, Jelenska J, Cecchini NM, et al. HopW1 from Pseudomonas syringae disrupts the actin cytoskeleton to promote virulence in Arabidopsis[J]. PLoS Pathogens, 2014, 10(6): e1004232. DOI:10.1371/journal.ppat.1004232 |




