2. 中国农业大学食品科学与营养工程学院,北京 100083
2.College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083
随着现代社会生活方式、饮食习惯等的改变,人类肠道疾病的发病率呈增长趋势,如何治疗肠道疾病,维持肠道健康已成为研究者关注的重点。由于肠道疾病的发病机理复杂,治疗手段局限,多为药物及手术治疗,效果欠佳,大部分肠道疾病目前无法完全治愈。令人欣喜的是,近年来的研究表明,肠道中的微生物对肠道的免疫调节、营养物质代谢等有至关重要的作用,研究者以此为基础,开发出利用微生物制剂调节肠道菌群,改善肠道状况的方法,如利用天然益生菌对肠道菌群进行调节[1-2],或者利用菌群移植的方法将健康的人的菌群移植到病人体内[3-4],这些方法对部分病人的肠道疾病具有一定的改善作用,但由于机理上不明确,方法上不可控,这些方法的广谱有效性受到质疑。因此这些方法往往只用于辅助治疗,并不能作为治疗特定肠道疾病的针对性方法。
合成生物学的出现,给这些问题带来了转机。合成生物学利用现代分子生物学技术,把“基因”连接成可以实现特定功能的电路网络并导入细胞基因组中,让细胞来完成设计人员设想的各种任务。现阶段,合成生物学研究范围广泛,研究方向多样,并与其他学科结合,给可再生燃料生产[5]、环境治理[6-7]、药物生产[8-10]、新型材料合成[11]、天然产物生产[12-15]等领域提供了新的思路。在生物学领域,合成生物学制备的工程菌株,可以实现抑制特定类型菌群,靶向特定状态细胞,调控肠道上皮细胞状态等功能。
与传统的益生菌干预和肠道菌群移植相比,合成生物学用于调控肠道菌群具有更多维的调控靶标和更好的调控针对性。将合成生物学与肠道疾病的治疗结合起来,可以有效的改善机体健康状况。以溃疡性结肠炎(Ulcerative colitis,UC)为例,该病是由人体遗传基因变异、肠道菌群失调、饮食结构改变等多种病因所引起的肠道慢性及复发性炎症[16-17]。其中肠道菌群失衡和饮食结构改变导致肠道内抗原成分的变化,宿主遗传基因变异导致肠壁细胞对肠道内抗原成分识别状态的变化,二者共同作用导致宿主机体长期处于炎性状态,从而导致疾病发生。通过合成生物学设计改造工程菌株,使其能感知饮食成分、肠道菌群组成及肠壁细胞状态的变化,随之启动自身药物的表达,从而帮助肠道营养素代谢、肠道菌群改善、修复受损细胞,就可以有效的治疗溃疡性结肠炎,控制其恶化。
近年来,合成生物学方法与肠道疾病治疗方面出现了许多交集,但该方向的各研究之间相对比较独立松散,并未形成一套完整的技术理论体系。本文对合成生物学在肠道菌群调节、肠道代谢改善、肠壁细胞状态调控3个方面的实践进行了总结,在此基础上对未来合成生物学方法用于肠道疾病治疗的技术体系进行展望。
1 合成生物学在调节肠道菌群中的应用正常肠道菌群对肠道健康的维持有着至关重要的作用。肠道菌群失衡或食源性病原菌的侵入,会引发宿主疾病,损害机体。现阶段,针对肠道菌群失衡,医学上主要采用益生菌干预或者肠道菌群移植的方法进行改善,因这些方法都是非靶向性的,故治疗效果不佳,且可能会有副作用。针对病原菌侵入,医学上大多采用口服抗生素进行治疗,大量研究结果表明,长期、高剂量的抗生素会使病原菌产生耐药性,并且破坏正常的肠道菌群结构,出现内源性感染、二重感染及免疫力下降等问题[18]。因此,如何开发新型的肠道菌群调控的技术是当前肠道菌群领域研究亟需解决的问题。
合成生物学可以实现肠道微生物靶向调控,抗菌物质的诱导性分泌,抗菌物质剂量降低等。在合成生物学中,靶向调控通过对致病菌群体感应分子的检测后,启动运动蛋白以及黏附素的表达,使其能够趋向于病原体生长并黏附在靶细胞上。诱导性分泌同样是在检测到致病菌存在的信号后,激活相应的启动子表达药物分子。由于其具有靶向性和诱导性,从而降低抗菌物质的用量。并且药物在微生物体内合成,可以使用分子量小、光谱抗菌、热稳定性好、抗菌机理独特的抗菌肽替代人工体外合成的抗生素[19],进一步降低了致病菌的耐药性。
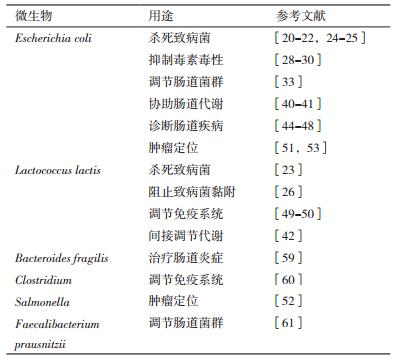
1.1 直接杀死致病菌合成微生物可以通过分泌抗菌物质杀死肠道内的特定致病菌。Saeidi等[20]将抗菌物质的基因序列插入到可以被铜绿假单胞菌分泌物激活的启动子下游。工程大肠杆菌luxR启动子可以被铜绿假单胞菌分泌的LasR复合物激活,开启其下游的E7裂解蛋白和S5脓菌素在细胞内的表达,当E7蛋白的浓度达到阈值时会引起大肠杆菌裂解,这样积累的脓菌素S5就会释放,杀死铜绿假单胞菌。Gupta等[21]利用大肠杆菌建立的装置同样是通过结合P. aeruginosa(PAO1)的QS分子3OC12HSL来开启。研究优化了抗菌物质,使用杀伤效果更好的FlgM-CoPy。同时,增加了一个表达GFP的报告系统,使大肠杆菌同时具有诊断和治疗两个功能。
Hwang等[22]改造的大肠杆菌进一步完善,可以经过识别、迁移、释放抗菌物质等一系列步骤靶向杀死游离或者产生生物膜的铜绿假单胞菌。在大肠杆菌中建立了群体感知(Quorum sensing,QS)装置(感知模块),可以识别游离的和生物膜包裹的铜绿假单胞菌细胞分泌的QS分子酰基高丝氨酸内酯(N-Acyl homoserine lactone,AHL),在感知到铜绿假单胞菌的存在下,细胞的运动和杀伤模块被激活。AHL反应的趋化性使得大肠杆菌在自身表达的CheZ下沿AHL的浓度梯度生长,从而细胞将更接近铜绿假单胞菌(运动模块)。此外,AHL激活了密码子优化后的抗菌肽(MccS)和抗菌膜酶(DNaseI)的表达和分泌,在靶向针对病原体的情况下,通过杀死病原体和破坏生物膜的双重作用消灭致病菌(杀伤模块)。
为了消除肠道内革兰氏阴性致病菌,研究者将两种针对革兰氏阴性菌的抗菌肽A3APO和Alyteserin经过密码子优化后,与信号肽Usp45融合,插入到乳链球菌素诱导启动子PsinA的下游,将该序列转入到乳酸乳球菌IL1403中。该微生物表达相应的抗菌肽,能杀灭革兰氏阴性菌大肠杆菌、沙门氏菌,并对乳酸菌几乎无杀伤作用[23]。
同样机理也适用于杀伤霍乱弧菌。在野生型大肠杆菌中建立一个感应和杀死系统,使其能够通过QS分子CAI-1检测霍乱弧菌,并激活表达裂解蛋白YebF-Art-085,机体裂解后使体内的Art-085释放,杀死霍乱弧菌[24]。
与表达抗菌物质不同,噬菌体的受控递送系统是另一种杀伤病原体的方式。将编码T4 lysozyme、T4 holin、T4 antiholin的序列置于pLacO启动子的控制之下,转入到E. coli K-12中,使其可以诱导表达噬菌体。工程细菌(EB)通过表达噬菌体实现两个功能:调节自身的数量增长和裂解释放噬菌体来消灭霍乱弧菌[25]。
1.2 阻止病原体的黏附Bacillus cereus CH是一种益生菌菌株,其表面的鞭毛蛋白可以与黏蛋白(Mucin)结合。将鞭毛蛋白基因插入乳链菌肽诱导型启动子的下游,转入乳酸乳球菌中。改造的菌株在用乳链菌肽诱导6h后产生表面相关的鞭毛蛋白。重组乳球菌菌株强烈黏附在涂覆黏蛋白的聚苯乙烯板上,同时可以抑制病原体E. coli LMG2092和肠炎沙门氏菌的黏附[26]。
在胃肠道中,李斯特黏附蛋白(LAP)帮助L. monocytogenes由细胞旁路穿过上皮屏障。使用表达LAP的重组益生菌将有助于从肠道中有针对性地清除李斯特菌,并保护高危人群免受感染[27]。
1.3 抑制毒素毒性除了直接杀死病原体外,微生物还能通过降低致病菌毒素的毒力来维持肠道健康。将脑膜炎奈瑟菌或空肠弯曲杆菌的糖基转移酶基因转入无致病性的大肠杆菌,作用于致病性大肠杆菌分泌的肠毒素,治疗和预防肠毒素引起的腹泻症状[28]。同样,将来自淋病奈瑟氏球菌和空肠弯曲杆菌的糖基转移酶基因转入到无害的大肠杆菌中,使其模拟神经节苷脂GM(1)的方式生产嵌合的脂多糖。重组大肠杆菌表达的脂多糖能以高亲合力结合霍乱毒素,能在体外吸附其自身重量5%的毒素。在小鼠感染之后进行,也能保护幼鼠免受毒性霍乱弧菌的攻击[29]。
研究发现,CAI-1也可以降低霍乱毒素的毒性。CAI-1是一种自体诱导分子,当它与另一种分子CAI-2在高浓度下共存时,可以防止霍乱毒素的毒力作用。将编码最终CAI-1合成酶的基因cqsA插入到天然组成型启动子fliC的下游,转入大肠杆菌中表达。用表达CAI-1(Nissle-cqsA)的大肠杆菌预处理小鼠,增加了小鼠摄入霍乱弧菌后的存活率(92%)[30]。
1.4 调控肠道菌群组成许多微生物通过表达群体感应分子进行细胞间交流,通过这种相互作用可以人为调节菌群密度。费希尔氏弧菌是典型的群体感应细菌,lux box、luxR和luxI是已知的群体感应关键元件[31]。为了维持一个相对稳定的细胞密度,利用LuxI/LuxR与自由扩散的信号分子AHL共同构成一个装置。当细胞数量增加时,AHL在细胞和培养基中逐渐积累。当AHL的浓度达到一个较高的数值后,会与LuxR的转录因子结合活化,并激活在luxI启动子(pluxI)之下killer gene(E)的表达。足够高的杀伤蛋白水平会导致细胞死亡,以此来维持一个较为稳定的微生物密度[32]。
同样利用来自费希尔氏弧菌的lux box、luxR和luxI将几种程序性cell-death BioBricks和quorums sensing基因置于lux pR和PlacO-1启动子的控制下,转入大肠杆菌细胞中表达。研究表明,改造后细菌成功地相互感知并相互通信,以巧妙地控制其群体密度[33]。以群体感应信号转导电路为基础,不同的抗生素水平和初始细胞密度会引起相关的菌群关系变化,如灭绝、强制性共生、兼性共生和共生[34]。
微生物间的菌群调节往往不是单向的,也存在双方共同决定的情况。“捕食者-猎物系统”就是这样一个有趣的菌群调节系统。它由两个大肠杆菌菌群组成,通过群体感知的双向通信、工程基因回路来调节彼此的基因表达和存活。捕食者通过诱导猎物中杀伤蛋白的表达来杀死猎物,而猎物通过引发捕食者中解毒剂蛋白的表达来自我拯救[35]。假若将这种机制引入到肠道中,就可以使肠道菌群处于动态平衡之中。
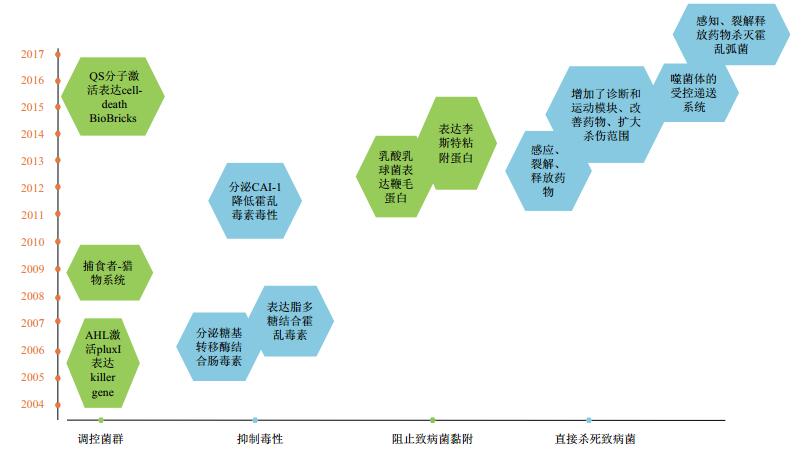
利用合成生物学改造的微生物可以通过抑制病原体黏附于肠壁来防止其入侵,也可以直接杀死病原体或减弱其毒素毒性降低致病性,也能够通过细胞间交流调节肠道菌群,从而维持肠道健康(图 1)。微生物作为药用载体的方法,不用担心药物口服之后在胃酸环境下会被分解代谢,有效的降低抗菌物质的使用量而达到相同的治疗效果,并且省去了体外合成和纯化的步骤。同时,一种微生物可以表达多种抗菌物质,提高了治疗的效率[36]。但是研究发现,抗菌肽对哺乳动物细胞也有一定的细胞毒性。所以在之后的研究中,选择合适的抗菌物质,及时停止微生物表达抗菌肽,并消除在体内的残留,副作用更低,使治疗更加安全是研究的重点。
|
| 图 1 合成生物学调节肠道菌群研究时间轴 |
在医疗上,代谢紊乱是一种状态,即身体的供需平衡被打破,引起相应的疾病,如脂肪代谢紊乱引起肥胖症,糖代谢紊乱引起糖尿病,这些病症的进一步恶化会导致严重的心血管疾病。现今,肥胖症多通过控制饮食加强运动、药物和手术治疗。但是改变生活习惯需要患者强大的意志力,见效缓慢;药物治疗的副作用大且医学上对于使用何种药物没有明确的阐述,用药应十分谨慎;手术治疗会引起并发症。对于糖尿病的治疗主要是控制饮食、注射胰岛素、服用药物等。这种治疗手段无法根治糖尿病且影响患者正常生活,药物的长期使用会对其他器官产生损伤。这些不完善的治疗手段驱使着科学家为了人类的健康继续探索。
研究发现,虽然在肠道中没有发现这些疾病的直接症状,但与肠道的代谢息息相关。摄入食物后,小肠主要进行大分子营养物质的消化吸收;小肠内未被消化的蛋白质、碳水化合物等进入大肠,被大肠内微生物发酵从而进一步利用[37]。微生物既可以帮助肠道内的脂质代谢[38],也可以促进后肠中钙、磷等矿物质的吸收,部分微生物还可以合成多种维生素[39]。肠道进行消化吸收时需要肠道微生物的参与,合成生物学以此为切入点,构建了治疗代谢紊乱的新模型。改造后的微生物可以直接控制营养物质的合成分解,也可以通过调控体内激素的表达来平衡机体内的代谢过程。在合成生物学中,使工程微生物表达代谢途径中的中间产物或终产物来改变其代谢路线;而机体内的反馈作用使得微生物调节激素水平成为可能,通过表达一种激素来促进或抑制其他激素的产生,从而利用激素调节代谢。这种方法不会限制患者的饮食,不必考虑手术的并发症,也没有药物副作用的产生,而且微生物会在肠道中定居繁殖,使得治疗可以持久有效。
2.1 直接代谢N-酰基磷脂酰乙醇胺(NAPE)是N-酰基乙醇酰胺(NAE)脂质家族的前体,其在小肠中合成以减少食物摄取和肥胖。将拟南芥中催化NAPE合成的At1g78690(一种N-酰基转移酶)转入E. coli C41-DE3菌株。这些细菌中的NAPE水平显著增加。实验证明,在饮用水中加入工程化的表达NAPE的E. coli Nissle1917细菌8周后,降低了饲喂高脂肪饮食的小鼠的肥胖水平。与接受标准水或对照细菌的小鼠相比,接受改良细菌的小鼠显著降低了食物摄取量、肥胖、胰岛素抵抗和肝硬化的发生几率。此外,向TallyHo小鼠(肥胖的多基因小鼠模型)施用表达NAPE的细菌,可以抑制体重的增加[40]。
将两个基因序列插入到大肠杆菌中。果糖脱氢酶(fdh)基因,用于将果糖转化为5-酮-D-果糖(Fructose to 5-keto-D-fructose),甘露醇-2-脱氢酶(Mannitol-2-dehydrogenase,mtlK)基因将果糖转化为甘露醇。高果糖喂养的大鼠体重、血清葡萄糖、肝损伤标志物、血清和肝脏脂质谱均显著增加,抗氧化酶活性降低。然而,用EcN(pqq-glf-mtlK)和EcN(pqq-fdh)处理的大鼠,随着血清和肝脏抗氧化酶活性的增加,表现出脂质过氧化作用明显降低,同时观察到肝损伤标记酶的恢复。改造的E. coli Nissle 1917可以减少饮食果糖慢性消耗引起的代谢影响[41]。
2.2 调控宿主代谢无活性全长形式的GLP-1(1-37)可以刺激大鼠和人肠上皮细胞转化成胰岛素分泌细胞。使用SlpA启动子和USP45-LEISS分泌信号肽,下游插入GLP-1(1-37)序列,转入到乳酸杆菌,使其表达分泌GLP-1(1-37)。口服分泌GLP-1(1-37)的共生细菌可以改善大鼠糖尿病模型中的高血糖。每天给予糖尿病大鼠分泌GLP-1(1-37)的乳酸杆菌,观察到大鼠胰岛素水平显著增加,并且葡萄糖耐受性更高。这些大鼠在上肠内发育出足以代替非糖尿病健康大鼠的胰岛素生成细胞。具有重编程细胞的大鼠中的肠组织表达MafA、PDX-1和FoxA2。这些结果证明了安全有效的非吸收性糖尿病口服治疗的潜力,并支持了设计的共生细菌信号传导在体内介导肠细胞功能的概念[42]。
合成生物学通过口服工程微生物的方式,对代谢进行直接或间接调控。这一方法避免了用药的依赖性和手术的副作用,是未来医学的主要发展方向。但是因为身体是一个复杂而且平衡的环境,人为改变其激素水平和代谢途径是否会产生其他损伤或者腺体功能是否因此而降低还无法给出确切的答案。对于身体机理的不明确性,是合成微生物应用的限制,也是将来主要的研究问题。
3 合成生物学与肠道细胞状态的诊断及调节现阶段对肠道病症的诊断多通过肠镜、X线钡餐、放射性核素、选择性血管造影、CT等,这些手段价格高昂,存在安全性问题。近几年来进入人们视野的超声检测,虽然无创伤、无辐射,但其显像效果欠佳,检测前准备工作多、时间长,对操作者的技术要求高[43]。对于疾病及肿瘤的诊断治疗,也因为靶向性的缺乏,用药或者化疗等手段在杀死病变细胞的同时,也对健康细胞产生了副作用,这种“两败俱伤”的治疗方法急需合适的替代。
细胞状态是肠道健康与否的直接反映。在细胞发生病变时,其表达水平也随之产生变化。利用合成微生物,可以对肠道细胞状态进行快速准确的检测。将能被疾病特有信号激活的启动子置于报告分子的上端,就可以在疾病发生的情况下,表达相应的输出信号,进行快速的诊断治疗。合成微生物感知的信号,不局限于蛋白质、糖类或其他活性物质,也可对部分环境变化(缺氧)做出反应。将诊断与治疗起来,有效的解决了现阶段医疗中靶向性不足的问题,对患者健康组织起到了很好的保护作用,是更为完善的一种医疗方法。
3.1 疾病诊断的生物传感器因为一氧化氮水平升高是肠道炎症发生的重要标志,所以选择能够识别NO信号的大肠杆菌,利用其自身的DNA重组酶FimE和催化重组的序列fims共同构成一个报告装置。基因改造后的大肠杆菌,在NO缺乏的情况下,NorR蛋白阻碍NO还原酶NorV的启动子(PnorV)的转录,从而没有重组酶的表达,fimS向右转录表达黄色荧光蛋白(Yellow fluorescent protein,YFP);当NO充足时,NorR与转录因子结合,开启PnorV的转录,表达重组酶FimE,使得fimS序列重组,向左侧转录表达青色荧光蛋白(Cyan fluorescent protein,CFP)。经过试验验证,该报告系统对化学和生物来源一氧化氮都有很好的灵敏度[44]。这一报告装置不仅可以完成检测任务,对于后期加入药物表达,完成由诊断到治疗的一系列任务,是意义重大的进展。
类似的工程细菌诊断疾病的应用还有一些。鼠伤寒沙门氏菌和耶尔森氏菌诱导的小肠结肠炎症中,硫代硫酸盐会在小鼠肠中转化为四硫酸[45]。将海洋细菌中的识别硫代硫酸盐和四硫酸的序列转入到大肠杆菌中,用于诊断结肠炎症[46]。设计的共生鼠大肠杆菌检测在炎症期间产生的四硫酸,通过粪便测试诊断[47]。
Kotula等[48]构建的一个两部分系统,“触发元件”lambda Cro基因由四环素诱导启动子控制转录,“记忆元件”是源自λ噬菌体的cI/Cro区域。用脱水四环素处理含有改造大肠杆菌的小鼠时,所回收的细菌都均为Cro状态,而未处理小鼠的细胞仍处于cI状态。这个机制通过更换相应的启动子,能够诊断更多的肠道疾病。
3.2 调节肠道免疫系统合成生物学在增强肠道免疫作用上也有所尝试。Robert等[49]为了解决口服蛋白质药物会被降解的问题,建立了乳酸乳球菌耐受性细菌传播技术。用于控制分泌type 1型糖尿病自身抗原(GAD)-65和肠道内抗炎症细胞因子IL-10。研究证明,与短期低剂量anti-CD3联合使用,即使在诊断时高血糖严重的情况下,这种治疗仍可以稳定胰岛炎,维持功能性β细胞的数量,使近期发作的NOD小鼠血糖恢复正常。联合治疗不能消除致病效应T细胞,但增加了功能性CD4+ Foxp3+ CD25+调节性T细胞的数量。
del Carmen等[50]建立了2种新型表达系统,重组乳球菌通过递送IL-10在模拟IBD复发的鼠结肠炎模型中具有保护作用。第一个系统基于粘膜表面产生和递送异源蛋白质的应激诱导控制表达系统,第二个系统将il-10 cDNA盒包含在真核DNA表达载体(pValac)中递送至宿主细胞。提供IL-10蛋白或cDNA的乳球菌菌株降低TNBS诱导的慢性结肠炎的严重程度。研究结果证实IL-10作为蛋白质或作为cDNA在模拟IBD复发性质的结肠炎模型中的具有保护作用。
3.3 肿瘤细胞的定位将来自耶尔森氏杆菌假性肺炎的侵入物作为输出模块,使得改造后的大肠杆菌能够侵入癌细胞。为了环境限制入侵,以费希尔氏弧菌lux群体感应回路为背景,将该模块置于缺氧反应性fdhF启动子或阿拉伯糖诱导型araBAD启动子的控制下。这种细菌可以感测肿瘤的微环境,并通过侵入癌细胞和释放细胞毒性剂来发挥杀伤作用[51]。
去毒后的鼠伤寒沙门氏菌(S. typhimurium)菌株可以选择性地生长和表达肿瘤中的外源基因用于靶向治疗。Chen等[52]的设计鼠伤寒沙门氏菌菌株VNP20009,可以在缺氧诱导的nirB启动子的控制下分泌肿瘤坏死因子相关的凋亡诱导配体(TRAIL)。改造后的VNP优先在肿瘤组织中累积,并且nirB启动子有效驱动TRAIL的靶向表达。研究证明,表达TRAIL的VNP20009通过诱导黑素瘤细胞凋亡,产生其抗肿瘤作用。使用表达TRAIL的沙门氏菌介导的肿瘤靶向治疗可以减少肿瘤生长并延长宿主的存活时间。
黏附素的使用也有效的解决了肿瘤定位和治疗靶向性的问题。细菌自身表达的黏附素(adhesins),对其生物膜形成、宿主定植和细胞侵袭有着重要的作用。利用已知的黏附素序列,保留其本身的信号肽和β结构域,只将其Ig结构域替换为heavy-chain-only antibody(VHH),利用自杀质粒将该序列转入大肠杆菌基因组中,构造出一个产生合成黏附素的大肠杆菌。实验证明,该大肠杆菌在随机运动的过程中,紧密黏附于表达相应抗原的肿瘤细胞上,并且,该工程大肠杆菌在肝脏和脾脏的残留量低。这一研究有效的解决了肿瘤定位的问题,将其与药物结合起来,是一种有效的治疗方法[53]。
利用RNA也可以确定肿瘤细胞。构建由siRNA靶向转录Lac阻遏物(LacI)和LacI-controlled启动子CAGop(CAG启动子,随后是具有两个LacO位点的内含子)组成的细胞分型器。当细胞内瞬时表达时,分类器确定六种内源性miRNA的表达谱是否符合HeLa宫颈癌细胞系特征的预定参考谱。将匹配细胞识别为HeLa并触发细胞凋亡[54]。这些肿瘤检测技术在肠道内的尝试与应用,将开启癌症诊断的新篇章。
通过微生物与细胞间的交流,合成生物学对肠道疾病进行监测和定位。人为改造的细菌能够感知环境,区分细胞类型,递送蛋白质,建立由环境信号决定的细菌和癌细胞之间的相互作用,使其完成相应的定位任务。微生物对细胞的膜上和胞外分子,特异性结合并激活相应的启动子开启转录,完成相应的任务。后续研究需要扩大微生物感知信号的类型,并寻找相应的启动子;将细胞状态的检测和定位,与药物治疗相结合是进一步的研究重点。
4 总结与展望 4.1 合成生物学在改善肠道健康状态中存在的问题合成生物学应用于维护肠道健康有着巨大的优势和可塑性,前景十分广阔。按照意愿改造微生物,使其完成指定的检测、黏附、调节、分泌、裂解等任务,能够在不对其他细胞、组织产生损伤的情况下,治疗肠道疾病,维护肠道健康。尽管合成生物学优点明显,依然有很多问题有待解决。
首先,当前合成生物学可以使用的功能基因模块种类和数量有限,许多功能无法完成。合成生物学改造的微生物应该越来越功能化、标准化。它不应只单纯的完成一项任务,而是可以从识别到给药等一系列的“自动化”过程。那就要求研究人员不断丰富合成微生物的序列,建立功能模块,多个模块组合完成信号收集、诊断、表达、分泌、自身裂解等指令;模块自由组合,针对特定的病人完成相应的任务。
其次,宿主的微生物群落和宿主细胞相互作用的机制不够清晰,而这些机制对于微生物组合治疗的合理设计至关重要。现阶段的研究多在啮齿动物中进行验证,但动物与人肠道微生物群落的数量结构和复杂程度仍有差异,所以合成微生物在人体中能否稳定遗传和发挥作用,需要进一步试验确定[55]。
另外,现阶段合成生物学改造的微生物种类选择较少,多局限于大肠杆菌、乳杆菌[56]和乳酸乳球菌[57-58]等(表 1)。研究人员在合成生物学的微生物种类上应有更多尝试,利用各自的特性进行不同的改造。
同时,尽管肠道疾病治疗靶向性的问题在合成生物学的参与下,有了很好的改善,但是治疗药物存在杀伤力不够或杀伤健康细胞的问题。药物是治疗的关键,种类和用量都要严格的考量,防止其对健康的肠道细胞产生不可逆转的伤害。
最后,合成微生物在离开体内后对环境可能会造成影响,在研究过程中避免发生基因的迁移,充分消除其隐患,使合成生物学的应用更加安全。
4.2 应用合成生物学方法的建立新型益生菌体系“个性化医疗”的理念受到越来越多的重视,然而在肠道健康调控的领域,应用益生菌进行肠道健康干预时,其“个性化”却是相当薄弱的。当不同症状、不同疾病的患者,只能服用相同或者相似的几种益生菌制剂时,其治疗或者辅助治疗的效果通常无法保证。应用合成生物学,针对不同肠道疾病的发病机制,开发出针对性的益生菌,将开启益生菌调控肠道健康的新纪元。为此,我们提出了以下技术上和管理上的建议。
数据库层面:合成生物学在肠道疾病诊断和治疗的应用,是调控转录、翻译的结果。这一过程需要复杂的合成基因电路,建立合成生物学工具数据库可以简化这些步骤。数据库根据控制RNA水平、控制蛋白质翻译和转录翻译外的调控3个主要方向分类。调节RNA转录的元件多为启动子,活化子、阻遏蛋白等[62];调节蛋白质翻译的主要为核糖体结合位点、taRNAs等;也可以通过修饰蛋白质等进行调控。除了生物信号识别元件,还包括各式各样的信号输出元件[63]。按照设计需求从数据库中选择相应的电路元件,经过简单的拼接、切割构成完整的基因通路,完成相应的任务[64]。
分子生物学技术层面:选取适合的序列和元件后,要将其连接起来,并转入到微生物体内,这一切依托于分子生物学的方法。常用DNA酶对序列进行合理剪切、连接,后用PCR扩增,克隆的基因利用琼脂糖凝胶电泳和DNA测序验证。将设计的DNA序列插入到载体质粒中,通过制备感受态细胞等方法将质粒转入细胞中或利用Crispr-Cas9技术将基因整合到细胞基因组中,使其能表达出新的性状。
产品有效性评价层面:合成的基因序列需要以微生物为载体发挥作用。优先在培养基中诱导其表达,确定合成电路的可行性;之后将工程细菌置于细胞中培养,验证其是否可以完成指定任务;经过细胞试验的微生物为了确定能在动物体内稳定发挥作用,需要进一步的动物试验;动物试验表现出良好效果并不能完全适用于人体中,所以最终需要将微生物移植到人的肠道中确定其有效性。
产品安全性评价层面:验证工程微生物可以完成指定任务之后,要对其安全性进行评价,这是该产品能否应用于临床治疗的决定步骤。合成生物学所构造的微生物可以通过新表达蛋白及菌株整体的致敏性评价、致畸性评价畸形毒性评价、亚慢性毒性评价等一系列安全评价试验评价其安全性。
产品认证管理层面:《农业转基因生物安全管理条例》第六条规定“国家对农业转基因生物安全实行分级管理评价制度”。合成生物学开发的新型菌株通过食用安全评价和环境安全评价后,会获得安全证书,即证明该生物是安全的。消费者对合成生物学开发的益生菌产品具有知情权和自主选择的权利。在我国,新《食品安全法》第六十九条规定“生产经营转基因食品应当按照规定显著标示”。对于合成生物学开发的益生菌产品可以参考转基因食品的标示管理体系。
合成生物学通过改造微生物,在其体内构建基因回路,使微生物能够完成诊断、靶向治疗、运输、报告等任务,给肠道疾病的治疗带来了希望。合成生物学的出现同样对食品加工、医疗诊断、环境检测治理等领域起到了很好的推进作用。尽管合成生物学有很多的优点,但伦理道德和生物安全问题可能会成为限制其发展的重要因素。随着科技的不断进步,降低风险、发挥优势才能给合成生物学的创造更大的发展空间。与此同时,自然界的一些成就对于人类来说很难再现。而在该领域的进一步发展中成功的从业者将是那些明白如何理解自然生物学作品的,具有工程意识的人[65]。
| [1] |
蔡凯凯, 黄占旺, 叶德军, 等. 益生菌调节肠道菌群及免疫调节作用机理[J]. 中国饲料, 2011, 18: 34-37. |
| [2] |
黄重发. 益生菌对腹泻型肠易激综合征患者肠道菌群调节作用的研究[J]. 疑难病杂志, 2008, 7(6): 362-364. |
| [3] |
韦瑶, 龚剑峰, 朱维铭, 等. 粪便菌群移植治疗溃疡性结肠炎9例临床分析[J]. 中国实用外科杂志, 2014, 34(10): 970-973. DOI:10.7504/CJPS.ISSN1005-2208.2014.10.21 |
| [4] |
李宁. 肠道菌群紊乱与粪菌移植[J]. 肠外与肠内营养, 2014(4): 193-197. |
| [5] |
Steen EJ, Kang Y, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature, 2010, 463(7280): 559. DOI:10.1038/nature08721 |
| [6] |
Sinha J, Reyes SJ, Gallivan JP. Reprogramming bacteria to seek and destroy an herbicide[J]. Nature Chemical Biology, 2010, 6(6): 464-470. DOI:10.1038/nchembio.369 |
| [7] |
唐鸿志, 王伟伟, 张莉鸽, 等. 合成生物学在环境修复中的应用[J]. 生物工程学报, 2017, 3: 018. |
| [8] |
Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440(7086): 940-943. DOI:10.1038/nature04640 |
| [9] |
彭曙光, 谢震. 基于合成基因线路的智能药物[J]. 生物工程学报, 2017, 33(3): 456-466. |
| [10] |
王勇. 新本草计划——基于合成生物学的药用植物活性代谢物研究[J]. 生物工程学报, 2017, 33(3): 478-485. |
| [11] |
赵田鑫, 钟超. 合成生物学技术在材料科学中的应用[J]. 生物工程学报, 2017, 33(3): 494-505. |
| [12] |
孙丽超, 李淑英, 王凤忠, 等. 萜类化合物的合成生物学研究进展[J]. 生物技术通报, 2017, 33(1): 64-75. |
| [13] |
匡雪君, 邹丽秋, 孙超, 等. 天然产物合成生物学体系的优化策略[J]. 生物技术通报, 2017, 33(1): 48-57. |
| [14] |
申晓林, 袁其朋. 生物合成芳香族氨基酸及其衍生物的研究进展[J]. 生物技术通报, 2017, 33(1): 24-34. |
| [15] |
王丽苹, 罗云孜. 合成生物学在天然产物研究中的应用[J]. 生物技术通报, 2017, 33(1): 35-47. |
| [16] |
欧阳钦, 梁红亮. 溃疡性结肠炎[J]. 继续医学教育, 2006, 20(3): 30-34. |
| [17] |
Stenson WF, Tremaine WJ, Cohen RD. Ulcerative Colitis:Clinical Manifestations and Management[J]. Yamada's Atlas of Gastroenterology, 2016, 216-224. |
| [18] |
刘伶俐. 传统抗生素的可能替代物——抗菌肽的研究进展[J]. 江西饲料, 2004(5): 1-3. |
| [19] |
孙科, 邹晓庭. 抗菌肽研究进展及在畜牧生产上的应用[J]. 甘肃畜牧兽医, 2008, 38(3): 43-46. |
| [20] |
Saeidi N, Wong CK, Lo TM, et al. Engineering microbes to sense and eradicate Pseudomonas aeruginosa, a human pathogen[J]. Molecular Systems Biology, 2011, 7(1): 521. |
| [21] |
Gupta S, Bram EE, Weiss R. Genetically programmable pathogen sense and destroy[J]. ACS Synthetic Biology, 2013, 2(12): 715-723. DOI:10.1021/sb4000417 |
| [22] |
Hwang IY, Tan MH, Koh E, et al. Reprogramming microbes to be pathogen-seeking killers[J]. ACS Synthetic Biology, 2013, 3(4): 228-237. |
| [23] |
Volzing K, Borrero J, Sadowsky MJ, et al. Antimicrobial peptides targeting Gram-negative pathogens, produced and delivered by lactic acid bacteria[J]. ACS Synthetic Biology, 2013, 2(11): 643-650. DOI:10.1021/sb4000367 |
| [24] |
Jayaraman P, Holowko MB, Yeoh JW, et al. Repurposing a two-component system-based biosensor for the killing of Vibrio cholera[J]. ACS Synthetic Biology, 2017, 6(7): 1403-1415. DOI:10.1021/acssynbio.7b00058 |
| [25] |
Florjanczyk UA. Design of engineered bacteria for regulated phage release: Progress towards targeted elimination of pathogens[D]. Canada: University of Toronto, 2014.
|
| [26] |
Sánchez B, López P, González-Rodríguez I, et al. A flagellin-producing Lactococcus strain:interactions with mucin and enteropathogens[J]. FEMS Microbiology Letters, 2011, 318(2): 101-107. DOI:10.1111/fml.2011.318.issue-2 |
| [27] |
Koo OK, Amalaradjou MAR, Bhunia AK. Recombinant probiotic expressing Listeria adhesion protein attenuates Listeria monocytogenes virulence in vitro[J]. PLoS One, 2012, 7(1): e29277. DOI:10.1371/journal.pone.0029277 |
| [28] |
Paton AW, Jennings MP, Morona R, et al. Recombinant probiotics for treatment and prevention of enterotoxigenic Escherichia coli diarrhea[J]. Gastroenterology, 2005, 128(5): 1219-1228. DOI:10.1053/j.gastro.2005.01.050 |
| [29] |
Focareta A, Paton JC, Morona R, et al. A recombinant probiotic for treatment and prevention of cholera[J]. Gastroenterology, 2006, 130(6): 1688-1695. DOI:10.1053/j.gastro.2006.02.005 |
| [30] |
Duan F, March JC. Engineered bacterial communication prevents Vibrio cholerae virulence in an infant mouse model[J]. Proc Natl Acad Sci, 2010, 107(25): 11260-11264. DOI:10.1073/pnas.1001294107 |
| [31] |
庞庆霄, 梁泉峰, 祁庆生. 合成生物学开关在代谢工程中的应用[J]. 生物技术通报, 2017, 33(1): 58-63. |
| [32] |
You L, Cox Ⅲ RS, Weiss R, et al. Programmed population control by cell-cell communication and regulated killing[J]. Nature, 2004, 428(6985): 868. DOI:10.1038/nature02491 |
| [33] |
Wang Z, Wu X, Peng J, et al. Artificially constructed quorum-sensing circuits are used for subtle control of bacterial population density[J]. PLoS One, 2014, 9(8): e104578. DOI:10.1371/journal.pone.0104578 |
| [34] |
Hu B, Du J, Zou R, et al. An environment-sensitive synthetic microbial ecosystem[J]. PLoS One, 2010, 5(5): e10619. DOI:10.1371/journal.pone.0010619 |
| [35] |
Balagaddé FK, Song H, Ozaki J, et al. A synthetic Escherichia coli predator-prey ecosystem[J]. Molecular Systems Biology, 2008, 4(1): 187. |
| [36] |
Claesen J, Fischbach MA. Synthetic microbes as drug delivery systems[J]. ACS Synthetic Biology, 2014, 4(4): 358-364. |
| [37] |
朱伟云, 余凯凡, 慕春龙, 等. 猪的肠道微生物与宿主营养代谢[J]. 动物营养学报, 2014, 26(10): 3046-3051. DOI:10.3969/j.issn.1006-267x.2014.10.016 |
| [38] |
黄小燕, 王根虎. 肠道微生物调控脂质代谢的分子机制[J]. 饲料工业, 2012(18): 59-62. |
| [39] |
张日俊. 消化道微生物与宿主营养素的吸收和代谢研究[J]. 中国饲料, 2003(2): 11-14. |
| [40] |
Chen Z, Guo L, Zhang Y, et al. Incorporation of therapeutically modified bacteria into gut microbiota inhibits obesity[J]. The Journal of clinical investigation, 2014, 124(8): 3391. DOI:10.1172/JCI72517 |
| [41] |
Somabhai CA, Raghuvanshi R, Nareshkumar G. Genetically engineered Escherichia coli Nissle 1917 synbiotics reduce metabolic effects induced by chronic consumption of dietaryfructose[J]. PLoS One, 2016, 11(10): e0164860. DOI:10.1371/journal.pone.0164860 |
| [42] |
Duan FF, Liu JH, March JC. Engineered commensal bacteria reprogram intestinal cells into glucose-responsive insulin-secreting cells for the treatment of diabetes[J]. Diabetes, 2015, 64(5): 1794-1803. DOI:10.2337/db14-0635 |
| [43] |
黄争, 范一宏, 吕宾, 等. 超声在肠道疾病诊断中的应用价值[J]. 世界华人消化杂志, 2010, 18(23): 2401-2404. |
| [44] |
Archer EJ, Robinson AB, Süel GM. Engineered E. coli that detect and respond to gut inflammation through nitric oxide sensing[J]. ACS Synthetic Biology, 2012, 1(10): 451-457. DOI:10.1021/sb3000595 |
| [45] |
Riglar DT, Baym M, Kerns SJ, et al. Long-term monitoring of inflammation in the mammalian gut using programmable commensal bacteria[J]. BioRxiv, 2016, 075051. |
| [46] |
Daeffler KNM, Galley JD, Sheth RU, et al. Engineering bacterial thiosulfate and tetrathionate sensors for detecting gut inflammation[J]. Molecular Systems Biology, 2017, 13(4): 923. DOI:10.15252/msb.20167416 |
| [47] |
Riglar DT, Giessen TW, Baym M, et al. Engineered bacteria can function in the mammalian gut long-term as live diagnostics of inflammation[J]. Nat Biotechnol, 2017, 35: 653-658. DOI:10.1038/nbt.3879 |
| [48] |
Kotula JW, Kerns SJ, Shaket LA, et al. Programmable bacteria detect and record an environmental signal in the mammalian gut[J]. Proc Natl Acad Sci, 2014, 111(13): 4838-4843. DOI:10.1073/pnas.1321321111 |
| [49] |
Robert S, Gysemans C, Takiishi T, et al. Oral delivery of glutamic acid decarboxylase(GAD)-65 and IL10 by Lactococcus lactis reverses diabetes in recent-onset NOD mice[J]. Diabetes, 2014, 63(8): 2876-2887. DOI:10.2337/db13-1236 |
| [50] |
del Carmen S, Rosique RM, Saraiva T, et al. Protective effects of lactococci strains delivering either IL-10 protein or cDNA in a TNBS-induced chronic colitis model[J]. Journal of Clinical Gastroenterology, 2014, 48: S12-S17. DOI:10.1097/MCG.0000000000000235 |
| [51] |
Anderson JC, Clarke EJ, Arkin AP, et al. Environmentally controlled invasion of cancer cells by engineered bacteria[J]. Journal of Molecular Biology, 2006, 355(4): 619-627. DOI:10.1016/j.jmb.2005.10.076 |
| [52] |
Chen J, Yang B, Cheng X, et al. Salmonella-mediated tumor-targeting TRAIL gene therapy significantly suppresses melanoma growth in mouse model[J]. Cancer Science, 2012, 103(2): 325-333. DOI:10.1111/cas.2012.103.issue-2 |
| [53] |
Piñero-Lambea C, Bodelón G, Fernández-Periáñez R, et al. Programming controlled adhesion of E. coli to target surfaces, cells, and tumors with synthetic adhesins[J]. ACS Synthetic Biology, 2014, 4(4): 463-473. |
| [54] |
Xie Z, Wroblewska L, Prochazka L, et al. Multi-input RNAi-based logic circuit for identification of specific cancer cells[J]. Science, 2011, 333(6047): 1307-1311. DOI:10.1126/science.1205527 |
| [55] |
Mimee M, Citorik RJ, Lu TK. Microbiome therapeutics—Advances and challenges[J]. Advanced Drug Delivery Reviews, 2016, 105: 44-54. DOI:10.1016/j.addr.2016.04.032 |
| [56] |
Lagenaur LA, Sanders-Beer BE, Brichacek B, et al. Prevention of vaginal SHIV transmission in macaques by a live recombinant Lactobacillus[J]. Mucosal Immunology, 2011, 4(6): 648. DOI:10.1038/mi.2011.30 |
| [57] |
Motta JP, Bermúdez-Humarán LG, Deraison C, et al. Food-grade bacteria expressing elafin protect against inflammation and restore colon homeostasis[J]. Science Translational Medicine, 2012, 4(158): 158ra144. |
| [58] |
Limaye SA, Haddad RI, Cilli F, et al. Phase 1b, multicenter, single blinded, placebo-controlled, sequential dose escalation study to assess the safety and tolerability of topically applied AG013 in subjects with locally advanced head and neck cancer receiving induction chemotherapy[J]. Cancer, 2013, 119(24): 4268-4276. DOI:10.1002/cncr.28365 |
| [59] |
Mazmanian SK, Round JL, Kasper DL. A microbial symbiosis factor prevents intestinal inflammatory disease[J]. Nature, 2008, 453(7195): 620. DOI:10.1038/nature07008 |
| [60] |
Atarashi K, Tanoue T, Shima T, et al. Induction of colonic regulatory T cells by indigenous Clostridium species[J]. Science, 2011, 331(6015): 337-341. DOI:10.1126/science.1198469 |
| [61] |
Sokol H, Pigneur B, Watterlot L, et al. Faecalibacterium prausnitzii is an anti-inflammatory commensal bacterium identified by gut microbiota analysis of Crohn disease patients[J]. Proc Natl Acad Sci, 2008, 105(43): 16731-16736. DOI:10.1073/pnas.0804812105 |
| [62] |
吕永坤, 堵国成, 陈坚, 等. 合成生物学技术研究进展[J]. 生物技术通报, 2015, 31(4): 134-148. |
| [63] |
周益康, 吴亦楠, 王天民, 等. 代谢物生物传感器:微生物细胞工厂构建中的合成生物学工具[J]. 生物技术通报, 2017, 33(1): 1-11. |
| [64] |
Bradley R W, Buck M, Wang B. Tools and principles for microbial gene circuit engineering[J]. Journal of Molecular Biology, 2016, 428(5): 862-888. DOI:10.1016/j.jmb.2015.10.004 |
| [65] |
Way JC, Collins JJ, Keasling JD, et al. Integrating biological redesign:where synthetic biology came from and where it needs to go[J]. Cell, 2014, 157(1): 151-161. DOI:10.1016/j.cell.2014.02.039 |