植物对人类的生存至关重要,它不仅为人类的食物、饲料以及生物能源生产奠定了基础,还能通过光合作用为人类提供氧气。自然条件下,植物的生长会受到内部或外部的刺激,面对这些胁迫,生物体已经进化出多种调节机制来协调其生命活动[1]。其中F-box蛋白参与的泛素-蛋白酶体途径是最重要的生物调节体系之一[2]。
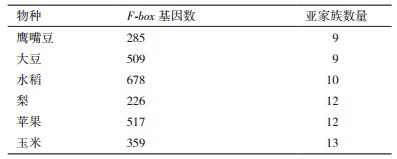
首个F-box蛋白是由Kumar和Paietta[3]两个科学家在1995年研究WD(Trp-Asp)结构域时发现,但当时并未命名,随后由Bai等[4]科学家研究细胞周期蛋白Cyclin F时发现这一类蛋白都含有F-box结构域,将其命名为F-box蛋白。随后的几十年,越来越多的真核F-box基因被发现。令人感兴趣的是,不同物种中的F-box基因的数量差距较大。动物体内的F-box基因数量相对较少,秀丽线虫是目前发现含有F-box基因最多的动物,具有326个F-box基因,而人体内目前已知含有38个,果蝇只含有22个F-box基因[5];植物中F-box基因家族成员个数远高于动物,目前已经鉴定出模式植物苜蓿和拟南芥分别含有972和694个F-box基因[6-7],重要农作物水稻、大豆、玉米和鹰嘴豆分别含有678、509、359和285个F-box基因[8-11],经济作物苹果和梨分别含有517和226个F-box基因[12-13]。数以百计的F-box基因存在于各种植物中,这使其成为植物最大的基因家族之一。
目前的研究表明,F-box基因能够参与调节植物的生长发育以及响应逆境胁迫[14-16]。但是,近年来,对F-box基因家族在植物生长发育以及逆境胁迫中作用的全面综述较少。本文主要就其蛋白结构及其参与植物的生长发育、生物和非生物胁迫进行概述,探讨了F-box基因参与的生命活动,旨为以后F-box的深入研究奠定基础。
1 F-box蛋白结构F-box蛋白的N端F-box结构域大约含有40-50个氨基酸,它通常介导蛋白质-蛋白质相互作用,其保守序列较少,仅有几个位置的氨基酸残基比较保守[17]。C端通常是介导底物识别特异性的二级结构,它也是F-box蛋白分类的主要依据。在动物中,根据C末端二级结构的不同,F-box蛋白被分为3类,即FBXL、FBXW、FBXO[18]。C端含有亮氨酸重复序列(Leucine rich repeats,LRRs)的蛋白质称为FBXL;C端含有WD重复序列的蛋白质称为FBXW;FBXO是指C端含有其他二级结构的蛋白质,如含有亮氨酸拉链(Leucine zipper)、34肽重复序列(Tetratricopeptide repeats,TPRs)、Kelch、环指结构、锌指结构等的蛋白质[19]。除了这些序列外,F-box蛋白基本上没有保守序列[20]。然而在植物中,由于其家族过于庞大,且不同物种之间数量差距很大,所以亚家族分类数量有所不同(表 1)。但一般都包含以下9个亚家族,即FBU:C末端具有未知结构域的F-box蛋白;FBL:C末端富含亮氨酸的重复序列域(LRRs);FBK:C末端具有Kelch结构域;FBA:C末端具有F-box相关域;FBD:C末端具有F-box域;FBT:C末端具有TUB结构域;FBP:C末端具有韧皮部蛋白2(phloem protein 2,PP2)的结构域;FBW:C末端具有WD40域;FBO:具有其他已知结构域或多个C端结构域的F-box蛋白[10-13]。
庞大的F-box基因家族的蛋白成员遍布于植物的各个组织,其中某些蛋白具有组织特异性[12]。这使它具有多种多样的生物学功能,其行使功能的两种主要方式是形成SCF复合体和非SCF复合体。
2.1 F-box蛋白以SCF复合体形式参与泛素-蛋白酶体途径(Ubiquitin-proteasome pathway,UPP)目前国内外研究F-box蛋白的功能主要致力于泛素-蛋白酶体途径。在植物各个组织中,大多数细胞内蛋白质都是经泛素-蛋白酶体途径降解[21-22],所以说泛素-蛋白酶体途径是机体重要的调节机制之一。它主要包括泛素(Ubiquitin,Ub)、E1泛素激活酶(Ubiquitin-activating enzymes)、E2泛素结合酶(Ubiquitin-conjugating enzyme)、E3泛素连接酶(Ubiquitin ligase)及26S蛋白酶体。泛素(Ub)是一种广泛存在于各种细胞相对分子量为8 500 Da的多肽[23],所以又称遍在蛋白。它一般以两种形式存在于细胞中,一种以单体的形式存在;另一种是和其他蛋白结合存在[21]。
泛素-蛋白酶体途径降解蛋白质分为4步(图 1)[24-25]:(1)在ATP存在的情况下,泛素通过泛素激活酶E1活化,在泛素的C末端Gly(甘氨酸)残基和E1内部半胱氨酸残基之间现成高能硫羟酸酯键;(2)E1将活化的泛素转移到E2泛素结合酶,形成新的硫酯键;(3)泛素连接酶E3催化在泛素的C末端的羧基和底物的ε-胺基之间形成异肽键(极少数情况下,泛素转移到底物的氨基末端形成肽键);(4)最终,26S蛋白酶体将其降解成多肽和单体泛素。

|
| 图 1 泛素—蛋白酶途径 |
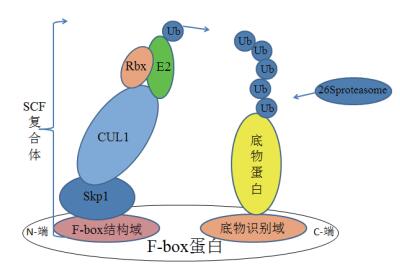
在泛素-蛋白酶体系统(Ubiquitin-proteasome system,UPS)中,E3决定着底物的特异性,它能够靶向具有不同底物识别结构域的底物蛋白用于泛素化[26]。在植物中,根据其作用机制不同,E3可以分为3种主要类型[27-28]:HECT结构域蛋白(与E6-AP同源羧基末端)、RING结构域蛋白(Really interesting new gene)及U-box结构域蛋白。这3种类型中RING结构域的E3在SCF中又称Rbx,它能帮助E2募集到SCF复合体[29]。SCF另外的3个亚基分别为:Skp1(结合蛋白、在植物中称为ASK)、骨架蛋白(Cullin 1、CUL1)和F-box蛋白[30]。Zheng等[31]通过悬滴漫射等方法分析出SCF复合物的晶体结构图(图 2)。其中Skp1、CUL1、Rbx1构成SCF的基本骨架且和F-box蛋白中的F-box结构域特异性结合,F-box蛋白通过C端的底物识别域和不同的底物蛋白结合(图 3)。
|
| 图 3 F-box蛋白作用示意图 |
大部分的F-box蛋白以SCF复合物行使其功能,也有少部分的F-box蛋白以非SCF复合体发挥作用。然而,与广泛研究的SCF型F-box蛋白相比,非SCF型F-box蛋白的功能仍然很大程度上是未知的[32]。非SCF型F-box蛋白与SCF型F-box蛋白不同之处一般在于,非SCF型F-box蛋白N末端区域具有保守的F-box结构域,其可与Skp1相互作用,但不能与Cul1和Rbx1形成复合物。例如,Rcy1蛋白与Skp1结合不依赖SCF复合体,且它们的底物不被泛素化降解[33]。另外,Kim等[34]研究发现F-box蛋白DIA2也是通过非SCF复合体发挥作用。
3 植物中F-box基因家族的功能 3.1 F-box与植物的生长发育植物中第一个鉴定出的F-box基因是拟南芥的AtUFO(Unusual floral orangs)基因,它能够调节花分生组织和花器官发育[35-36]。Zhao等[37]研究表明:UFO与ASK1和LEAFY基因相互作用正调节B功能基因(APETALA3,AP3)的表达,当拟南芥中的AtUFO突变时能导致花发育存在很多缺陷。FOF2是一个受自主通路调节的光诱导F-box基因,它能够促进FLC表达来调控开花。AtFOF2在光照条件下的表达量显著高于黑暗条件,当黑暗处理2 h后其转录水平降低11倍。正常植株在花过度期间(7-19 d)AtFOF2表达水平较低,当其超表达时,能够延迟植株开花,但是这种抑制作用能够通过对植物进行春化来解决[38]。
叶片是植物的主要光合器官,在植物生长发育阶段,叶片非常重要。F-box基因家族中有很多能作用于叶片的基因。例如,FBX92能够影响植物叶片的大小,Baute等[14]研究发现:ZmFBX92在拟南芥中异位表达能导致拟南芥的叶片增大,然而拟南芥中同源基因AtFBX92超表达之后,拟南芥的叶片变小,降低AtFBX92表达水平之后,则观察到相反的作用。这主要是F-box蛋白FBX92影响细胞分裂导致细胞数量不同最终影响叶片大小。番茄中的SIGID2在所有组织中都普遍表达,当其沉默后植株表现出矮化和深绿叶。实验表明,在相同栽培条件下SIGID2i转基因株系表现出明显较短节间的严重矮化;在同一发育阶段,SIGID2i转基因株系的叶片细胞更小更紧凑。植物的矮化一般和GA代谢异常有关,通过喷洒GA能够使其恢复正常。然而SIGID2i转基因株对GA不敏感,其内源性GA含量显著高于野生型(WT),喷洒GA3后植株矮化也未见改善。SIGID2i植株深绿也表形是由于其总叶绿素含量比WT增加了1.5-1.8倍[39]。
Chen等[40]研究发现,水稻中的F-box蛋白OsFBK12在泛素-蛋白体途径(UPP)中能够和S相激酶相关蛋白相互作用,并靶向降解底物OsSAMS1,导致水稻体内乙烯水平发生变化,进而调节叶片衰老和籽粒的大小。该实验采用了超表达和干扰两种方法进行研究,超表达OsFBK12植株体内乙烯含量降低,叶片衰老和种子萌发延迟,籽粒增大;OsFBK12i的乙烯水平含量升高,促进植物的衰老进程。Hong等[41]报道小麦的F-box基因TaCFBD在早起花序发育阶段表达量非常高,并且受冷胁迫诱导。其蛋白TaCFBD通过泛素-蛋白酶体途径调节小麦的生长发育并能调节麦穗的大小。
3.2 F-box与植物的非生物胁迫非生物胁迫是影响植物生长发育的关键因素,主要有干旱胁迫、盐碱(盐、碱)胁迫、低温胁迫和重金属胁迫等[42]。当植物面对这些非生物胁迫,它能够通过自身调节来适应环境[43]。研究表明,小麦TaFBA1基因能响应多种非生物胁迫[15, 44-45],它能在植物不同器官中调控多个靶位点来优化植物生长发育。在干旱、氧化及盐胁迫条件下,超表达TaFBA1的转基因烟草发芽率、根长、叶绿素含量、净光合速率和体内抗氧化酶活性等指标都高于野生型;而且,超表达TaFBA1的转基因烟草体内的ROS积累、MDA含量、膜损伤程度及植物的生长抑制均低于WT。另外,在干旱胁迫条件下,转基因植株的蒸腾速率和气孔导度都高于WT[44, 46];在氧化胁迫条件下,转基因植株体内的蛋白质损伤小于WT,且抗氧化酶活性更高[45];在长时间盐胁迫条件下,转基因植株具有更大的生物量积累,且叶片中Na+含量较低,K+含量高于WT[15]。
拟南芥F-box基因AtPP2-B11不仅作为负调节因子在干旱胁迫中起到重要作用,作为盐胁迫的正调节因子[47-48]。在干旱胁迫条件下,F-box基因AtPP2-B11的表达被诱导,超表达AtPP2-B11植株体内的AtLEA14表达量降低,导致植株对干旱的耐受性降低[47]。在盐胁迫条件下,AtPP2-B11在转录和翻译水平的表达量均显著增加,超表达AtPP2-B11通过上调膜蛋白的表达水平,抑制了ROS积累和影响体内Na+平衡来提高植株的耐盐性[48]。
MAX2是一种多功能的F-box蛋白,它不仅在多种信号传导中起调节作用,还能调控光形态发生和响应生物及非生物胁迫[49-51]。2014年,Bu和Ha等[51-52]发现拟南芥max2突变体对干旱和盐非常敏感。MdMAX2是MAX2的同源蛋白,An等[53]研究发现MdMAX2超表达能够提高植株的耐盐和耐旱性。在盐(150 mmol/L NaCl)、干旱(300 mmol/L甘露醇)条件下,转基因种子的萌发率和幼苗存活率都显著高于WT。在盐胁迫条件下,MdMAX2超表达株系中SOS相关基因的表达水平显著高于WT;在干旱胁迫条件下,MdMAX2超表达株系通过降低失水来增强耐旱性。
3.3 F-box与植物的生物胁迫生物胁迫也是影响植物生长发育的重要因素。在生长发育过程中,植物经常受到潜在的病原菌侵袭,它们也产生许多防御途径保护自己。气孔闭合功能是植物抵抗细菌先天免疫反应的一个重要的组成部分,当病原体细菌接触植物叶片时,植物能够通过关闭气孔来防御伤害[54],而MAX2是病原体触发的气孔关闭所必需的一种蛋白。研究显示,拟南芥F-box蛋白MAX2能够提高其对细菌的抗性,而max2突变体植株的气孔导度增强,气孔闭合受损,有利于病原菌侵染[55]。拟南芥中At2g02360(F-box-Nicta)基因编码一个糖凝结F-box蛋白,当植物受到丁香假单胞菌感染时,F-box-Nicta基因的表达增强;超表达F-box-Nicta转基因拟南芥能够抑制PstDC3000定植,减少叶片损伤[16]。水稻的F-box基因OsDRF1能够上调防御相关基因(PR-1a、Sar8.2b)的表达来增强植株的抗病性。超表达OsDRF1转基因烟草对番茄花叶病毒和丁香假单胞菌的抗性增强,在接种番茄花叶病毒(Tobacco mosaic virus,To MV)4 d后,接种丁香假单胞菌(Pseudo monas syringae,Psp)7 d后,转基因植株的病害程度明显小于WT[56]。Paquis等[57]研究发现BIG-24.1是一种调控葡萄防御灰霉菌的F-box基因,当葡萄受到灰霉菌感染时,BIG-24.1表达量显著上升。
4 展望F-box基因作为植物最大的基因家族之一,在植物的生长发育和逆境胁迫中起到重要的作用。随着基因组测序的不断完善,越来越多物种的F-box基因被鉴定出。目前的文库和表达谱数据分析方法,虽然有助于我们预测F-box基因的功能,但对于其具体功能和作用机制需要我们通过实验来证明。到目前为止,人们已经鉴定出部分F-box基因的功能,但有些F-box基因的作用机制尚不清晰。例如,能够增强转基因烟草抗旱、耐盐及抗氧化的小麦TaFBA1基因[44-46],拟南芥中干旱胁迫的负调节因子AtPP2-B11基因[47]等。然而,现在我们了解的这些F-box基因对于整个植物F-box基因家族来说只是冰山一角。因此,在F-box基因家族方面的研究有待进一步深入。随着人们对其家族更加全面的认识,把在拟南芥和烟草中已经验证过对植物生长发育和抗逆境胁迫的基因运用到水稻、小麦、大豆等重要农作物中,这将对培育作物新品种以及提高粮食产量具有重大意义。
| [1] |
Fahad S, Bajwa AA, Nazir U, et al. Crop production under drought and heat stress:plant responses and management options[J]. Frontiers in Plant Science, 2017, 01147. |
| [2] |
Duplan V, Rivas S. E3 ubiquitin-ligases and their target proteins during the regulation of plant innate immunity[J]. Frontiers in Plant Science, 2014, 00042. |
| [3] |
Kumar A, Paietta JV. The sulfur controller-2 negative regulatory gene of Neurospora crassa encodes a protein with beta-transducin repeats[J]. Proc Natl Acad Sci USA, 1995, 92(8): 3343-3347. DOI:10.1073/pnas.92.8.3343 |
| [4] |
Bai C, Sen P, Hofmann K, et al. SKP1 connects cell cycle regulators to the ubiquitin proteolysis machinery through a novel motif, the F-box[J]. Cell, 1996, 86(2): 263-274. DOI:10.1016/S0092-8674(00)80098-7 |
| [5] |
Kipreos ET, Pagano M. The F-box protein family[J]. Genome Biology, 2000, 1(5): 1-7. |
| [6] |
Song JB, Wang YX, Li HB, et al. The F-box family genes as key elements in response to salt, heavy mental, and drought stresses in Medicago truncatula[J]. Functional & Integrative Genomics, 2015, 15(4): 495-507. |
| [7] |
Jia Q, Xiao ZX, Wong FL, et al. Genome-wide analyses of the soybean F-box gene family in response to salt stress[J]. Int J Mol Sci, 2017, 18(4): 818-835. DOI:10.3390/ijms18040818 |
| [8] |
Gagne JM, Downes BP, Shiu SH, et al. The F-box subunit of the SCF E3 complex is encoded by a diverse superfamily of genes in Arabidopsis[J]. Proc Natl Acad Sci, 2002, 99(17): 11519-11524. DOI:10.1073/pnas.162339999 |
| [9] |
Jain M, Nijhawan A, Arora R, et al. F-box proteins in rice. Genome-wide analysis, classification, temporal and spatial gene expression during panicle and seed development, and regulation by light and abiotic stress[J]. Plant Physiology, 2007, 143(4): 1467-1483. DOI:10.1104/pp.106.091900 |
| [10] |
Cui HR, Zhang ZR, Lv W, et al. Genome-wide characterization and analysis of F-box protein-encoding genes in the Malus domestica genome[J]. Mol Genet Genomics, 2015, 290(4): 1435-1446. DOI:10.1007/s00438-015-1004-z |
| [11] |
Jia F, Wu B, Li H, et al. Genome-wide identification and characterisation of F-box family in maize[J]. Mol Genet Genomics, 2013, 288(11): 559-577. DOI:10.1007/s00438-013-0769-1 |
| [12] |
Gupta S, Garg V, Kant C, et al. Genome-wide survey and expression analysis of F-box genes in chickpea[J]. BMC Genomics, 2015, 16(1): 1-15. DOI:10.1186/1471-2164-16-1 |
| [13] |
Wang GM, Yin H, Qiao X, et al. F-box genes:Genome-wide expansion, evolution and their contribution to pollen growth in pear(Pyrus bretschneideri)[J]. Plant Science, 2016, 253: 164-175. DOI:10.1016/j.plantsci.2016.09.009 |
| [14] |
Baute J, Polyn S, De BJ, et al. F-Box Protein FBX92 Affects Leaf size in Arabidopsis thaliana[J]. Plant & Cell Physiology, 2017, 58(5): 962-975. |
| [15] |
Zhao Z, Zhang G, Zhou S, et al. The improvement of salt tolerance in transgenic tobacco by overexpression of wheat F-box gene TaFBA1[J]. Plant Science, 2017, 259: 71-85. DOI:10.1016/j.plantsci.2017.03.010 |
| [16] |
Stefanowicz K, Lannoo N, Zhao Y, et al. Glycan-binding F-box protein from Arabidopsis thaliana protects plants from Pseudomonas syringae infection[J]. BMC Plant Biol, 2016, 16(1): 213-226. DOI:10.1186/s12870-016-0905-2 |
| [17] |
Lechner E, Achard P, Vansiri A, et al. F-box proteins everywhere[J]. Curr Opin Plant Biol, 2006, 9(6): 631-638. DOI:10.1016/j.pbi.2006.09.003 |
| [18] |
王秀燕, 孙莉萍, 张建锋, 等. F-box蛋白家族及其功能[J]. 生命科学, 2008(5): 807-811. |
| [19] |
Kipreos E, Pagano M. The F-box protein family[J]. Genome Biology, 2000, 1(5): 1-7. |
| [20] |
Xue F, Cooley L. Kelch encodes a component of intercellular bridges in Drosophila egg chambers[J]. Cell, 1993, 72(5): 681-693. DOI:10.1016/0092-8674(93)90397-9 |
| [21] |
Rogel MR, Jaitovich A, Ridge KM. The role of the ubiquitin proteasome pathway in keratin intermediate filament protein degradation[J]. Proc Ame Thorac Soc, 2010, 7(1): 71-76. DOI:10.1513/pats.200908-089JS |
| [22] |
Shang F, Taylor A. Roles for the ubiquitin-proteasome pathway in protein quality control and signaling in the retina:implications in the pathogenesis of age-related macular degeneration[J]. Molecular Aspects of Medicine, 2012, 33(4): 446-466. DOI:10.1016/j.mam.2012.04.001 |
| [23] |
李杨, 李栋. 泛素连接酶-底物选择关系的研究进展[J]. 生物技术通报, 2015, 31(1): 11-20. |
| [24] |
Wang F, Deng XW. Plant ubiquitin-proteasome pathway and its role in gibberellin signaling[J]. Cell Res, 2011, 9: 1286-1294. |
| [25] |
Buckley DL, Crews CM. Small-molecule control of intracellular protein levels through modulation of the ubiquitin proteasome system[J]. Angew Chem Int Ed Engl, 2014, 9: 2312-2330. |
| [26] |
Sang Y, Yan F, Ren X. The role and mechanism of CRL4 E3 ubiquitin ligase in cancer and its potential therapy implications[J]. Oncotarget, 2015, 6(40): 42590-42602. |
| [27] |
Yu F, Wu Y, Xie Q. Ubiquitin-proteasome system in ABA signaling:from perception to action[J]. Mol Plant, 2016, 9(1): 21-23. DOI:10.1016/j.molp.2015.09.015 |
| [28] |
Yee D, Goring DR. The diversity of plant U-box E3 ubiquitin ligases:from upstream activators to downstream target substrates[J]. J Exp Bot, 2009, 60(4): 1109-1121. DOI:10.1093/jxb/ern369 |
| [29] |
Kawakami T, Chiba T, et al. NEDD8 recruits E2-ubiquitin to SCF E3 ligase[J]. EMBO J, 2001, 20(15): 4003-4012. DOI:10.1093/emboj/20.15.4003 |
| [30] |
Chen L, Hellmann H. Plant E3 ligases:flexible enzymes in a sessile world[J]. Mol Plant, 2013, 6(5): 1388-1404. DOI:10.1093/mp/sst005 |
| [31] |
Meng X, Wang C, Rahman SU, et al. Genome-wide identification and evolution of HECT genes in soybean[J]. Int J Mol Sci, 2015, 16(4): 8517-8535. DOI:10.3390/ijms16048517 |
| [32] |
Liu Y, Nakatsukasa K, et al. Non-SCF-type F-box protein Roy1/Ymr258c interacts with a Rab5-like GTPase Ypt52 and inhibits Ypt52 function[J]. Mol Biol Cell, 2011, 22(9): 1575-1584. DOI:10.1091/mbc.E10-08-0716 |
| [33] |
Hermand D. F-box proteins:more than baits for the SCF?[J]. Cell Division, 2006, 1(1): 30-36. DOI:10.1186/1747-1028-1-30 |
| [34] |
Kim DH, Zhang W, Koepp DM. The Hect domain E3 ligase Tom1 and the F-box protein Dia2 control Cdc6 degradation in G1 phase[J]. J Biol Chem, 2012, 287(53): 44212-44220. DOI:10.1074/jbc.M112.401778 |
| [35] |
Levin JZ, Meyerowitz EM. UFO:an Arabidopsis gene involved in both floral meristem and floral organ development[J]. Plant Cell, 1995, 7(5): 529-548. DOI:10.1105/tpc.7.5.529 |
| [36] |
Ni W, Xie D, Hobbie L, et al. Regulation of flower development in Arabidopsis by SCF complexes[J]. Plant Physiology, 2004, 134(4): 1574-1585. DOI:10.1104/pp.103.031971 |
| [37] |
Zhao D, Yu Q, Chen M, et al. The ASK1 gene regulates B function gene expression in cooperation with UFO and LEAFY in Arabidopsis[J]. Development, 2001, 128(14): 2735-2746. |
| [38] |
He R, Li X, Zhong M, et al. A photo-responsive F-box protein FOF2 regulates floral initiation by promoting FLC expression in Arabidopsis[J]. Plant Journal, 2017, 13607. |
| [39] |
Liu Q, Guo X, Chen G, et al. Silencing SlGID2, a putative F-box protein gene, generates a dwarf plant and dark-green leaves in tomato[J]. Plant Physiol Biochem, 2016, 109: 491-501. DOI:10.1016/j.plaphy.2016.10.030 |
| [40] |
Chen Y, Xu Y, Luo W, et al. The F-box protein OsFBK12 targets OsSAMS1 for degradation and affects pleiotropic phenotypes, including leaf senescence, in rice[J]. Plant Physiology, 2013, 163(4): 1673-1685. DOI:10.1104/pp.113.224527 |
| [41] |
Hong MJ, Kim DY, Kang SY, et al. Wheat F-box protein recruits proteins and regulates their abundance during wheat spike development[J]. Mol Biol Rep, 2012, 39(10): 9681-9696. DOI:10.1007/s11033-012-1833-3 |
| [42] |
Sarkar T, Mogili T, Sivaprasad V. Improvement of abiotic stress adaptive traits in mulberry(Morus spp.):an update on biotechnological interventions[J]. Biotech, 2017, 7(3): 214-228. |
| [43] |
Nawaz G, Kang H. Chloroplast-or mitochondria-targeted DEAD-Box RNA helicases play essential roles in organellar RNA metabolism and abiotic stress responses[J]. Frontiers in Plant Science, 2017, 8: 00871. DOI:10.3389/fpls.2017.00871 |
| [44] |
Zhou S, Sun X, Yin S, et al. The role of the F-box gene TaFBA1 from wheat(Triticum aestivum L.)in drought tolerance[J]. Plant Physiol Biochem, 2014, 84: 213-223. DOI:10.1016/j.plaphy.2014.09.017 |
| [45] |
Zhou SM, Kong XZ, Kang HH, et al. The involvement of wheat F-box protein gene TaFBA1 in the oxidative stress tolerance of plants[J]. PLoS One, 2015, 10(4): e0122117. DOI:10.1371/journal.pone.0122117 |
| [46] |
Kong X, Zhou S, Yin S, et al. Stress-inducible expression of an F-box gene TaFBA1 from wheat enhanced the drought tolerance in transgenic tobacco plants without impacting growth and development[J]. Frontiers in Plant Science, 2016, 7: 01295. |
| [47] |
Li Y, Jia F, Yu Y, et al. The SCF E3 ligase AtPP2-B11 plays a negative role in response to drought stress in Arabidopsis[J]. Plant Molecular Biology Reporter, 2014, 32(5): 943-956. DOI:10.1007/s11105-014-0705-5 |
| [48] |
Jia F, Wang C, Huang J, et al. SCF E3 ligase PP2-B11 plays a positive role in response to salt stress in Arabidopsis[J]. J Exp Bot, 2015, 66(15): 4683-4697. DOI:10.1093/jxb/erv245 |
| [49] |
Nelson DC, Smith SM. F-box protein MAX2 has dual roles in karrikin and strigolactone signaling in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2011, 108(21): 8897-8902. DOI:10.1073/pnas.1100987108 |
| [50] |
Shen H, Zhu L, Bu QY, et al. MAX2 affects multiple hormones to promote photomorphogenesis[J]. Mol Plant, 2012, 3: 750-762. |
| [51] |
Bu Q, Lv T, Shen H, et al. Regulation of drought tolerance by the F-box protein MAX2 in Arabidopsis[J]. Plant Physiology, 2014, 164(1): 424-439. DOI:10.1104/pp.113.226837 |
| [52] |
Ha CV, Leyva-González MA, Osakabe Y, et al. Positive regulatory role of strigolactone in plant responses to drought and salt stress[J]. Proc Natl Acad Sci USA, 2014, 111(2): 851-856. DOI:10.1073/pnas.1322135111 |
| [53] |
An JP, Li R, Qu FJ, et al. Apple F-Box protein MdMAX2 regulates plant photomorphogenesis and stress response[J]. Frontiers in Plant Science, 2016, 7: 01685. |
| [54] |
Melotto M, Underwood W, Koczan J, et al. Plant stomata function in innate immunity against bacterial invasion[J]. Cell, 2006, 126: 969-980. DOI:10.1016/j.cell.2006.06.054 |
| [55] |
Piisilä M, Keceli MA, Brader G, et al. The F-box protein MAX2 contributes to resistance to bacterial phytopathogens in Arabidopsis thaliana[J]. BMC Plant Biology, 2015, 15(1): 53-70. DOI:10.1186/s12870-015-0434-4 |
| [56] |
Cao Y, Yang Y, Zhang H, et al. Overexpression of a rice defense-related F-box protein gene OsDRF1 in tobacco improves disease resistance through potentiation of defense gene expression[J]. Physiol Plant, 2008, 134(3): 440-452. DOI:10.1111/ppl.2008.134.issue-3 |
| [57] |
Paquis S, Mazeyrat-Gourbeyre F, Fernandez O, et al. Characteriza-tion of a F-box gene up-regulated by phytohormones and upon biotic and abiotic stresses in grapevine[J]. Mol Biol Rep, 2011, 38(5): 3327-3337. DOI:10.1007/s11033-010-0438-y |