2. 华南农业大学昆虫毒理研究室,广州 510642
2. Laboratory of Insect Toxicology, South China Agricultural University, Guangzhou 510642
昆虫组织蛋白酶,尤其是Cathepsin B类酶,在不同物种中被研究,包括双翅目,鞘翅目,半翅目,鳞翅目等。在大多数鞘翅目昆虫中,组织蛋白酶B是肠内的一类蛋白水解酶,属于木瓜蛋白酶类[1]。大量的研究表明,组织蛋白酶与昆虫的消化作用、昆虫的个体发育、组织分化以及变态反应密切相关,因此已被考虑为该目害虫防治的靶蛋白。在某些双翅目和半翅目昆虫中,半胱氨酸蛋白酶也是重要的消化蛋白酶[2]。许多研究证实组织蛋白酶参与了昆虫的胚前期卵子发生以及卵子发生过程中某些卵子细胞的退化,蚊子(Culex pipiens pallens)产卵前的卵子细胞解体过程中,出现了Cathepsin B和Cathepsin L的活化[3]。在棉铃虫(Helicoverpa armigera)成虫的脂肪体、卵巢、卵中含有组织蛋白酶B[4-5]。经证实蚊子的卵子退化实际上是一种细胞凋亡,也即程序性细胞死亡过程(program cell death,PCD)[6]。这说明组织蛋白酶参与了卵子细胞的程序化死亡。
昆虫组织蛋白酶通过其蛋白降解活性和维持正常的细胞程序性死亡功能,在昆虫生长发育和代谢过程中起着重要的作用。在各种卵生动物中,胚胎发育处于外源营养缺乏的条件下,因此发育的胚胎必须从卵储存物中获取所需的营养,组织蛋白酶作为重要的消化蛋白酶[7],在这一过程中通过分解氨基酸组分高度丰富的卵黄蛋白提供营养物质。大量实验证实,组织蛋白酶通过降解卵黄蛋白为胚胎发育提供营养物质,从而参与胚胎发生过程[8-9]。全变态昆虫的变态发育是一个包含了大量组织降解与重建的关键发育事件。此期间,大多数幼虫组织逐步被破坏分解,而成虫组织则从成虫盘中重新发育分化形成。棉铃虫组织蛋白酶B在成虫期表达,参与成虫脂肪的解体,为卵母细胞发育提供养料,在胚胎发育中水解卵黄蛋白供给胚胎发育必需的氨基酸[10]。用抑制剂抑制组织蛋白酶活性,可以扰乱昆虫的正常代谢、导致昆虫发育不正常或终止胚胎发育,进而杀死虫体[11-12],达到防治害虫的目的。因此,组织蛋白酶可以成为害虫防治的重要靶标蛋白,尤其是Cathepsin B(SCB)这种组织蛋白酶,它们在昆虫体内含量丰富,功能多样,已在多种昆虫中被研究,其基因序列被克隆和深入分析,这些研究为进一步探索组蛋白酶的功能奠定基础。
综上所述,组织蛋白酶参与昆虫发育过程正常的细胞程序性死亡是昆虫完成变态发育所必需的,因此组织蛋白酶可以成为农药作用的新靶标,任何抑制组织蛋白酶活性的化合物都有可能干扰昆虫正常发育,研究组织蛋白酶及其与抑制化合物的相互作用,对新农药开发有重要理论指导意义。本研究以草地贪夜蛾离体细胞Sf9为对象开展研究,克隆组织蛋白酶SCB基因,分析序列特征,再采用分子模拟软件包以同源建模的方法模拟预测SCB三维结构和活性中心,以及其与对应抑制剂的结合模式,旨为基于组织蛋白酶靶标的新农药开发及其作用机理研究奠定基础。
1 材料与方法 1.1 材料草地贪夜蛾(Spodoptera frugiperda)Sf9细胞系(中山大学生命科学学院生物防治重点实验室惠赠)。trans1-T1感受态细胞和pEASY-T1克隆载体购自北京全式金生物技术有限公司。ExTaq DNA聚合酶(5 U/μL)、dNTPs(each 15 mmol/L)购自东盛生物公司; 凝胶DNA回收试剂盒购自天根生化科技有限公司; DL2000、AMV反转录酶(10 U/μL)购自TaKaRa公司; CA-074me(CA)购自默克公司; EB自北京百泰克生物科技有限公司; 质粒抽提试剂盒购自天根生化科技有限公司; RNA提取试剂Trizol购自Invitrogen公司; 基因组DNA小样抽提试剂盒购自碧云天生物技术研究所; 引物合成和测序均由上海英骏生物技术有限公司完成。其它常规试剂为分析纯试剂; DU-640核酸蛋白检测仪(Beckman公司); PCR仪(Bio-Red公司); Power Pac1000电泳仪(Bio-Red公司); Tanon-4200凝胶成像系统(上海天能科技有限公司); Discovery Studio 2.1分析软件购自创藤科技有限公司。
1.2 方法 1.2.1 PCR引物的设计与合成借助NCBI信息库的BLAST程序和DNAStar软件对NCBI公布的棉铃虫(Helicoverpa armigera)、烟夜蛾(Helicoverpa assulta)和甜菜夜蛾(Spodoptera exigua)三种夜蛾科昆虫Cathepsin B cDNA序列进行辅助分析,发现三者的ORF序列保守性很强,同源性在90%以上。同时前人研究表明可以利用棉铃虫Cathepsin B ORF序列两端直接设计引物克隆烟夜蛾Cathepsin B ORF序列[12],本研究中Sf9细胞从草地贪夜蛾(S. frugiperda)分离获得,而甜菜夜蛾与草地贪夜蛾同属灰翅夜蛾属,所以甜菜夜蛾与草地贪夜蛾Cathepsin B基因的同源性应该更高,利用甜菜夜蛾的Cathepsin B ORF序列两端设计引物更能直接克隆出草地贪夜蛾的Cathepsin B ORF序列,引物为:SexiCB P1:5'-atggtcgcttcgcgt-3'(正向引物); SexiCB P2:5'-ttaaacatcaacaag-3'(反向引物)。
1.2.2 RNA和DNA的提取Sf9细胞总RNA和基因组DNA的提取,cDNA的反转录合成,均参照对应的试剂盒说明和步骤进行。
1.2.3 SCB ORF序列的克隆与分析以反转录合成的Sf9细胞cDNA为模板,利用引物SexiCB P1和SexiCB P2分别进行PCR扩增,反应条件为:95℃预变性5 min; 95℃变性1 min,54℃退火1 min,72℃延伸1 min,35个循环。最后72℃延伸10 min。电泳分析PCR产物,出现一条1 000 bp左右的条带,DNA回收试剂盒纯化PCR产物,产物序列,利用NCBI BLAST程序把测定结果与甜菜夜蛾的Cathepsin B基因序列进行比对分析,确定目的基因SCB片段序列。
为了获得SCB基因ORF全长序列,将所获的PCR产物切胶回收克隆到pEASY-T1克隆载体上构建SCB-pEASY-T1重组质粒,将重组质粒转化到大肠杆菌trans1-T1,挑取阳性克隆先进行菌落PCR鉴定和质粒PCR鉴定,确定阳性克隆。把阳性克隆菌液送测序,通过序列比对分析,确定SCB基因ORF全长序列。采用NCBI的BLAST,ClustalX2等生物信息学软件对SCB ORF序列进行特征分析。
1.2.4 SCB编码区内含子的确定根据已获得的SCB ORF cDNA序列两端设计对应的特异性引物:
SCB P1:5'-ATGGTCGCTTCGCGTGCAAC-3'(正向引物),SCB P2:5'-TTAAACATCAACAAGGAGTG-3'(反向引物)。
以提取的Sf9细胞基因组DNA为模板进行PCR扩增,PCR条带与基因克隆过程的PCR产物电泳检测结果比较,如结果一致,将PCR产物测序,继而把基因组DNA为模板的扩增产物序列与SCB ORF进行序列比对,如结果一致,表明SCB编码区没有内含子。
1.2.5 SCB同源建模预测三维结构同源建模是以已知三维结构的高同源性蛋白为模板,所需模板序列相似度超过30%[13]。步骤包括:(1)模板蛋白的确定:采用BLAST把SCB氨基酸序列在PDB蛋白结构数据库中进行序列相似性搜索,确定用于SCB三维结构建模的同源蛋白。(2)序列联配:采用Discovery Studio/Homology软件包中的Clustal W程序对目的蛋白与模板蛋白序列进行比对[14],然后根据晶体结构知识手动调整序列联配结构,以确定模建蛋白的SCRs(Structural Conserved Regions,结构保守区)和Loop区。(3)蛋白质三维结构的模建和动力学优化:在序列比对基础上,利用软件包的Homology模块中的Modeler程序构建SCB的三维结构。模建后对各模型以PDF total energy进行排序,选取能量最低的构象作为目的蛋白的初始三维结构。利用软件包的Standard Dynamics Cascade程序对SCB的初始三维结构模型进行分子力学和分子动力学优化,以获得蛋白更接近其在溶液环境的构象。首先采用最陡下降法(Steepset descent method,SD)对初始结构进行500步的优化,然后采用500步的共轭梯度法(Conjugate gradient method,CG)进行能量优化。完成上述优化后,在300 K的温度下对模型进行分子动力学模拟[15-16]。(4)蛋白结构合理性评价:为了验证所模拟和预测的模建蛋白三维结构模型的合理性。采用软件包中的Profile-3D程序和拉曼图Ramachandran对分子力学和分子动力学优化后的蛋白结构进行合理性评价[17]。
1.2.6 SCB的活性位点分析及其与抑制剂的对接 1.2.6.1 SCB的活性位点分析应用Discovery Studio软件包中的Binding Site模块搜索SCB可能的活性位点,位点分析过程采用CVFF力场。Binding Site模块提供两种方法寻找蛋白质中可能的活性位点:通过对蛋白质表面的几何形状分析寻找能与底物结合的口袋,预测可能的结合位点; 通过对蛋白质家族在进化过程中的保守氨基酸进行研究,确定这些氨基酸残基在三维结构中的位置,预测蛋白质的活性位点。
1.2.6.2 SCB与抑制剂的对接首先应用Discovery Studio软件包中Builder程序构建抑制剂小分子并同时进行能量最小化优化。采用Cdock对接程序将蛋白与化合物对接,对接中参数设置为对接后配体自动产生10个不同的对接构象,采用score ligand pose程序的不同打分函数对这些构象进行评价[18],并采用consensus score程序对其进行打分排列[19]; 蛋白与化合物小分子的最佳对接复合物将由对接能量、氢键结合数和一致性打分结果决定。同时,采用软件包中Calculate Interaction Energy程序进行抑制剂小分子与活性位点每个氨基酸的结合自由能计算,以确定蛋白与抑制剂小分子对过程中的关键氨基酸。
2 结果 2.1 获取Sf9细胞总RNA本研究抽提Sf9细胞总RNA,采用1.5%琼脂糖凝胶电泳检测RNA,紫外灯下可见到清晰18S rRNA带型(图 1-A),结果表明RNA抽提质量较好,未发生明显降解,再进行RT-PCR反应获得Sf9细胞总cDNA,以其为模板,利用SexiCB P1和SexiCB P2引物进行扩增(图 1-B),分别获得1 000 bp左右的片段序列,进行序列测定,序列比对分析发现所获的扩增片段与甜菜夜蛾的Cathepsin B基因序列同源性高达90%,表明所获PCR产物为Sf9细胞即草地贪夜蛾的SCB基因序列。
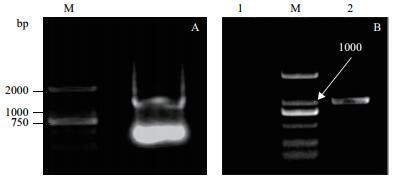
|
| 图 1 Sf9细胞总RNA的琼脂糖电泳图(A)和SCB PCR产物琼脂糖电泳分析图(B) M:DNA Marker DL2000;1:ddH2O为模板; 2:Sf9细胞cDNA为模板 |
挑取SCB-pEASY-T1重组质粒转化后的大肠杆菌trans1-T1阳性克隆,进行菌落PCR和质粒PCR鉴定均扩增出1 000 bp左右条带(图 2),表明挑取的克隆为含有重组质粒的真阳性克隆。把阳性克隆菌液送测序,将测序结果与上述目的片段进行比对,结果一致,即得到SCB基因ORF全长序列。SCB基因ORF为1 026 bp,编码341个氨基酸。SCB ORF的cDNA全长序列已经登录GenBank,登录号为HQ110064。
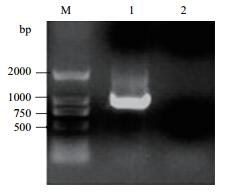
|
| 图 2 SCB-pEASY-T1重组质粒PCR鉴定 M:DNA Marker DL2000;1:重组质粒作为模板; 2:ddH2O作为模板 |
以基因组DNA为模板,分别利用SCB P1和SCB P2引物进行PCR扩增,由图 3可知,扩增产物均为1 000 bp左右,与基因克隆过程的PCR产物电泳检测结果一致。将PCR产物送测序,比对分析发现,基因组DNA为模板的扩增产物与对应的SCB ORF序列一致,表明SCB编码区没有内含子。

|
| 图 3 SCB PCR产物琼脂糖电泳分析 M:DNA Marker DL2000;1:基因组DNA为模板; 2:ddH2O为模板 |
用程序SignalP 3.0分析蛋白质SCB信号肽剪切位点(图 4),SCB信号肽的最有可能的剪切位点在20到21位之间,预测其蛋白信号肽序列为:MVASRATFVALVCALALASA。蛋白质切割信号肽后经过简单的加工过程而成为成熟肽。SCB成熟肽含有321个氨基酸。

|
| 图 4 SCB推导的氨基酸序列信号肽预测 |
SCB氨基酸成分分析结果(表 1)显示,SCB分别由321个氨基酸组成,蛋白质中酸性氨基酸为Asp,在SCB中占蛋白分子量的12.68%,利用频率为11.53%;碱性氨基酸为Lys,占蛋白分子量的12.23%,利用频率为9.97%;极性氨基酸为Asn、Cys、Gln、Ser、Thr和Tyr,占蛋白分子量的28.76%,利用率为28.66%;疏水氨基酸为Ala、Ile、Leu、Phe、Trp和Val,占蛋白分子量的29.35%,利用频率为28.66%。各种氨基酸在同一蛋白质组成中利用次数不一样,SCB利用最多的是Gly,利用32次,占蛋白分子量的5.13%,利用频率为9.97%;其次为Leu,利用25次,占蛋白分子量的7.95%,利用频率为7.79%,Met利用最少为3次,占蛋白分子量的1.11%,利用频率为0.93%。
用ExPASy Proteomics Server软件的Kyte-Dooli-ttle法(Window size设为9)对SCB编码的氨基酸序列进行亲水性分析,结果(图 5)表明SCB整个蛋白分子呈亲水性,但在0线以上的A、B、C、D、E五个区域表现出较强的亲脂性,其中A区域为信号肽。
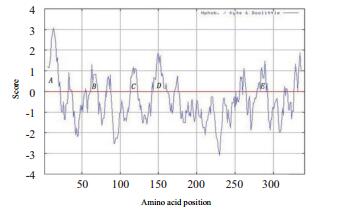
|
| 图 5 SCB氨基酸亲脂性分析 正值代表亲脂性,负值代表亲水性 |
将SCB氨基酸序列在NCBI信息库中进行同源性搜索,在鳞翅目、鞘翅目、双翅目、半翅目4个目7个科选出12种昆虫以及家鼠(Rattus norvegicus)和猕猴(Rhesus monkey)两种哺乳动物的组蛋白酶Cathepsin B基因氨基酸序列。应用DNAStar软件对SCB氨基酸序列用Clustal W方法进行一致性比对,结果见图 7,包括草地贪夜蛾在内的15个物种的Cathepsin B氨基酸序列有12个保守的半胱氨酸残基(Cys,C)位点,而保守性最强的氨基酸残基为甘氨酸(Gly,G),有23个保守位点,酸性氨基酸(Asp,D)有5个保守位点,另外,Gly是SCB氨基酸序列中含量最丰富的氨基酸。计算各序列的遗传距离(图 6),可知SCB与甜菜夜蛾的Cathepsin B基因相似度最高,相似度达96.2%,其次与烟夜蛾和棉铃虫的相似度也较高,分别为88.8%和88.5%,与半翅目的锥猎蝽(Triatoma infestans)相似度最低为51.2%,与两个哺乳动物家鼠和猕猴的Cathepsin B基因相似度分别为57.7%和55.2%,高于半翅目昆虫锥猎蝽。
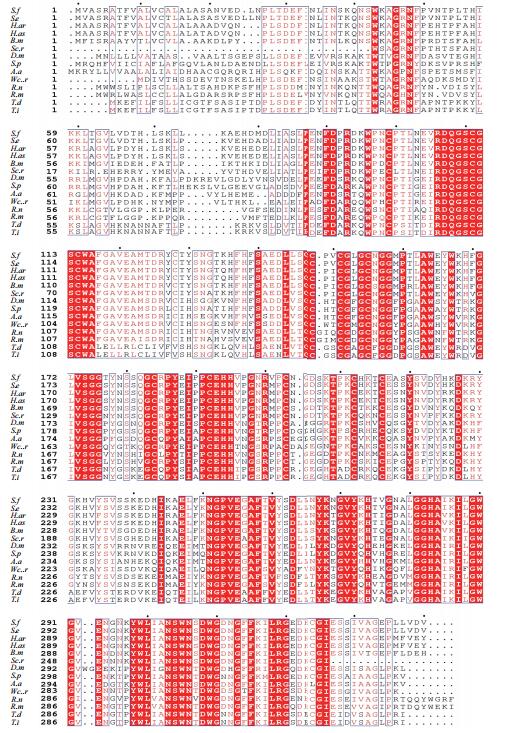
|
| 图 7 昆虫SCB氨基酸序列比对 棉铃虫Helicoverpa armigera(H.ar),烟夜蛾Helicoverpa assulta(H.as),草地贪夜蛾Spodoptera frugiperda(S.f),甜菜夜蛾Spodoptera exigua(S.e),家蚕Bombyx mori(B.m),蓖麻蚕Samia cynthia ricini(Sc.r),琥珀蚕Antheraea assama(A.a),家鼠Rattus norvegicus(R.n),猕猴Rhesus monkey(R.m),二分锥蝽Triatoma dimidiata(T.d),锥猎蝽Triatoma infestans(T.i),果蝇Drosophila melanogaster(D.m),麻蝇 Sarcophaga peregrine(S.p),埃及伊蚊Stegomyia aegypti(S.a),西部玉米根虫Western corn rootworm(Wc.r); (红色显示主要保守氨基酸残基) |

|
| 图 6 昆虫SCB一致性和差异度矩阵 |
根据距离建立系统进化树[20]将15个Cathepsin B基因的氨基酸序列分为四大类(图 8):第1类主要为鳞翅目昆虫的Cathepsin B,本研究中的SCB也聚到这类,与同属的甜菜夜蛾Cathepsin B一致性最高。鳞翅目各昆虫间Cathepsin B相对距离根据科属的不同差异较大,同属距离最近,不同属或不同科距离较远。第2类为半翅目,哺乳动物的家鼠和猕猴聚到这类; 第3和4类分别为双翅目和鞘翅目。
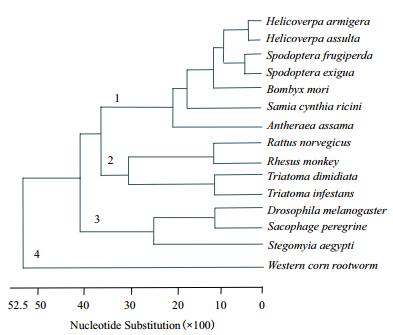
|
| 图 8 昆虫SCB的系统进化树 棉铃虫Helicoverpa armigera,烟夜蛾Helicoverpa assulta,草地贪夜蛾Spodoptera frugiperda,甜菜夜蛾Spodoptera exigua,家蚕Bombyx mori,蓖麻蚕Samia cynthia ricini,琥珀蚕Antheraea assama,家鼠Rattus norvegicus,猕猴Rhesus monkey,二分锥蝽Triatoma dimidiata,锥猎蝽Triatoma infestans,黑腹果蝇Drosophila melanogaster,麻蝇 Sarcophaga peregrine,埃及伊蚊Stegomyia aegypti,西部玉米根虫Western corn rootworm |
蛋白质二级结构是氨基酸序列和三维构象之间的桥梁,对蛋白质二级结构的预测能得到许多重要的结构信息[21]。SCB成熟肽氨基酸序列用DNAStar软件的Garnier-Robson法进行结构预测。由图 9可知,SCB的二级结构主要由19个α螺旋,10个β折叠,32个β转角以及26个不规则转曲结构组成。
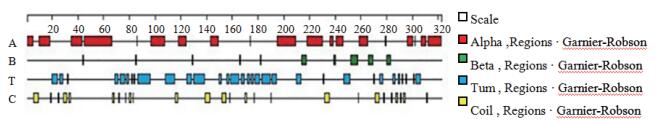
|
| 图 9 Garnier-Robson方法预测SCB二级结构 |
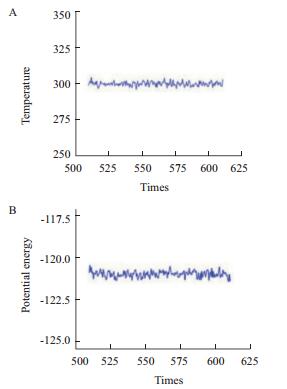
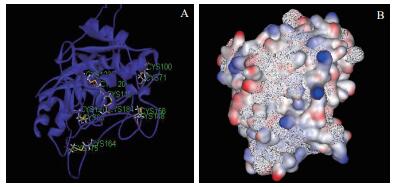
(1) 模板蛋白的确定:将SCB对序列与PDB数据库中的蛋白质序列比对,结果发现编号为1MIR的蛋白与SCB的氨基酸序列同源性达到61%。(2)序列联配:SCB蛋白与模板蛋白的同源性满足同源建模最低同源性30%的要求。因此1MIR适合作为SCB三维结构同源建模的模板。SCB与1MIR序列联配比对结果表明,两者同源性非常高(图 10),以1MIR为模板进行SCB三维结构同源建模可靠性高。(3)三维结构建模和动力学优化:对SCB三维结构进行了同源建模,选取模型的总能量较低的模型作为初始三维结构模型,并对SCB初始三维结构模型进行分子力学和分子动力学优化,优化过程中使用CVFF力场,并在300 K的温度下对SCB模型进行625 ps的分子动力学模拟,模拟步长为1 ps,蛋白的温度和能量变化曲线,由图 11可知,SCB 500 ps后温度和能量均一直处于稳定状态。选择动力学优化后能量最低的构象用于后续的对接实验。经动力学优化后的SCB的三维结构分别如图 12-A。由图可知,SCB三维结构共有6个二硫键,Cys71-Cys100,Cys164-Cys175;Cys156-Cys188等6个。同时,研究中对SCB三维结构中的亲疏水性氨基酸进行分析。由图 12-B可知,SCB折叠成非常紧密而稳定的球状结构,亲水性氨基酸主要包被在蛋白的表面(如图固体状表面),而疏水性氨基酸主要分布在蛋白三维结构的内部(白色网状表面),形成一个疏水性空穴。

|
| 图 10 SCB和1MIR氨基酸序列比对结果 红色区域表示氨基酸一致; 蓝色区域表示氨基酸非常同源; 黄色区域表示氨基酸比较同源; 白色区域表示氨基酸同源性较差 |
|
| 图 11 SCB蛋白温度(A)和内能(B)在500 ps动力学模拟中随时间变化情况 |
|
| 图 12 SCB三维结构模型(A)和亲疏水性三维结构模型(B) |
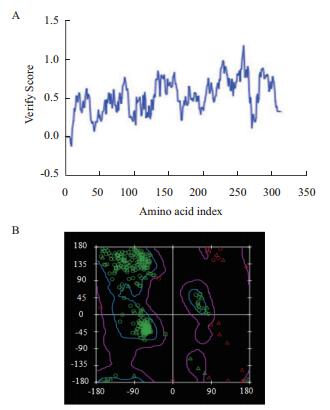
利用Discovery Studio软件的Verify Protein(Profile-3D)程序对已构建的SCB的三维结构模型进行可靠性分析,图 13-A显示除了N端和C端(肽链两端的得分与许小于0),两个模型绝大部分氨基酸残基的Profile-3D得分都大于0,说明几乎所有残基都位于合理位置。为了进一步直观地对SCB的三维结构模型进行其合理性分析,运用拉曼图检测所模拟蛋白结构的合理性。由图 13-B可知,绝大部分氨基酸残基落在非常合理的蓝色区域内。由上述Profile-3D分析和拉曼图分析表明构建的SCB的三维结构的二面角分布和立体构象均较为合理,符合立体化学φ、ψ二面角分布的要求。
|
| 图 13 SCB三维模型的Profile-3D分析(A)和拉曼图分析(B) 氨基酸残基二面角位于蓝色区域内表示最允许范围,紫色区域以外代表不允许范围。二面角位于蓝色区域外紫色区域内表示允许范围 |
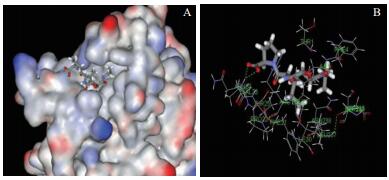
使用Discovery Studio软件包中的Binding Site程序对SCB三维结构进行可能活性位点搜索,得到15个结合位点。选取软件中排列在第1位的作为最佳活性位点用于下一步的模拟对接。采用软件包中的Cdock程序进行SCB与抑制剂(CA)的模拟对接,对接后每个抑制剂将产生10个与蛋白结合的不同构象,根据程序对不同构象的打分评价,选出最优构象。SCB与CA对接后形成的最优复合物结构模型。由图 14-A可知,SCB结合口袋比较浅,形状不规则、袋口较宽。不同化合物结构不相同,进入蛋白活性中心后,与活性中心位点相互作用的氨基酸,以及作用强弱、作用方式等也不同,本研究对CA与SCB活性位点的相互作用进行模拟。由图 14-B可知,CA与SCB活性位点的ILE5、VAL23等多个氨基酸相互作用,其中分别与VAL23和ASN24形成两个氢键。由此可见CA和与SCB具有较强的相互作用。
|
| 图 14 CA与SCB对接复合物(A)和CA与SCB活性位点的结合模式(B) 绿色虚线表示氢键 |
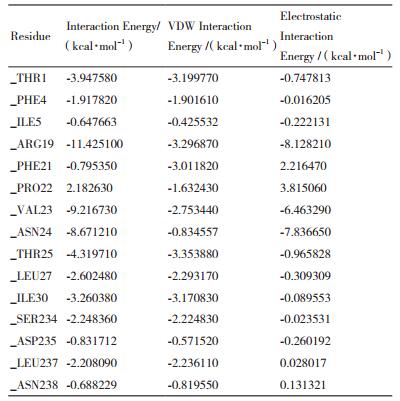
蛋白与不同化合物形成的复合物中起主要作用的氨基酸残基可能不同。本研究计算了抑制剂分子CA与SCB对应蛋白酶活性位点中单个氨基酸残基的相互作用能。根据这个相互作用能,进一步确定SCB与CA分子相互作用的关键氨基酸残基。由表 2可知CA与SCB活性位点单个氨基酸残基相互作用的能量包括:总相互作用能、范德华力相互作用能和静电相互作用能(表中只显示总能量小于-0.5 kcal/mol的氨基酸残基)。CA与SCB活性位点的ARG19、VAL23、ASN24有较强的相互作用,是关键的氨基酸,总作用能分别为-11.43、-9.22、-8.67 kcal/mol,3个残基对复合物形成的贡献最大,且均主要表现为静电相互作用能,其中VAL23、ASN24与CA形成氢键。
研究蛋白质的结构是了解蛋白质的生物学功能的重要途径。氨基酸序列对蛋白质构象起着决定性的作用,很多同源性的氨基酸序列就可以决定同一蛋白构象,这是蛋白质三维结构预测的理论基础[22]。目前研究蛋白质空间结构方法包括X射线晶体衍射技术和核磁共振技术[23]均属于生物学实验方法,它们能够准确测定蛋白三维结构,但同时也存在许多技术难点,使得测定进度性对于大量未知蛋白来说显得十分缓慢。为了加快新蛋白质的结构测定及其与化合物的相互作用方式,当前通过PDB蛋白结构信息库中搜索已知结构的同源蛋白质作为模板,利用同源建模软件对新蛋白进行结构建模是最为常用且有效的一种蛋白质结构预测方法[22]。随着生物信息学的快速发展及其在生物学研究中的突出作用,蛋白质同源模建技术已经成为一种可靠性高且被广泛认可的蛋白结构预测技术。任何一对氨基酸序列长度大于80的蛋白质,如果两者的序列同源性超30 %,则可认为它们具有相似的三维结构,即蛋白质的基本折叠相同[24]。本研究中克隆的组织蛋白酶基因SCB的ORF,编码区能够分别编码321个氨基酸,在PDB数据库中搜索发现蛋白1MIR与SCB的氨基酸序列同源性高达60%以上。因此符合蛋白同源建模的条件,可以以1MIR为模板分别对SCB的三维结构模型进行预测。
有关组织蛋白酶的结构分析及其与化合物相互作用的机理,在各种物种中做了较多的研究。晶体学分析结果表明锥虫Cathespin B在Pro93和Leu94断裂形成成熟蛋白,而且在N端活性位点之前还有16个额外的氨基酸残基。锥虫Cathespin B与抑制剂CA-074[25]相互作用形成的复合物结构,以人类Cathepsin B(1GMY),老鼠Cathepsin B(1CTE)and斑马Cathepsin B(1F2A),作为模板蛋白用MODELLER[26]同源建模技术以及MODELLER分析替换技术进行了分析研究。CA-074通过与Cathespin B结合位点的氨基酸残基Gly165、Gys122等形成多个氢键而牢固结合与Cathespin B,同时大量的疏水氨基酸残基在S2位点上提供结合能。有研究还发现锥虫Cathespin B活性位点有一个横跨的‘occluding loop’结构,在Cathepsin L同源物rhodesain上没有发现这个结构,这与‘occluding loop’是Cathespin B区别Cathepsin L类组织蛋白酶特征的观点一致[27-28],然而该研究发现锥虫Cathespin B的‘ occluding loop ’结构比哺乳动物的Cathespin B多3个氨基酸残基,而且在His 194 and His195之间形成一个双重构造。这些区别的发现对设计锥虫组织蛋白酶专一性抑制剂类药物,防治锥虫病很有意义的。本研究用Discovery Studio同源建模软件对SCB蛋白与其对应抑制剂CA的相互作用模式进行模拟,结果表明SCB结合口袋比较浅,形状不规则、袋口较宽。结合能预测发现CA与SCB活性位点的ARG 19、VAL 23、ASN 24有较高的相互作用能,是关键的氨基酸。这些研究对进一步运用生物实验手段研究其结构和功能,为设计和开发昆虫组织蛋白酶类抑制剂杀虫剂有重要意义。
4 结论本研究克隆获得草地贪夜蛾Cathepsin B基因ORF序列,该基因的ORF为1 026 bp的片段,编码341个氨基酸。同源建模预测该组织蛋白酶的三维结构,确定其折叠成紧密而稳定的球状结构,含6个二硫键。分子对接模拟表明该酶活性中心的ARG19、VAL23和ASN24为关键氨基酸残基,以氢键与抑制剂分子为结合。
| [1] |
Li Y, Wang K, Xie H, et al. Cathepsin B cysteine proteinase is esse-ntial for the development and pathogenesis of the plant parasitic nem-atode Radopholus similis[J]. International Journal of Biological Sciences, 2015, 11(9): 1073-1087. DOI:10.7150/ijbs.12065 |
| [2] |
Cristofoletti PT, Ribeiro AF, Deraison C, et al. Midgut adaptation and digestive enzyme distribution in a phloem feeding insect, the pea aphid Acyrthosiphom pisum[J]. Journal of Insect Physiology, 2003, 49(1): 11-24. DOI:10.1016/S0022-1910(02)00222-6 |
| [3] |
Uchida K, Ohmori D, Ueno T, et al. Preoviposition activation of cathepsin-like proteinases in degeneratingovarian follicles of the mosquito Cuxel pipiens pallens[J]. Developmental Biology, 2001, 237(1): 68-78. DOI:10.1006/dbio.2001.0357 |
| [4] |
徐夏莲, 赵小凡, 王金星. 棉铃虫组织蛋白酶B组织分布与合成部位研究[J]. 动物学研究, 2001, 22(3): 242-245. |
| [5] |
罗梅, 董章勇, 宾淑英, 等. 扶桑绵粉蚧组织蛋白酶B基因的克隆、原核表达和不同发育阶段表达分析[J]. 昆虫学报, 2012, 55(3): 276-283. |
| [6] |
Uchida K, Nishizuka M, Ohmori D, et al. Follicular epithelial cell apoptosis of atretic of follicles within developing varies of the mosquito Cuxel pipiens pallen[J]. Journal of Insect Physiology, 2004, 50(10): 903-912. DOI:10.1016/j.jinsphys.2004.07.002 |
| [7] |
Cristofoletti PT, Ribeiro AF, Deraison C, et al. Midgut adaptation and digestive enzyme distribution in a phloem feeding insect, the pea aphid Acyrthosiphon pisum[J]. Insect Physiol, 2003, 49(1): 11-24. DOI:10.1016/S0022-1910(02)00222-6 |
| [8] |
Cho WL, Tsao SM, Hyas AR. Mosquito cathepsin B-like Protease involved in embryonic degradation of vitelin is produced as alatent extraovarian precursor[J]. Journal of Biochemistry, 1999, 274(19): 13311-13321. |
| [9] |
Zhao XF, An XM, Wang JX, et al. Expression of the Helicoverpa cathepsin B like proteinase during embryonic development[J]. Archives of Insect Biochemistry and Physiology, 2005, 58(1): 39-46. DOI:10.1002/(ISSN)1520-6327 |
| [10] |
张新昌, 刘杨, 王金星, 等. 从大豆中纯化棉铃虫组织蛋白酶B抑制因子HCB-SoyI[J]. 昆虫学报, 2005, 48(6): 849-853. |
| [11] |
Luo M, Dong ZY, Bin SY, et al. Molecular cloning, prokaryotic expression and expression at different developmental stages of cathepsin B gene in mealybug Phenacoccus solenopsis Tinsley(Hemiptera:Pseudococcidae)[J]. Acta Entomologica Sinica, 2012, 55(3): 276-283. |
| [12] |
赵艳艳, 刘建兵, 罗梅浩, 等. 烟夜蛾组织蛋白酶B酶原基因的克隆、序列分析和原核表达[J]. 昆虫学报, 2008, 51(11): 1121-1128. DOI:10.3321/j.issn:0454-6296.2008.11.003 |
| [13] |
林春娇, 杨立荣, 徐刚, 等. R-2-卤代酸脱卤酶的同源模建及底物对映体选择性研究[J]. 生物技术通报, 2011, 0(10): 191-198. |
| [14] |
张乃心, 张玉娟, 余果, 等. 双翅目昆虫线粒体基因组结构特点及其测序通用引物的设计和应用[J]. 昆虫学报, 2013, 56(4): 398-407. |
| [15] |
冯磊, 花慧, 邱丽颖, 等. 白藜芦醇靶点蛋白质的研究[J]. 生物技术通报, 2009(8): 128-133. |
| [16] |
朱威, 张莲芬, 雷楗勇, 等. 羟喜树碱结合肽的研究[J]. 生物技术通报, 2009(1): 126-129. |
| [17] |
吴玲, 何佩勋, 孙磊, 等. G蛋白偶联受体81同源建模及其与二羟基苯甲酸对接研究[J]. 化学研究与应用, 2017, 29(6): 841-846. |
| [18] |
Ruvinsky A, Watson C. Intron Phase Patterns in Genes:Preserv-ation and Evolutionary Changes[J]. Open Evolution Journal, 2007, 107(1): 1-14. |
| [19] |
Teramoto R, Fukunishi H. Supervised Consensus Scoring for Docking and Virtual Screening[J]. Journal of Chemical Information & Modeling, 2007, 47(2): 526. |
| [20] |
滕飞翔, 崔玉宝. 昆虫AQPs系统进化树构建及分析[J]. 西南农业学报, 2013, 26(6): 2642-2647. |
| [21] |
李任峰, 田香勤, 何启盖, 等. 大肠杆菌P4 FliC蛋白结构分析及同源建模[J]. 生物技术通报, 2010, 0(10): 143-148. |
| [22] |
邓海游, 贾亚, 张阳. 蛋白质结构预测[J]. 物理学报, 2016, 65(17): 169-179. |
| [23] |
樊盛博, 吴妍洁, 杨兵, 等. 蛋白质结构与相互作用研究新方法——交联质谱技术[J]. 生物化学与生物物理进展, 2014(11): 1109-1125. |
| [24] |
Gunasekaran K, Nussinov R. How different are sttiructurally flexible and rigid binding sites? Sequence and structural features discriminating proteins that do and do not undergo conformational change upon ligand binding[J]. Journal of Molecular Biology, 2007, 365(1): 257-273. DOI:10.1016/j.jmb.2006.09.062 |
| [25] |
Xu M, Yang L, Rong JG, et al. Inhibition of cysteine cathepsin B and L activation in astrocytes contributes to neuroprotection against cerebral ischemia via blocking the tBid-mitochondrial apoptotic signaling pathway[J]. Glia, 2014, 62(6): 855-880. DOI:10.1002/glia.v62.6 |
| [26] |
Webb B, Sali A. Comparative protein structure modeling using MODELLER[J]. Current Protocols in Protein Science, 2014, 47(5, 6): 1-32. |
| [27] |
Sajid M, McKerrow JH. Cysteine proteases of parsitic organisms[J]. Molecular and Biochemical Parasitology, 2002, 120(1): 1-21. DOI:10.1016/S0166-6851(01)00438-8 |
| [28] |
Abdulla MH, Lim KC, Sajid M, et al. Schistosomiasis mansoni:novel chemotherapy using a cysteine protease inhibitor[J]. PLoS Medicine, 2007, 4(1): e14. DOI:10.1371/journal.pmed.0040014 |