2. 福建农林大学动物 科学学院,福州 350002
2. College of Animal Science, Fujian Agriculture and Forestry University, Fuzhou 350002
与哺乳动物类似,鱼类也存在天然和获得性两种免疫应答方式。其中,天然免疫既是抵御病原微生物侵害的第一道防线,同时也具有诱导获得性免疫应答反应的作用[1]。Toll样受体(Toll-like receptor,TLR)是一类重要的模式识别受体(Pattern recognition receptor,PRR),在天然免疫和获得性免疫中起着桥梁作用,主要是通过识别具有保守微生物结构的病原相关分子模式(Pathogen-associated molecular pattern,PAMPs),包括脂多糖(Lipopolysaccharides,LPS)、肽聚糖(Peptidoglycan,PGN)、鞭毛蛋白、CpG-DNA、poly I:C以及酵母聚糖等引发先天免疫应答[2]。目前,在哺乳动物中有13种不同类型TLRs,而鱼类中发现17种,在结构与功能方面具有高度相似性[3]。TLRs家族中,Toll样受体3(TLR3)在识别双链RNA(dsRNA)或病毒类似物poly I:C中发挥重要作用[4-5]。从低等动物到高等动物,TLR3在结构上非常保守,具有多个亮氨酸重复序列(Leucine-rich repeat,LRR)的胞外结构域、N和C末端侧翼区的跨膜结构域以及TIR(Toll-like/IL-1 receptor,TIR)结构域[6]。TLR3的信号传导方式主要通过两种途径:一种是髓样分化因子88(Myeloid differentiation factor 88,MyD88)依赖途径,即TIR结构域与接头蛋白MyD88或其类似物相互作用后将信号传递到细胞内,引起免疫应答[7]; 另一种是MyD88非依赖途径,主要通过TIR结构域募集的另一接头蛋白TRIF(TIR-domain-containing adaptor inducing interferon-β,TRIF)传递信号,TRIF招募不同信号通路下游的蛋白分子并与其结合,激活不同的转录因子和转录激酶,如NF-κB、干扰素调节因子3(Interferon regulatory factor 3,IRF-3)和MAP激酶等,进而激活相应的信号通路[4]。
迄今为止,已有多种鱼类的TLR3基因序列被克隆和报道,包括斑马鱼(Danio rerio)[8]、斑点叉尾鮰(Ictalurus punctatus)[9]、红鳍东方鲀(Takifugu rubripes)[10]、鲤鱼(Cyprinus carpio)[11]、稀有鮈鲫(Gobiocypris rarus)[12]、草鱼(Ctenopharyngodon idella)[13]以及虹鳟(Oncorhynchus mykiss)[14]等。越来越多的研究表明TLR3在鱼类抗病毒免疫应答中发挥重要作用[15]。经poly I﹕C免疫注射后,斑点叉尾鮰[9]、鲤鱼[11]以及大黄鱼(Larimichthys crocea)[16]的TLR3基因表达量显著上调。Su等[12]用草鱼呼肠孤病毒(Grass carp reovirus,GCRV)感染稀有鮈鲫、团头鲂(Megalobrama amblycephala)[17]后,TLR3基因表达水平均有明显提高,提示TLR3与抗病毒免疫应答紧密相关。近年来的研究也发现TLR3在抗细菌免疫应答中扮演重要角色。大黄鱼[16]和斑马鱼[8]分别经副溶血弧菌(Vibrio Parahaemolyticus)和迟缓爱德华菌(Edwardsiella tarda)感染后,TLR3基因表达水平均有显著提高。最近的研究发现,LPS和PGN能显著增强草鱼头肾细胞TLR3基因的表达[18],提示该分子参与了抗细菌感染的免疫反应。
日本鳗鲡(Anguilla japonica)是中国、日本和韩国等东亚国家的重要养殖种类,味道鲜美、营养丰富,具有极高的经济价值[19]。然而,由于养殖过程中的病害频繁发生,给日本鳗鲡养殖业造成巨大的经济损失[20]。因此,对于日本鳗鲡抵抗外源微生物免疫机制的研究显得尤为重要。本实验克隆了日本鳗鲡AjTLR3全长cDNA序列,利用荧光定量PCR技术研究了经poly I:C和LPS免疫刺激后的日本鳗鲡肝、脾、肠、鳃、肾、皮肤、心脏及肌肉AjTLR3基因表达水平变化。此外,选择病毒和细菌不同的病原体相关分子模式(LPS,poly I:C,CpG-DNA和PGN)以及不同浓度的嗜水气单胞菌(Aeromonas hydrophila),对体外培养的日本鳗鲡肝脏细胞进行免疫刺激,检测不同时相AjTLR3基因表达谱的变化,以期为深入了解日本鳗鲡AjTLR3在抗病毒和抗细菌免疫应答中的作用机制提供参考。
1 材料与方法 1.1 材料日本鳗鲡体重约45-50 g,购自中国福清鳗鱼养殖场,于25℃,1 000 L循环水中暂养一周以适应实验室环境。解剖取各组织器官,包括肝脏、脾脏、鳃、肾、肠、心脏、皮肤和肌肉,放入液氮冷冻后置于-80℃保存,用于RNA提取。体外实验采用的日本鳗鲡肝脏细胞由本实验室制备,培养方法参照文献[21]。
1.2 方法 1.2.1 实验动物免疫刺激将LPS和poly I:C(均购自SIGMA公司)用PBS溶解稀释,至终浓度分别为4 mg/mL和2 mg/mL。日本鳗鲡被分为3组进行腹腔注射,实验组分别注射250 µL LPS和250 µL poly I:C,对照组注射250 µL灭菌PBS溶液。免疫刺激24 h后,每组随机选择6尾日本鳗鲡,取血、肝脏、脾脏、鳃、肾、肠、心脏、皮肤和肌肉,液氮迅速冷冻,置于-80℃冰箱保存。
1.2.2 细胞免疫刺激当细胞贴壁覆盖率达到90%以上用含0.25%胰酶的消化液进行消化,随后转入3 cm的细胞培养板,细胞长满后更换细胞培养液,分别加入poly I:C(50 μg/mL)、LPS(30 μg/mL)、CpG(10 μg/mL)、PGN(30 μg/mL)[21],以及终浓度分别为106CFU/mL、107 CFU/mL和108 CFU/mL的嗜水气单胞菌,对照组加入等体积细胞培养液。在免疫刺激后0 h、3 h、6 h、12 h、24 h和48 h取样,每个时相取4个细胞样品重复。
1.2.3 总RNA的提取以及cDNA的合成日本鳗鲡不同组织样品以及肝脏细胞样品总RNA的提取均采用E.Z.N.A.TM总RNA试剂盒Ⅱ(Omega),具体方法参照试剂盒说明书。取1μg的RNA通过PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒(TaKaRa,China)制备得到cDNA模板,稀释100倍后用于日本鳗鲡组织和细胞荧光定量PCR。另取部分肝脏总RNA,通过SMART RACE cDNA扩增试剂盒(TaKaRa,China)合成RACE反应的第一链cDNA,用于AjTLR3基因全长的克隆。
1.2.4 日本鳗鲡AjTLR3基因全长的克隆根据本实验室日本鳗鲡转录组数据分析获得AjTLR3基因片段序列,利用Premier 5.0软件设计工具(http://www.premierbiosoft.com/primerdesign/index.html)(表 1)设计引物,随后进行PCR,扩增日本鳗鲡AjTLR3部分基因片段。将纯化产物插入pMD19-T载体(TaKaRa,China),并转化至感受态中,随后将阳性克隆的质粒克隆并由上海生工进行测序。基于测序正确的AjTLR3部分基因序列,使用表 1中列出的特异性引物扩增日本鳗鲡AjTLR3基因全长的cDNA序列,并通过头至趾引物验证其开放阅读框(Open reading frame,ORF)的正确性。
选择β-actin作为内参基因(引物5'-β-actin和3'-β-actin,表 1),选择引物5' real-TLR3和3' real-TLR3(表 1)特异性扩增AjTLR3基因片段,进行预实验,以确保没有引物二聚体的单个离散带的扩增。将PCR产物,纯化后测序分析。实时荧光定量PCR总反应体系为20 μL,含有cDNA模板9 μL,2×SYBR Green Master Mix(Vazyme TM,Q111-02)10 μL以及引物各0.5 μL,在Roche Light Cycler 480机器(Roche,Sussex,UK)上进行扩增,反应条件如下:95℃,1 min,40个循环(94℃,20 s,60℃,1 min)。荧光定量PCR数据结果根据Roche480自动给出的每个样品的Cp值,并使用比较CT方法(即2-ΔΔ CT方法)计算出样品的RQ值。
1.2.6 统计学分析使用SPSS 15.0版(SPSS Inc,Chicago,IL,USA)分析实验数据。天然日本鳗鲡不同组织基因表达水平差异采用方差分析以及Duncan多重比较法; 不同实验处理组之间的基因水平差异采用成组T-检验分析。
1.2.7 生物信息学分析序列相似性分析采用BLAST程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi),用ORF Finder(http://www.ncbi.nlm.nih.gov/projects/gorf/)分析AjTLR3的全长cDNA序列; 在(http://cn.expasy.org/tools/pi_tool.html)网站上进行等电点、分子量预测; 蛋白质域特征由Simple Modula Architecture Reach Tool(SMART)(http://smart.embl-heidelberg.de/); 使用BioEdit程序进行蛋白质序列的多重比对; 通过MEGA 5.0软件构建N-J系统发育树。
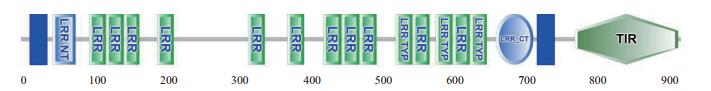
2 结果 2.1 AjTLR3基因全长克隆以及序列分析日本鳗鲡AjTLR3(GenBank登录号:KJ726740)的cDNA全长为3 383 bp,包含93 bp的5'非翻译区(Untranslated regions,UTR),524 bp的3'-UTR以及2 766 bp的ORF,编码921个氨基酸(图 1)。预测蛋白质分子量为103.0 kD,理论等电点为pI 8.84。通过SMART程序预测的AjTLR3的结构域显示:AjTLR3分子由16个富含亮氨酸重复序列(Leucine-rich repeat,LRR)的胞外结构域、TIR结构域(773-917 aa)以及在N末端(13-25 aa)与C末端(721-743 aa)的两个跨膜结构域组成(图 1-2)。其中胞外结构域含有11个保守LRR结构域、3个特殊LRR结构域(LRR-TYP)、1个N末端区域的LRR-NT结构域(44-76 aa)以及1个C末端的LRR-CT结构域(663-715 aa)。

|
| 图 1 AjTLR3 cDNA的核苷酸序列和推导的氨基酸序列 起始密码子(ATG)和终止密码子(TGA)被圆圈标记; LRR结构域显示为下划线(95-108 aa,119-143 aa,144-165 aa,190-213 aa,317-340 aa,372-395 aa,422-447 aa,448-472 aa,473-496 aa,547-570 aa,603-626 aa)。粗体下划线是LRR-TYP结构域(523-546 aa,579-602 aa,627-650 aa); 方框圈住LRR-NT结构域(44-76 aa)和LRR-CT结构域(663-715 aa); 跨膜区域用粗体突出(13-25 aa)(721-743 aa); 阴影部分为TIR结构域(773-917 aa) |
|
| 图 2 AjTLR3结构域示意图 |
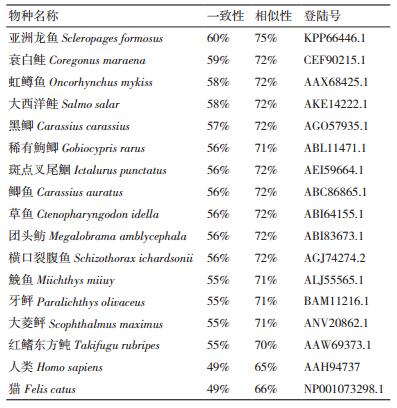
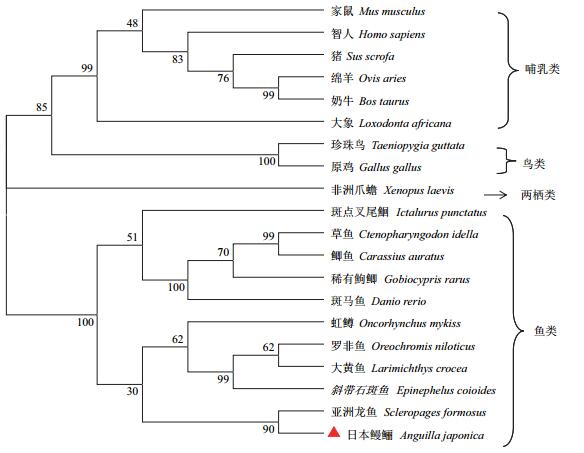
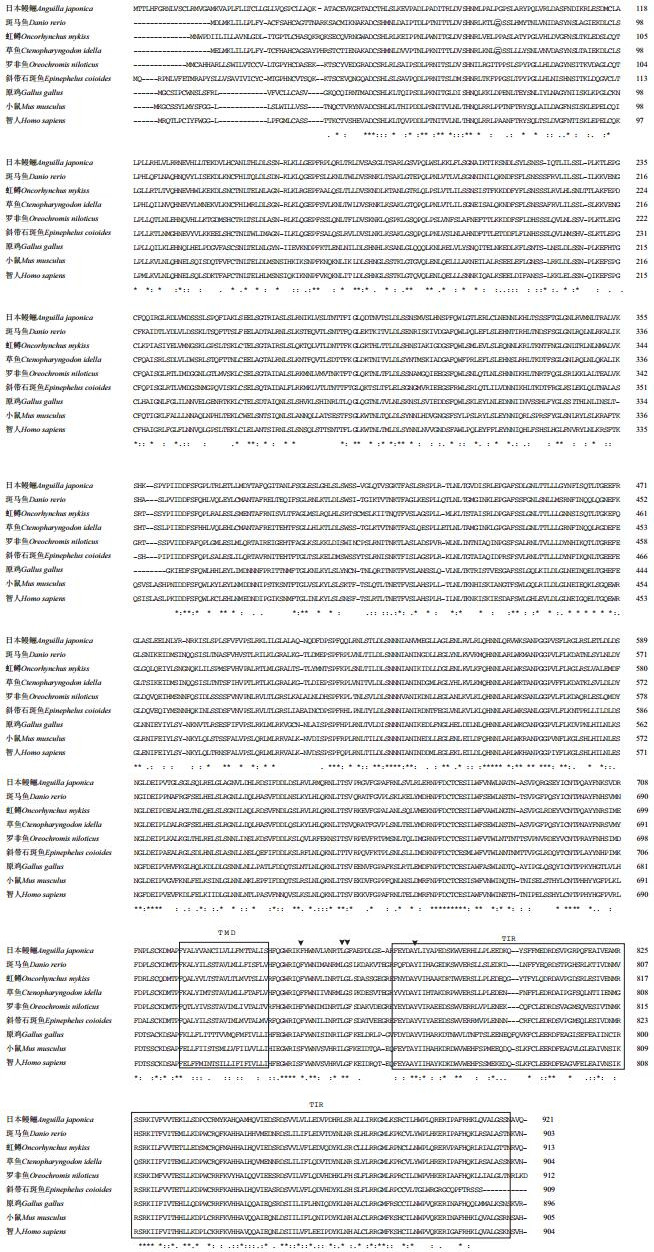
系统发育树(图 3)显示,日本鳗鲡与其他鱼类TLR3聚为一支,哺乳类、鸟类、两栖类各聚为一支。通过比较不同物种的氨基酸序列显示(表 2),AjTLR3氨基酸序列分别与亚洲龙鱼(Scleropages formosus)、衰白鲑(Coregonus maraena)、虹鳟鱼、大西洋鲑(Salmo salar)、黑鲫(Carassius carassius)、稀有鮈鲫、斑点叉尾鮰、鲫鱼、草鱼、团头鲂、横口裂腹鱼(Schizothorax richardsonii)、鮸鱼(Miicht-hys miiuy)、牙鲆(Paralichthys olivaceus)、大菱鲆(Scophthalmus maximus)、红鳍东方鲀、人类(Homo sapiens)和猫(Felis catus)有60%、59%、58%、58%、57%、56%、56%、56%、56%、56%、56%、55%、55%、55%、55%、49%和49%的一致性。AjTLR3氨基酸序列与鱼类一致性较高(55%-60%),而与哺乳动物基因的一致性较低(49%)。将日本鳗鲡AjTLR3基因氨基酸序列与其他物种进行多重序列比对分析见图 4,结果表明日本鳗鲡AjTLR3与鸟类和哺乳类一样,均具有跨膜结构域和TIR结构域。此外,在哺乳动物TLR3基因上有4个高度保守的氨基酸残基,也在日本鳗鲡的AjTLR3的序列中发现,分别为Phe750、Leu761、Gly762和Tyr778。
|
| 图 3 日本鳗鲡AjTLR3和其他物种TLR3氨基酸序列的NJ系统发育树 物种名称和GenBank登录号:家鼠Mus musculus:AAH99937;智人Homo sapiens:AAH94737;猪Sus scrofa:BAG12312;绵羊Ovis aries:NP_001129400;奶牛Bos taurus:ABN71659;大象Loxodonta africana:ABC95781;珍珠鸟Taeniopygia guttata:XP_002190888;原鸡Gallus gallus:ABL74502;非洲爪蟾Xenopus laevis:BAF57488;斑点叉尾鮰Ictalurus punctatus:ABD93872;草鱼Ctenopharyngodon idella:ABI64155;鲫鱼Carassius auratus:ABC86865.1;稀有鮈鲫Gobiocypris rarus:ABL11471.1;斑马鱼Danio rerio:NP_001013287;虹鳟Oncorhynchus mykiss:AAX68425;罗非鱼Oreochromis niloticus:XP_019215641.1;大黄鱼Larimichthys crocea:GCA_000972845;斜带石斑鱼Epinephelus coioides:ADZ76423.1;亚洲龙鱼Scleropages formosus:KPP66446.1(AjTLR3用三角标出) |
|
| 图 4 日本鳗鲡和其他物种TLR3氨基酸序列多重比对 物种名称和GenBank登录号:斑马鱼Danio rerio:NP_001013287;虹鳟Oncorhynchus mykiss:AAX68425;草鱼Ctenopharyngodon idella:ABI64155;罗非鱼Oreochromis niloticus:XP_019215641.1;斜带石斑鱼Epinephelus coioides:ADZ76423.1;原鸡Gallus gallus:ABL74502;小鼠Mus musculus:AAH99937;智人Homo sapiens:AAH94737。跨膜区和TIR结构域分别方框圈出。高度保守的氨基酸(Phe750,Leu761,Gly762,Tyr778)用三角表示 |
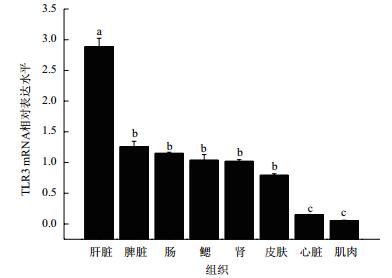
日本鳗鲡AjTLR3基因为组成型表达模式。其中肝脏表达量显著高于其他组织(P < 0.05);其次是脾脏、肠、鳃、肾及皮肤,在心脏和肌肉中的表达水平较低(图 5)。
|
| 图 5 AjTLR3基因在健康日本鳗鲡各组织中的相对表达水平 标注不同字母表示组间差异显著(P < 0.05) |
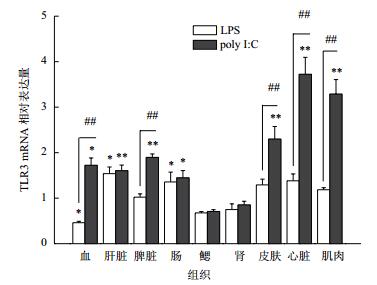
为了探讨AjTLR3对抗病毒和抗细菌免疫应答的作用,利用荧光定量PCR技术检测了在poly I:C和LPS感染后AjTLR3基因表达水平见图 6。
|
| 图 6 LPS和poly I:C刺激下24 h后不同组织中TLR3基因的相对表达水平 数据表示为4个独立个体的平均值±SE。*表示该时相刺激组和空白对照组即PBS刺激组存在显著性差异(P < 0.05);**表示该时相刺激组和空白对照组存在极显著性差异(P < 0.01);##表示LPS和poly I:C两种刺激组之间存在极显著性差异(P < 0.01) |
日本鳗鲡经poly I:C免疫刺激后,AjTLR3基因的表达量在肝脏组织(1.6倍,P < 0.01)、脾脏(1.9倍,P < 0.01)、肠(1.5倍,P < 0.05)、血(1.7倍,P < 0.05)、皮肤(2.4倍,P < 0.01)、心脏(3.7倍,P < 0.01)和肌肉(3.3倍,P < 0.01)中显著上调,而在鳃和肾脏中无明显变化。同时,在LPS的刺激下,仅肝脏(1.7倍,P < 0.05)和肠(1.3倍,P < 0.05)的AjTLR3基因水平显著提高,而血液的中的表达量(0.4倍,P < 0.05)显著降低,其他组织无明显变化。此外,Poly I:C免疫注射组中的脾脏、皮肤、心脏和肌肉组织的AjTLR3基因表达水平显著高于LPS注射组(P < 0.05)。
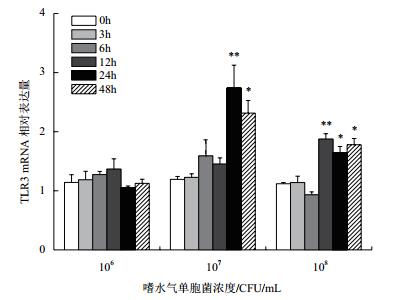
2.5 嗜水气单胞菌感染日本鳗鲡肝脏细胞后的AjTLR3基因表达变化三种不同浓度的嗜水气单胞菌感染日本鳗鲡肝脏细胞,其AjTLR3基因表达水平见图 7。如图 7所示:浓度为106 CFU/mL的嗜水气单胞菌未能引起日本鳗鲡肝脏细胞AjTLR3基因表达水平的变化; 浓度为107 CFU/mL的嗜水气单胞菌处理日本鳗鲡肝脏细胞后,AjTLR3的基因表达水平在24 h(2.7倍,P < 0.01)和48 h(2.3倍,P < 0.05)显著升高,于24 h达到峰值; 当嗜水气单胞菌浓度达到108 CFU/mL时,AjTLR3的基因表达量在12 h(1.9倍,P < 0.01),24 h(1.7倍,P < 0.05)及48 h(1.8倍,P < 0.05)均有显著增加,并于12 h达到峰值。
|
| 图 7 三种浓度嗜水气单胞菌作用下TLR3在鳗鲡肝细胞系不同时相的表达变化 *表示该时相刺激组和空白对照组即PBS刺激组存在显著性差异(P < 0.05),**表示该时相刺激组和空白对照组存在极显著性差异(P < 0.01) |
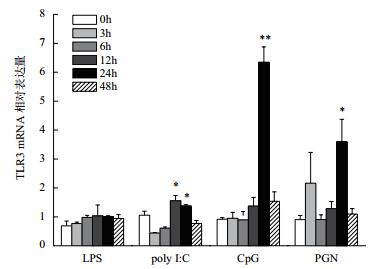
不同病原相关分子模式LPS、poly I:C、CpG-DNA和PGN刺激下日本鳗鲡肝脏细胞AjTLR3基因的表达水平见图 8。如图所示,LPS刺激未能引起AjTLR3基因表达变化; poly I﹕C刺激后的12 h(1.6倍,P < 0.05)及24 h(1.5倍,P < 0.05),AjTLR3表达量显著提高; CpG处理组中的AjTLR3基因表达水平在24 h极显著(P < 0.01)提高,是对照的6.4倍; PGN免疫组在24 h时基因表达水平极显著升高(3.7倍,P < 0.01)。
|
| 图 8 LPS,poly I:C,CpG-DNA和PGN对鳗鲡肝细胞AjTLR3基因表达的影响 *表示该时相刺激组和空白对照组即PBS刺激组存在显著性差异(P < 0.05),**表示该时相刺激组和空白对照组存在极显著性差异(P < 0.01) |
日本鳗鲡AjTLR3基因编码921个氨基酸,具有类似高等哺乳类TLR3结构域,包括亮氨酸的重复序列LRR、跨膜结构域以及TIR结构域,系TLRs家族基因的典型结构特征[22]。LRR结构域参与了特定病原微生物分子模式的识别,诱导Ⅰ型干扰素(Interferon,IFN)以及其他细胞因子的产生,进而引发下游的免疫信号分子传导[2]。AjTLR3有16个LRR结构域,与虹鳟TLR3完全一致[14],其中位于C端以及N端的LRR的结构有利于包埋暴露的疏水性氨基酸,使胞外结构域(Extracellular domain,ECD)趋于稳定[22-23]。人类TLR3中的TIR结构域上有两个保守的酪氨酸残基,磷酸化后能诱导TLR3分子识别dsRNA,激发免疫信号通路的传导[24]。其中,一个保守的氨基酸残基(Tyr777)在日本鳗鲡AjTLR3中被发现,而另一个被组氨酸替代。此外,在人类TLR3的TIR结构域与跨膜结构域之间存在3个保守氨基酸残基(Phe732、Leu742和Gly743),与NF-κB和IFN-β启动子激活密切相关[25]。这3个保守氨基酸在日本鳗鲡(Phe750、Leu761和Gly762)和斜带石斑鱼(Phe748、Leu758和Gly759)(图 4)均有发现。推测日本鳗鲡AjTLR3与人类TLR3类似,具有诱导NF-κB/ IFN-β的表达和细胞内信号传导的功能。
日本鳗鲡AjTLR3基因在各组织中均有不同程度的表达,与大黄鱼[16]、斜带石斑鱼[26]、鲤鱼[11]和黄颡鱼[27]等鱼类表达情况相似。鱼类TLR3基因组成型表达模式反映该分子在宿主不同组织的免疫监测中发挥作用。日本鳗鲡肝脏AjTLR3基因表达水平明显高于其他组织器官,在其他多种鱼类如大黄鱼、斜带石斑鱼、鲤鱼和黄颡鱼的肝脏中也均有高表达,表明肝脏是日本鳗鲡重要的免疫器官,在天然免疫中具有重要作用[28]。
肖调义等[29]研究发现,赤眼鳟(Squaliobarbus curriculus)感染GCRV后其肝脏、脾脏、肾脏的TLR3表达水平显著升高,而Rodriguez等[14]发现感染传染性造血器官坏死病病毒(Infectious hematopoietic necrosis virus,IHNV)可以引起虹鳟TLR3的表达水平显著上调,表明TLR3分子与鱼体抗病毒的免疫应答密切相关。已有的研究表明,作为病毒类似物的poly I:C免疫注射大黄鱼[16]、日本牙鲆[30]、虹鳟[14]以及稀有鮈鲫[12]后,TLR3基因的表达量均有显著提高。在本研究中,日本鳗鲡免疫注射poly I:C后,AjTLR3基因表达量在肝脏、脾脏、皮肤、心脏、肌肉和肠中显著上调(P < 0.05)。此外,日本鳗鲡肝脏细胞经poly I﹕C刺激12 h和24 h后AjTLR3表达量显著升高(P < 0.05),表明poly I:C作为特异性配体被AjTLR3识别,这与草鱼头肾细胞在poly I:C刺激下的TLR3表达量显著增加的结果一致[18]。以上的日本鳗鲡体内和体外研究结果表明AjTLR3分子在抗病毒免疫应答中起重要作用。
嗜水气单胞菌属于革兰氏阴性细菌,是养殖鱼类主要病原菌之一[31]。最近,Zhang等[27]研究发现黄颡鱼经嗜水气单胞菌免疫刺激后的TLR3基因水平在肝脏、肾脏及脾脏免疫器官均有显著提高。本研究发现高浓度的嗜水气单胞菌能够引起日本鳗鲡肝脏细胞AjTLR3的表达量显著增加,与迟缓爱德华菌感染后的斑马鱼以及副溶血弧菌免疫刺激后的大黄鱼中的研究结果相似[8, 16],这可能与AjTLR3参与了肝脏细胞抗细菌免疫应答反应有关。LPS作为革兰氏阴性细菌的主要病原模式分子,能够诱导日本鳗鲡肝脏和肠中AjTLR3表达水平升高,表明AjTLR3参与了相关组织器官对细菌的免疫识别与应答反应。此外,本实验发现LPS刺激并未引起AjTLR3基因在日本鳗鲡肝脏细胞中的明显变化,其原因可能是由于机体内以及单独肝脏细胞针对LPS的免疫应答机制不同所致。PGN作为革兰阳性菌细胞壁的主要组分,免疫刺激日本鳗鲡肝脏细胞24 h后,其AjTLR3的表达量显著增高,且达到峰值,这与草鱼头肾细胞的经PGN刺激后的结果相一致[18]。以上结果提示,AjTLR3在日本鳗鲡抗细菌免疫应答中扮演重要角色。
细菌基因组DNA存在富含CpG的寡脱氧核苷酸(ODNs)序列,可以激活脊椎动物的天然免疫和适应性免疫应答[32]。Tsujimoto等[33]研究发现CpG-ODN能够提高小鼠的抗肿瘤细胞的免疫能力。在本实验中,CpG-ODN也能诱导日本鳗鲡肝脏细胞的AjTLR3基因表达水平显著升高,这与日本牙鲆淋巴外周血细胞研究结果一致[30],推测CpG-ODN在促进细胞抗病毒炎症反应方面发挥作用。
综上所述,经poly I:C免疫刺激后,AjTLR3基因表达水平在日本鳗鲡多个组织中显著提高,同时也能增强日本鳗鲡肝脏细胞AjTLR3基因的表达,表明AjTLR3在日本鳗鲡抗病毒免疫应答中具有重要作用。此外,嗜水气单胞菌以及病原相关分子模式CpG和PGN均能诱导AjTLR3基因在日本鳗鲡肝脏细胞中的高水平表达,显示该分子也与日本鳗鲡抗细菌免疫反应密切相关。本研究结果为日本鳗鲡针对不同病原微生物免疫防御机制的深入探讨提供了参考。
4 结论本研究中克隆了日本鳗鲡AjTLR3基因,其全长3 383 bp,编码921个氨基酸,包含16个LRR的胞外结构域、跨膜结构域以及TIR结构域,其中在TIR结构域含有一个高度保守的氨基酸残基Tyr778。在不同病原分子模式刺激下,体内以及体外实验的Real-time PCR结果充分证明,AjTLR3在日本鳗鲡抵御病毒及细菌的免疫应答过程中具有重要作用。
| [1] |
Takeda K, Akira S. Toll-like receptors in innate immunity[J]. International Immunology, 2005, 17(1): 1-14. |
| [2] |
Rauta PR, Samanta M, Dash HR, et al. Toll-like receptors(TLRs)in aquatic animals:signaling pathways, expressions and immune responses[J]. Immunology Letters, 1900, 158(2): 14-24. |
| [3] |
范泽军, 邹鹏飞, 姚翠鸾. 鱼类Toll样受体及其信号传导的研究进展[J]. 水生生物学报, 2015, 39(1): 173-184. DOI:10.7541/2015.22 |
| [4] |
Yan K, Zhu W, Yu L, et al. Toll-like receptor 3 and RIG-I-like receptor activation induces innate antiviral responses in mouse ovarian granulosa cells[J]. Molecular & Cellular Endocrinology, 2013, 372(1-2): 73-85. |
| [5] |
Bell JK, Askins J, Hall PR, et al. The dsRNA binding site of human Toll-like receptor 3[J]. Proc Natl Acad Sci USA, 2006, 103(23): 8792-8797. DOI:10.1073/pnas.0603245103 |
| [6] |
Coscia MR, Giacomelli S, Oreste U. Toll-like receptors:an overview from invertebrates to vertebrates[J]. Invertebrate Survival Journal, 2011, 8(2): 210-226. |
| [7] |
Jenkins KA, Mansell A. TIR-containing adaptors in Toll-like receptor signalling[J]. Cytokine, 2010, 49(3): 237-244. DOI:10.1016/j.cyto.2009.01.009 |
| [8] |
Phelan PE, Mellon MT, Kim CH. Functional characterization of full-length TLR3, IRAK-4, and TRAF6 in zebrafish(Danio rerio)[J]. Molecular Immunology, 2005, 42(9): 1057-1071. DOI:10.1016/j.molimm.2004.11.005 |
| [9] |
Bilodeau AL, Waldbieser GC. Activation of TLR3 and TLR5 in channel catfish exposed to virulent Edwardsiella ictaluri[J]. Developmental & Comparative Immunology, 2005, 29(8): 713-721. |
| [10] |
Oshiumi H, Tsujita T, Shida K, et al. Prediction of the prototype of the human Toll-like receptor gene family from the pufferfish, Fugu rubripes, genome[J]. Immunogenetics, 2003, 54(11): 791-800. |
| [11] |
Yang C, Su J. Molecular identification and expression analysis of Toll-like receptor 3 in common carp Cyprinus carpio[J]. Journal of Fish Biology, 2010, 76(8): 1926-1939. DOI:10.1111/jfb.2010.76.issue-8 |
| [12] |
Su JG, Zhu ZY, Wang YP, et al. Toll-like receptor 3 regulates Mx expression in rare minnow Gobiocypris rarus after viral infection[J]. Immunogenetics, 2008, 60(3): 195-205. |
| [13] |
Su JG, Jang SH, Yang CR, et al. Genomic organization and expression analysis of Toll-like receptor 3 in grass carp(Ctenopharyngodon idella)[J]. Fish & Shellfish Immunology, 2009, 27(3): 433-499. |
| [14] |
Rodriguez MF, Wiens GD, Purcell MK, et al. Characterization of Toll-like receptor 3 gene in rainbow trout(Oncorhynchus mykiss)[J]. Immunogenetics, 2005, 57(7): 510-519. DOI:10.1007/s00251-005-0013-1 |
| [15] |
Matsumoto M, Oshiumi H, Seya T. Antiviral responses induced by the TLR3 pathway[J]. Reviews in Medical Virology, 2011, 21(2): 67-77. DOI:10.1002/rmv.680 |
| [16] |
Huang XN, Wang ZY, Yao CL. Characterization of Toll-like receptor 3 gene in large yellow croaker, Pseudosciaena crocea[J]. Fish & Shellfish Immunology, 2011, 31(1): 98-106. |
| [17] |
苏建国, 朱作言, 汪亚平. 团头鲂Toll样受体3基因的克隆及特征研究[J]. 水生生物学报, 2009, 33(5): 986-993. |
| [18] |
Chen LJ, Li QM, Su JG, et al. Trunk kidney of grass carp(Ctenopharyngodon idella)mediates immune responses against GCRV and viral/bacterial PAMPs in vivo and in vitro[J]. Fish & Shellfish Immunology, 2013, 34(3): 909-919. |
| [19] |
Lee WC, Chen YH, Lee YC, et al. The competitiveness of the eel aquaculture in Taiwan(China), Japan, and China[J]. Aquaculture, 2003, 221(1-4): 115-124. DOI:10.1016/S0044-8486(03)00004-8 |
| [20] |
Joh SJ, Kim MJ, Kwon HM, et al. Characterization of Edwardsiella tarda isolated from farm-cultured eels, Anguilla japonica, in the Republic of Korea[J]. Journal of Veterinary Medical Science, 2011, 73(1): 7-11. DOI:10.1292/jvms.10-0252 |
| [21] |
Feng JJ, Guo SL, Lin P, et al. Identification of a retinoic acid-inducible gene I from Japanese eel(Anguilla japonica)and expression analysis in vivo and in vitro[J]. Fish & Shellfish Immunology, 2016, 55: 249-256. |
| [22] |
Gay NJ, Gangloff M. Structure and function of Toll receptors and their ligands[J]. Annual Review of Biochemistry, 2007, 76(1): 141-165. DOI:10.1146/annurev.biochem.76.060305.151318 |
| [23] |
Samanta M, Basu M, Swain B, et al. Molecular cloning and characterization of Toll-like receptor 3, and inductive expression analysis of type Ⅰ IFN, Mx and pro-inflammatory cytokines in the Indian carp, rohu(Labeo rohita)[J]. Mol Biol Rep, 2013, 40(1): 225-235. DOI:10.1007/s11033-012-2053-6 |
| [24] |
Sarkar SN, Smith HL, Rowe TM, et al. Double-stranded RNA signaling by Toll-like receptor 3 requires specific tyrosine residues in its cytoplasmic domain[J]. J Biol Chem, 2003, 278(7): 4393-4396. DOI:10.1074/jbc.C200655200 |
| [25] |
Funami K, Matsumoto M, Oshiumi H, et al. The cytoplasmic 'linker region' in Toll-like receptor 3 controls receptor localization and signaling[J]. International Immunology, 2004, 16(8): 1143-1154. DOI:10.1093/intimm/dxh115 |
| [26] |
Lin KB, Ge H, Lin Q, et al. Molecular characterization and functional analysis of Toll-like receptor 3 gene in orange-spotted grouper(Epinephelus coioides)[J]. Gene, 2013, 527(1): 174-182. DOI:10.1016/j.gene.2013.06.014 |
| [27] |
Zhang XT, Zhang GR, Shi Z C, et al. Expression analysis of nine Toll-like receptors in yellow catfish(Pelteobagrus fulvidraco)responding to Aeromonas hydrophila challenge[J]. Fish & Shellfish Immunology, 2017, 63: 384-393. |
| [28] |
Feng JJ, Guan RZ, Guo SL, et al. Molecular cloning of japanese eel Anguilla japonica TNF-α and characterization of its expression in response to LPS, polyI:C and Aeromonas hydrophila infection[J]. Chinese Journal of Oceanology and Limnology, 2014, 32(5): 1046-1059. DOI:10.1007/s00343-014-3231-2 |
| [29] |
肖调义, 李伟, 王荣华, 等. 赤眼鳟Toll样受体3基因cDNA全长克隆及表达分析[J]. 水生生物学报, 2016, 40(5): 894-901. DOI:10.7541/2016.115 |
| [30] |
Hwang SD, Ohtani M, Hikima J, et al. Molecular cloning and characterization of Toll-like receptor 3 in Japanese flounder, Paralichthys olivaceus[J]. Developmental & Comparative Immunology, 2012, 37(1): 87-96. |
| [31] |
郭松林. 鳗鲡细菌性疾病的研究概况[J]. 科学养鱼, 2011(7): 48-49. |
| [32] |
Krieg AM, Kline JN. Immune effects and therapeutic applications of CpG motifs in bacterial DNA[J]. Immunopharmacology, 2000, 48(3): 303-305. DOI:10.1016/S0162-3109(00)00228-9 |
| [33] |
Tsujimoto H, Efron PA, Matsumoto T, et al. Maturation of murine bone marrow-derived dendritic cells with poly(I﹕C)produces altered TLR-9 expression and response to CpG DNA[J]. Immunology Letters, 2006, 107(2): 155-162. DOI:10.1016/j.imlet.2006.09.001 |