2. 中国科学院大学,北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049
蛋白质是参与生命体代谢、各种信号传递、生长发育的重要大分子,因此,其合成、正常的折叠修饰并根据信号肽到达发挥功能的相应细胞器也是同样重要的命题。分泌蛋白和膜蛋白在核糖体上开始合成后很快进入内质网,并在内质网继续合成新肽链以及对新肽链进行初步的修饰。然而已有的研究显示,内质网中的蛋白质浓度是100 g/L(大约是2 mmol/L),而一个α螺旋或β折叠正确折叠完成时间是0.1-1 μs,因此,在如此高蛋白浓度的场所进行如此快速的折叠是非常复杂且易错的[1-2]。为了保证只有正确折叠的蛋白质进入高尔基体,内质网中有一个专门负责监测蛋白质折叠水平的系统,称为内质网质量监控系统(Endoplasmic reticulum quality control,ERQC)[3-4]。如果不能通过ERQC的监测,蛋白质会继续留在内质网中并在多种分子伴侣的帮助下进行再折叠,直到达到正确构象运出内质网或者被内质网相关的降解系统(Endoplasmic reticulum associated degradation,ERAD)降解。积累在内质网中的错误折叠蛋白超出了内质网处理的范围就会造成内质网胁迫(ER stress),而一些外源或者内源的胁迫压力,如植物面临的冷、热、盐和干旱等会加剧ER stress[5-8]。ER stress也会诱导非折叠蛋白反应(Unfolded protein response,UPR),UPR进一步诱导分子伴侣及胁迫相关基因的表达,帮助细胞度过难关。
ER stress、ERQC、UPR、ERAD等过程在酵母和动物方面研究较多,已经有了被认可的成熟机制。对植物的研究虽然起步较晚,但是近几年发展迅速,多个相关基因及编码蛋白的功能被一一揭示[9]。在本综述中,我们将详细总结蛋白质在内质网中的糖基化修饰过程、ERQC、UPR及ERAD识别错误折叠蛋白并发挥功能的过程,以及它们在植物生长发育过程中的作用。
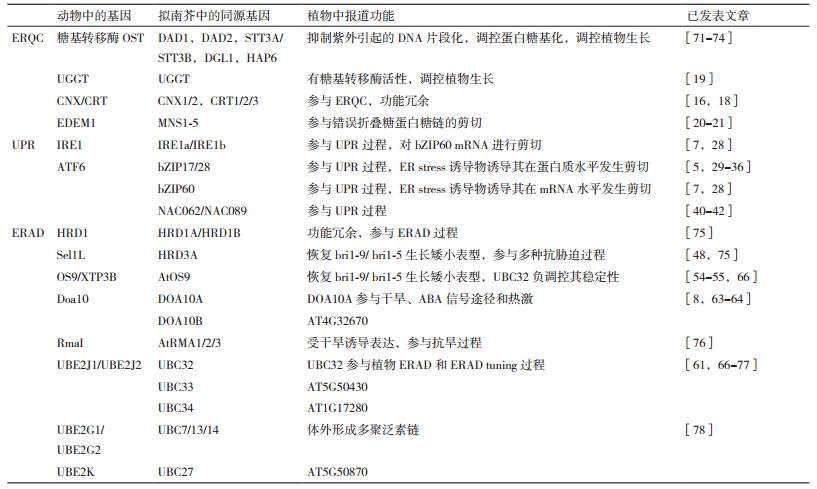
1 内质网质量监控系统(ERQC)ERQC的概念最早由Hurtley和Helenius[4]在1898年提出,主要涉及监控内质网中蛋白质的折叠状态。蛋白质的折叠与初生肽链的N-链接蛋白质糖基化修饰过程息息相关。加在蛋白质初生肽链上的糖结构称为G寡糖,它由两个葡糖酰胺,9个甘露糖和3个葡萄糖组成(Glc3Man9GlcNAc2)。G寡糖加在肽链特定序列(Asn-X-Ser或Asn-X-Thr)的天冬酰胺上,然后在内质网以及高尔基体一系列酶的作用下剪切成最终结构[10]。
肽链上的G寡糖依次经过葡萄糖苷酶Ⅰ和葡萄糖苷酶Ⅱ的酶切作用形成Glc1Man9GlcNAc2结构,该结构可以被内质网膜上类分子伴侣CNX以及其位于内质网腔中的同源蛋白CRT识别。CNX/CRT能够大量招募帮助蛋白质折叠的分子伴侣,如蛋白二硫键异构酶(Protein disulfide isomerases,PDIs)可以形成或者断裂分子内以及分子间二硫键,既保证了蛋白质的正确折叠又避免蛋白质凝聚。因此,进入CNX/CRT循环的糖蛋白可以在多种分子伴侣帮助下进行折叠,同时,在CNX/CRT循环中糖链的最后一个葡萄糖继续被葡萄糖苷酶Ⅱ剪切[11-12]。如果肽链能够完成正确折叠,则保留在内质网,或被分泌出内质网进入高尔基体;如果肽链没有完成正确折叠,则在UDP葡萄糖糖蛋白糖基转移酶(UDP-glucose:glycoprotein glucosyltransferase,UGGT)的作用下重新加上一个葡萄糖分子,继续在CNX/CRT循环中进行再折叠[13]。UGGT是一个定位于内质网腔的蛋白,其N端结构可以和非正确折叠的蛋白结构结合,C端是糖基转移酶催化结构域。作为蛋白折叠状态的检测员,UGGT能够保证蛋白质最大可能完成折叠。然而,CNX/CRT并不是无限循环的,如果蛋白质最终仍然不能得到其正确构象,蛋白也会从循环中分离。在内质网1,2-甘露糖苷酶Ⅰ和EDEM(酵母中为Htm1)的催化下切掉侧链上的两个甘露糖,形成能被ERAD系统组分OS9识别的α-1,6糖苷键,最终错误折叠蛋白通过ERAD系统清除[14-15]。到目前为止,错误折叠蛋白经过多久的再折叠循环后才从CNX/CRT循环中脱离还不清楚。
由于其功能的重要性,ERQC系统在高等生物中高度保守。在植物中已经鉴定到了多个参与ERQC和内质网中糖基化修饰相关的蛋白,并且由于植物要适应各种复杂生存环境,植物中ERQC的同源蛋白数目一般不止一个。在拟南芥中,CNX有两个拷贝,而CRT有3个同源蛋白。研究显示,在正常生长条件下,cnx1 cnx2双突变体以及crt1 crt2 crt3三突变体均没有明显表型,而cnx1 crt1 crt2 crt3四突变体表现出植株矮小,主根变短,根毛变密以及花粉活力变差,花粉管生长异常等表型。但是有意思的是,cnx2 crt1 crt2 crt3并没有表现出类似的表型,但是CNX2能够恢复cnx1 crt1 crt2 crt3突变体一系列生长异常的表型。说明CNX1和CNX2存在功能冗余,但是CNX1比CNX2作用更强。如果CNX/CRT循环中的5个基因同时功能缺失,则会导致植物致死[16]。植物甾醇受体BRI的突变体形式bri1-9和bri1-5被证实是ERAD的底物,正常的BRI蛋白在质膜上接受信号,而突变形式的bri1-9和bri1-5积累在内质网中,导致植物出现生长矮小的表型[17]。如果ERAD途径受阻,大量的bri1-9/ bri1-5在内质网中积累并溢出到质膜,接收信号后植物能够恢复表型。通过筛选恢复bri1-9矮小表型的基因,筛选到了CRT3,crt3 bri1-9双突变体能够很好地恢复bri1-9生长矮小的表型[18]。
UGGT在拟南芥中只有一个同源蛋白,uggt突变体植物中以UDP-葡萄糖为底物的糖基转移酶活性明显下降,同时突变体植物表现出明显的生长矮小,主根变短表型。在uggt突变体中,BiP1/2、BiP3、PDIL等UPR响应基因的表达明显上调,说明在没有外源ER stress时,UGGT突变能够诱导UPR的产生[19]。
哺乳动物中α-甘露糖苷酶EDEM在拟南芥中的同源家族有5个成员,分别称为MNS1-5,其中MNS1和MNS2是高尔基体甘露糖苷酶,MNS3是内质网甘露糖苷酶,在MNS1/2的上游发挥作用。在MNS1-3的作用下,寡糖由Man9GlcNAc2结构剪切为Man5GlcNAc2结构[20]。MNS4和MNS5是内质网定位的蛋白质,对折叠正确的分泌蛋白上的甘露糖没有作用,通过烟草瞬时表达实验发现,它们能够顺利剪切内质网滞留的错误折叠糖蛋白上的甘露糖,产生被ERAD系统识别的α-1,6糖苷键,说明MNS4和MNS5参与拟南芥ERQC过程[21]。同时,mns4 mns5突变体表现出对盐胁迫处理敏感的表型。植物中越来越多的研究使得植物蛋白在内质网中的加工折叠过程越来越明朗,其中大部分过程和酵母,动物中的研究是高度保守的。
2 非折叠蛋白应答(UPR)错误折叠蛋白质在内质网中的大量积累会诱导UPR的产生。UPR过程也是酵母、动物以及植物领域保守存在但具体的作用机制又有所差异的。酵母中的UPR只有一条通路,由内质网膜蛋白IRE1p(Inositol-Requiring Transmembrane Kinase/Endonuclease p)介导。在动物UPR中除了IRE1外还存在另外两条通路,分别是ATF6和PERK通路。热激蛋白HSP70家族成员BiP起着调控3个跨膜受体活性的作用。BiP蛋白由N端的ATP酶活结构域和C端的肽段结合结构域组成,其与错误折叠蛋白暴露的疏水残基之间的结合受ATP酶活调控。在没有受胁迫的细胞中,BiP和这3个受体结合,抑制它们的活性;而在受到胁迫时,BiP即与3个受体解离,受体被激活[22-23]。
IRE1是一个约100 kD的内质网膜定位蛋白,该蛋白主要由核糖核酸酶结构域和激酶结构域组成,在动物中存在两个同源蛋白,分别是IRE1α和IRE1β。内质网腔内的部分与BiP相互作用并在受到胁迫时解离。解离以后IRE1首先通过自身激酶活性进行自身磷酸化,进而形成同源二聚体,完成自身激活。激活的IRE1通过其核糖核酸酶活性剪切掉XBP1u中一个约26个核苷酸的内含子,形成XBP1s,通过XBP1s发挥缓解ER stress的功能[24]。另一方面,近来的研究显示,没有胁迫出现时,IRE1α作为ERAD底物被调控,并且BiP参与了该过程[25]。胁迫出现后,BiP与IRE1α解离,IRE1α蛋白质稳定性增强,发挥UPR功能。以上研究说明BiP可以从活性和蛋白质稳定性两方面调控IRE1α[25]。
ATF6是一个90 kD大小的蛋白,定位在内质网膜上。在动物中也存在两个同源蛋白,分别是ATF6α和ATF6β。研究认为ATF6α在缓解ER stress中发挥着更重要的作用。与IRE1类似,在没有胁迫时,ATF6和BiP相互作用;一旦出现胁迫,BiP与ATF6解离,ATF6激活且定位从内质网转移到高尔基体中。ATF6在高尔基体中接受进一步的膜内蛋白剪切,首先被丝氨酸蛋白酶位点1(S1P)剪切,随后被金属蛋白酶位点2(S2P)剪切。经过两步剪切,ATF6由原来的90 kD变成只具有50 kD大小转录激活结构域的蛋白并进入细胞核中,通过调控UPR相关基因或者ERAD组分基因的表达,发挥缓解内质网胁迫的作用[26]。
PERK的激活类似于IRE1,也是与BiP解离后形成二聚体并自我激活。活化后的PERK可以进一步磷酸化eIF2α(Eukaryotic translation Initiation Factor 2α),磷酸化的eIF2α继续保持与eIF2B的结合因而不能与GDP结合,造成翻译不能正常进行。因此,减少了进入内质网中的蛋白量,达到缓解ER stress的目的[27]。
植物中的UPR主要有两条通路,一条是类似于IRE1的bZIP60转录因子介导的通路,另外一条是类似于ATF6的bZIP28转录因子介导的通路。目前植物中还没有动物中PERK同源基因被报道,但是除了膜定位的bZIP类转录因子外,植物特有的NAC转录因子也参与到缓解ER stress中。2011年,Deng等[7]发现Tm和DTT等ER stress诱导剂可以诱导bZIP60发生mRNA剪切。bZIP60的mRNA在跨膜域上游被剪切掉23 bp,新组合序列编码的蛋白在剪切识别位点下游有两个预测的核定位信号[7]。进一步研究发现IRE1在bZIP60的剪切中发挥功能。IRE1在拟南芥中有两个同源蛋白,分别称为IRE1a和IRE1b。Nagashima等[28]发现在ire1b单突变体中bZIP60的剪切几乎不受影响;而在ire1a/ire1b双突变体中,bZIP60的剪切完全消失,说明植物中IRE1a和IRE1b在负责bZIP60的剪切上存在功能冗余。bZIP60和XBP1序列同源性不高,但都具有双茎环结构,对其成功剪接非常重要。
与bZIP60在mRNA水平进行活化不同,bZIP17/28在蛋白质水平进行活化。bZIP17/28均是定位在内质网膜上的转录因子,与ATF6序列相似性虽然不高,但是在结构域排列上有相似性。例如,在朝向细胞质侧的N端有一个预测的bZIP结构域,并且在朝向内质网腔的C端有一个S1P酶切位点。bZIP17和bZIP28的作用机制相似,以下以bZIP28为例来说明。与ATF6不同,bZIP28的激活虽然也受到了BiP的影响,但是并不直接与BiP相关。正常情况下,bZIP28通过与BiP相互作用滞留在内质网。当体内错误折叠的蛋白积累时,这些错误折叠蛋白竞争性的与BiP相互作用,因此使bZIP28从内质网得到释放[29-31]。而bZIP28本身在细胞质侧靠近跨膜域的部分,以及内质网腔内均有高尔基体定位信号[32-33]。当被释放的bZIP28从内质网转移到高尔基体后被S1P和S2P两个酶剪切,使包含DNA结合结构域和转录激活结构域的蛋白胞质侧从膜系统脱离进入细胞核,调控相应胁迫响应基因的表达[34-36]。研究显示不仅TM和DTT这类典型的ER stress诱导物能诱导bZIP28的活化途径,环境胁迫比如热胁迫也能够发挥作用[37-39]。此外,盐胁迫能够诱导bZIP17发生类似的活化过程[5]。
NAC类转录因子是植物中特有的一类转录因子。近期研究表明,NAC062和NAC089均可以受到ER stress的诱导表达,面对ER stress,内质网定位的NAC089和质膜定位的NAC062会转移到细胞核内,在植物的UPR中发挥重要作用[40-41]。它们均是二类单次跨膜膜蛋白,与bZIP28在内质网腔内还有大的蛋白结构域不同,NAC062和NAC089仅通过C末端插在膜上,功能结构域均位于胞质侧。因此目前关于二者是如何定位到细胞核的具体机制还不甚清晰。CHIP-qPCR结果显示NAC062在一些UPR响应基因如BiP2的启动子区域富集。下调体内NAC062时,植物表现对胁迫敏感,而在bzip28/bzip60双突背景下过表达核定位的NAC062D-Myc时植物对胁迫表现出抗性[41]。冷胁迫也可以诱导NAC062的核定位[42]。关于植物体内的UPR系统除了膜相关转录因子外可能还有更多蛋白参与其中,相信随着研究的深入会有更多参与因子被发掘,同时,更详细的功能机制也会被解析。
为了缓解ER stress,直接将错误折叠蛋白清除对于生物体同样至关重要。那么接下来就介绍参与这个过程的重要因子:ERAD和ERQC autophagy。这两个途径的主要区别是ERAD机制将错误折叠蛋白底物反转运到细胞质然后进行泛素化修饰,最后通过细胞质的26S蛋白酶体清除[43];而集聚在一起不能被反转运到细胞质中的错误折叠蛋白或者不能被ERAD降解的蛋白质则通过ERQC autophagy途径降解[44-45]。首先总结ERAD途径的研究进展。
3 内质网相关的蛋白质降解ERAD系统有效的行使其功能对于细胞正常的生命活动非常重要[43, 46]。在人中,ERAD与一些疾病相关,如帕金森综合症。Parkin蛋白是一个RING-type的E3连接酶,家族性帕金森综合症患者的致病原因之一是Parkin蛋白的突变,突变的Parkin丧失了E3活性,不能有效缓解错误折叠蛋白积累造成的胁迫,这种胁迫持续的积累会产生神经毒性,从而造成帕金森综合症[47]。植物中ERAD也与耐受逆境胁迫过程有关,研究工作表明用300 mM NaCl处理野生型拟南芥6 h后,BiP、CNX1的表达量均明显上调[48]。除了在降解这些错误折叠蛋白中发挥作用外,研究显示ERAD在正常的生长发育过程中也发挥着重要的作用。哺乳动物中,UBE2J1功能缺失会导致小鼠精子发育缺陷,从而影响生育[49]。同样在小鼠体内敲除SEL1L,能够引起小鼠体内脂类代谢的异常,最终导致小鼠体重下降[50]。而在植物当中,通过比较番茄成熟缺陷突变体rin和野生型差异表达蛋白发现了一个与酵母ERAD中Ubc6p同源的番茄UBC32,该基因的功能缺失导致番茄果实晚熟[51]。综上所述,ERAD过程中的组分蛋白既可以在外界胁迫存在的情况下,通过降解错误折叠蛋白来缓解ER stress,同时也在动植物的正常生长发育中发挥重要功能。
ERAD过程主要由四步组成:(1)对错误折叠蛋白的识别;(2)位于内质网腔和内质网膜上的蛋白质反转运到细胞质;(3)在特定的E1,E2和E3连接酶作用下进行泛素化修饰;(4)由26S蛋白酶体降解。对错误折叠蛋白的识别主要涉及两种,一种是对错误折叠糖蛋白的识别,这种识别主要依赖于糖蛋白经过剪切后暴露的α-1,6糖苷键。OS9(植物中是AtOS9)结合α-1,6糖苷键后通过与SEL1L(植物中HRD3A)互作将底物拉进HRD1复合体。另一种是对非糖蛋白的识别,通过BiP识别错误折叠蛋白暴露的疏水肽段完成。蛋白与BiP结合后并不进入CNX/CRT循环,而是被其传递给二硫键还原酶ERdj5,进而传递给SEL1L进入ERAD系统[52-53]。如果将糖蛋白的糖链加工过程进行人为抑制,那么糖蛋白也会通过BiP识别进入ERAD降解系统[52]。拟南芥中已证实AtOS9参与ERAD过程,atos9突变体表现出对盐胁迫和衣霉素处理敏感的表型,并且atos9 bri1-9/ atos9 bri1-5能够恢复bri1-9/ bri1-5生长矮小的表型。但是AtOS9并不能恢复酵母中OS9p对于底物CPY*的降解,说明植物中和酵母中OS9在介导不同的底物中发挥作用[54-55]。
大部分经过ERAD系统清除的错误蛋白都是内质网腔蛋白或者内质网膜蛋白,这部分蛋白需要转运到细胞质中才能被细胞质中的蛋白酶体降解。目前关于反转运通道研究成熟的主要有3种。一种是SEC61转运通道,关于SEC61,我们更熟悉的是其作为通道介导分泌蛋白进入内质网中。但是酵母和动物中的研究表明缺失SEC61可以使多个ERAD底物蛋白,如酵母配对信息素pαF、细菌毒素的催化亚基等稳定性增强[56-57]。此外,酵母中的DER1(动物中为Derlin1)也认为在底物转运中发挥重要功能。DER1缺失可以完全阻止CPY*和PrA*的降解[58]。但是DER1只有四个跨膜域,很难单独发挥通道功能,因此更认为它作为通道的重要组成部分。第三种通道即是介导底物蛋白泛素化修饰的E3连接酶,研究表明HRD1通常通过跨膜域与底物蛋白互作,并介导底物蛋白的反转运[59-60]。拟南芥中的E2 UBC32是HRD1的底物,研究显示UBC32与HRD1互作即是通过跨膜域实现[61]。
反转运到细胞质后即进行底物的泛素化修饰。有一类特殊的E2和E3介导ERAD底物的降解。在酵母中有两个E3连接酶,分别为Hrd1p和Doa10p,E2分别有Ubc6p和Ubc7p。动物中目前已经报道的参与ERAD的E3有16个,而Ubc6p在人类细胞中的同源蛋白有UBE2J1和UBE2J2;Ubc7p在人类细胞中的同源蛋白有UBE2G1和UBE2G2。植物中已经报道的有E3连接酶HRD1、DOA10及Rma1,接头蛋白HRD3A,泛素结合酶UBC32等。HRD1复合体中的HRD1蛋白,HRD3A以及AtOS9在拟南芥中均已有报道。HRD1在拟南芥中有两个功能冗余的同源蛋白HRD1A和HRD1B。双突变体表现出衣霉素处理敏感的表型,并且HRD1通过直接介导UBC32的泛素化参与ERAD活性的调节[61]。hrd3a突变体对盐、百草枯等一系列处理均是超敏感的,并且突变体体内积累更多的ROS[48],在其突变体中过表达UPR相关基因bZIP17,bZIP28,bZIP60可以弥补hrd3a突变造成的胁迫表型[62]。通过筛选干旱敏感突变体dry2的抑制子得到SUD1(Suppressor of dry2 defects1),SUD1即酵母中Doa10p的同源蛋白。突变的DOA10(SUD1)能够显著恢复dry2干旱敏感的表型,但是DOA10只能下调羟甲基戊二酸单酰辅酶A还原酶(HMG-CoA)的活性并不影响HMG-CoA的蛋白稳定性[8]。此外,doa10突变体在ABA上表现出超敏感表型,突变体干种子中的ABA含量明显增高,添加ABA抑制剂后突变体表型得到恢复。因此,DOA10是ABA合成和信号通路中的负调控因子[63]。而最近又有研究报道doa10hrd1ahrd1b三突变体对热激处理表现不敏感表型[64]。已报道的动物中参与ERQC、UPR、ERAD相关基因在拟南芥中的同源基因见表 1,相信随着植物领域ERAD研究的深入,越来越多的ERAD组分以及这些组分参与的生物学过程被发掘。
鉴于ERAD系统如此重要,ERAD活性受到了生命体的精细调控。如果体内的ERAD活性紊乱,内质网中的ERAD组分含量过高,当ERAD总体活性过高时,就会非特异性的识别一些蛋白折叠中间体,将这些正在折叠中的蛋白作为错误折叠蛋白降解,这显然对生物体是不利的。在这种情况下,细胞通过降解体内ERAD组分使ERAD活性保持在一个较低水平,进而保证细胞内的蛋白正确折叠的过程,这种机制就称为ERAD平衡(ERAD tuning)[65]。被调控的这些ERAD组分本身又是发挥功能的组分,如果细胞遭受外来侵害而使细胞内错误折叠蛋白增多,这些蛋白又会稳定下来发挥其相应的功能。因此,ERAD tuning活性是受体内错误折叠蛋白水平调控的。但是目前胁迫使ERAD组分稳定的具体机制研究还非常少见。一种假设认为,错误折叠蛋白可能在这个过程中起到重要作用。在错误折叠蛋白很少的情况下,ERAD组分大部分处于未组装的状态,因此使某些组分被降解;而大量错误折叠蛋白存在时,可以促使ERAD组分组装,形成有功能的ERAD机器,同时保护那些原来被降解的组分不再被降解。最近一篇关于HRD1介导IRE1降解的文章介绍,胁迫可以很大程度上减弱HRD1与IRE1之间的相互作用。另外,胁迫处理与不处理条件下,胰蛋白酶对IRE1的剪切带型也不同,因此,作者认为胁迫可能从影响蛋白相互作用和改变构象两方面使ERAD组分稳定[24]。胁迫消除后,被过量诱导表达的各种伴侣分子和ERAD组分需要被清除掉,分子伴侣的消除时间更长,如PDI、BIP、CNX等的半衰期均大于24 h。而ERAD组分的消除时间相对较快。因此,ERAD tuning在面对胁迫时对ERAD活性的调节相比UPR更加迅速,同时UPR一部分下游基因编码ERAD相关蛋白。但是目前关于ERAD平衡的研究还很少。Chen等[61]最近发现UBC32的蛋白稳定性受胁迫处理的调控,在没有胁迫时HRD1可以直接介导UBC32通过蛋白酶体途径降解;胁迫处理时,UBC32稳定性增强。并且HRD1对UBC32的调控在动物细胞中保守存在。同时,他们还发现UBC32可以负调控HRD1复合体中的AtOS9,通过相互调控实现ERAD活性的平衡[66]。而这个结果也和动物中OS9在UBE2J1突变体中积累的结果是一致的,说明ERAD平衡像ERAD过程一样在高等生物中保守存在。
4 ERQC自噬途径(autophagy)很多内质网膜上的蛋白由于本身是膜蛋白,多个蛋白分子集聚到一起后体积较大,不能反转运到细胞质中由蛋白酶体来降解,这些蛋白一般由另外一种重要的调控蛋白稳定性的途径——自噬途径降解。但是目前植物中相关领域研究还很少,动物中有较多报道,如α1抗胰蛋白酶Z(ATZ)是α1抗胰蛋白酶的一个点突形式,ATZ能够影响蛋白的正常糖基化,形成不可溶的聚集体。由于其不能反转运到细胞质中,因此会通过类内吞的作用形成自噬体,进而通过自噬途径降解。这个过程不需要泛素化修饰,也没有ERAD相关组分参与[67]。Houck等[45]在2014年报道某些蛋白是可溶的,但是由于其不能被ERAD底物识别系统识别,也会通过ERQC autophagy途径降解。如一种G蛋白偶联受体GnRHR,其部分折叠的突变形式E90K在蛋白酶体抑制剂处理时不受影响,而在自噬途径抑制剂处理时蛋白积累。作者认为这是因为部分错误折叠使ERAD识别系统不能很好地识别该底物,从而诱导了自噬途径。完全错误折叠的突变体蛋白S168R主要是通过蛋白酶体途径降解,但是如果将ERAD途径阻止,S168R就会进入ERQC autophagy途径。以上两种自噬都是不依赖于泛素化修饰的,而有一类选择性自噬途径依赖于泛素化修饰,然后由一个中间受体介导进入自噬途径。这个中间受体需要同时具有泛素结合域和LC3(定位于自噬体)结合域,目前已经知道的有SQSTM1/P62,NBR1等。2017年Feng等[68]报道HRD1可以介导SERPINAE342K进行K48位的泛素化修饰,部分通过蛋白酶体途径降解,而另外大部分则通过SQSTM1/P62介导的选择性自噬途径降解。
研究显示在拟南芥中ER stress可以诱导自噬过程,进而造成内质网膜及内含物的降解。其中IRE1b在该过程中发挥重要作用,而同源基因IRE1a没有此作用[69]。进一步研究揭示了ER stress造成的积累的错误折叠蛋白作为信号激活了自噬过程[70]。虽然目前植物领域这部分详细的报道还较少,但是鉴于植物领域近几年的蓬勃发展我们相信在不久的将来肯定会有更多相关报道。图 1总结了动植物中内质网质量检测系统过程。
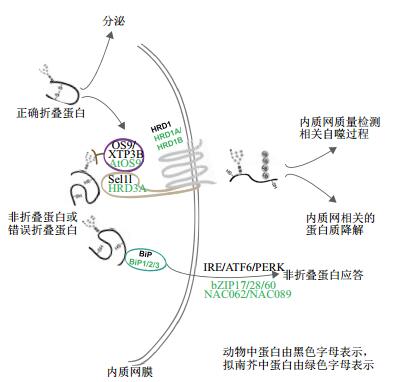
|
| 图 1 动植物中的内质网质量监控系统 |
目前关于ERAD的研究仍然是酵母和动物领域研究的最详尽,植物中的相关研究是近几年才逐渐增多。植物整个生命过程中受到精细的激素调控,同时也会遭受各种生物或非生物胁迫,解析ERAD如何在植物面对胁迫时发挥功能是非常重要的。虽然已经报道ERAD参与盐胁迫,热胁迫以及ABA信号通路等过程,但是距离解析ERAD在更多胁迫或者激素信号中详细的作用机理还有很大距离。此外,自噬途径与ERAD介导的蛋白酶体途径在介导底物降解之间的协同合作方面的研究在植物领域也知之甚少,值得科学家投入更多精力进行研究,对这些问题的深入研究将有助于我们进一步揭示ERAD调控植物生长发育的奥秘。
| [1] |
Castellani F, van Rossum B, Diehl A, et al. Structure of a protein determined by solid-state magic-angle-spinning NMR spectroscopy[J]. Nature, 2002, 420(6911): 98-102. DOI:10.1038/nature01070 |
| [2] |
Schroder M, Kaufman RJ. ER stress and the unfolded protein response[J]. Mutat Res, 2005, 569(1-2): 29-63. DOI:10.1016/j.mrfmmm.2004.06.056 |
| [3] |
Ellgaard L, Helenius A. Quality control in the endoplasmic reticulum[J]. Nat Rev Mol Cell Biol, 2003, 4(3): 181-191. DOI:10.1038/nrm1052 |
| [4] |
Hurtley SM, Helenius A. Protein oligomerization in the endoplasmic reticulum[J]. Annu Rev Cell Biol, 1989, 5: 277-307. DOI:10.1146/annurev.cb.05.110189.001425 |
| [5] |
Liu JX, Srivastava R, Che P, et al. Salt stress responses in Arabidopsis utilize a signal transduction pathway related to endoplasmic reticulum stress signaling[J]. Plant J, 2007, 51(5): 897-909. DOI:10.1111/j.1365-313X.2007.03195.x |
| [6] |
Thomashow MF. PLANT COLD ACCLIMATION:freezing tolerance genes and regulatory mechanisms[J]. Annu Rev Plant Physiol Plant Mol Biol, 1999, 50: 571-599. DOI:10.1146/annurev.arplant.50.1.571 |
| [7] |
Deng Y, Humbert S, Liu JX, et al. Heat induces the splicing by IRE1 of a mRNA encoding a transcription factor involved in the unfolded protein response in Arabidopsis[J]. Proc Natl Acad Sci USA, 2011, 108(17): 7247-7252. DOI:10.1073/pnas.1102117108 |
| [8] |
Doblas VG, Amorim-Silva V, Pose D, et al. The SUD1 gene encodes a putative E3 ubiquitin ligase and is a positive regulator of 3-hydroxy-3-methylglutaryl coenzyme a reductase activity in Arabidopsis[J]. Plant Cell, 2013, 25(2): 728-743. DOI:10.1105/tpc.112.108696 |
| [9] |
杨正婷, 刘建祥. 植物内质网胁迫应答研究进展[J]. 生物技术通报, 2016, 32(10): 84-96. |
| [10] |
Kanehara KS, Kawaguchi, Ng DT. The EDEM and Yos9p families of lectin-like ERAD factors[J]. Semin Cell Dev Biol, 2007, 18(6): 743-750. DOI:10.1016/j.semcdb.2007.09.007 |
| [11] |
Clerc S, Hirsch C, Oggier DM, et al. Htm1 protein generates the N-glycan signal for glycoprotein degradation in the endoplasmic reticulum[J]. J Cell Biol, 2009, 184(1): 159-172. DOI:10.1083/jcb.200809198 |
| [12] |
Hammond C, Braakman I, Helenius A. Role of N-linked oligosaccharide recognition, glucose trimming, and calnexin in glycoprotein folding and quality control[J]. Proc Natl Acad Sci USA, 1994, 91(3): 913-917. DOI:10.1073/pnas.91.3.913 |
| [13] |
Parodi AJ. Protein glucosylation and its role in protein folding[J]. Annu Rev Biochem, 2000, 69: 69-93. DOI:10.1146/annurev.biochem.69.1.69 |
| [14] |
Jakob CA, Burda P, Roth J, et al. Degradation of misfolded endoplasmic reticulum glycoproteins in Saccharomyces cerevisiae is determined by a specific oligosaccharide structure[J]. J Cell Biol, 1998, 142(5): 1223-1233. DOI:10.1083/jcb.142.5.1223 |
| [15] |
Oda Y, Hosokawa N, Wada I, et al. EDEM as an acceptor of terminally misfolded glycoproteins released from calnexin[J]. Science, 2003, 299(5611): 1394-1397. DOI:10.1126/science.1079181 |
| [16] |
Vu KV, Nguyen NT, Jeong CY, et al. Systematic deletion of the ER lectin chaperone genes reveals their roles in vegetative growth and male gametophyte development in Arabidopsis[J]. Plant J, 2017, 89(5): 972-983. DOI:10.1111/tpj.2017.89.issue-5 |
| [17] |
Noguchi T, Fujioka S, Choe S, et al. Brassinosteroid-insensitive dwarf mutants of Arabidopsis accumulate brassinosteroids[J]. Plant Physiol, 1999, 121(3): 743-752. DOI:10.1104/pp.121.3.743 |
| [18] |
Jin H, Hong Z, Su W, et al. A plant-specific calreticulin is a key retention factor for a defective brassinosteroid receptor in the endoplasmic reticulum[J]. Proc Natl Acad Sci USA, 2009, 106(32): 13612-13617. DOI:10.1073/pnas.0906144106 |
| [19] |
Blanco-Herrera F, Moreno AA, Tapia R, et al. The UDP-glucose:glycoprotein glucosyltransferase(UGGT), a key enzyme in ER quality control, plays a significant role in plant growth as well as biotic and abiotic stress in Arabidopsis thaliana[J]. BMC Plant Biol, 2015, 15: 127. DOI:10.1186/s12870-015-0525-2 |
| [20] |
Liebminger E, Huttner S, Vavra U, et al. Class I alpha-mannosidases are required for N-glycan processing and root development in Arabidopsis thaliana[J]. Plant Cell, 2009, 21(12): 3850-3867. DOI:10.1105/tpc.109.072363 |
| [21] |
Huttner S, Veit C, Vavra U, et al. Arabidopsis class I alpha-manno-sidases MNS4 and MNS5 are involved in endoplasmic reticulum-associated degradation of misfolded glycoproteins[J]. Plant Cell, 2014, 26(4): 1712-1728. DOI:10.1105/tpc.114.123216 |
| [22] |
Gething MJ. Role and regulation of the ER chaperone BiP[J]. Semin Cell Dev Biol, 1999, 10(5): 465-472. DOI:10.1006/scdb.1999.0318 |
| [23] |
Howell SH. Endoplasmic reticulum stress responses in plants[J]. Annu Rev Plant Biol, 2013, 64: 477-499. DOI:10.1146/annurev-arplant-050312-120053 |
| [24] |
Chakrabarti A, Chen AW, Varner JD. A review of the mammalian unfolded protein response[J]. Biotechnol Bioeng, 2011, 108(12): 2777-2793. DOI:10.1002/bit.v108.12 |
| [25] |
Sun SY, Shi GJ, Sha HB, et al. IRE1 alpha is an endogenous substrate of endoplasmic-reticulum-associated degradation[J]. Nature Cell Biology, 2015, 17(12): 1546-1555. DOI:10.1038/ncb3266 |
| [26] |
Chen X, Shen J, Prywes R. The luminal domain of ATF6 senses endoplasmic reticulum(ER)stress and causes translocation of ATF6 from the ER to the Golgi[J]. J Biol Chem, 2002, 277(15): 13045-13052. DOI:10.1074/jbc.M110636200 |
| [27] |
Kebache S, Cardin E, Nguyen DT, et al. Nck-1 antagonizes the endoplasmic reticulum stress-induced inhibition of translation[J]. J Biol Chem, 2004, 279(10): 9662-9671. DOI:10.1074/jbc.M310535200 |
| [28] |
Nagashima Y, Mishiba K, Suzuki E, et al. Arabidopsis IRE1 catalyses unconventional splicing of bZIP60 mRNA to produce the active transcription factor[J]. Sci Rep, 2011, 1: 29. DOI:10.1038/srep00029 |
| [29] |
Harding HP, Calfon M, Urano F, et al. Transcriptional and translational control in the Mammalian unfolded protein response[J]. Annu Rev Cell Dev Biol, 2002, 18: 575-599. DOI:10.1146/annurev.cellbio.18.011402.160624 |
| [30] |
Kaufman RJ, Scheuner D, Schroder M, et al. The unfolded protein response in nutrient sensing and differentiation[J]. Nat Rev Mol Cell Biol, 2002, 3(6): 411-421. DOI:10.1038/nrm829 |
| [31] |
Kimata Y, Kimata YI, Shimizu Y, et al. Genetic evidence for a role of BiP/Kar2 that regulates Ire1 in response to accumulation of unfolded proteins[J]. Mol Biol Cell, 2003, 14(6): 2559-2569. DOI:10.1091/mbc.E02-11-0708 |
| [32] |
Srivastava R, Chen YN, Deng Y, et al. Elements proximal to and within the transmembrane domain mediate the organelle-to-organelle movement of bZIP28 under ER stress conditions[J]. Plant Journal, 2012, 70(6): 1033-1042. DOI:10.1111/tpj.2012.70.issue-6 |
| [33] |
Sun L, Lu SJ, Zhang SS, et al. The Lumen-facing domain is important for the biological function and organelle-to-organelle movement of bZIP28 during ER stress in Arabidopsis[J]. Mol Plant, 2013, 6(5): 1605-1615. DOI:10.1093/mp/sst059 |
| [34] |
Liu JX, Srivastava R., Che P, et al. An endoplasmic reticulum stress response in Arabidopsis is mediated by proteolytic processing and nuclear relocation of a membrane-associated transcription factor, bZIP28[J]. Plant Cell, 2007, 19(12): 4111-4119. DOI:10.1105/tpc.106.050021 |
| [35] |
Che P, Bussell JD, Zhou W, et al. Signaling from the endoplasmic reticulum activates brassinosteroid signaling and promotes acclimation to stress in Arabidopsis[J]. Sci Signal, 2010, 3(141): ra69. |
| [36] |
Liu JX, Howell SH. Managing the protein folding demands in the endoplasmic reticulum of plants[J]. New Phytol, 2016, 211(2): 418-428. DOI:10.1111/nph.13915 |
| [37] |
Gao H, Brandizzi F, Benning C, et al. A membrane-tethered transcription factor defines a branch of the heat stress response in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2008, 105(42): 16398-16403. DOI:10.1073/pnas.0808463105 |
| [38] |
Liu JX, Howell SH. Endoplasmic reticulum protein quality control and its relationship to environmental stress responses in plants[J]. Plant Cell, 2010, 22(9): 2930-2942. DOI:10.1105/tpc.110.078154 |
| [39] |
Zhang SS, Yang H, Ding L, et al. Tissue-specific transcriptomics reveals an important role of the unfolded protein response in maintaining fertility upon heat stress in Arabidopsis[J]. Plant Cell, 2017, 29: 1007-1023. DOI:10.1105/tpc.16.00916 |
| [40] |
Yang ZT, Wang MJ, Sun L, et al. The membrane-associated transcription factor NAC089 controls ER-stress-induced programmed cell death in plants[J]. PLoS Genet, 2014, 10(3): e1004243. DOI:10.1371/journal.pgen.1004243 |
| [41] |
Yang ZT, Lu SJ, Wang MJ, et al. A plasma membrane-tethered transcription factor, NAC062/ANAC062/NTL6, mediates the unfolded protein response in Arabidopsis[J]. Plant J, 2014, 79(6): 1033-1043. DOI:10.1111/tpj.12604 |
| [42] |
Seo PJ, Kim MJ, Park JY, et al. Cold activation of a plasma membrane-tethered NAC transcription factor induces a pathogen resistance response in Arabidopsis[J]. Plant J, 2010, 61(4): 661-671. DOI:10.1111/tpj.2010.61.issue-4 |
| [43] |
McCracken AA, Brodsky JL. Assembly of ER-associated protein degradation in vitro:dependence on cytosol, calnexin, and ATP[J]. J Cell Biol, 1996, 132(3): 291-298. DOI:10.1083/jcb.132.3.291 |
| [44] |
Buchberger A. ERQC Autophagy:yet another way to die[J]. Mol Cell, 2014, 54(1): 3-4. DOI:10.1016/j.molcel.2014.03.037 |
| [45] |
Houck SA, Ren HY, Madden VJ, et al. Quality control autophagy degrades soluble erad-resistant conformers of the misfolded membrane protein GnRHR[J]. Mol Cell, 2014, 54(1): 166-179. DOI:10.1016/j.molcel.2014.02.025 |
| [46] |
Vembar SS, Brodsky JL. One step at a time:endoplasmic reticulum-associated degradation[J]. Nat Rev Mol Cell Biol, 2008, 9(12): 944-957. DOI:10.1038/nrm2546 |
| [47] |
Imai Y. Parkin suppresses unfolded protein stress-induced cell death through Its E3 Ubiquitin-protein ligase activity[J]. Journal of Biological Chemistry, 2000, 275(46): 35661-35664. DOI:10.1074/jbc.C000447200 |
| [48] |
Liu LJ, Cui F, Li QL, et al. The endoplasmic reticulum-associated degradation is necessary for plant salt tolerance[J]. Cell Res, 2011, 21(6): 957-969. DOI:10.1038/cr.2010.181 |
| [49] |
Koenig PA, Nicholls PK, Schmidt FI, et al. The E2 ubiquitin-conjugating enzyme UBE2J1 is required for spermiogenesis in mice[J]. Journal of Biological Chemistry, 2014, 289(50): 34490-34502. DOI:10.1074/jbc.M114.604132 |
| [50] |
Sun S, Shi G, Han X, et al. Sel1L is indispensable for mammalian endoplasmic reticulum-associated degradation, endoplasmic reticulum homeostasis, and survival[J]. Proc Natl Acad Sci USA, 2014, 111(5): E582-591. DOI:10.1073/pnas.1318114111 |
| [51] |
Wang YY, Wang WH, Cai JH, et al. Tomato nuclear proteome reveals the involvement of specific E2 ubiquitin-conjugating enzymes in fruit ripening[J]. Genome Biology, 2014, 15(12). |
| [52] |
Ushioda R, Hoseki J, Nagata K. Glycosylation-independent ERAD pathway serves as a backup system under ER stress[J]. Mol Biol Cell, 2013, 24(20): 3155-3163. DOI:10.1091/mbc.E13-03-0138 |
| [53] |
Fu XL, Gao DS. Endoplasmic reticulum proteins quality control and the unfolded protein response:the regulative mechanism of organisms against stress injuries[J]. Biofactors, 2014, 40(6): 569-585. DOI:10.1002/biof.1194 |
| [54] |
Su W, Liu Y, Xia Y, et al. The Arabidopsis homolog of the mammalian OS-9 protein plays a key role in the endoplasmic reticulum-associated degradation of misfolded receptor-like kinases[J]. Mol Plant, 2012, 5(4): 929-940. DOI:10.1093/mp/sss042 |
| [55] |
Huttner S, Veit C, Schoberer J, et al. Unraveling the function of Arabidopsis thaliana OS9 in the endoplasmic reticulum-associated degradation of glycoproteins[J]. Plant Mol Biol, 2012, 79(1-2): 21-33. DOI:10.1007/s11103-012-9891-4 |
| [56] |
Wiertz EJ, Tortorella D, Bogyo M, et al. Sec61-mediated transfer of a membrane protein from the endoplasmic reticulum to the proteasome for destruction[J]. Nature, 1996, 384(6608): 432-438. DOI:10.1038/384432a0 |
| [57] |
Pilon M, Schekman R, Romisch K. Sec61p mediates export of a misfolded secretory protein from the endoplasmic reticulum to the cytosol for degradation[J]. EMBO J, 1997, 16(15): 4540-4548. DOI:10.1093/emboj/16.15.4540 |
| [58] |
Knop M, Finger A, Braun T, et al. Der1, a novel protein specifically required for endoplasmic reticulum degradation in yeast[J]. EMBO J, 1996, 15(4): 753-763. |
| [59] |
Carvalho P, Stanley AM, Rapoport TA. Retrotranslocation of a misfolded luminal ER protein by the ubiquitin-ligase Hrd1p[J]. Cell, 2010, 143(4): 579-591. DOI:10.1016/j.cell.2010.10.028 |
| [60] |
Schoebel S, Mi W, Stein A, et al. Cryo-EM structure of the protein-conducting ERAD channel Hrd1 in complex with Hrd3[J]. Nature, 2017, 548(7667): 352-355. DOI:10.1038/nature23314 |
| [61] |
Chen Q, Zhong YW, Wu YR, et al. HRD1-mediated ERAD tuning of ER-bound E2 is conserved between plants and mammals[J]. Nat Plants, 2016, 2: 16094. DOI:10.1038/nplants.2016.94 |
| [62] |
Li QL, Wei H, Liu LJ, et al. Unfolded protein response activation compensates endoplasmic reticulum-associated degradation deficiency in Arabidopsis[J]. J Integr Plant Biol, 2017, 59(7): 506-521. DOI:10.1111/jipb.v59.7 |
| [63] |
Zhao H, Zhang H, Cui P, et al. The Putative E3 ubiquitin ligase ECERIFERUM9 regulates abscisic acid biosynthesis and response during seed germination and postgermination growth in Arabidopsis[J]. Plant Physiol, 2014, 165(3): 1255-1268. DOI:10.1104/pp.114.239699 |
| [64] |
Li LM, Lu SY, Li RJ. The Arabidopsis endoplasmic reticulum associated degradation pathways are involved in the regulation of heat stress response[J]. Biochem Biophys Res Commun, 2017, 487(2): 362-367. DOI:10.1016/j.bbrc.2017.04.066 |
| [65] |
Merulla J, Fasana E, Solda T, et al. Specificity and regulation of the endoplasmic reticulum-associated degradation machinery[J]. Traffic, 2013, 14(7): 767-777. DOI:10.1111/tra.12068 |
| [66] |
Chen Q, Liu RJ, Wang Q, et al. ERAD Tuning of the HRD1 complex component AtOS9 is modulated by an ER-Bound E2, UBC32[J]. Mol Plant, 2017, 10(6): 891-894. DOI:10.1016/j.molp.2016.12.011 |
| [67] |
Perlmutter DH. Alpha-1-antitrypsin deficiency:importance of proteasomal and autophagic degradative pathways in disposal of liver disease-associated protein aggregates[J]. Annu Rev Med, 2011, 62: 333-345. DOI:10.1146/annurev-med-042409-151920 |
| [68] |
Feng L, et al. Ubiquitin ligase SYVN1/HRD1 facilitates degradation of the SERPINA1 Z variant/alpha-1-antitrypsin Z variant via SQSTM1/p62-dependent selective autophagy[J]. Autophagy, 2017, 13(4): 686-702. DOI:10.1080/15548627.2017.1280207 |
| [69] |
Yang X, Srivastava R, Howell SH, et al. Activation of autophagy by unfolded proteins during endoplasmic reticulum stress[J]. Plant J, 2016, 85(1): 83-95. DOI:10.1111/tpj.13091 |
| [70] |
Liu Y, Burgos JS, Deng Y, et al. Degradation of the endoplasmic reticulum by autophagy during endoplasmic reticulum stress in Arabidopsis[J]. Plant Cell, 2012, 24(11): 4635-4651. DOI:10.1105/tpc.112.101535 |
| [71] |
Gallois P, Makishima T, Hecht V, et al. An Arabidopsis thaliana cDNA complementing a hamster apoptosis suppressor mutant[J]. Plant J, 1997, 11(6): 1325-1331. DOI:10.1046/j.1365-313X.1997.11061325.x |
| [72] |
Danon A, Rotari VI, Gordon A, et al. Ultraviolet-C overexposure induces programmed cell death in Arabidopsis, which is mediated by caspase-like activities and which can be suppressed by caspase inhibitors, p35 and defender against apoptotic death[J]. J Biol Chem, 2004, 279(1): 779-787. DOI:10.1074/jbc.M304468200 |
| [73] |
Lerouxel O, Mouille G, Andeme-Onzighi C, et al. Mutants in DEFECTIVE GLYCOSYLATION, an Arabidopsis homolog of an oligosaccharyltransferase complex subunit, show protein underglycosylation and defects in cell differentiation and growth[J]. Plant J, 2005, 42(4): 455-468. DOI:10.1111/tpj.2005.42.issue-4 |
| [74] |
Koiwa H, Li F, McCully MG, et al. The STT3a subunit isoform of the Arabidopsis oligosaccharyltransferase controls adaptive responses to salt/osmotic stress[J]. Plant Cell, 2003, 15(10): 2273-2284. DOI:10.1105/tpc.013862 |
| [75] |
Su W, Liu Y, Xia Y, et al. Conserved endoplasmic reticulum-associated degradation system to eliminate mutated receptor-like kinases in Arabidopsis[J]. Proc Natl Acad Sci USA, 2011, 108(2): 870-875. DOI:10.1073/pnas.1013251108 |
| [76] |
Lee HK, Cho SK, Son O, et al. Drought stress-induced Rma1H1, a RING membrane-anchor E3 ubiquitin ligase homolog, regulates aquaporin levels via ubiquitination in transgenic plants[J]. Plant Cell, 2013, 21(2): 622-641. |
| [77] |
Cui F, Liu LJ, Zhao QZ, et al. Arabidopsis ubiquitin conjugase UBC32 is an ERAD component that functions in brassinosteroid-mediated salt stress tolerance[J]. Plant Cell, 2012, 24(1): 233-244. DOI:10.1105/tpc.111.093062 |
| [78] |
van Nocker S, Walker JM, Vierstra RD. The Arabidopsis thaliana UBC7/13/14 genes encode a family of multiubiquitin chain-forming E2 enzymes[J]. J Biol Chem, 1996, 271(21): 12150-12158. DOI:10.1074/jbc.271.21.12150 |