2. 内蒙古民族大学农学院,通辽 028042
2. College of Agriculture, Inner Mongolia University for Nationalities, Tongliao 028042
干旱、高盐和低温是植物频繁遭受的主要非生物胁迫,在很大程度上限制植物时空分布,造成作物产量降低和品质下降,严重时甚至绝收[1-3]。植物在长期进化过程中形成了一系列应答保护机制去忍受或适应各种极端环境条件[3-4]。通过特异的信号传导通路感知非生物胁迫,植物启动相关胁迫基因的表达,调节细胞内的各种新陈代谢反应去适应逆境胁迫。其中,蔗糖非发酵相关的蛋白激酶(Su-crose non-fermenting-1-related protein kinase,SnRK)调控靶蛋白的可逆磷酸化修饰在这一系列防御机制过程中发挥着重要的作用。
SnRK2蛋白激酶成员通过ABA非依赖和ABA依赖的两种调节途径参与渗透胁迫信号通路。拟南芥10个SnRK2成员中,有9个(除AtSnRK2.9外)能响应NaCl和甘露醇胁迫,但只有AtSnRK2.2、AtSnRK2.3、AtSnRK2.6、AtSnRK2.7和AtSnRK2.8五个成员参与ABA应答反应[5]。类似地,水稻10个SnRK2成员均能响应NaCl胁迫,但仅有3个成员(SAPK8、SAPK9和SAPK10)能应答ABA胁迫[6]。现在对于SnRK2非依赖ABA调控通路的研究还不太深入,而对ABA依赖信号通路的研究已经取得一定进展。高盐、干旱等逆境胁迫能导致植物内源激素ABA大量合成,植物细胞膜上的含有START结构域的受体(PYR/PYL/RCAR)能感受ABA含量的变化[7]。细胞内缺少ABA时,PP2C会抑制依赖ABA调控通路中SnRK2蛋白激酶活性; 相反,细胞产生ABA时,PP2C的活性则被PYR/PYL/RCAR受体与ABA结合形成的复合体所抑制,SnRK2蛋白激酶得以释放[7-8]。所以,SnRK2可通过ABA非依赖通路直接作用于相关基因,开启一系列胁迫响应基因表达; 也可通过ABA依赖通路调节下游底物的活性来激活相关胁迫响应基因的表达。SnRK2作用的底物至少有58个,涉及表型遗传调控、RNA拼接、离子通道、ROS稳定和叶绿体过程等多种生物过程[9]。SnRK2可通过SnRK2-AREB/ABF调控通路引发一系列ABA应答基因的转录。ABA应答基因的启动子上含有多个保守的响应ABA的顺式作用元件ABRE,SnRK2蛋白激酶可以磷酸化转录因子如ABRE/AREB/bZIP,活性被磷酸化调节的反式因子随后与ABRE顺式作用元件结合,启动ABA响应基因的表达[10, 11]。
菘蓝(Isatis tinctoria L.)作为一种重要的中药资源,其有效成分是它的次生代谢产物靛玉红。由于非生物胁迫会导致植物体内产生大量的次生代谢产物,所以靛玉红生成和累积与菘蓝忍受或适应非生物胁迫的分子应答机制存在某种相关性。菘蓝具有很强的抵御环境胁迫的能力,包含众多的优良性状及抗逆基因源。但是,相对于其它十字花科蔬菜,对菘蓝抵抗逆境胁迫的研究较少,到目前为止,还未见有对SnRK2蛋白激酶成员的研究与报道。本研究基于同源克隆的方法,从菘蓝中分离了一个SnRK2成员,对其基因结构、理化性质、系统发生等进行了生物信息学分析。同时,利用实时定量PCR分析了菘蓝SnRK2基因的组织表达模式和胁迫表达模式。本研究期望对菘蓝SnRK2基因的抗逆功能研究提供理论依据,为揭示菘蓝抗逆机制以及有效成分的积累奠定理论基础。
1 材料与方法 1.1 材料本研究所用的菘蓝种子购于迈格威农业开发有限公司。将菘蓝种子用70%乙醇处理杀菌,用水漂洗几次后播种到土壤中,培养温度为26℃,光照强度2 000 Lx,光照16 h/d。选取30 d苗龄的菘蓝种子幼苗进行高盐胁迫处理(200 mmol/L NaCl)、干旱胁迫处理(10% PEG6000)和ABA胁迫处理(100 μmol/L ABA),以无离子水处理的菘蓝幼苗作为对照。收集对照幼苗的全株用于基因克隆。分别收集对照幼苗的根、茎和叶组织用作基因组织表达模式分析。分别收取胁迫处理前(0 h)和胁迫处理后1 h、3 h、6 h、12 h、24 h、48 h和72 h的菘蓝叶片用于基因胁迫表达模式分析。样品经液氮速冻后,置于-70℃冰箱保存,用于总RNA制备。
1.2 方法 1.2.1 ItSnRK2.1基因克隆以拟南芥AtSnRK2.1 mRNA序列(NM _120946.5)作为检索序列,进行Nucleotide BLAST,下载以下7条代表性序列:At-SnRK2.1(NM_120946.5)、AlSRK2G(XM_0028713-03.2)、CsSRK2G(XM_010424744.2)、BrSRK2G(X-M_009132845.2)、BoSRK2G(XM_013773862.1)、SRK2G(XM_018591452.1)和BnSRK2G(XM_013-824876.1),多序列比对后,在保守区域设计一对简并引物(5'-GGRTYTCACCAKCTCNTCTTCTTAT-3'和5'-TAGTTTACMGCBTWTGGCTTTGACA-3')。总RNA提取按照TaKaRa公司MiniBEST Plant RNA Extraction Kit说明书进行。第一链cDNA合成:在冰浴的RNase Free离心管中加入3 μg总RNA,1 μL Random 6 mers(50 μmol/L),1 μL dNTP Mixture(10 mmol/L),加RNase free ddH2O至10 μL,65℃保温5 min,迅速冰浴5 min,短暂离心后,向管中加入4 μL 5×PrimeScript Buffer,0.5 μL RNase Inhibitor(40 U/μL),1 μL PrimeScript RTase(200 U/μL),加RNase free ddH2O至20 μL,混匀后,30℃保温10 min,42℃ 30 min,95℃保温5 min后迅速至冰上冷却。以第一链cDNA为模板进行PCR扩增。扩增体系为:2 μL 1st-Strand cDNA溶液,4 μL dNTP Mixture(各2.5 mmol/L),1 μL上游兼并引物(10 μmol/L),1 μL下游兼并引物(10 μmol/L),10 μL 5×Prime STAR Buffer(Mg2+ Plus),0.5 μL Prime STAR HS DNA Polymerase(2.5 U/μL),31.5 μL ddH2O。扩增程序为:95℃ 4 min; 95℃ 30 s,55℃ 30 s,72℃ 90 s,32个循环; 72℃ 10 min。PCR产物经回收、末端加A和纯化后克隆至T-Vector pMD20(TaKaRa公司)上进行测序。
1.2.2 ItSnRK2.1基因的生物信息学分析基因的编码区的预测、序列提交、蛋白质的理化性质分析,分别采用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)、Bankit(https://www.ncbi.nlm.nih.gov/WebSub/?tool=genbank)和ProtParam tool(http://web.expasy.org/protparam/)在线程序完成。利用ScanProsite(http://prosite.expasy.org/scanprosite/)、TMpred Server(http://www.ch.embnet.org/software/TMPRED_form.html)、NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)、ProtScale(http://web.expasy.org/protscale/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)、SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)在线程序分别对ItSnRK2.1蛋白进行保守功能域、跨膜区、磷酸化位点、亲水性/疏水性、二级结构和信号肽分析预测。Blastp搜索与ItSnRK2.1同源的蛋白序列,基于MEGA5.0软件以邻近法完成蛋白系统发育树构建,利用GENEDOC软件生成蛋白多序列比对。基因表达水平采用Miura等[12]方法进行分析。
1.2.3 ItSnRK2.1基因的实时定量RT-PCR检测利用TaKaRa公司PrimeScriptTM RT reagent Kit(Perfect Real Time)合成cDNA第一链。
参照TaKaRa公司SYBR® Fast qPCR Mix试剂盒说明书进行实时定量RT-PCR检测ItSnRK2.1基因表达模式。ItSnRK2.1基因特异性引物为5'-GGAGTA-TGCTTCTGGAGGAG-3'和5'-GAGCAGGGCTCCCAT-CAAGC-3'。用Itactin(AY870652)作为持家基因,扩增引物为5'-ATTCCGTTGCCCTGAAATAC-3'和5'-CTTTGCTCATACGGTCAGCG-3'。扩增程序为94℃ 30 s; 94℃ 5 s,60℃ 10 s,40个循环。
2 结果 2.1 菘蓝ItSnRK2.1基因的克隆菘蓝幼苗的总RNA提取结果(图 1-A)显示,所提取的总RNA 5S、18S和28S条带较完整和清晰,可以用于合成cDNA第一链的模板。以第一链cDNA为模板进行RT-PCR反应,扩增产物经琼脂糖凝胶电泳显示,产生1 300 bp左右的的特异性片段(图 1-B)。扩增产物经测序得到1 305 bp cDNA序列。ORF Finder预测结果(图 2)显示,1305 bp的cDNA具有一个112 bp的3'-非编码区(3'-UTR),一个1 071 bp的开放阅读框(ORF)和一个122 bp的5'-非编码区(5'-UTR),编码356个氨基酸。将该cDNA全长序列命名为ItSnRK2.1,提交至Genbank,获得登录号为MF423122。
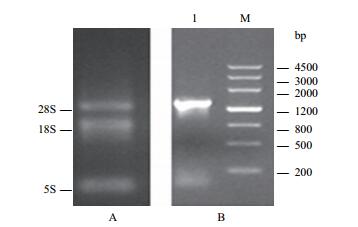
|
| 图 1 总RNA的提取和ItSnRK2.1基因的RT-PCR扩增 |

|
| 图 2 ItSnRK2.1基因cDNA序列及其推导的氨基酸序列 |
ItSnRK2.1全长cDNA编码356 aa,理论等电点为5.64,理论分子量为40.6 kD; ItSnRK2.1蛋白中Glu、Lys、Ser和Leu含量较高,分别为10.4%、7.9%、7.9%和7.6%;ItSnRK2.1蛋白中带负电荷的残基(Asp+Glu)总数为58个,带正荷的残基(Arg+Lys)总数为47个; ItSnRK2.1蛋白分子式为C1791H2823N489O555S15,平均疏水性(GRAVY)为-0.515,脂肪氨基酸指数(AI)为82.16。ItSnRK2.1蛋白的不稳定指数为44.94,属于不稳定蛋白,与拟南芥AtSnRK2.1蛋白激酶的氨基酸组成极为相似。
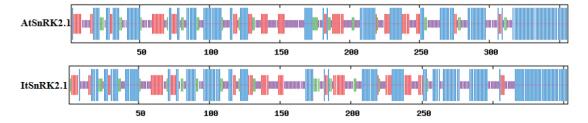
2.3 ItSnRK2.1蛋白激酶的多序列比对和进化分析为了明确ItSnRK2.1蛋白激酶的结构特征,对推导的ItSnRK2.1蛋白激酶的氨基酸序列同拟南芥、水稻、大麦、玉米、水稻、燕麦和二穗短柄草的SnRK2氨基酸序列进行多序列比对和ScanProsite在线预测。结果显示,ItSnRK2.1蛋白包含一个由酸性氨基酸序列组成的高度特异的C端激酶调控域及一个高度保守的N端蛋白激酶催化域(图 3-A)。ItSnRK2.1蛋白高度保守的N端催化域(4-260 aa)具有一个24 aa的ATP结合位点(图 3-B,黑框Ⅰ)和一个13 aa的丝氨酸/苏氨酸蛋白激酶激活位点(图 3-B,黑框Ⅱ)。C端包含一个34 aa的结构域Ⅰ(Do-main Ⅰ)(图 3-B,黑框Ⅲ)。但是,同AtSnRK2.1蛋白激酶相同,C端缺少44 aa的结构域Ⅱ(Domain Ⅱ)(图 3-B,黑框Ⅳ)。

|
| 图 3 ItSnRK2.1蛋白激酶保守结构预测和多序列比对 A:ItSnRK2.1蛋白激酶结构;B:ItSnRK2.1同相关物种的同源氨基酸序列比对 |
多序列比对结果(图 3-B)显示,ItSnRK2.1蛋白与其它8个SnRK2成员高度同源。ItSnRK2.1氨基酸序列与拟南芥AtSnRK2.1(NP_196476.1)、水稻OsSAPK7(BAD18003.1)、大麦HvPKABA1(BAB61735.1)、拟南芥AtSnRK2.6(NP_567945.1)、玉米ZmSnRK2.1(ACG50005.1)、水稻OsSAPK2(BAD17998.1)、燕麦TaSnRK2.8(AKZ18234.1)和二穗短柄草BdSAPK8(AJR27164.1)氨基酸序列的相似性分别为95.18%、74.16%、66.67%、64.33%、64.91%、65.50%、62.92%和62.90%。
为了明确ItSnRK2.1与其它nRK2蛋白激酶的进化关系,用菘蓝、水稻、玉米、燕麦等30个SnRK2成员的氨基酸序列构建进化树。结果(图 4)显示,30个SnRK2.1蛋白激酶可以分为3组,菘蓝ItSnRK2.1蛋白激酶同拟南芥AtSnRK2.1、马铃薯StSnRK2.4、玉米ZmSnRK2.6、水稻OsSAPK7等12个SnRK2成员聚为一组,说明这些基因在进化上高度保守,可能在抗逆功能上具有相似性。

|
| 图 4 ItSnRK2.1蛋白聚类分析 |
氨基酸的亲水性和疏水性是蛋白质固有的特征,氨基酸的亲水性和疏水性之间的平衡决定着蛋白质的结构特征,而氨基酸内的疏水作用在很大程度上决定着蛋白质结构的稳定性。所以,分析和预测蛋白质的亲水性和疏水性可以为蛋白质结构分析和功能预测提供重要的理论依据。氨基酸的亲水性和疏水性分析结果(图 5)显示,ItSnRK2.1蛋白激酶最大疏水性氨基酸值为2.189,最小疏水性氨基酸值为-3.300,存在多个明显的亲水区域,而且组成ItSnRK2.1蛋白激酶的大部分的氨基酸属于亲水性氨基酸。据此推断,菘蓝ItSnRK2.1蛋白激酶属于亲水性蛋白。
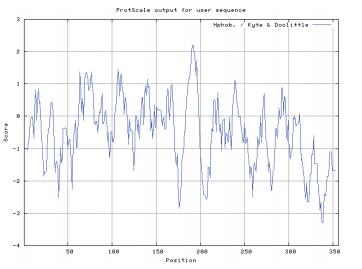
|
| 图 5 ItSnRK2.1蛋白激酶亲水性/疏水性分析 |
蛋白质二级结构预测结果(图 6)显示,菘蓝ItSnRK2.1蛋白激酶的二级结构具有151个α-螺旋(Alpha helix)、109个无规则卷曲(Random coil)、66个延伸链(Extended strand)和30个β-转角(Beta turn)。与拟南芥AtSnRK2.1蛋白激酶的155个α-螺旋(Alpha helix)、109个无规则卷曲(Random coil)、62个延伸链(Extended strand)和27个β-转角(Beta turn)极为相似,暗示菘蓝ItSnRK2.1蛋白激酶可能与拟南芥AtSnRK2.1蛋白激酶可能具有相似的功能。
|
| 图 6 ItSnRK2.1同AtSnRK2E二级结构比较 β-转角、延伸链、α-螺旋和自由卷曲分别用依次增多的竖线表示 |
信号肽是由长度5-30个N-末端氨基酸组成的短肽链(有时不一定在N端),其功能是引导新合成的蛋白质向分泌通路转移。利用SignalP 4.1 Server对ItSnRK2.1进行预测。结果(图 7)显示,可能没有信号肽区域存在于ItSnRK2.1蛋白激酶中。
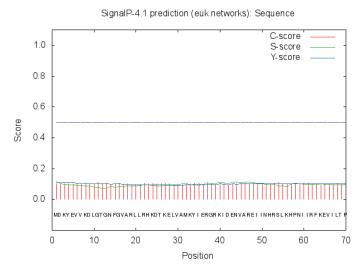
|
| 图 7 ItSnRK2.1蛋白信号肽的预测 |
为了掌握菘蓝ItSnRK2.1蛋白的表达和作用位置,利用在线预测软件PSORT Prediction对ItSnRK2.1蛋白进行亚细胞定位。预测结果表明,ItSnRK2.1蛋白的可能定位在细胞质(Cytoplasm)、微体(Microbody)、线粒体基质空间(Mitochondrial matrix space)和溶酶体(Lysosome)上。这4个位置的决定性系数(Certainty)分别为0.450、0.300、0.100和0.100,所以推测ItSnRK2.1蛋白最可能定位的位置是在细胞质上。
2.8 ItSnRK2.1蛋白激酶的的跨膜区分析α-螺旋结构通常作为蛋白质序列中跨越细胞膜的区域,构成跨膜区的氨基酸大部分必须是疏水性氨基酸,这样才能使膜蛋白顺利通过细胞膜的磷脂双分子层。TMpred预测结果(图 8)显示,可能有3个跨膜结构域存在于ItSnRK2.1蛋白中。由外向内的跨膜结构域有一个,位于183-200 aa,长度为18 aa,分数为892。由内向外的跨膜结构域有二个:一个位于98-121 aa,长度为24 aa,分数为182;另一个位于183-200 aa,长度为18 aa,分数为1 109。所以,ItSnRK2.1蛋白激酶可能存在2个显著跨膜结构域(分数大于500)。
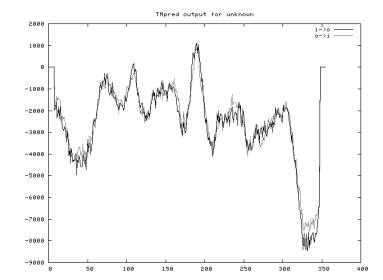
|
| 图 8 蛋白跨膜结构域预测 |
大部分前体蛋白需要经过翻译后修饰才能成为有功能的蛋白质,而蛋白质翻译后修饰中最重要、最常见的方式之一是蛋白质的磷酸化。磷酸化位点预测结果(图 9)显示,ItSnRK2.1蛋白激酶可能有8个酪氨酸(Tyr),11个苏氨酸(Thr),23个丝氨酸(Ser)成为磷酸化位点。拟南芥AtSnRK2.1蛋白激酶可能有7个酪氨酸(Tyr),10个苏氨酸(Thr),21个丝氨酸(Ser)成为磷酸化位点。二者可能的磷酸化位点的数目和位置极为相似,它们可能具有类似的磷酸化过程。
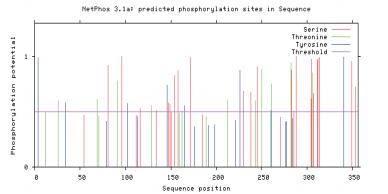
|
| 图 9 ItSnRK2.1蛋白磷酸化位点预测 |
为了探明ItSnRK2.1基因在雪菜根、茎和叶组织中的表达模式,利用实时定量RT-PCR对ItSnRK2.1基因的转录情况进行了分析。检测结果(图 10)显示,在3个供试组织中都能检测到ItSnRK2.1基因的表达,但基因表达水平在不同组织中存在一定差异。ItSnRK2.1基因表达量最高的组织是根,其次为叶片,表达量最低的组织是茎,说明ItSnRK2.1基因的表达具有组织特异性。
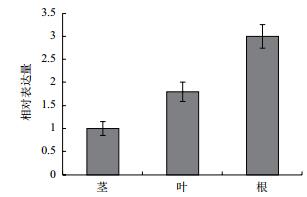
|
| 图 10 ItSnRK2.1基因的的组织表达分析 |
实时定量RT-PCR检测结果(图 11)显示,ItSnRK2.1基因积极参与高盐和干旱胁迫应答反应。当菘蓝幼苗受到高盐胁迫时,ItSnRK2.1基因表达量迅速上升,在胁迫3 h时,表达量达到最高,为处理前(0 h)的4.8倍,随后表达量逐渐降低,72 h时表达量仅为对照的1.3倍(图 11-A)。当菘蓝幼苗遭受PEG胁迫时,ItSnRK2.1基因表达量也呈现出先升高、后下降的表达规律,但出现两个波峰值,分别出现在3 h时和12 h时,表达量分别是对照的2.2和2.3倍(图 11-B)。当用ABA处理菘蓝幼苗时,各时间段ItSnRK2.1基因表达水平无明显差异(图 11-C),类似于去离子水(对照)处理(图 11-D)。
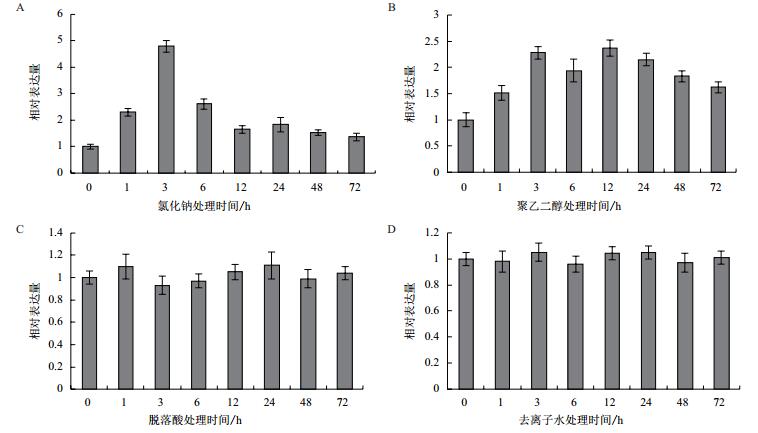
|
| 图 11 ItSnRK2.1基因的的胁迫表达分析 |
SnRK2基因编码的蛋白质包含一段27 aa的保守序列(DFGYSKSSVLHSQPKSTVGTPAYIAPE),即丝氨酸/苏氨酸蛋白激酶激活环。对拟南芥AtSn-RK2.6和水稻SAPK2研究结果表明,丝氨酸/苏氨酸蛋白激酶激活环上的3个丝氨酸/苏氨酸残基(下划线处)对SnRK2蛋白激酶的酶活起到关键的作用[6, 13]。用天冬氨酸替代这3个氨基酸中的前两个后,AtSnRK2.6蛋白激酶活性远远低于对照,只能使底物蛋白轻微磷酸化。同样水稻SAPK2丝氨酸/苏氨酸蛋白激酶激活环上的3个丝氨酸/苏氨酸残基全部、两两或逐一被天冬氨酸取代后,无法检测到SAPK2蛋白激酶活性。多序列比对结果显示,菘蓝ItSnRK2.1蛋白激酶激活环(DFGYSKSSILHSTPKSTVGTPAYIAPE)与拟南芥AtSnRK2.6的该序列有两个氨基酸差异(着重号处氨基酸,I取代了V,T取代了Q),分享92.6%的相似性,但是二者3个丝氨酸/苏氨酸残基完全一致,推测菘蓝ItSnRK2.1蛋白激酶与拟南芥AtSnRK2.1蛋白激酶具有相似的磷酸化作用。
SnRK2家族基因编码植物体内特有的参与多种信号传导的一类Ser/Thr蛋白激酶,是影响植物抗逆境胁迫能力的关键调控基因,其在不同胁迫下和不同组织中的表达具有一定的差异性。实时定量和半定量分析结果显示,烟草NtSnRK2.1基因在根部有最高表达水平,叶片中的次之,茎部的表达量最低[14]。Northern blot检测结果表明,在ABA、NaCl和甘露醇胁迫下,水稻基因组中10个SnRK2成员(SAPK1-SAPK10)在根、叶鞘和叶片不同组织中的表达存在差异性。[6]本研究发现ItSnRK2.1基因的表达也具有一定的组织差异性,与水稻和烟草中的组织表达模式基本一致。
多序列比对结果显示,不同SnRK2蛋白激酶成员在N末端高度保守,而在C末端高度特异。SnRK2家族成员在C末端包含一个酸性补丁(D/E),一般富含天冬氨酸(D)或谷氨酸(E),依据这一特点将SnRK2成员分为二组:SnRK2a(酸性补丁中富含D)和SnRK2b(酸性补丁中富含E)。[15]序列分析显示,菘蓝ItSnRK2.1蛋白的C末端也包含一个谷氨酸(E)的酸性补丁,因此,ItSnRK2.1属于SnRK2b家族中的一员。基于C末端结构组成和系统发生,又可将SnRK2家族成员分为3组:Group Ⅰ-Group Ⅲ[16]。进化树构建结果表明,菘蓝ItSnRK2.1被归为Group Ⅰ成员,这与Group Ⅱ和Group Ⅲ属于SnRK2a,而Group Ⅰ属于SnRK2b的研究结果一致。SnRK2蛋白激酶家族的C末端主要包含两个结构域:一个是Domain Ⅰ,该区域是SnRK2蛋白激酶响应非生物胁迫的必要元件,Group Ⅰ-Group Ⅲ成员都具备这一结构域; 另一个是Domain Ⅱ,为Group Ⅲ成员所特有的一段序列,是响应ABA应答不可缺少元件,在与PP2C、ABI1等磷酸酶的互作及响应ABA应答等方面发挥着重要的作用[6, 13, 17-18]。除SnRK2.9外,拟南芥其余9个SnRK2成员都响应高渗胁迫,同时研究发现Group Ⅰ成员不响应ABA胁迫,Group Ⅱ成员对ABA胁迫不响应或具有微弱的响应,Group Ⅲ成员强烈响应ABA胁迫[5, 10, 16]。10个水稻SnRK2成员也都响应高渗胁迫,其中Group Ⅰ和Group Ⅱ中的3个成员(SAPK8、SAPK9和SAPK10)也响应ABA胁迫[6]。这些研究结果与SnRK2家族成员的高度保守的N端催化域和特异的C端激酶调控域是相关的,SnRK2家族成员在功能上的相似性与差异性、响应胁迫的相似性与差异性等都可能归因于SnRK2s序列的一致性和多样性。与拟南芥AtSnRK2.1和水稻SAPK7的研究结果及其相似,菘蓝ItSnRK2.1蛋白激酶的C末端仅含有一个34 aa的Domain Ⅰ,缺少44 aa的Domain Ⅱ,并且响应高渗胁迫但对ABA胁迫不敏感,暗示菘蓝ItSnRK2.1基因与拟南芥AtSnRK2.1和水稻SAPK7有类似的功能,在参与非生物胁迫过程中很可能也是不依赖ABA调控通路的。
4 结论从菘蓝中克隆了1个编码蔗糖非发酵相关的蛋白激酶的基因ItSnRK2.1,包含一个1 071 bp的完整开放阅读框,编码356个氨基酸,理论等电点为5.64,理论分子量为40.6 kD。
ItSnRK2.1蛋白激酶包含一个ATP结合位点和一个丝氨酸/苏氨酸酶活结构域。预测了ItSnRK2.1蛋白激酶的亚细胞定位、亲水性/疏水性、信号肽、二级结构、磷酸化位点和跨膜域。ItSnRK2.1氨基酸序列与其它SnRK2成员分享较高的相似性,同AtSnRK2.1、AtSnRK2.5和StSnRK2.6蛋白亲缘关系最近,处在同一进化分枝。ItSnRK2.1基因表达具有组织特异性,积极参与高盐和干旱胁迫应答反应,但对ABA胁迫不敏感,表明该基因可能与植物抗逆境胁迫有关,且不依赖ABA调控通路。
| [1] |
Duan K, Sun G, Zhang Y, et al. Impact of air pollution induced climate change on water availability and ecosystem productivity in the conterminous United States[J]. Climatic Change, 2017, 140(2): 259-272. DOI:10.1007/s10584-016-1850-7 |
| [2] |
Man L, Xiang D, Wang L, et al. Stress-responsive gene RsICE1 from Raphanus sativus increases cold tolerance in rice[J]. Protoplasma, 2017, 254(2): 945-956. DOI:10.1007/s00709-016-1004-9 |
| [3] |
Ye Y, Ding Y, Jiang Q, Wang F, et al. The role of receptor-like protein kinases(RLKs)in abiotic stress response in plants[J]. Plant Cell Reports, 2017, 36(2): 235-242. DOI:10.1007/s00299-016-2084-x |
| [4] |
Forni C, Duca D, Glick BR. Mechanisms of plant response to salt and drought stress and their alteration by rhizobacteria[J]. Plant and Soil, 2017, 410(1-2): 335-356. DOI:10.1007/s11104-016-3007-x |
| [5] |
Boudsocq M, Barbier-Brygoo H, Laurière C. Identification of nine sucrose nonfermenting 1-related protein kinases 2 activated by hyperosmotic and saline stresses in Arabidopsis thaliana[J]. J Biol Chem, 2004, 279(40): 41758-41766. DOI:10.1074/jbc.M405259200 |
| [6] |
Kobayashi Y, Yamamoto S, Minami H, et al. Differential activation of the rice sucrose nonfermenting1-related protein kinase2 family by hyperosmotic stress and abscisic acid[J]. The Plant Cell, 2004, 16(5): 1163-1177. DOI:10.1105/tpc.019943 |
| [7] |
Park SY, Fung P, Nishimura N, et al. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins[J]. Science, 2009, 324(5930): 1068-1071. |
| [8] |
Santiago J, Dupeux F, Betz K, et al. Structural insights into PYR/PYL/RCAR ABA receptors and PP2Cs[J]. Plant Sci, 2012, 182: 3-11. DOI:10.1016/j.plantsci.2010.11.014 |
| [9] |
Wang P, Xue L, Batelli G, et al. Quantitative phosphoproteomics identifies SnRK2 protein kinase substrates and reveals the effectors of abscisic acid action[J]. Proc Natl Acad Sci USA, 2013, 110(27): 11205-11210. DOI:10.1073/pnas.1308974110 |
| [10] |
Fujita Y, Yoshida T, Yamaguchi-Shinozaki K. Pivotal role of the AREB/ABF-SnRK2 pathway in ABRE-mediated transcription in response to osmotic stress in plants[J]. Physiol Plant, 2013, 147(1): 15-27. DOI:10.1111/ppl.2013.147.issue-1 |
| [11] |
Yoshida T, Fujita Y, et al. Four Arabidopsis AREB/ABF transcrip-tion factors function predominantly in gene expression downstream of SnRK2 kinases in abscisic acid signalling in response to osmotic stress[J]. Plant Cell Environ, 2015, 38(1): 35-49. DOI:10.1111/pce.2015.38.issue-1 |
| [12] |
Miura K, Jin JB, Lee J, et al. SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis[J]. The Plant Cell, 2007, 19(4): 1403-1414. DOI:10.1105/tpc.106.048397 |
| [13] |
Belin C, De Franco PO, Bourbousse C, et al. Identification of features regulating OST1 kinase activity and OST1 function in guard cells[J]. Plant Physiology, 2006, 141(4): 1316-1327. DOI:10.1104/pp.106.079327 |
| [14] |
张洪映, 贾宏昉, 张松涛, 等. 烟草NtSnRK2. 1基因的克隆及其在非生物胁迫条件下的表达[J]. 中国烟草学报, 2014, 20(4): 94-100. |
| [15] |
Yoshida R, Hobo T, Ichimura K, et al. ABA-activated SnRK2 protein kinase is required for dehydration stress signaling in Arabidopsis[J]. Plant Cell Physiol, 2002, 43(12): 1473-1483. DOI:10.1093/pcp/pcf188 |
| [16] |
Kulik A, Wawer I, Krzywińska E, et al. SnRK2 protein kinases—key regulators of plant response to abiotic stresses[J]. OMICS, 2011, 15(12): 859-872. DOI:10.1089/omi.2011.0091 |
| [17] |
Yoshida R, Umezawa T, Mizoguchi T, et al. The regulatory domain of SRK2E/OST1/SnRK2. 6 interacts with ABI1 and integrates abscisic acid(ABA)and osmotic stress signals controlling stomatal closure in Arabidopsis[J]. J Biol Chem, 2006, 281(8): 5310-5318. DOI:10.1074/jbc.M509820200 |
| [18] |
王永波, 高世庆, 唐益苗, 等. 植物蔗糖非发酵-1相关蛋白激酶家族研究进展[J]. 生物技术通报, 2011(11): 7-18. |




