2. 西北农林科技大学动物科技学院,杨陵 712100
2. College of Animal Science and Technology, Northwest A & F University, Yangling 712100
基因转录的表观遗传调控主要通过DNA甲基化和组蛋白修饰,组蛋白修饰包括甲基化、乙酰化和磷酸化等。组蛋白特异赖氨酸位点的甲基化修饰参与细胞分化、衰老、组织修复,在肿瘤及代谢性疾病的发生、发展中发挥了重要的作用[1-4]。组蛋白特异赖氨酸位点的甲基化修饰主要是由组蛋白赖氨酸去甲基化酶和组蛋白赖氨酸甲基转移酶来调节的。组蛋白去甲基化酶主要分为LSD1和JumonjiC(JmjC)家族两类。LSD1主要催化一甲基化和二甲基化的H3K4和H3K9去甲基化,JmjC家族的成员能催化一甲基化、二甲基化、三甲基化去甲基化[5]。H3K27的甲基化在基因组中分布广泛,呈现一甲基化、二甲基化及三甲基化3种状态。质谱研究表明,在胚胎干细胞中15%的H3K27呈现单甲基化状态,50%呈现二甲基化状态,15%呈现三甲基化状态[6]。H3K27三甲基化是基因转录抑制的标志,其去甲基化后可以激活基因表达[1, 7],目前共鉴定出JMJD3与UTX两种H3K27去甲基化酶,二者结构相似。本文综述了JMJD3在细胞分化、衰老、胚胎发育及肿瘤发生、发展中的作用及其调节机制,对在肿瘤诊断和治疗方面的应用前景进行展望,旨为今后的研究工作奠定理论基础。
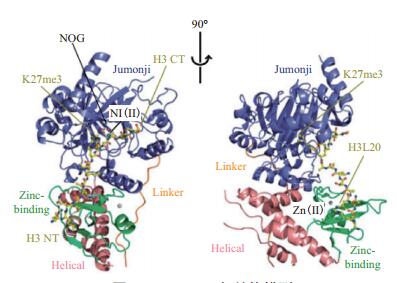
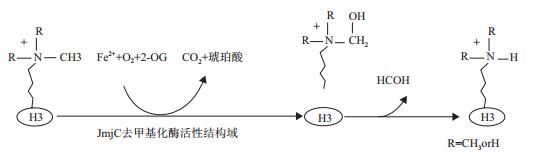
1 JMJD3的结构与催化机制JMJD家族成员的C端和N端都包含一个转录因子家族Jumonji小的特征结构域,分别称为JmjC和JmjN。JmjC是JMJD家族酶活性中心的组成之一,JmjN结构域与转录调节相关,因此该家族又被称为包含JmjC结构域的组蛋白去甲基化酶家族[1]。JMJD家族成员包含一个锌指结构,一个Fe2+离子结合位点和一个含有JmjC结构域的酮戊二酸依赖性的去甲基化酶活性结构域(图 1),属于双加氧酶家族[8]。目前,JmjC结构域的组蛋白赖氨酸去甲基化酶活性的催化机制已比较清楚。JmjC结构域在Fe2+、α-酮戊二酸(2-OG)和O2的参与下,催化组蛋白赖氨酸残基上甲基化氨基生成羟基化中间体,同时产生1分子琥珀酸盐和1分子CO2,随后该中间体生成不稳定的半氨醛,并进一步分解为去甲基化赖氨酸和甲醛[8](图 2)。
|
| 图 1 JMJD3三级结构模型 |
|
| 图 2 JmjC结构域的催化机制 |
在人中,大约有30种蛋白含有JmjC结构域,根据整体的序列比对大致可以分成JARID1、JHDM3、JHDM1、PHF8、JHDM2、UTX/UTY以及仅含JmjC结构域的蛋白质等7个亚家族。一些含有JmjC结构域的JMJD有着不同的底物特异性(表 1)。
JMJD3(Jumonji domain-containing protein 3),又被称作KDM6B,是JMJD家族成员之一,JMJD3是含JmjC结构域的组蛋白去甲基化酶UTX/UTY家族成员之一,与UTX有84%的氨基酸序列相似度。人的JMJD3基因定位于17p13.1,编码1个含1682个氨基酸残基的多肽[9]。JMJD3和UTX均具有组蛋白去甲基化酶活性[10]。大量体内外研究发现,在细胞分化过程中,JMJD3和UTX表达量增加,许多发育相关基因(如HOX、Mashl、SP-B等)的启动子区H3K27me3含量显著减少,从而促进细胞分化;而当JMJD3的表达被抑制后,H3K27me3表达增加,部分分化相关基因(如SP-B、RUNX2、Bhlhb4) 的表达被抑制,造成组织器官发育缺陷[2, 11-12]。这说明JMJD3与H3K27位点的去甲基化密切相关,可能具有H3K27me2/3去甲基化酶活性。
JMJD3具有使非组蛋白去甲基化的去甲基化酶作用。JMJD3能够催化Set7/9介导的肿瘤抑制蛋白RB第810位赖氨酸甲基化位点去甲基化,抑制细胞周期蛋白激酶CDK4与RB蛋白的结合,降低RB蛋白的磷酸化水平,促进细胞衰老[3],该研究成果表明JMJD3也可以催化非组蛋白去甲基化。
此外,JMJD3还可以作为转录因子直接调控靶标基因的转录。植物同源异型结构域锌指蛋白20(PHF20) 是细胞重编程的必要条件,缺乏PHF20的细胞无法去分化形成多能干细胞。Zhao等[13]发现JMJD3可以介导PHF20的泛素化降解失活,从而降低小鼠体细胞重编程为多能干细胞的效率。目前,关于JMJD3的非去甲基化酶作用机制尚不完善,需进一步研究。
2 JMJD3与胚胎发育H3K27me3被看作多能干细胞的标志之一,H3K27的去甲基化可作为基因转录去抑制化的一种重要方式[1, 7]。目前,H3K27的去甲基化酶共鉴定出JMJD3与UTX两种。H3K27的甲基化调节酶在胚胎发育的不同分化阶段呈现动态变化[31-33],通过对不同基因的激活和抑制,调控细胞分化正常进行,从而保证胚胎的正常发育。
2.1 JMJD3与桑葚胚和囊胚形成在猪胚胎发育过程中,H3K27组蛋白甲基转移酶EZH2的表达在桑葚胚期达到高峰,H3K27me3表达较高,随着细胞分化的进行,JMJD3表达增加,H3K27甲基化水平降低,进而激活与细胞发育相关Hox基因表达。在外胚层植入前的囊胚期,EZH2的表达量增高,导致JMJD3和UTX表达降低,H3K27甲基化水平增加[32],促进细胞的分化。Canovas等[33]研究发现JMJD3在牛成熟卵母细胞中有较高的转录水平,受精后JMJD3蛋白含量增加。与此同时,其mRNA水平却降低,这种表达模式说明JMJD3为母体遗传基因。另外,他们在孤雌激活的MⅡ卵母细胞中,干扰JMJD3的表达抑制植入前胚胎H3K27me3的表达,降低囊胚发育的速度。这些结果说明JMJD3在不同动物桑葚胚和囊胚发育时表达水平呈动态变化,促进细胞分化与胚胎发育。
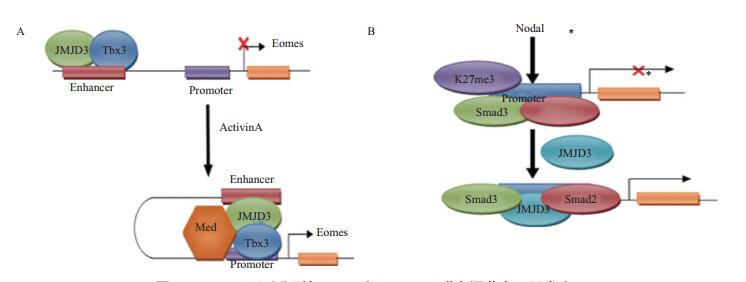
2.2 JMJD3与内胚层形成内胚层主要可以发育为胃肠道、呼吸道、内分泌腺和泌尿系统等组织器官,在胚胎分化发育过程中,JMJD3可以调控内胚层的形成。研究发现,敲除人类胚胎干细胞中的JMJD3和UTX基因后,可以抑制胚胎干细胞向内胚层细胞的分化[24],说明JMJD3和UTX在内胚层发育中具有重要的调控作用,而这一作用可能是通过JMJD3与一些内胚层形成过程中重要的因子(如T-box转录因子、Eomes和Tbx3等)相互作用来实现的。在内胚层分化早期,TBX3与JMJD3结合于Eomes增强子区域,使其增强子靠近启动子,激活Activin A信号通路,促使Eomes转录、表达,从而激活内胚层分化过程中的转录调控因子,促进内胚层分化[34](图 3-A)。在内胚层发育过程中,胚胎转化生长因子Nodal可以激活Smad2/3,进而结合到H3K27me3沉默的靶基因上,然后Smad2/3招募JMJD3从而使H3K27me3去甲基化,最终激活靶基因的表达[35](图 3-B)。在小鼠胚胎肺发育的过程中,敲除JMJD3基因后导致小鼠肺发育不全,从而引起围产期胎儿致死[3, 36],说明JMJD3对肺的健康发育有至关重要的作用。JMJD3表达量的下调能够明显的降低与肺发育相关的关键性基因的表达,特别是表面活化蛋白SP-B的表达。JMJD3通过与转录因子Nkx2.1和表观遗传蛋白Brg1相互作用来调节SP-B启动子区域H3K27甲基化状态,从而调节SP-B的表达,调节肺发育[2]。这些研究发现,表明了JMJD3在内胚层形成,特别是肺的发育过程中具有重要的调节作用。
|
| 图 3 JMJD3可以分别与Tbx3和Smad2/3联合调节内胚层发育 |
JMJD3与中胚层发育也有着重要的关系,在胚胎干细胞分化为内胚层细胞的过程中,JMJD3可以补偿部分UTX的功能[37]。中胚层可以发育为肌肉、脾脏、软骨、骨骼、淋巴结、性腺、淋巴管、皮肤、肾脏、心脏、血管和骨髓等组织器官。在胚胎干细胞中缺乏JMJD3,可以显著的增加中胚层调节因子Brachyury(T-box转录因子家族成员)启动子区H3K27me3的数量,降低Brachyury的表达量,从而导致β-链蛋白补充受损,而β-链蛋白是中胚层分化的首要调节因素[38],缺乏β-链蛋白后将导致无法形成中胚层。
在晚期中胚层分化期间,JMJD3对器官的发育有着至关重要的作用。将胚胎干细胞中JMJD3敲除,可以降低内皮细胞和心肌祖细胞的分化形成[38],使小鼠脾脏发育受损还可影响骨形成。在软骨骨形成过程中,JMJD3可以通过去甲基化酶活性降低调节因子RUNX2启动子区域H3K27me3的含量,激活RUNX2转录活性,从而促进骨形成[11]。在软骨骨化期间,JMJD3在肥大前细胞和肥大软骨细胞中表达较高,将JMJD3敲除后,可以导致骨化速度降低[39],但会增加成脂分化[40]。Huszar等[41]在骨髓间充质干细胞(BMSCs)分化过程中研究发现,miR-146a能抑制JMJD3的表达,是JMJD3的负调控因子。调节miR-146a表达水平,可以改变JMJD3和RUNX2表达水平,从而影响成骨分化。在牙齿发育的过程中,JMJD3可以与骨形态生成蛋白BMP2启动子结合,解除H3K27me3表达抑制,促进牙向分化,敲除JMJD3可以减弱牙向分化[42]。这些研究均表明JMJD3在骨形成中必不可少。
免疫细胞(如B细胞、T细胞和巨噬细胞等)广泛存在于由中胚层发育的骨髓、淋巴结、淋巴管等免疫组织器官中。在免疫细胞分化过程中,JMJD3扮演着重要的角色。JMJD3与B细胞分化有密切的联系,与处于分化阶段的记忆性B细胞相比,JMJD3转录靶基因在生发中心B细胞的表达水平显著增加[43]。在T细胞分化过程中,JMJD3也有着重要的作用。Liu等[44]发现T细胞抗原受体(TCR)信号可以快速诱导JMJD3表达,敲除JMJD3基因可以抑制早期CD4+T细胞向Th17细胞分化,而过表达JMJD3能够恢复Th17细胞的分化缺陷。另外,JMJD3能够直接结合到Th17细胞的特征性转录因子Rorgt的启动子区域,降低H3K27me3水平,激活Rorgt基因的表达。
巨噬细胞可以分为M1和M2两个亚群,M1型具有清除病原体与促炎作用,而M2型具有免疫调节作用和组织修复能力。JMJD3对M1型巨噬细胞没有明显的影响,但在M2型巨噬细胞分化中是必不可少的。在巨噬细胞中JMJD3可以调节IRF4启动子区域结合的组蛋白甲基化水平,调控IRF4的表达,从而调节M2型巨噬细胞相关的表面标记分子[36]。另外,在巨噬细胞中,炎症相关信号的(如脂多糖LPS)刺激能够激活NF-κB信号通路,后NF-κB信号通路关键蛋白与JMJD3启动子区域保守的NF-κB结合位点结合,促进JMJD3基因表达。JMJD3通过对H3K27甲基化水平的调节,影响下游靶基因的转录表达,进而调节巨噬细胞的表型使其对炎症刺激作出相应的反应[45]。
2.4 JMJD3与外胚层发育外胚层的形成同样受到JMJD3的调节。外胚层主要分化发育为神经系统和表皮。在胚胎干细胞分化成神经细胞时需要JMJD3的参与,在神经祖细胞分化的第8天JMJD3表达上调,在第26天时表达水平降低[32]。神经形成主要调控因子有Pax6、Sox1、Nestin等,JMJD3可以直接调控这3个因子的表达[32]。JMJD3也可以调控Shh表达,从而调控神经管的形成[5]。在RA诱导P19细胞向神经细胞方向分化过程中,JMJD3被募集到神经分化关键基因Mashl启动子上使H3K27me3去甲基化,提高Mashl的表达量,促进神经分化[46]。在脊髓发育过程中,JMJD3与转录因子SMAD1/4相互作用,激活BMP信号的直接拮抗剂Noggin的表达,从而抑制BMP信号通路,确保BMP在脊髓发育过程中保持在合适的水平[47]。在视网膜神经元分化过程中JMJD3可以使Bhlhb4基因启动子H3K27me3得含量降低,促进视网膜神经元分化[12]。TAp63是神经发育过程中的一种重要的促凋亡蛋白,JMJD3的超表达可以促进TAp63γ的表达,进而增强神经标记物β-Ⅲ微管蛋白表达[48]。这些研究表明JMJD3在神经系统发育中有重要的作用,不可或缺。
外胚层发育为表皮过程中,JMJD3也能够调控相关基因的表达,从而调节表皮细胞的分化。在未分化细胞中,许多表皮分化相关基因(如IVL、S100A8和KRT1等)的启动子区域富含H3K27me3标记。在Ca2+诱导分化的过程中,这些基因的启动子区域结合上JMJD3或缺失多梳蛋白SUZ12可以使H3K27me3去甲基化,激活基因表达,促进角质细胞分化,促进表皮成熟。JMJD3过表达可导致表皮分化的提前成熟,而缺失会阻碍表皮分化[49]。
3 JMJD3与细胞衰老细胞衰老是机体新陈代谢的自然现象,细胞衰老可能是由DNA损伤、癌基因诱导、染色质紊乱等因素引起。复制性衰老或者癌基因的诱导可以引起人二倍体成纤维细胞的细胞核内染色质重排,形成异染色质凝集,这一现象被称为衰老相关的异染色质凝集(SAHF)[3]。复制性衰老与多梳蛋白复合物PcG蛋白(如EZH2) 表达量下降、JMJD3表达量上调等因素相关。INK4b-ARF-INK4a基因共编码p16(INK4a)、p15(INK4b)和p14(ARF)3个成员,3种蛋白均参与了细胞的衰老调节。而PcG蛋白和JMJD3参与调节p16基因的表达。在正常生长的细胞中,PcG蛋白可以使H3K27三甲基化从而抑制INK4a/Arf基因编码的肿瘤抑制蛋白p16和p14表达[50]。在原癌基因BRAF和HRASV12的压力下,JMJD3的表达水平大幅提高,2 h内提高大约4倍,在5 d的时候提高到12倍[51]。JMJD3可以与p16INK4a基因转录起始位点上游结合,降低H3K27甲基化水平和促进PcG蛋白CBX7、Bmil和SUZ12的解离,逆转PcG蛋白对基因的抑制作用,激活基因表达,使肿瘤抑制蛋白p16表达量增加,进而抑制细胞增殖,促进细胞衰老[51-53]。
SAHF的形成可以稳定细胞衰老的状态,抑制肿瘤的发生。采用ChIP-seq方法研究发现,在形成SAHF的过程中,许多组蛋白的修饰都参与了这一过程,并且呈现出不同的分布情形,抑制性的组蛋白修饰H3K9me3位于SAHF凝集点的核心区域,H3K27me3位于H3K9me3凝集点的外侧,形成一个“环”环绕着H3K9me3凝集点[54-56]。Zhao等[3]研究发现JMJD3是一个新的SAHF的诱导因子,其可以催化非组蛋白RB蛋白去甲基化,抑制细胞周期蛋白依赖性激酶4(CDK4) 与RB蛋白的结合,降低了RB蛋白磷酸化水平,之后RB蛋白可与细胞周期调控蛋白E2F结合,使E2F靶基因不能表达,从而促进细胞衰老形成SAHF(图 4),说明JMJD3诱导细胞衰老存在一条不依赖于p16的途径。E2F能通过诱导ARF表达,其可抑制p53特异性泛素连接酶MDM2活性,激活p53的表达,促进细胞衰老。
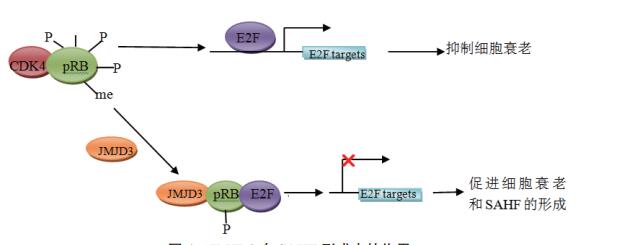
|
| 图 4 JMJD3在SAHF形成中的作用 |
细胞本身的组蛋白去乙酰化酶可以调节EZH2和JMJD3的表达。随着人类多潜能干细胞的衰老,组蛋白去乙酰化酶的含量下降,伴随而来的是JMJD3表达增多和EZH2表达减少,Jung等[50]研究发现,使用组蛋白去乙酰化酶抑制剂也可增加JMJD3的表达和衰老的发生,JMJD3表达过多可加速细胞衰老。
4 JMJD3与肿瘤发生正常的细胞衰老可以控制因DNA损伤或者癌基因刺激造成的生长失控和无限分裂增殖,防止肿瘤的发生。但当衰老调节失控时,细胞则会出现无限增殖,最终引发肿瘤发生[57]。多种人类肿瘤中存在频繁的INK4b-ARF-INK4a位点丧失现象,INK4b-ARF-INK4a功能缺失是造成肿瘤发生的一个重要因素[58],JMJD3可以促进其编码的抑癌性蛋白p16和p14表达。这说明JMJD3与肿瘤的发生发展有密切的关系。大量研究表明,在多种肿瘤的临床标本和细胞系中出现H3K27me3含量增加的现象,如乳腺癌[59]、鼻咽癌[59]、前列腺癌[60]、多发性骨髓瘤[60]和胃癌[61]等。一方面提示H3K27me3水平增高可能成为一种新的肿瘤预测信号;另一方面则暗示H3K27去甲基化酶将具有癌症抑制效应。杨琳等[62]证明了在急性髓性白血病HL-60、U937、NB4三种细胞中H3K27me3的含量均较高,去甲基化酶JMJD3低表达。在HL-60细胞中过表达JMJD3后,H3K27me3含量降低,抑癌蛋白P15、P16分子的表达上调,细胞增殖效率显著降低。这说明在肿瘤细胞中上调JMJD3蛋白水平或酶活性能显著降低H3K27me3水平,这会成为肿瘤治疗的一个新方向。
但有研究发现在前列腺癌[8]、肾细胞癌[62]、原发性霍奇金淋巴瘤[63]、乳腺癌[64]、非霍奇金氏淋巴瘤[43]等肿瘤细胞中,出现了JMJD3过表达现象,这意味着JMJD3基因在不同肿瘤中可能发挥不同的生物学功能。在肾透明细胞癌中,敲除JMJD3可以抑制肿瘤发生,其可以通过激活转录因子SLUG诱导上皮间质转化(EMT)表达[62]。在乳腺癌中,JMJD3能够调节抗凋亡基因BCL2启动子区域的H3K27甲基化水平,从而提高BCL2的表达,促进肿瘤细胞增殖[64]。在原发的霍奇金淋巴瘤中,EBV原癌基因是导致霍奇金淋巴瘤发生的一个重要的诱导因素,能诱导生发中心B细胞中JMJD3的表达,促使JMJD3的转录靶基因高度富集,沉默JMJD3基因能恢复这些基因的H3K27甲基化水平[65]。这提示在这些肿瘤中JMJD3可能起着原癌基因的作用。
JMJD3作为H3K27me2/3的去甲基化酶,作用和EZH2相拮抗,理论上应该起到抑制肿瘤发生的作用。但部分研究发现,在一些肿瘤组织中JMJD3、UTX和EZH2表达均有增高的现象,出现同一种肿瘤中H3K27甲基转移酶和去甲基化酶表达同时升高的“矛盾”现象。Xiang等[1]在前列腺癌中发现JMJD3和EZH2均高表达,其认为JMJD3和EZH2可能通过“代偿”机制调节H3K27的甲基化水平,即当甲基转移酶表达水平过度时,去甲基化酶就通过“代偿”调节H3K27甲基化水平,两者表达失调从而引起肿瘤的发生。在小鼠神经干细胞中,JMJD3可直接和p53和ARF外显子相结合,但H3K27me3表达水平不变,这提示JMJD3并未通过其对H3K27me3的去甲基化酶作用来调节p53和ARF的表达[66]。因此说明JMJD3可能存在去甲基化酶作用以外的其他功能,参与更加广泛的调节网络。
5 JMJD3研究前景研究表明,H3K27去甲基化酶JMJD3,在细胞分化、衰老、胚胎发育及肿瘤发生、发展等生理活动中有着重要的作用。但是,关于JMJD3在参与各种生理活动的作用和调节机理尚不太清楚,如JMJD3在哪些生理活动中通过其非组蛋白去甲基化酶活性或作为转录因子去调节,其是否还存在其他作用。在肿瘤发生过程中,研究JMJD3在哪些肿瘤中发挥了抑癌作用,在哪些肿瘤中发挥了原癌基因作用,探索其调节的靶基因及它们在肿瘤发生中的作用,为进一步研究针对癌症治疗的组蛋白去甲基化酶抑制剂或激活剂奠定理论基础,为肿瘤发生机制的阐明、预防、诊断和治疗发挥重大的推动作用。
| [1] | Xiang Y, Zhu Z, Han G, et al. JMJD3 is a histone H3K27 demethylase[J]. Cell Res, 2007, 17 (10): 850–857. DOI:10.1038/cr.2007.83 |
| [2] | Li Q, Wang HY, Chepelev I, et al. Stage-dependent and locus-specific role of histone demethylase Jumonji D3(JMJD3) in the embryonic stages of lung development[J]. PLoS One, 2014, 10 (7): e1004524. |
| [3] | Zhao L, Zhang Y, Gao Y, et al. JMJD3 promotes SAHF formation in senescent WI38 cells by triggering an interplay between demethylation and phosphorylation of RB protein[J]. Cell Death Differ, 2015, 22 (10): 1630–1640. DOI:10.1038/cdd.2015.6 |
| [4] | Burchfield JS, Li Q, Wang HY, Wang RF. JMJD3 as an epigenetic regulator in development and disease[J]. Int J Biochem Cell Biol, 2015, 67 : 148–157. DOI:10.1016/j.biocel.2015.07.006 |
| [5] | Shi Y, Lan F, et al. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1[J]. Cell, 2004, 119 (7): 941–953. DOI:10.1016/j.cell.2004.12.012 |
| [6] | Peters AH, KubicekS, Mechtler K, et al. Partitioning and plasticity of repressive histone methylation states in mammalian chromatin[J]. Molecular Cell, 2003, 12 : 1577–1589. DOI:10.1016/S1097-2765(03)00477-5 |
| [7] | Barski A, Cuddapah S, Cui K, et al. High-resolution profiling of histone methylations in the human genome[J]. Cell, 2007, 129 : 823–837. DOI:10.1016/j.cell.2007.05.009 |
| [8] | Klose RJ, Kallin EM, Zhang Y. JmjC-domain-containing proteins and histone demethylation[J]. Nat Rev Genet, 2006, 7 (9): 715–727. DOI:10.1038/nrg1945 |
| [9] | Ke XS, Qu Y, Rostad K, et al. Genome-wide profiling of histone h3 lysine 4 and lysine 27 trimethylation reveals an epigenetic signature in prostate carcinogenesis[J]. PLoS One, 2009, 4 (3): e4687. DOI:10.1371/journal.pone.0004687 |
| [10] | Swigut T, Wysocka J. H3K27 demethylases, at long last[J]. Cell, 2007, 131 (1): 29–32. DOI:10.1016/j.cell.2007.09.026 |
| [11] | Zhang F, Xu L, Xu L, et al. Histone demethylase JMJD3 is required for osteoblast differentiation in mice[J]. Sci Rep, 2015, 5 : 13418. DOI:10.1038/srep13418 |
| [12] | Iida A, Iwagawa T, Kuribayashi H, et al. Histone demethylase Jmjd3is required for the development of subsets of retinal bipolar cells[J]. Proc Natl Acad Sci USA, 2014, 111 : 3751–3756. DOI:10.1073/pnas.1311480111 |
| [13] | Zhao W, Li Q, Ayers S, et al. Jmjd3 inhibits reprogramming by up regulating expression of INK4a/Arf and targeting PHF20 for ubiquitination[J]. Cell, 2013, 152 (5): 1073–1050. |
| [14] | Tanaka Y, Yano H, Ogasawara S, et al. Mild glucose starvation induces KDM2A-mediated H3K36me2 demethylation through AMPK to reduce rRNA transcription and cell proliferation[J]. Mol Cell Biol, 2015, 35 (24): 4170–4184. DOI:10.1128/MCB.00579-15 |
| [15] | Janzer A, Stamm K, Becker A, et al. The H3K4me3 histone demethylase fbxl10 is a regulator of chemokine expression, cellular morphology, and the metabolome of fibroblasts[J]. Journal of Biological Chemistry, 2012, 287 (37): 30984–3092. DOI:10.1074/jbc.M112.341040 |
| [16] | Herzog M, Josseaux E, et al. The histone demethylase Kdm3a is essential to progression through differentiation[J]. Nucleic Acids Res, 2012, 40 (15): 7219–7232. DOI:10.1093/nar/gks399 |
| [17] | Ling Y, Fan Z, Bo Y, et al. Histone Demethylases KDM4B and KDM6B Promotes Osteogenic Differentiation of Human MSCs[J]. Cell Stem Cell, 2012, 11 (1): 50. DOI:10.1016/j.stem.2012.04.009 |
| [18] | Su Z, Wang F, Lee JH, et al. Reader domain specificity and lysine demethylase-4 family function[J]. Nature Communications, 2016, 7 : 13387. DOI:10.1038/ncomms13387 |
| [19] | Pack LR, Yamamoto KR, Fujimori DG. Opposing Chromatin Signals Direct and Regulate the Demethylase Activity of KDM4C[J]. J Biol Chem, 2016, 291 (12): 6060–6070. DOI:10.1074/jbc.M115.696864 |
| [20] | Kupershmit I, Khouryhaddad H, Awwad S W, et al. KDM4C(GASC1) lysine demethylase is associated with mitotic chromatin and regulates chromosome segregation during mitosis[J]. Nucleic Acids Research, 2014, 42 (10): 6168–6182. DOI:10.1093/nar/gku253 |
| [21] | Zoabi M, Nadar-Ponniah PT, Khoury-Haddad H, et al. RNA-dependent chromatin localization of KDM4D lysine demethylase promotes H3K9me3 demethylation[J]. Nucleic Acids Research, 2014, 42 (21): 13026. DOI:10.1093/nar/gku1021 |
| [22] | Wang C, Wang J, et al. KDM5A controls bone morphogenic protein 2-induced osteogenic differentiation of bone mesenchymal stem cells during osteoporosis[J]. Cell Death Dis, 2016, 7 (8): e2335. DOI:10.1038/cddis.2016.238 |
| [23] | Wang D, Han S, Peng R, et al. Depletion of histone demethylase KDM5B inhibits cell proliferation of hepatocellular carcinoma by regulation of cell cycle checkpoint proteins p15 and p27[J]. J Exp Clini Cancer Res, 2016, 35 (1): 1–13. DOI:10.1186/s13046-015-0276-9 |
| [24] | Jiang W, Wang J, Zhang Y. Histone H3K27me3 demethylases KDM6A and KDM6B modulate definitive endoderm differentiation from human ESCs by regulating WNT signaling pathway[J]. Cell Research, 2013, 23 (1): 122–130. DOI:10.1038/cr.2012.119 |
| [25] | Pan MR, Hsu MC, Chen LT, et al. G9a orchestrates PCL3 and KDM7A to promote histone H3K27 methylation[J]. Scientific Reports, 2015, 5 : 18709. |
| [26] | Feng W, Yonezawa M, Ye J, et al. PHF8 activates transcription of rRNA genes through H3K4me3 binding and H3K9me1/2 demethylation[J]. Nat Struct Mol Biol, 2010, 17 : 445–450. DOI:10.1038/nsmb.1778 |
| [27] | Qi HH, Sarkissian M, Hu GQ, et al. Histone H4K20/H3K9 demethylase PHF8 regulates zebrafish brain and craniofacial development[J]. Nature, 2010, 466 : 503–507. DOI:10.1038/nature09261 |
| [28] | Chen B, Yu M, et al. Mdig de-represses H19 large intergenic non-coding RNA(lincRNA)by down-regulating H3K9me3 and heterochromatin[J]. Oncotarget, 2013, 4 (9): 1427. DOI:10.18632/oncotarget.v4i9 |
| [29] | Lee KH, Ju UI, Song JY, et al. The histone demethylase PHF2 promotes fat cell differentiation as an epigenetic activator of both C/EBPα and C/EBPδ[J]. Mol Cells, 2014, 37 (10): 734. DOI:10.14348/molcells.2014.0180 |
| [30] | Chen Q, Sinha K, Deng JM, et al. Mesenchymal deletion of histone demethylase NO66 in mice promotes bone formation[J]. J Bone Miner Res, 2015, 30 (9): 1608–1617. DOI:10.1002/jbmr.2494 |
| [31] | Burgold T, Spreafico F, De Santa F, et al. The histone H3 lysine 27-specific demethylase JMJD3 is required for neural commitment[J]. PLoS One, 2008, 3 (8): e3034. DOI:10.1371/journal.pone.0003034 |
| [32] | Ga oY, Hyttel P, Hall VJ. Regulation of H3K27me3 and H3K4me3 during early porcine embryonic development[J]. Mol Reprod Dev, 2010, 77 (6): 540–549. DOI:10.1002/mrd.v77:6 |
| [33] | Canovas S, Cibelli JB, Ross PJ. Jumonji domain-containing protein 3 regulates histone3 lysine 27 methylation during bovine preimplantation development[J]. Proc Natl Acad Sci USA, 2012, 109 (7): 2400–2405. DOI:10.1073/pnas.1119112109 |
| [34] | Kartikasari AE, Zhou JX, Kanji MS, et al. The histone demethylase Jmjd3 sequentially associates with the transcription factors Tbx3 and Eomes to drive endoderm differentiation[J]. Embo J, 2013, 32 (10): 1393–1408. DOI:10.1038/emboj.2013.78 |
| [35] | Dahle, Kumar A, Kuehn MR. Nodal signaling recruits the histone demethylase Jmjd3 to counteract polycomb-mediated repression at target genes[J]. Sci Signal, 2010, 3 (127): ra48. |
| [36] | Satoh T, Takeuchi O, Vandenbon A, et al. The Jmjd3-Irf4 axisregulates M2 macrophage polarization and host responses against helminthinfection[J]. Nat Immun, 2010, 11 : 936–944. DOI:10.1038/ni.1920 |
| [37] | Morales Torres C, Laugesen A, Helin K. Utx is required for proper induction of ectoderm andmesoderm during differentiation of embryonic stem cells[J]. PLoS One, 2013, 8 : e60020. DOI:10.1371/journal.pone.0060020 |
| [38] | Ohtani K, Zhao C, Dobreva G, et al. Jmjd3 controls mesodermal andcardiovascular differentiation of embryonic stem cells[J]. Circ Res, 2013, 113 : 856–862. DOI:10.1161/CIRCRESAHA.113.302035 |
| [39] | Zhang F, Xu L, Xu L, et al. JMJD3 promotes chondrocyte proliferation and hypertrophy during endochondral bone formation in mice[J]. J Mol Cell Biol, 2015, 7 (1): 23–34. DOI:10.1093/jmcb/mjv003 |
| [40] | Gomez-Sanchez JA, Gomis-Coloma C, Morenilla-Palao C, et al. Epigenetic induction of the Ink4a/Arf locus prevents Schwann cell overproliferation during nerveregeneration and after tumorigenic challenge[J]. Brain:J Neurol, 2013, 136 : 2262–2278. DOI:10.1093/brain/awt130 |
| [41] | Huszar JM, Payne CJ. MIR146A inhibits JMJD3 expression and osteogenic differentiation in human mesenchymal stem cells[J]. FEBS Lett, 2014, 588 (9): 1850–1856. DOI:10.1016/j.febslet.2014.03.057 |
| [42] | Xu J, Yu B, Hong C, Wang CY. KDM6B epigenetically regulates odontogenic differentiation of dentalmesenchymal stem cells[J]. Int J Oral Sci, 2013, 5 : 200–205. DOI:10.1038/ijos.2013.77 |
| [43] | 张妍. JMJD3调控弥漫性大B细胞淋巴瘤进展的作用机制研究[D]. 天津: 南开大学, 2014. |
| [44] | Liu Z, Cao W, Xu L, et al. The histone H3 lysine-27 demethylase Jmjd3 plays a critical role in specific regulation of Th17 cell differentiation[J]. J Mol Cell Biol, 2015, 7 (6): 505–516. DOI:10.1093/jmcb/mjv022 |
| [45] | De Santa F, Totaro MG, et al. The histone H3 lysine-27 demethylase Jmjd3 links inflammation to inhibition of polycomb-mediated gene silencing[J]. Cell, 2007, 130 (6): 1083–1094. DOI:10.1016/j.cell.2007.08.019 |
| [46] | Dai JP, Lu JY, Zhang Y, et al. Jmjd3 activates Mash1 gene in RA induced neuronal differentiation of P19 cell[J]. J Cell Biochem, 2010, 110 : 1457–1463. DOI:10.1002/jcb.22703 |
| [47] | Akizu N, Estaras C, Guerrero L, et al. H3K27me3 regulates BMP activity indeveloping spinal cord[J]. Development(Cambridge, England), 2010, 137 : 2915–2925. |
| [48] | Fonseca MB, Nunes AF, Morgado AL, et al. TAp63γ demethylation regulates protein stability and cellular distribution during neural stem cell differentiation[J]. PLoS One, 2012, 7 (12): e52417. DOI:10.1371/journal.pone.0052417 |
| [49] | Sen GL, Webster DE, Barragan DI, et al. Control of differentiationin a self-renewing mammaliantissue by the histonedemethylase JMJD3[J]. Genes Dev, 2008, 22 (14): 1865–1870. DOI:10.1101/gad.1673508 |
| [50] | Jung JW, et al. Histone deacetylase controls adult stem cell aging by balancing the expression of polycomb genes and jumonji domain containing 3[J]. Cell Mol Life Sci, 2010, 67 (7): 1165–1176. DOI:10.1007/s00018-009-0242-9 |
| [51] | Agger K, Cloos PA, Rudkjaer L, et al. The H3K27me3 demethylase JMJD3 contributes to the activation of the INK4A-ARF locus in response to oncogene-and stress-induced senescence[J]. Genes & Development, 2009, 23 (10): 1171–1176. |
| [52] | Barradas M, Anderton E, Acosta JC, et al. Histone demethylase JMJD3 contributes to epigenetic control of INK4a/ARF by oncogenic RAS[J]. Genes Dev, 2009, 23 (10): 1177–1182. DOI:10.1101/gad.511109 |
| [53] | Agherbi H, Gaussmann-Wenger A, et al. Polycomb mediated epigenetic silencing and replication timing at the INK4a/ARF locus during senescence[J]. PLoS One, 2009, 4 (5): e5622. DOI:10.1371/journal.pone.0005622 |
| [54] | Chandra T, Kirschner K, Thuret JY, et al. Independence of repressive histone marks and chromatin compaction during senescent heterochromatic layer formation[J]. Mol Cell, 2012, 47 (2): 203–214. DOI:10.1016/j.molcel.2012.06.010 |
| [55] | Di Micco R, Sulli G, Dobreva M, et al. Interplay between oncogene-induced DNA damage response and heterochromatin in senescence and cancer[J]. Nat Cell Boil, 2011, 13 (3): 292–302. DOI:10.1038/ncb2170 |
| [56] | Wang Z, Liu Y, Takahashi M, et al. N terminus of ASPP2 binds to Ras and enhances Ras/Raf/MEK/ERK activation to promote oncogene-induced senescence[J]. Proc Natl Acad Sci USA, 2013, 110 (1): 312–317. DOI:10.1073/pnas.1201514110 |
| [57] | Campisi J, d'AddadiFagagna F. Cellular senescence:when bad things happen to good cells[J]. Nat Rev Mol Cell Biol, 2007, 8 (9): 729–740. DOI:10.1038/nrm2233 |
| [58] | Kim WY, Sharpless NE. The regulation of INK4/ARF in cancer and aging[J]. Cell, 2006, 127 (2): 265–275. DOI:10.1016/j.cell.2006.10.003 |
| [59] | Yang Q, Tian Y, Ostler KR, et al. Epigenetic alterations differ in phenotypically distinct human neuroblastoma cell lines[J]. BMC Cancer, 2010, 10 : 286. DOI:10.1186/1471-2407-10-286 |
| [60] | He LR, Liu MZ, et al. Prognostic impact of H3K27me3 expression-onlocoregional progression after chemoradiotherapy in esophageal squamous cellcarcinoma[J]. BMC Cancer, 2009, 9 : 461. DOI:10.1186/1471-2407-9-461 |
| [61] | Lu TY, Kao CF, Lin CT, et al. DNA methylation and histone modif-icationregulate silencing of OPG during tumor progression[J]. J Cell Biochem, 2009, 108 (1): 315–325. DOI:10.1002/jcb.v108:1 |
| [62] | 杨琳. 真核表达载体过表达JMJD3基因对急性白血病HL-60细胞株影响的实验研究[D]. 福州: 福建医科大学, 2014. |
| [63] | Shen Y, Guo X, Wang Y, et al. Expression and significance of histone H3K27 demethylases in renal cell carcinoma[J]. BMC Cancer, 2012, 12 : 470. DOI:10.1186/1471-2407-12-470 |
| [64] | Anderton JA, Bose S, et al. The H3K27me3 demethylase, KDM6B, is induced by Epstein-Barr virus and over-expressed in Hodgkin's Lymphoma[J]. Oncogene, 2011, 30 (17): 2037–2043. DOI:10.1038/onc.2010.579 |
| [65] | Svotelis A, Bianco S, Madore J, et al. H3K27 demethylation by JMJD3 at a poised enhancer of anti-apoptotic gene BCL2 determines ER alpha ligand dependency[J]. Embo J, 2011, 30 (19): 3947–3961. DOI:10.1038/emboj.2011.284 |
| [66] | Sola S, Xavier JM, Santos DM, et al. p53 interaction with JMJD3 results in its nuclear distribution during mouse neural stem cell differentiation[J]. PLoS One, 2011, 6 (3): el8421. |