哺乳动物出生后,由于其获得性免疫的防御系统并不成熟和完善,对外界病原微生物的抵抗能力非常弱,因此从初乳中获得抗病原微生物的活性蛋白,便成了其对抗疾病侵袭的主要防御手段。乳铁蛋白(Lactoferrin,LF)是发现于动物初乳中具有广谱抗菌能力的单体糖蛋白,在初乳中的含量是常乳的十几倍甚至几十倍[1],这种高浓度的蛋白表达对新生动物的天然免疫具有重要作用。LF蛋白和其酶解肽段具有普通抗生素所不具备的一系列优点,包括具有广谱抗菌、抗病毒、抗真菌和抑杀肿瘤细胞的功能,对动物细胞几乎没有毒性,不含稀有氨基酸和外源化学成分[2]。另外,抗菌肽不具有固定的抗菌模式,这使致病菌不易针对其抗菌作用机制产生耐药性。以上优点显示了LF蛋白替代抗生素的巨大潜能,使它被认为是一种新型的抗菌、抗病毒、抗癌药物和极具开发潜力的食品和饲料添加剂。
本文对近年来LF蛋白的抗菌研究进行了归纳和总结,以LF蛋白的膜结合能力和LF蛋白酶解产物的抗菌活性为切入点,对LF蛋白的抗菌机理进行深入探讨。对LF蛋白抗菌机理的剖析将使我们更为深入的了解LF蛋白及其抗菌肽的构效关系,根据这些LF蛋白分子结构信息,可以人为的设计出抗菌功能更强的活性蛋白,为进一步开发新型的抗菌类药物奠定理论基础。
1 LF蛋白概述LF蛋白是一种分子质量为80 kD左右的铁结合糖蛋白,产生于约1.25亿年前胎盘类动物和有袋类动物分化之时,因与转铁蛋白有较高的同源性而被归于转铁蛋白家族,是转铁蛋白家族中最年轻的成员[3]。LF蛋白广泛存在于外分泌液(乳汁、唾液、泪液、鼻分泌物)、血浆、羊水、子宫分泌物、尿液和中性粒细胞中,以初乳中含量最高[1]。研究发现,LF蛋白具有多种生物学功能,包括广谱抗菌、抗病毒、抗肿瘤[4]作用,并且与体内的许多重要生理过程有关,如能调节体内铁的平衡、调节骨髓细胞生成、促进细胞生长、调节机体免疫功能、增强机体抗病能力、抗高血压活性、与多种抗生素及抗真菌制剂协同作用[5-7]。
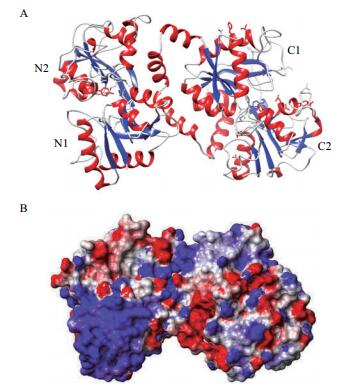
LF蛋白由两个对称的N-叶和C-叶组成,两叶间在氨基酸序列上有显著的同源性,每叶又分别由两个结构域构成(图 1),每一叶可以结合一个Fe3+离子,每个铁结合部位由4个氨基酸残基组成。LF蛋白表面带有大量的正电荷(图 1),这种特征有利于其与带负电的细胞膜相结合。在大多数细胞中,LF蛋白以无铁的形式(apo-LF)被分泌到胞外,当与过量的三价铁离子结合后变成铁饱和LF蛋白(holo-LF)。人类LF蛋白有多个表达后修饰位点,如磷酸化[9]和N-聚糖修饰[10],这些修饰可能使LF蛋白免受水解酶作用和参与受体识别[11]。
作为抑菌剂的LF蛋白具有广谱性,即能抑制革兰氏阴性菌,如大肠杆菌(Escherichia coli)、幽门螺旋杆菌(Helicobacter pylori)、克雷白氏杆菌(Klebsiella)、沙门氏菌(Salmonella)和志贺氏菌(Shigella)等,又对革兰氏阳性菌如金黄色葡萄球菌(Staphylococcus aureus)、枯草芽孢杆菌(Bacillus subtilis)和单增李斯特菌(Listeria monocytogens)具有抗菌作用[12-13]。
不同状态下LF蛋白具有不同的抗菌能力[14],而且基因工程表达的LF蛋白也具有与天然状态相似的抗菌活性[15]。对不同物种的LF蛋白抗菌活性的研究显示,牛LF蛋白的抗菌能力在哺乳动物中是最强的[16]。LF蛋白主要通过铁剥夺、膜渗透和酶抑制3种途径发挥抑菌作用[6, 17],但其具体抗菌行使的分子机制尚不明确,具体作用方式如下:(1) 铁剥夺是LF蛋白最基本的抑菌机制。不饱和LF蛋白具有极强的铁结合性,它可以与病原性微生物竞争性地结合铁离子,使病原性微生物因失去生长所需的基本元素铁而停止生长甚至死亡;或使微生物由于缺乏铁离子而不能形成致病性生物膜,降低细菌浓度,减少发病率[18]。(2) 膜渗透的产生机制是LF蛋白依靠本身带有的正电荷与革兰氏阳性菌细胞壁上的磷壁酸(Lipoteichoic acid,LTA)或革兰氏阴性菌的脂多糖(Lipopolysaccharide,LPS)产生静电吸引,破坏微生物细胞膜的正常生理功能、通透性增加和脂质双分子层破坏,增加细胞膜通透性,使细菌脂多糖渗出,造成内容物流失而达到杀死病原菌的目的[17, 19]。LF蛋白这种杀菌作用还可能与LF蛋白结合钙、镁离子的能力有关[20]。(3) 酶抑制机制是LF蛋白通过其蛋白酶活性降解一些细菌毒力因子或黏附蛋白,降低病原体结合和侵入哺乳动物细胞的几率。LF蛋白表面所携带的碱性高密度电荷,很容易与细菌或宿主细胞的一些生物分子发生非特异性结合,如LF蛋白可以与双岐杆菌和假单胞菌属细胞膜上的结合蛋白相结合,从而降低细菌的感染力[21]。
铁剥夺可以通过对铁离子的剥夺而抑制细菌的生长,酶抑制可以减少细菌对宿主的侵染和减弱毒性分子的毒力,真正可以直接导致细菌的死亡的还是其膜渗透的抗菌途径,也是LF蛋白行使抗菌能力的主要途径。由于膜渗透抗菌活性行使的前提是LF蛋白必须首先与细菌的细胞膜结合,LF蛋白的表位与细菌的膜表面特征对这种结合起着至关重要的作用。
3 LF蛋白与细菌细胞膜的结合能力研究表明,LF蛋白与多种病原微生物的细胞膜表面存在相互作用,这种膜结合能力是由LF蛋白与细菌膜上不同组分的亲和力所决定的[22]。由于病原微生物的细胞膜由多种组分构成,其中最主要的有脂质、脂多糖和膜蛋白,我们分别对这些组分与LF蛋白的作用进行探讨。
3.1 LF蛋白与细菌细胞膜上脂质的作用体外实验显示,当缺少胃蛋白酶时,LF蛋白不能被降解为小的肽段,它通过静电作用与脂肪乳剂的磷脂界面结合,有稳定脂滴的作用。LF蛋白和其酶解释放的抗菌肽Lfcin都可以抑制引起牙周炎的牙龈卟啉单胞菌和中间普雷沃菌的生长,但只有LF蛋白可以通过与膜上脂质的相互作用而抑制这两种菌细胞膜的形成[23]。LF蛋白还可以通过铁螯合作用使绿脓杆菌的胞膜变得不稳定[24],进而可以增加绿脓甲单胞菌的通透性,与木糖醇共同作用可以在一定程度上破坏细胞膜的结构,进而降低绿脓甲单胞菌的生存能力[25]。
3.2 LF蛋白与细菌脂多糖作用革兰氏阴性菌的外膜是含有大量的脂多糖(在沙门氏菌和大肠杆菌中的含量分别为3/16和7/19),LF蛋白便可以与这些脂多糖相结合,利用这种结合能力使大肠杆菌和鼠沙门氏菌的脂多糖从胞膜上释放[26]。通过抗原表位在空间上较远的两个抗体的免疫结合实验显示,LF蛋白对革兰氏阴性菌的脂多糖和脂质A具有结合能力,两者具有空间上相近的结合位点,并且对脂质A的结合能力大于脂多糖[27]。LF蛋白的这种结合往往是通过其自身表位上的肽段实现的,如其多肽片段LF11与脂多糖通过静电力和疏水力相互作用,这种作用可以减弱脂多糖对机体的刺激[28]。
3.3 LF蛋白与细菌膜蛋白的结合除了与脂质和脂多糖的相互作用外,通过细菌表面的结合蛋白也是LF蛋白与膜结合的重要作用方式。这种与细菌细胞膜的结合方式在多种细菌中得到体现,如LF蛋白与甲单胞菌细胞表面的LF结合蛋白相结合,这种结合对细菌的生长起抑制作用[29]。晶体结构分析显示,带负电荷肺炎球菌的表面蛋白PspA的N端螺旋结构可以与LF蛋白的N叶带正电荷的部分相结合[30],这种结合阻碍了抗菌肽进入胞膜而对细菌的杀伤作用[31]。LF蛋白对溶血性曼氏杆菌有杀菌作用,通过覆盖检测法和双向电泳分析发现LF蛋白可以结合于溶血性曼氏杆菌的两个外膜蛋白OmpA和孔蛋白,这两个LF结合蛋白在菌体内可以与两种状态的LF蛋白(缺铁LF蛋白和铁饱和LF蛋白)相结合[32]。另外,奈瑟氏菌的LF蛋白结合蛋白A和B结合于LF蛋白的C叶,它可能通过这种结合来获得铁离子[33-34]。
3.4 LF蛋白糖基化对膜结合能力的影响LF蛋白的翻译后修饰同样对其活性起着非常重要的作用,如糖基化[10, 35-36]和酰胺化[37]。关于牛LF蛋白糖基化位点存在不同的报道。Baker等[38]认为牛LF蛋白有4个糖基化位点,分别在第233、368、76、545位的天冬酰胺,而Yu[36]报道牛LF蛋白只有第138和479位的天冬酰胺被糖基化。这种糖基化的变化可能因不同泌乳期各种糖基转移酶的表达量不同所决定[39]。天然状态下的人LF蛋白和转基因牛奶中得到的人LF蛋白存在不同的糖基化[40],重组LF蛋白含有较多的甘露糖,较少的N-乙酰神经氨酸和岩藻糖,有些含有乙酰半乳糖胺-乙酰氨基葡萄糖二糖。糖基化与细菌的膜结合密切相关[35],如龈卟啉单胞菌的菌毛只能和糖基化的LF蛋白结合,只有N-乙酰半乳糖胺和岩藻糖是对结合有抑制作用的[41]。人类LF蛋白能够阻止病原菌的粘附,LF蛋白上结合的多糖可以降低沙门氏菌对结肠上皮细胞的侵染。LF蛋白的糖基化被基因表达严格调控,糖基化的不同与病原菌的调节密切相关,在整个泌乳期岩藻糖转移酶表达量升高,在泌乳第二周寡糖基转移酶复合物表达量开始下降[10]。
LF蛋白虽然可以与多种病源菌的表面物质相结合,但其本身不能充分发挥杀菌作用,如LF蛋白影响肽聚糖的合成和/或结合于肽聚糖上的乙酰氨基葡萄糖,并且可与金黄色葡萄球菌的细胞壁相结合,影响分裂后的子细胞散播,但并不能使细菌裂解。LF蛋白抑制临床实验分离的绿脓杆菌细胞膜的形成,这种抑制作用与其杀菌作用不相关,因为体外实验显示多种细菌对LF蛋白不敏感[42]。然而,LF蛋白的酶解肽段却可致使金黄色葡萄球菌的细胞壁变形,进而通过细胞破裂杀死细菌[43]。
4 LF蛋白酶解多肽的抗菌能力LF蛋白降解后其抗菌活性会进一步增加,这主要是因为LF蛋白降解后产生多种比其本身抗菌能力更强的肽段[13],如牛LF蛋白的胃蛋白酶水解产物具有比LF蛋白本身更强的抗双歧杆菌、单核细胞增多性双歧杆菌、李斯特氏菌和金黄色葡萄球菌的抗菌能力,同时对奶酪上的假单孢菌和大肠杆菌具有明显的抑制作用[44]。LF蛋白的N端的降解片段对耐药性的金黄色葡萄球菌和肺炎链球菌引起的感染有治疗作用,主要是由于其对细菌细胞膜膜渗透的破坏作用,肽段上的前两个R氨基酸对这种破坏作用起着至关重要的的作用[45]。
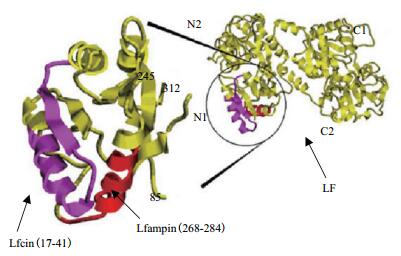
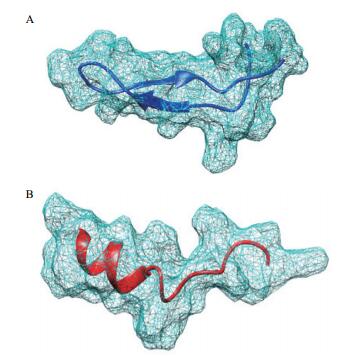
4.1 抗菌肽Lfcin乳铁蛋白素(Lactoferricin,Lfcin)是LF蛋白在酸性环境下经胃蛋白酶作用,从N端释放的一段25个氨基酸残基的小肽。有人认为它是LF蛋白酶解产物中活性最强的抗菌肽。Lfcin除了不能结合铁离子外,几乎具备LF蛋白的所有生物学活性[46]。在LF蛋白结构内部时,Lfcin会在其表面形成一个α-螺旋的二级结构(图 2),而当它处于水相溶液时,则主要以两个反相平行的β-折叠结构存在(图 3)。造成这种结构的变化是因为Lfcin在疏水性分子(如脂质分子)的作用下发生了变构,由β-折叠结构变回α-螺旋结构。这种α-螺旋结构具有与细胞膜的脂质更好的亲和性,因此,位于表面的Lfcin对LF蛋白的膜结合能力做出了一定的贡献。Lfcin多肽两亲性二级结构和阳离子特性是其具有抗菌功能的基础,主要通过与细菌胞膜结合以膜渗透的途径发挥抗菌活性[1]。
Lfcin对病原菌的致死作用机制与抗生素的亲膜特性相似,依靠其自身带有的正电荷与革兰氏阳性菌细胞壁上的LTA或革兰氏阴性菌的LPS产生静电吸引,使抗菌肽附着于外膜表面,然后利用疏水结构插入细胞膜,造成细胞膜脂质双分子层结构改变,形成环状孔道,实现抗菌肽跨膜进入细胞内;或者多个抗菌肽聚合到膜区引发通道形成,增强细胞膜通透性,使细胞膜失去跨膜电荷梯度和pH梯度,或在膜上形成孔洞导致细胞内容物流失达到杀菌目的[47]。利用荧光信号分子的实验也证实,Lfcin可破坏大肠杆菌的细菌膜致使荧光信号分子流入,还也可以使大型脂质单层囊泡破裂造成荧光分子的释放[48]。
4.2 抗菌肽LfampinLfampin也是由LF蛋白酶解产生的肽段,位于LF蛋白的268-284部位氨基酸(图 2),肽段的N端形成两亲性的α-螺旋结构(图 3)[51],带正电荷的C端对其行使抗菌活性具有重要作用[52]。与许多抗菌相似,Lfampin含有的色氨酸和带正电荷的氨基酸使它具有广谱抗菌活性,并且为两亲性分子。Lfampin与细菌细胞膜也具有一定的结合能力,其C端所带的正电荷先与细菌脂双层膜相吸引,然后N端的螺旋结构结合于膜表面[52]。增加Lfampin的C端的正电荷氨基酸的数目可以明显提高其抗菌活性[53],差示扫描量热法确定它插入膜的疏水核心区域,荧光光谱学证实多肽上的氨基酸残基决定了肽段与磷脂双分子层上核心疏水区的相互作用[53]。Lfampin抗菌肽进行适当的人为改造可以进一步提高其抗菌能力,如由Lfcin和Lfampin抗菌肽串联组成的LFchimera具有比两者更强的与带负电荷细胞膜结合的能力和抗菌活性[54-55]。
除Lfcin和Lfampin多肽外,LF蛋白的酶解产物中还存在其它一些具有明显抗菌活性的小肽。源于人类LF蛋白的抗菌肽HLR1r具有抗革兰氏阴性菌和革兰氏阳性菌的作用,如耐甲氧金黄色葡萄球菌,同时具有抗感染的功效[56]。多肽LF11对细菌胞膜具有一定的破坏作用,去除多肽LF11的不带电荷残基、增加LF11多肽N端的疏水氨基酸或N端的酰化作用都可以提高其抗大肠杆菌的能力,N端酰化的LF11可以结合革兰氏阴性菌表面的脂多糖[57]。源于LF蛋白序列的多肽L10对超广谱β内酰胺酶阳性的革兰氏阴性菌和多药耐药性的真菌均有杀伤作用,电镜观察确定了L10肽段可以与两种菌胞膜的结合,进一步研究确定它可以结合革兰氏阴性菌的脂质A和脂多糖结合,并且对假丝酵母的细胞具有透化作用[58]。
部分LF蛋白的抗菌多肽对真菌也有一定的抑制作用,如多肽Lfpep和Kaliocin-1对白色念珠菌的细胞膜具有渗透作用,同样Lfpep也是通过破坏生物膜结构来达到杀菌的目的[59],LF11也可以抑制白色念珠菌生物膜的形成[60]。由LF蛋白序列衍生出的两个肽段HLopt2和HLBD1对假丝酵母有杀伤作用,HLopt2可以使酵母的生物膜表面形成凹陷,进而破坏膜结构达到杀菌的目的[61]
5 小结综上所述,LF蛋白的总体抗菌活性由其膜结合能力和其酶解多肽的抗菌活性所决定,因此,作为抗菌蛋白的LF不但是潜在抗菌肽的前体,而且是膜结合功能的执行者和降解酶的优秀底物。在此,我们认为LF蛋白行使抗菌功能的过程是,整体LF蛋白先与细菌的细胞膜结合,这种结合能力的强弱与细菌细胞膜的组分和LF蛋白的糖基化密切相关;膜结合可以使LF蛋白发生结构改变,进而改变其对蛋白酶的敏感性;LF蛋白变构之后被酶解,产生出具有比LF蛋白细菌细胞膜破坏能力更强的多种抗菌肽;这些抗菌肽通过增加细胞膜的通透性使细胞内溶物流失,最终达到杀菌的效果。
| [1] | Wakabayashi H, Yamauchi K, Takase M. Lactoferrin research, technology and applications[J]. International Dairy Journal, 2006, 16 (11): 1241–1251. DOI:10.1016/j.idairyj.2006.06.013 |
| [2] | 朱艳萍, 滕达, 田子罡, 等. 乳铁蛋白分子结构及其抗菌机制[J]. 生物技术通报, 2010(6): 37–42. |
| [3] | Lambert LA. Molecular evolution of the transferrin family and associated receptors[J]. Biochimica Et Biophysica Acta-General Subjects, 2012, 1820 (3): 244–255. DOI:10.1016/j.bbagen.2011.06.002 |
| [4] | Duarte DC, Nicolau A, Teixeira JA, et al. The effect of bovine milk lactoferrin on human breast cancer cell lines[J]. Journal of Dairy Science, 2011, 94 (1): 66–76. DOI:10.3168/jds.2010-3629 |
| [5] | Pierce A, Legrand D. Advances in lactoferrin research Introduction[J]. Biochimie, 2009, 91 (1): 1–2. DOI:10.1016/j.biochi.2008.10.002 |
| [6] | Ochoa TJ, Sizonenko SV. Lactoferrin and prematurity:a promising milk protein?[J]. Biochemistry and Cell Biology, 2017, 95 (1): 22–30. DOI:10.1139/bcb-2016-0066 |
| [7] | Wang X, Wang X, Hao Y, et al. Research and development on lactoferrin and its derivatives in China from 2011-2015[J]. Biochemistry and Cell Biology, 2017, 95 (1): 162–170. DOI:10.1139/bcb-2016-0073 |
| [8] | Moore SA, Anderson BF, Groom CR, et al. Three-dimensional struc-ture of diferric bovine lactoferrin at 2.8 angstrom resolution[J]. Journal of Molecular Biology, 1997, 274 (2): 222–236. DOI:10.1006/jmbi.1997.1386 |
| [9] | Rikova K, Guo A, Zeng Q, et al. Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer[J]. Cell, 2007, 131 (6): 1190–1203. DOI:10.1016/j.cell.2007.11.025 |
| [10] | Barboza M, Pinzon J, Wickramasinghe S, et al. Glycosylation of human milk lactoferrin exhibits dynamic changes during early lactation enhancing its role in pathogenic bacteria-host interactions[J]. Molecular & Cellular Proteomics, 2012, 11 (6). |
| [11] | Mayeur S, Spahis S, Pouliot Y, et al. Lactoferrin, a Pleiotropic Protein in Health and Disease[J]. Antioxidants & Redox Signaling, 2016, 24 (14): 813–835. |
| [12] | Valenti P, Antonini G. Lactoferrin:an important host defence against microbial and viral attack[J]. Cellular and Molecular Life Sciences, 2005, 62 (22): 2576–2587. DOI:10.1007/s00018-005-5372-0 |
| [13] | Del Olmo A, Calzada J, Nunez M. Effect of lactoferrin and its derivatives against gram-positive bacteria in vitro and, combined with high pressure, in chicken breast fillets[J]. Meat Science, 2012, 90 (1): 71–76. DOI:10.1016/j.meatsci.2011.06.003 |
| [14] | 文鹏程, 余丹丹, 汪昕昕, 等. 不同处理条件对乳铁蛋白构象的影响研究[J]. 光谱学与光谱分析, 2012(1): 162–165. |
| [15] | Zhang J, Li L, Cai Y, et al. Expression of active recombinant human lactoferrin in the milk of transgenic goats[J]. Protein Expression and Purification, 2008, 57 (2): 127–135. DOI:10.1016/j.pep.2007.10.015 |
| [16] | Jenssen H, Hancock REW. Antimicrobial properties of lactoferrin[J]. Biochimie, 2009, 91 (1): 19–29. DOI:10.1016/j.biochi.2008.05.015 |
| [17] | Gonzalez-Chavez SA, Arevalo-Gallegos S, Rascon-Cruz Q. Lactofe-rrin:structure, function and applications[J]. International Journal of Antimicrobial Agents, 2009, 33 (4): 301–306. |
| [18] | Yen C-C, Shen C-J, Hsu W-H, et al. Lactoferrin:an iron-binding antimicrobial protein against Escherichia coli infection[J]. Biometals, 2011, 24 (4): 585–594. DOI:10.1007/s10534-011-9423-8 |
| [19] | Leon-Sicairos N, Canizalez-Roman A, de la Garza M, et al. Bacteri-cidal effect of lactoferrin and lactoferrin chimera against halophilic Vibrio parahaemolyticus[J]. Biochimie, 2009, 91 (1): 133–140. DOI:10.1016/j.biochi.2008.06.009 |
| [20] | Mela I, Aumaitre E, Williamson A-M, et al. Charge reversal by salt-induced aggregation in aqueous lactoferrin solutions[J]. Colloids and Surfaces B-Biointerfaces, 2010, 78 (1): 53–60. DOI:10.1016/j.colsurfb.2010.02.011 |
| [21] | Rahman MM, Kim W-S, Ito T, et al. Growth promotion and cell bin-ding ability of bovine lactoferrin to Bifidobacterium longum[J]. Anaerobe, 2009, 15 (4): 133–137. DOI:10.1016/j.anaerobe.2009.01.003 |
| [22] | 张伟, 任发政, 葛绍阳, 等. 拉曼光谱研究乳铁蛋白及其肽段与DPPC、DPPG脂质体的相互作用[J]. 光谱学与光谱分析, 2011(6): 1533–1536. |
| [23] | Wakabayashi H, Yamauchi K, Kobayashi T, et al. Inhibitory effects of lactoferrin on growth and biofilm formation of Porphyromonas gingivalis and Prevotella intermedia[J]. Antimicrobial Agents and Chemotherapy, 2009, 53 (8): 3308–3316. DOI:10.1128/AAC.01688-08 |
| [24] | Ammons MCB, Ward LS, Dowd S, et al. Combined treatment of Pseudomonas aeruginosa biofilm with lactoferrin and xylitol inhibits the ability of bacteria to respond to damage resulting from lactoferrin iron chelation[J]. International Journal of Antimicrobial Agents, 2011, 37 (4): 316–323. DOI:10.1016/j.ijantimicag.2010.12.019 |
| [25] | Ammons MCB, Ward LS, Fisher ST, et al. In vitro susceptibility of established biofilms composed of a clinical wound isolate of Pseudomonas aeruginosa treated with lactoferrin and xylitol[J]. International Journal of Antimicrobial Agents, 2009, 33 (3): 230–236. DOI:10.1016/j.ijantimicag.2008.08.013 |
| [26] | Ellison RT, Ⅲ Giehl TJ, LaForce FM. Damage of the outer membrane of enteric gram-negative bacteria by lactoferrin and transferrin[J]. Infection and Immunity, 1988, 56 (11): 2774–2781. |
| [27] | Caccavo D, Afeltra A, Pece S, et al. Lactoferrin-lipid A-lipopolysaccharide interaction:Inhibition by anti-human lactoferrin monoclonal antibody AGM 10. 14[J]. Infection and Immunity, 1999, 67 (9): 4668–4672. |
| [28] | Japelj B, Pristovsek P, Majerle A, et al. Structural origin of endo-toxin neutralization and antimicrobial activity of a lactoferrin-based peptide[J]. Journal of Biological Chemistry, 2005, 280 (17): 16955–16961. DOI:10.1074/jbc.M500266200 |
| [29] | Kim WS, Rahman MM, Shimazaki KI. Antibacterial activity and binding ability of bovine lactoferrin against Pseudomonas spp[J]. Journal of Food Safety, 2008, 28 (1): 23–33. DOI:10.1111/jfs.2008.28.issue-1 |
| [30] | Andre GO, Politano WR, Mirza S, et al. Combined effects of lactoferrin and lysozyme on Streptococcus pneumoniae killing[J]. Microbial Pathogenesis, 2015, 89 : 7–17. DOI:10.1016/j.micpath.2015.08.008 |
| [31] | Senkovich O, Cook WJ, Mirza S, et al. Structure of a complex of human lactoferrin N-lobe with pneumococcal surface protein A provides insight into microbial defense mechanism[J]. Journal of Molecular Biology, 2007, 370 (4): 701–713. DOI:10.1016/j.jmb.2007.04.075 |
| [32] | Samaniego-Barron L, Luna-Castro S, Pina-Vazquez C, et al. Two outer membrane proteins are bovine lactoferrin-binding proteins in Mannheimia haemolytica A1[J]. Veterinary Research, 2016, 47 . |
| [33] | Wong H, Schryvers AB. Bacterial lactoferrin-binding protein A binds to both domains of the human lactoferrin C-lobe[J]. Microbiology-Sgm, 2003, 149 : 1729–1737. DOI:10.1099/mic.0.26281-0 |
| [34] | Ostan N, Morgenthau A, Yu RH, et al. A comparative, cross-species investigation of the properties and roles of transferrin-and lactoferrin-binding protein B from pathogenic bacteria[J]. Biochemistry and Cell Biology, 2017, 95 (1): 5–11. DOI:10.1139/bcb-2016-0055 |
| [35] | Zinger-Yosovich KD, Sudakevitz D, Iluz D, et al. Analyses of diverse mammals' milk and lactoferrin glycans using five pathogenic bacterial lectins[J]. Food Chemistry, 2011, 124 (4): 1335–1342. DOI:10.1016/j.foodchem.2010.07.084 |
| [36] | Yu T, Guo C, Wang J, et al. Comprehensive characterization of the site-specific N-glycosylation of wild-type and recombinant human lactoferrin expressed in the milk of transgenic cloned cattle[J]. Glycobiology, 2011, 21 (2): 206–224. DOI:10.1093/glycob/cwq151 |
| [37] | Del Olmo A, Calzada J, Nunez M. Short communication:Antimic-robial effect of lactoferrin and its amidated and pepsin-digested derivatives against Salmonella Enteritidis and Pseudomonas fluorescens[J]. Journal of Dairy Science, 2010, 93 (9): 3965–3969. DOI:10.3168/jds.2010-3152 |
| [38] | Baker EN, Baker HM. A structural framework for understanding the multifunctional character of lactoferrin[J]. Biochimie, 2009, 91 (1): 3–10. DOI:10.1016/j.biochi.2008.05.006 |
| [39] | Morgenthau A, Pogoutse A, Adamiak P, et al. Bacterial receptors for host transferrin and lactoferrin:molecular mechanisms and role in host-microbe interactions[J]. Future Microbiology, 2013, 8 (12): 1575–1585. DOI:10.2217/fmb.13.125 |
| [40] | Le Parc A, Karav S, Rouquie C, et al. Characterization of recombinant human lactoferrin N-glycans expressed in the milk of transgenic cows[J]. Plos One, 2017, 12 (2). |
| [41] | Sojar HT, Hamada N, Genco RJ. Structures involved in the interaction of Porphyromonas gingivalis fimbriae and human lactoferrin[J]. Febs Letters, 1998, 422 (2): 205–208. DOI:10.1016/S0014-5793(98)00002-7 |
| [42] | Kamiya H, Ehara T, Matsumoto T. Inhibitory effects of lactoferrin on biofilm formation in clinical isolates of Pseudomonas aeruginosa[J]. Journal of Infection and Chemotherapy, 2012, 18 (1): 47–52. DOI:10.1007/s10156-011-0287-1 |
| [43] | Diarra MS, Lacasse P, Deschenes E, et al. Ultrastructural and cytochemical study of cell wall modification by lactoferrin, lactoferricin and penicillin G against Staphylococcus aureus[J]. Journal of Electron Microscopy, 2003, 52 (2): 207–215. |
| [44] | Quintieri L, Caputo L, Monaci L, et al. Antimicrobial efficacy of pepsin-digested bovine lactoferrin on spoilage bacteria contaminating traditional Mozzarella cheese[J]. Food Microbiology, 2012, 31 (1): 64–71. DOI:10.1016/j.fm.2012.02.015 |
| [45] | Nibbering PH, Ravensbergen E, Welling MM, et al. Human lactoferrin and peptides derived from its N terminus are highly effective against infections with antibiotic-resistant bacteria[J]. Infection and Immunity, 2001, 69 (3): 1469–1476. DOI:10.1128/IAI.69.3.1469-1476.2001 |
| [46] | Tomita M, Wakabayashi H, Shin K, et al. Twenty-five years of research on bovine lactoferrin applications[J]. Biochimie, 2009, 91 (1): 52–57. DOI:10.1016/j.biochi.2008.05.021 |
| [47] | Duchardt F, Ruttekolk IR, Verdurmen WPR, et al. A cell-penetrating peptide derived from human lactoferrin with conformation-dependent uptake efficiency[J]. Journal of Biological Chemistry, 2009, 284 (52): 36099–36108. DOI:10.1074/jbc.M109.036426 |
| [48] | Moniruzzaman M, Alam JM, Dohra H, et al. Antimicrobial peptide lactoferricin B-induced rapid leakage of internal contents from single giant unilamellar vesicles[J]. Biochemistry, 2015, 54 (38): 5802–5814. DOI:10.1021/acs.biochem.5b00594 |
| [49] | Hwang PM, Zhou N, Shan X, et al. Three-dimensional solution structure of lactoferricin B, an antimicrobial peptide derived from bovine lactoferrin[J]. Biochemistry, 1998, 37 (12): 4288–4298. DOI:10.1021/bi972323m |
| [50] | Ghosh A, Datta A, Jana J, et al. Sequence context induced antimi-crobial activity:insight into lipopolysaccharide permeabilization[J]. Molecular Biosystems, 2014, 10 (6): 1596–1612. DOI:10.1039/C4MB00111G |
| [51] | van der Kraan MIA, Groenink J, Nazmi K, et al. Lactoferrampin:a novel antimicrobial peptide in the N1-domain of bovine lactoferrin[J]. Peptides, 2004, 25 (2): 177–183. DOI:10.1016/j.peptides.2003.12.006 |
| [52] | Haney EF, Lau F, Vogel HJ. Solution structures and model membrane interactions of lactoferrampin, an antimicrobial peptide derived from bovine lactoferrin[J]. Biochimica Et Biophysica Acta-Biomembranes, 2007, 1768 (10): 2355–2364. DOI:10.1016/j.bbamem.2007.04.018 |
| [53] | Haney EF, Nazmi K, Lau F, et al. Novel lactoferrampin antimicrobial peptides derived from human lactoferrin[J]. Biochimie, 2009, 91 (1): 141–154. DOI:10.1016/j.biochi.2008.04.013 |
| [54] | Bolscher JGM, Adao R, Nazmi K, et al. Bactericidal activity of Lfchimera is stronger and less sensitive to ionic strength than its constituent lactoferricin and lactoferrampin peptides[J]. Biochimie, 2009, 91 (1): 123–132. DOI:10.1016/j.biochi.2008.05.019 |
| [55] | Haney EF, Nazmi K, Bolscher JGM, et al. Structural and biophysical characterization of an antimicrobial peptide chimera comprised of lactoferricin and lactoferrampin[J]. Biochimica Et Biophysica Acta-Biomembranes, 2012, 1818 (3): 762–775. DOI:10.1016/j.bbamem.2011.11.023 |
| [56] | Bjorn C, Mahlapuu M, Mattsby-Baltzer I, et al. Anti-infective efficacy of the lactoferrin-derived antimicrobial peptide HLR1r[J]. Peptides, 2016, 81 : 21–28. DOI:10.1016/j.peptides.2016.04.005 |
| [57] | Zweytick D, Deutsch G, Andra J, et al. Studies on lactoferricin-derived Escherichia coli membrane-active peptides reveal differences in the mechanism of N-acylated versus nonacylated peptides[J]. Journal of Biological Chemistry, 2011, 286 (24): 21266–21276. DOI:10.1074/jbc.M110.195412 |
| [58] | Mishra B, Leishangthem GD, Gill K, et al. A novel antimicrobial peptide derived from modified N-terminal domain of bovine lactoferrin:Design, synthesis, activity against multidrug-resistant bacteria and Candida[J]. Biochimica Et Biophysica Acta-Biomembranes, 2013, 1828 (2): 677–686. DOI:10.1016/j.bbamem.2012.09.021 |
| [59] | Viejo-Diaz M, Andres MT, Fierro JF. Different anti-Candida activities of two human lactoferrin-derived peptides, Lfpep and kaliocin-1[J]. Antimicrobial Agents and Chemotherapy, 2005, 49 (7): 2583–2588. DOI:10.1128/AAC.49.7.2583-2588.2005 |
| [60] | Morici P, Fais R, Rizzato C, et al. Inhibition of Candida albicans biofilm formation by the synthetic lactoferricin derived peptide hLF1-11[J]. PLoS One, 2016, 11 (11). |
| [61] | Kondori N, Baltzer L, Dolphin GT, et al. Fungicidal activity of human lactoferrin-derived peptides based on the antimicrobial alpha beta region[J]. International Journal of Antimicrobial Agents, 2011, 37 (1): 51–57. DOI:10.1016/j.ijantimicag.2010.08.020 |