从1962年下村修等在名为Aequorea victoria的水母体内发现并分离得到最早的荧光蛋白绿色荧光蛋白(Green fluorescent protein,GFP)开始[1],人们对于荧光蛋白的进一步研究和应用都没有停止过。研究者通过一系列突变得到各种不同波长的突变体,大大丰富了荧光蛋白的家族成员。如此大量的荧光蛋白使得其在蛋白质分子标记和细胞内示踪等方面得到了大量的应用。
在红色荧光蛋白发现以前,GFP虽然能帮助生物科学家解决一些问题,但由于其发射光谱仅仅局限在440-529 nm,细胞内成像时背景较高,不能够解决活体生物皮下更深的荧光标记问题。1999年红色荧光蛋白首次被报道[2],红色荧光蛋白能够与GFP共用,激发和发射波长更长,最重要的是其在细胞内成像时背景低。这些优点促使科学家们对红色荧光蛋白进行了系列研究,极大的完善了其多样性。
研究者在应用红色荧光蛋白的时候也发现了一些问题,如野生型的红色荧光蛋白成熟速度慢且多以四聚体或二聚体形式出现,对细胞有一定的毒性作用[3-4]。红色荧光蛋白的这些问题影响了被标记物的功能和性质,因此对野生型红色荧光蛋白进行改造就成了必不可少的步骤,而改造的目的主要针对红色荧光蛋白的成熟慢,易聚合等缺点。此外,还要进一步研究荧光强度更高,能够进一步丰富荧光蛋白发射光谱的更适用于生物荧光标记的荧光蛋白。对以上内容进行简要综述,旨为以后红色荧光蛋白的进一步改造提供参考与帮助。
1 红色荧光蛋白的发现及其发展1962年,下村修等在一种名为维多利亚多管水母(Aequorea victoria)体内发现了绿色荧光蛋白(Green fluorescent protein,GFP),这是荧光蛋白首次被发现。从水母体内发现的GFP由238个氨基酸构成,其中65-67位氨基酸为发光团,可以被光激发产生荧光[5-6]。钱永健先生[7]在GFP的基础上进行了一系列的体外突变产生了荧光强度更强以及发射波长不同的多种突变体,如蓝色、青色、黄色荧光蛋白都是体外突变的产物。也因此在2008年,钱永健等三位科学家因发现并发展了GFP而获得了诺贝尔化学奖。
GFP作为能够在细胞内精确定位的遗传标签,结合如今飞速发展的成像技术和数据分析技术使得其在生物科学领域的应用飞速发展。GFP蛋白没有毒性,在目标生物体内能够高效表达的同时不影响生物体的功能,作为标记蛋白时GFP在不影响目标蛋白的活性的同时还能保持其荧光[8],这也是GFP能够被广泛应用于生命科学研究的重要原因之一。GFP各种突变体的出现又能够不同程度的满足科学家对于荧光蛋白的要求,目前应用最广的就是其中一个突变体EGFP。然而,这系列荧光蛋白的发射光谱局限在440-529 nm之间,在细胞内成像时能够激发细胞内的某些物质成像,造成细胞内成像时的背景较高。使得实验结果不够可靠。
1999年红色荧光蛋白首次被报道,与绿色荧光蛋白相比其优点显而易见。首先红色荧光蛋白可以和GFP共同使用解决一些GFP单独解决不了的科学问题;其次作为红色荧光蛋白其激发和发射波长更长可以覆盖GFP所不能涉及的波长段;最重要的是红色荧光蛋白在细胞内成像时背景低更适用于进行生物科学的研究。正因为红色荧光蛋白的这些优势,它在生物学研究中迅速得到了大量应用[9-10]。红色荧光蛋白分子与水母绿色荧光蛋白有中等的序列相似性,具有相似的结构基础[11],都是在由11个β-折叠形成的桶状结构中心的螺旋形成固有发色团。
最早用于研究的红色荧光蛋白是DsRed,它是一种从珊瑚中分离出来的红色荧光蛋白,具有荧光强、稳定性好等优势。但其本身也存在许多问题。例如,DsRed寡聚状态是四聚体,在进行蛋白融合时容易形成多聚体影响目标蛋白;DsRed在细胞内表达会产生毒性。这就促使科学家不得不对其进行结构改造,目前,至少6种不同的红色荧光蛋白类型,包括常规荧光蛋白、远红外荧光蛋白、具有斯托克斯位移的红色荧光蛋白、荧光定时器、可光活化红色荧光蛋白和可逆光开关红色荧光蛋白。这些类型中的每一种都能应用于不同的成像技术,但是每种红色荧光蛋白又都有局限性使其不能广泛应用。
2 红色荧光蛋白的种类及其相关应用的介绍目前已知的红色荧光蛋白类型就有6种之多,其中不少是由最初的红色荧光蛋白突变而来的,这里简要介绍了有关红色荧光蛋白突变体的结构研究,以及一些具有特殊性质的红色荧光蛋白的应用,相信以后对于这两方面的进一步研究能够使红色荧光蛋白的家族更加壮大。
2.1 常规红色荧光蛋白常规红色荧光蛋白也叫做永久荧光蛋白,可以分为橙色荧光蛋白(550-570 nm)红色荧光蛋白(570-620 nm)和远红外荧光蛋白( > 620 nm)3种。其中远红外荧光蛋白我们将分开讨论,以突显其在荧光成像中的重要性。
荧光蛋白在细胞标记中的适用性主要取决于其细胞毒性。只有降低荧光蛋白的细胞毒性保证高的细胞存活率,才能保证荧光蛋白稳定高效的表达。而目前荧光蛋白的细胞毒性主要是由于其在细胞内的非特异性聚合引起的,野生型的红色荧光蛋白就具有明显的寡聚现象。所以细胞毒性较小的突变体可以通过优化蛋白质的溶解性获得,这种方式对于四聚体的红色荧光蛋白同样适用,如DsRed、E2-Orange和DsRed-Express2已经通过这种方式得到了优化[12]。一般情况下红色荧光蛋白的寡聚确实会对细胞产生毒性使得实验结果不准确,但是这种特性也是可以开发和预测并加以利用的,Dash等[13]就利用这种性质优先将未配体的核受体从核转移到细胞质隔室然后再由其配体转入细胞核,这种能够将缺点进行开发和利用的做法就更加丰富了红色荧光蛋白的应用范围。
现代红色荧光蛋白与第一代常规红色荧光蛋白相比有增强的荧光特性。例如,mOrange2和TagRFP-T[14]与它们的祖先mOrange和TagRFP[15]相比其光稳定性更强。其他橙色荧光蛋白,如mKO2[16]和mKOk[17]与其祖先相比更明亮,具有更快的成熟时间,然而,它们的光稳定性较低。最近Bindels等[18]研究产生的mScarlet为单体红色荧光蛋白,与其他红色荧光蛋白相比更适合作为融合标签。2016年,Pandelieva等[19]在mRojoA的发色团之上引入芳香残基降低发色团的构象灵活性,这种突变使得其比前体的荧光蛋白量子产率提高了3倍以上。而且通过对突变体的晶体结构研究表明,发色团夹在芳环三层基序中的两个Tyr残基之间,这种基序的存在增加了发色团的刚性。Pandelieva提出的方法为具有较高量子产率和总体亮度的荧光蛋白的发展提供了一个选择。尽管现代红色荧光蛋白在某些特定应用方面有一定的优势,但一些早期的红色荧光蛋白是仍然存在竞争力的。2016年,Fan[20]报道的rxRFP1对于氧化还原反应敏感,能够用于研究各亚细胞域的氧化还原动力学。通常mCherry是作为标签的好选择,因其光稳定性更强,且其具备良好的单体特性,细胞毒性低[21]。2015年,Wannier等[22]通过计算设计的手段结合表面突变,创建了一种DsRed和mCherry的混合RFP不仅保持了单体性而且荧光不受损,相信这种方法对于以后的荧光蛋白改造有很大的意义。
从结构上看突变体mCherry能够具有如此强的竞争力有两个原因。第一个原因是,mCherry中的第163位Lys已经突变成了Gln,这个突变的氨基酸所在的侧链位于发色团苯环下方,在结构中能够清楚的看到163位的Lys与发色团的苯环之间由氢键相互作用。荧光蛋白中的发色团是去质子化的,主要原因在于发色团在蛋白质内部与蛋白所处环境的吸光度有所不同,据此我们相信163位的Lys是被质子化的,也因此通过氢键与发色团相互联系。这种相互作用能够稳定DsRed的电子密度,降低了发色团苯环的活跃性。而突变体中用中性的Glu替代酸性的Lys,这将导致这个位点与发色团的距离增加,从而维持一个相对较弱的氢键。这就引起发色团处于一个相对活跃的状态,从而能够产生更亮的荧光。第二个原因是83位的Lys突变为Leu。有一个荧光亮度增强的突变体,在这个位点上也发生了突变不过将Lys突变为Met,在后者突变体的结构中能够看出79位Lys向着Met的方向移动,这个方向刚好是远离发色团的方向。在mCherry中也观察到70位Lys有类似的变化,因此可以认为70位的Lys能够帮助发色团处于稳定状态,而Lys的远离能够使其处于一个活跃的状态,在激发荧光时产生更亮的荧光[23-24]。如图 1所示显示了突变体mCherry发色团与其周围环境的相互关系。
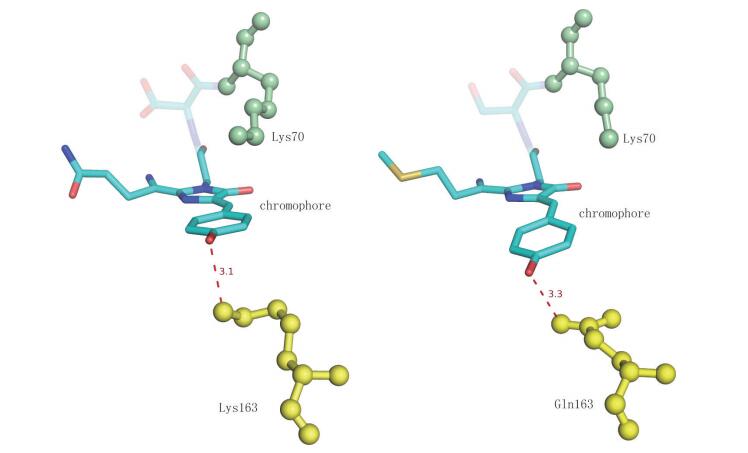
|
| 图 1 突变后的mCherry发色团与其环境相互关系 (左:DsRed;右:mCherry) |
研究具有600 nm以上发射波长的荧光蛋白主要有两个目的:首先,在生物成像技术中很需要能够在近红外(Near infrared,NIR)窗口(650-900 nm)内具有发射光的荧光蛋白,因为光照射穿透组织时,其中的生物分子如黑色素、血红蛋白和水,在近红外窗口的光具有最小的吸收;其次,这种荧光蛋白能为多波长成像技术提供更多的选择,其应用于生物体内的成像技术能产生更少的光毒性。荧光蛋白本身不能够在近红外窗口产生激发光,目前为止报道的天然红色荧光蛋白最长发射光波长为613 nm,是来自Nematostella vectensis体内的NvFP-7R[25]。
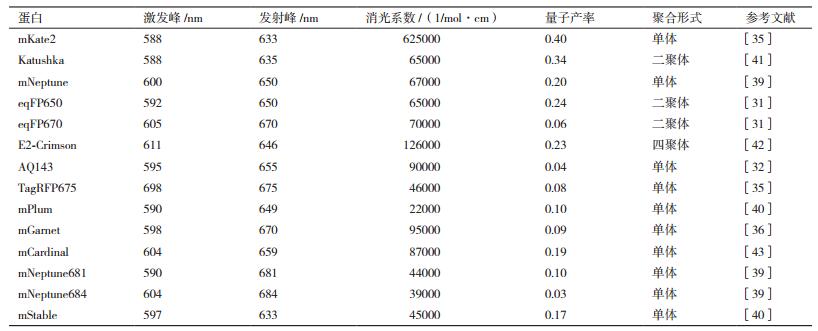
经过设计和突变得到了许多能够在近红外窗口产生激发光的荧光蛋白突变体,但都具有低产量或者低亮度等缺点[26-28]。这其中包括2005年从来自Actinia equine的aeCP597突变产生的AQ143,具有在595-655 nm的激发和发射最大值[29, 30]。2014年,由Archaerhodopsin-3突变产生的突变体具有迄今为止最远的红移发射[31]。2013年,经mKate突变产生的稳定性明显增强的突变体TagRFP675[32]。经mRuby突变产生的mGarnet在670 nm处产生激发光[33]。2015年,Yu等[34]通过对新近的单体红色荧光蛋白mIFP进行合理的设计突变使其产生新的突变体iBlueberry,这是一种蓝移突变体,其激发和发射光谱,约40 nm的光谱移动。iBlueberry除保持单体状态外,还具有比其母体稳定性高4倍的特性。2016年,Bajar等[35]经过26个突变最终得到mMaroon1,细胞毒性小,且具有红移吸光度,用于研究细胞周期的4个阶段取得了很好的效果。同年Li等[36]通过对mNeptune进行突变以开发新的荧光蛋白,在众多突变体中mNeptune681和mNeptune684相对于其母体显示出超过30 nm的红移发射,并且这两种突变体还保持着单体状态。Ren等[37]经过在mKate2的发色团附近引入半胱氨酸得到新的远红外红色荧光蛋白mStable,该突变体比其前体稳定性增强12倍,他们还对mPlum进行了同样的改变得到了稳定性增强23倍的突变体,这种突变代表了一种生产更好稳定性荧光蛋白的一种机制。表 1给出了目前常见的几种远红外红色荧光蛋白及其特征参数。
从结构方面分析这些突变体发生红移的原因主要有两方面。第一,发色团的平面化增强。从Neptune的结构来看其中的发色团平面性有明显的增强。在Neptune中,197位Arg的侧链与其上面的发色团中的两个环相互平行。这个变化可能是由于其中158位Cys突变成Ser引起的,突变后的氨基酸残基侧链占领了本来由197位Arg占有的空间。顺着Arg的延伸方向发色团中的两个环更接近共平面(图 2左侧),这种变化能够使电子位移容易发生,降低激发所需能量,从而使突变体的荧光范围发生红移。这种变化也能够在其他突变体中发现,比如eqFP650和eqFP670。第二,氢与酰基亚胺氧的结合。从Neptune的结构中可以看出,由于41位Met突变为Gly形成了一个含有水分子的空穴(图 2右侧),水分子与发色团中的酰基亚胺氧形成氢键,氢键的形成能够富集电子降低激发荧光所需能量。这种情况在eqFP650和eqFP670中同样也有发现,这说明41位Met突变为小侧链有助于红移的发生。
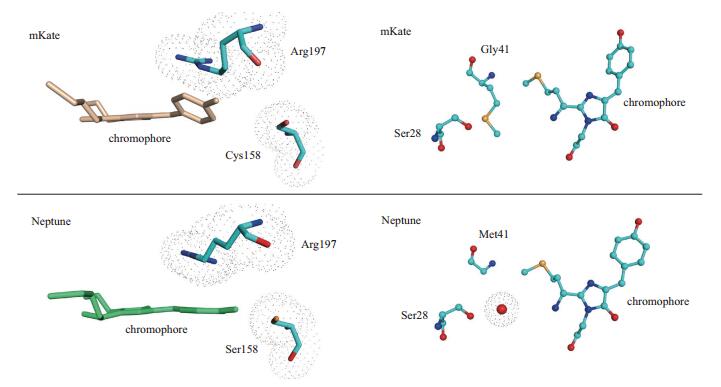
|
| 图 2 Neptine与mKate相比较发生红移的结构基础 |
此外,已经开发了几种近红外荧光蛋白,如最近在细菌植物色素的基础上突变得到的IFP1.4[41]和iRFP[42]。这些蛋白的荧光是依赖于外源四吡咯发色团的,称为胆绿素,在哺乳动物细胞中含量比较多。2013年产生的融合蛋白mPlum-IFP 1.4能够用于体内和体外的检测[43]。来自水母或珊瑚的荧光蛋白在产生激发光的同时需要氧气,并且在发色团形成时产生过氧化氢,因此需要对有氧环境具有耐受性[44]。发色团形成可能需要数小时,并且在其产物中绿色和红色荧光的混合物是常见的[45],在此过程中产生的过氧化氢是细胞存活、生长、分化和介导疾病的诱因[46],这些有可能导致实验结果不可信。因此,使用荧光蛋白内源发色团以消除需氧性和过氧化氢[47]的产生是有必要的。2016年,Rodriguez等[48]用TeAPCα经过突变产生了符合要求的突变体smURFP,smURFP在生物物理上是最亮的荧光蛋白,并能够激发近红外窗口内的激发光,且具有最小的毒性不产生过氧化氢。此外,不像其前体TeAPCα smURFP不需要裂解酶共价附着其发色团。经过一系列的体内或细胞实验,可以看出,与mCherry相比,smURFP没有出现聚合的情况,与目前常用的一些荧光蛋白相比,smURFP在细胞内能够更稳定的表达。目前smURFP能够有这些特性的原因还有待发现,希望能够通过解析smURFP的结构来解释这一变化。并且目前smURFP产生荧光依赖于其发色团胆绿素的结合,考虑到在原核生物(厌氧型)中smURFP不能方便的使用,对此进行研究和改进也是日后的一个目标。
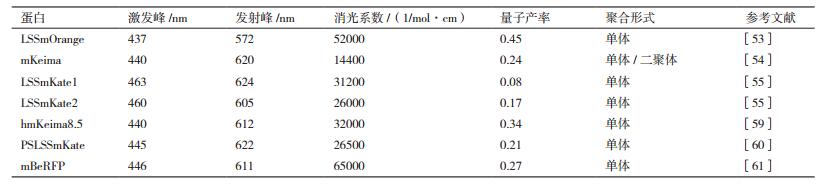
2.3 具有大斯托克斯频移的红色荧光蛋白大斯托克斯频移(Large stokes shift,LSS),是指激发和发射最大值之间的差大于100 nm(图 3),是荧光蛋白中具有独特特征的亚类,称为具有大色托克斯位移的荧光蛋白(Large stokes shift fluorescent protein,LSSFP)。LSSFP为多色成像应用提供光谱可分辨的颜色。除了绿色和黄色LSSFP[49]之外,还有橙色LSSFP-LSSmOrange[50]和3种红色LSSFP:mKeima、LSSmKate1和LSSmKate2[51-52]。2013年发现的青色荧光蛋白psamFP488也具有显著扩展的斯托克斯位移[53]。所有红移LSSFP光吸收值在440-460 nm并且激发荧光范围在光谱中红色部分。LSSmOrange在红色LSSFP中具有最高强度和最亮荧光[54]。LSSmKate2在pH稳定性、光稳定性和亮度方面胜过mKeima,它缺乏黄色光谱区的额外激发峰[55]。2015年,的突变体hmKeima8.5[56]具有更高的细胞内亮度,且其斯托克斯位移增加至约180 nm,这些性质使其更适用于生物科学研究。LSSmKate2具有较低的细胞毒性,能够在哺乳动物细胞内稳定的表达。2014年,LSSmKate的一个突变体PSLSSmKate具有可光活化的特性[57],相信通过对其结构的进一步研究能够指导我们进一步改善使其能够用于光活化成像。2013年,由mKate突变获得了一种新的LSSFP:mBeRFP,其中包括5个氨基酸突变。原始的mKate发色团中的68位Tyr比较靠近发生突变的147位Ser和162位Glu,假定发色团与162位Glu能够形成氢键从而使发色团处于相对稳定的中性环境,而突变可能改变了这种稳态,使得其性质发生变化,具体的原理还有待进一步研究[58]。2014年,Pletnev等[59]研究了LSSmOrange的晶体结构发现,其中唯一的突变Ile161Asp对于其性质的改进以及超大的斯托克斯位移有重要的意义。
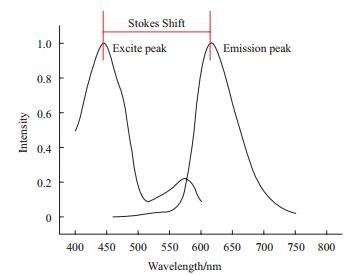
|
| 图 3 斯托克斯位移示意图 |
LSSFP的光谱特性对于使用单一的激发波长激发的多色应用是有利的。荧光互相关光谱(Fluorescence cross-correlation spectroscopy,FCCS)是一种可变的荧光相关光谱(Fluorescence Correlation Spectroscopy,FCS),其被优化用来研究相对分子量相似的蛋白质之间的相互作用。在双色FCCS中,两个信号之间的互相关程度与相互作用对的百分比相关。与FRET相反,FCCS不依赖于接近度和荧光蛋白之间的有利取向[60]。单激光双色FCCS比双激光器设置更有优势因为它不需要两个激光器精确对准共同共焦点。红色LSSFP,mKeima与青色荧光蛋白(Cyan fluorescent protein,CFP)一起能够被同一激光有效地激发产生双色FCCS。这一战略已经扩大到实现四色FCCS。对于FRET红色LSSFP是很好的探针。LSSFP是远红外荧光蛋白的假定供体和蓝色荧光蛋白的潜在受体。红色LSSFP供体激发光谱和红色LSSFP受体发射光谱的较大差异在相应的FRET组合中能够产生低背景和高灵敏度FRET测定[61]。基于LSSFP的FRET对也可以与其他FRET对结合使用,如LSSmOrange-mKate2 FRET对与CFP-YFP(Yellow fluorescent protein)FRET生物传感器一起使用在单个细胞中同时成像两个过程。2016年,Laviv等[62]对应用于FRET的荧光蛋白对进行了优化新开发的荧光蛋白对中采用能够激发红色荧光的LSSFP的mCyRFP1。LSSFP在生物学研究中的应用尤为重要,但是目前的LSSFP亮度普遍较低,而且在使用过程中只能与CFP联合使用,所以寻找能够与亮度最大的GFP联合使用的LSSFP成为了急需解决的问题。表 2给出了几种LSSFP及其特征参数。
荧光计时器(Fluorescent timers,FT)蛋白可以随着时间改变其荧光颜色。这个现象是基于其较慢的发色团形成过程,中间发色团和最终发色团在不同的光谱范围内形成荧光。可预测的颜色变化时间过程使得体内的时间和空间变化能够定量分析[63]。关于其颜色变化的机制还有待进一步的研究。与下节描述的适用于研究快速细胞内动力学研究的可光活化荧光蛋白相反,荧光计时器通常用于研究相对较慢的变化。
目前有两种类型的荧光计时器,第一种是mCherry衍生的单体快速(Fast-FT)、中等(Medium-FT)和慢速(Slow-FT)荧光计时器,随着时间的变化它们的荧光从蓝色变化到红色。蓝色和红色FT亮度较高,具有pH稳定性,并且能够完成蓝色到红色荧光的转换,但是这种FT需要在强紫外光下使用,这导致其在生物学研究中的使用变得复杂。2015年,Takamura等[64],通过体外重组FT碎片研究其颜色变化以及速率的方式对活细胞中的α-突触核蛋白的聚集过程进行了可视化研究。另一种类型的FT是单体Kusabira Green Orange(mK-GO)其颜色随时间从绿色变为橙色。与mCherry衍生的FT相反,两种形式的mK-GO独立成熟。绿色形式的mK-GO在橙色形式的mK-GO成熟后不消失。完全成熟后橙-绿两种形式的比例从约0.1变为0.7。图 4表示了这两种FT的基本原理。这种类型的FT在荧光的敏感程度不如第一种类型的FT。单体FT可以用于估计它所标记的目的蛋白的年龄。能用Medium-FT和mK-GO[65]确定参与自噬和内吞作用的组分的年龄。目前这两种类型的FT都存某些缺点,限制了其在生物科学研究领域中的顺利应用,因此接下来的研究任务是改进其应用的方便性及增加其荧光敏感性。
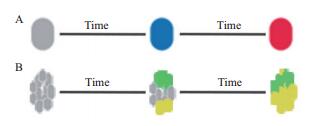
|
| 图 4 传统荧光计时器示意图 A:mCherry衍生型;B:mK-GO灰色代表未成熟荧光蛋白;蓝色、红色、绿色、橙色显示随着时间荧光蛋白的颜色变化 |
2012年,Khmelinskii等[66]通过串联荧光蛋白定时器(Tandem fluorescent protein timers,tFT)来测量细胞过程中的动力学反应。其原理是根据两个串联的荧光蛋白在发色团成熟过程中存在的时间差,整个检测过程中不同时间被检测细胞显示的颜色有所差异,也因此可以判定被检测蛋白的年龄及状态。图 5表示了这种串联荧光定时器的基本原理。tFT与定量双色荧光显微镜成像结合使用时能够提供从亚细胞到有机体的信号传导活动的详细“快照”读数,对于研究胚胎发育的研究具有重要的意义[67]。而在之后许多科学家也对此进行了研究[68-69],其中使用最多是成熟较慢的红色荧光蛋白mCherry与成熟较快的绿色荧光蛋白(Superfolder green fluorescent protein,sfGFP)串联形成的tFT。在2016年,Khmelinskii等[70]又通过不同的绿色荧光蛋白与mCherry串联研究了绿色荧光蛋白成熟动力学对tFT时间范围的影响,研究表明,实验过程中的不完全蛋白酶降解会影响到tFT的时间检测范围,所以在进行实验时有必要通过研究蛋白酶体降解来进一步精确tFT的检测范围。

|
| 图 5 串联荧光定时器示意图 黑色和灰色:未成熟荧光蛋白;绿色:成熟绿色荧光蛋白;红色:成熟红色荧光蛋白;绿色、红色、黑色曲线:绿色、红色荧光蛋白、tFT随时间荧光强度变化 |
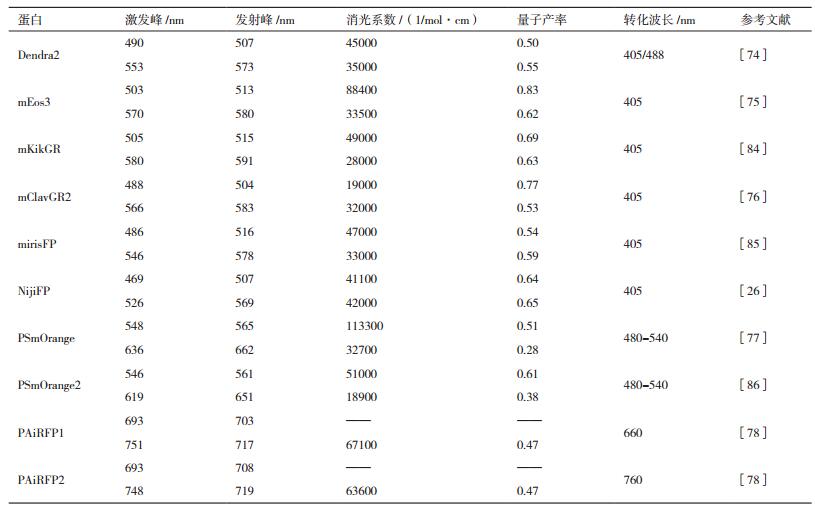
能够由光调节其荧光性质的荧光蛋白适合用于各种选择性成像标记技术,从粒子跟踪到超分辨率成像都适用,具有这种性质的荧光蛋白称为可光活化荧光蛋白(Photoactivatable FP,PAFP)。PAFP的重要特性主要包括两方面,首先是光活化对比度;其次是荧光蛋白在初始状态和光活化状态的亮度。光活化对比度是光活化后的荧光强度和光活化前的荧光强度的比率。较高的对比度能够产生更大的信号与背景比;另一个要考虑的参数是光活化时所需的光强度:不应该高到对细胞有害,同样也不能低到可以自发发生光活化。目前有3种类型的不可逆红色PAFP(图 6):从暗到红可光活化PAmCherry样红色荧光蛋白,如PAmCherry和PATagRFP,其中的PATagRFP在亮度和光稳定性方面比PAmCherry更好[71];从绿到红可光活化Kaede样红色荧光蛋白,如Dendra2[72]、mEos3[73]和mKikGR是目前比较常用的PAFP,在2010年的一个突变体mClavGR2 PSFP是一个亮度和光转化对比度更加优秀的PAFP[74];以及最近发现的从橙到远红可活化荧光蛋白PSmOrange[75],目前为止其具有最远的红色激发峰。2013年产生的PAiRFP1和PAiRFP2能够在近红外窗口产生激发光应用于组织内部检测[76]。
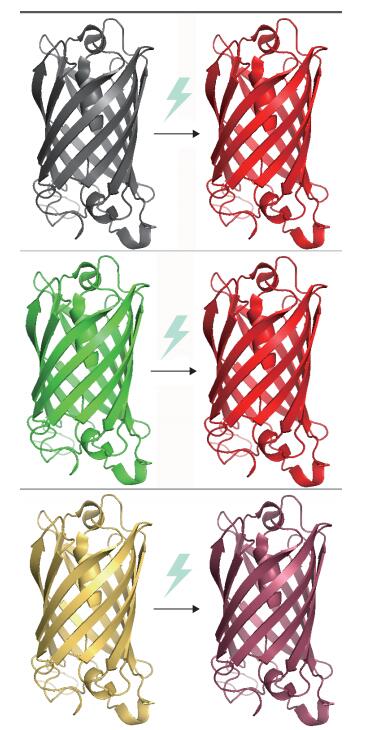
|
| 图 6 PAFP原理示意图 从上到下依次表示:从暗到红PAFP,从绿到红PAFP,从橙到红PAFP |
PAFP可以用来标记目标蛋白、特定细胞器或细胞。用PAFP标记蛋白一般用于研究蛋白动力学和转运。细胞器光标记能够监测裂变和融合。细胞追踪适用于研究发育、癌变和炎症反应[77]。PAFP的特性使其适合于在体内的长期跟踪检测。在FRET成像中也可以使用PAFP,可以基于一个简单的方法在一个细胞区室中特定PAFP的光活化然后监测PAFP供体和常规红色荧光蛋白受体之间不同的细胞区域FRET。PAFP表型对光活化定位显微镜(Photo-activated localization microscopy,PALM)[78-79]和荧光PALM(Fluorescent photo-activated localization mic-roscopy,FPALM)超分辨率方法至关重要。PALM依赖于单分子成像,单分子亮度以及背景和光活化后光子输出之间的对比度在这项技术中是关键的荧光蛋白特性。在PALM方法中,较高的对比度引起较高的空间分辨率,因此其成像伴随较低的自发荧光背景的红色荧光蛋白是优选PALM探针[80],这些蛋白不产生绿色荧光因此能够与绿色PAFP相结合使用达到更高的分辨率。值得一提的是,2014年的诺贝尔化学奖就表彰了Eric Bctzig等3位科学家在超高分辨率荧光显微镜上的贡献。PAFP的特性影响着PA-LM图像质量,主要包括每个周期发射的光子数量、蛋白的二聚化现象以及蛋白的信号效率。目前已知的各种PAFP都有很高程度的二聚化现象这就导致其信号效率变低影响PALM的图像质量[81],生产出没有二聚化现象的PAFP是今后改造工作的首要目标。表 3列举了几种常见的可活化红色荧光蛋白。
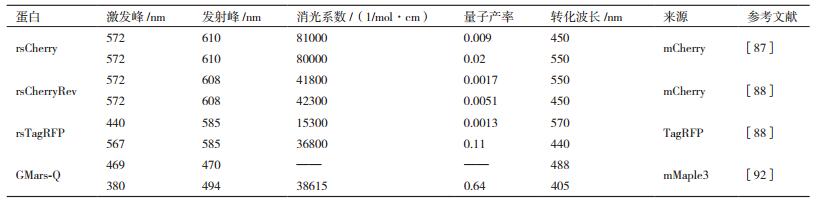
2.6 可逆光开关红色荧光蛋白可逆光开关荧光蛋白(Reversibly switchable fluorescent protein,rsFP)可以在两者之间重复光开关荧光和非荧光状态。这种属性能够促使产生新的成像技术。2009年发现的红色rsFP包括rsCherry[85]、rsCherryRev和rsTagRFP[86]。用550-570 nm光照射rsTagRFP能够关闭红色荧光状态使其在567 nm的光吸收特性变成在440 nm有光吸收的暗态,而照射430-450 nm光将暗态切换回荧光态。图 7显示了rsFP的基本原理。rsFP的这种特性有可能是由于用相应波长的光照射能够使其发色团发生顺-反光异构化,伴随着质子化与去质子化产生亮暗的转化。
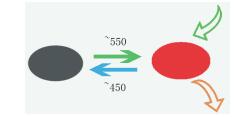
|
| 图 7 rsFP原理示意图 黑色:暗态;红色:荧光态 |
可逆地荧光转化表型可以为超分辨率技术提供优势。然而,在实践中使用rsFP的超分辨率成像受到荧光蛋白缺乏“耐疲劳”的限制。此外,即使亮度和对比度最好的可用红色rsFPs都不如那些红色PAFP和PSFP效果好[87]。到目前为止,已经报道了5种基于rsFP的超辨率技术,可逆饱和光学(荧光)转换(Reversible saturable optical fluorescence transitions,RESOLFT)、PALM运行采集(PALM independently running acquisition,PALMIRA)、活细胞纳米镜(Live-cell fluorescence nanoscopy)[88]和PALM与双色频闪照明(Two-color troboscopic illumination PALM,S-PALM)显微镜,以及2015年出现的光致变色随机光波动成像(Photochromic stochastic optical fluctuation imaging,pcSOFI)[89]。2016年,Wang等[90]报道了GMars-Q,这是一种具有低残留强度和较强的抗疲劳特性,这种抗疲劳特性归因于双相光漂白过程。这种rsFP的性质对于RESOLFT是非常有利的。红色rsFP已应用于RESOLFT对细菌成像,使用的是四聚体asFP595rsFP。2014年,Lavoie-Cardinal等[91]将一种新的红色荧光蛋白rsCherryRev1.4用于RESOLFT成像,获得了高出衍射屏障4倍的空间分辨率。表 4给出了几种常见的可逆光开关红色荧光蛋白及其特征参数。
目前红色rsFPs仍然具有限制其广泛应用的不足,包括其较低的耐疲劳性,较慢的光转化效率以及其较差的稳定性,但是科学家们已经通过改造得到了相对优秀的绿色rsFPs突变体[92]。例如,2016年出现的rsFolder和rsFolder2是由rsFolder是通过Superfolder-GFP与rsEGFP2的杂交设计的,能够用于有效的研究细胞室内的研究[93]。同样,由Smyrnova等[94]改进的突变体rsFastLime(Dronpa V157G)和rsKame(Dronpa V157L)在转化速度方面具有很大的优势,这两种突变体都通过异构化进行转化。并且通过功能模式分析得出其中的Val / Leu 157和α-螺旋中的氨基酸在异构化过程中有很重要的作用。而2015年出现的Skylan-S具有很高的光稳定性[95]。Morozov等[96]通过对Dronpa快速切换Met159Thr突变体的分子动力学模拟的研究指出,以后在进行新的rsFP的研究时除了关注结构方面的还要关注异构化方面的研究。有这些优秀的突变体以及其结构性质方面的研究作为参考,相信性质更加完善的红色rsFPs的改造能够很快的出现辅助于改善RESOLFT和PALMIRA性能,并使这些技术广泛应用于研究具有超分辨率精确度的细胞内过程。
3 发色团研究荧光蛋白颜色的多样性是因其发色团多样性以及蛋白所处化学环境的多样性导致的必然结果。由于科学家对于荧光蛋白的研究和改进,科学家们在研究先进荧光蛋白的同时也对其发色团的结构和形成过程进行了研究,而发色团自催化和光诱导化学的基本原理已经为人们所知[97]。这无疑为操纵发色团结构之间的跃迁提供了可能性,相信日后我们会获得新的荧光蛋白表型。这里主要介绍红色荧光蛋白的发光团。迅速发展的成像应用促进了红色荧光蛋白探针的改进。基于对红色荧光蛋白发色团的发色原理的了解,科学家们对红色荧光蛋白进行了有效的改进。用于红色荧光蛋白改造的方法有很多种包括有晶体学方法[98]、突变、同位素研究、质谱[99-101]等方法都应用于荧光蛋白的改进。我们完全可以应用这些手段制定合理的改进策略。
红色荧光蛋白的发色团中的三肽由固定的66位Tyr和67位Gly以及可变的65位氨基酸残基构成。其中核心4-对羟基亚苄基-5-咪唑啉酮结构在红色荧光蛋白是共有的发色团组成部分,而65位氨基酸残基的变化是导致其光谱范围变化的原因。在多种多样的发色团中还有一种称为光敏剂的发色团,这种发色团在被光照射时能够产生活性氧,可以用于靶蛋白的精确光诱导失活、DNA损伤和细胞杀伤。经过晶体结构的研究表明GFP样光敏剂中的独特结构特征是沿着β-桶轴从发色团延伸到筒体末端的充水通道[102]。2015年,Pletneva等[103]通过对二聚KillerOrange和单体mKillerOrange的晶体学研究发现在两种蛋白质中,发色团的Trp66通过与其附近的Gln159形成氢键来稳定一种不寻常的反式-顺式构象。这种反式-顺式构象和水通道是这两种橙色光敏剂产生明亮的橙色荧光和光毒性的关键结构特征。目前已知有两种类型的红色发光团:DsRed样和Kaede样发光团。有自催化或者三肽的光诱导转化,Kaede样发光团三肽只能产生光化学,而红色荧光蛋白的不同色调由DsRed样发色团的进一步转化产生。
目前,包含有DsRed样发色团的红色荧光蛋白家族主要有3个:DsRed的衍生物、eqFP611和eqFP578。研究显示,DsRed样发色团的成熟过程,首先包含有mTagBFP[104]发色团的N-酰基亚胺C = N的形成,然后Tyr66侧链的Cα-Cβ双键形成。只有一个DsRed样红色荧光蛋白通过GFP样生色团中间体成熟。关于几个具有不同的表型荧光蛋白的研究表明其具有mTagBFP样蓝色中间体的结构。
第二种类型的红色发色团,Kaede样发色团,发现于红色状态的PSFP(Photoswitchable fluorescent proteins)。在绿色态下,这些荧光蛋白具有GFP样发色团。用紫光照射后,β消除反应引起绿色至红色的光转化反应。这个过程中65位的His N-Cα键被切断,侧链的Cα-Cβ双键形成,导致π-共轭系统延伸并得到Kaede样红色发色团。
对于光可控的荧光蛋白发色团的研究目前有一些进展,光控的荧光蛋白分为可逆和不可逆的光活化,在可逆光转化中其发色团的原理包括顺反式异构化,质子化-去质子化和脱水缩合。而不可逆的光转化包括发色团内的共轭π系统,相邻Glu的脱羧和多肽主干的断裂。但是目前依然不清楚可逆光开关抗疲劳的化学基础,而在这方面的改进是现在生物科学研究技术中急需的。
基于目前已知的红色发色团、其衍生物以及其中的过渡态的结构数据可以得出以下结论:首先,发色团结构之间的化学转变可以发生自催化,光诱导或者被阻塞。第二,发色团可以处于荧光状态或色原态。第三,发色团中的氨基酸残基及其微环境决定发色团结构之间的转变以及所产生的荧光蛋白表型。电子结构计算和模型的化学合成与结晶学共同应用、质谱、生物化学和光物理表征等技术的进一步研究,将助力于进一步阐明发色团的化学和光谱行为。
4 展望人们对红色荧光蛋白结构和功能的理解在过去几年里已经大大提高了,但仍有许多争议和谜团需解决。在现有知识的基础上,可以设计和改进以得到具有新的光化学性质的红色荧光蛋白。目前对荧光蛋白的激发态的理论研究已经有许多了,我们仍在努力阐明发射荧光的调节机制。
对于红色荧光蛋白的研究不仅是理性工程,也有可能是将随机诱变与高通量筛选技术相结合来达到使红色荧光蛋白分子有效进化的目的。当然,不排除新的克隆或者新发现的红色荧光蛋白具有前所未有的光化学性质。正如Tsien[105]博士在1999年所说:“这些分子工具的普及使得欣赏阳光和珊瑚虫动物体内的荧光蛋白之间的相互作用变得重要,并且促使我们考虑如何使这些独特的蛋白在生物学研究中得到最佳应用”。通过一些优秀突变体的结构分析我们了解了一些能够引起荧光蛋白性质变化的位点,结合种类繁多的红色荧光蛋白,配合红色荧光蛋白的应用相信可以设计和生产出令人满意的突变体。
新出现的红色荧光蛋白肯定会刺激许多生物学家的想象力,反之也激发了生物学家们对于开发新的突变体和新技术的需求,使他们有高涨的研究热情。当然与此同时,荧光显微镜将不可避免地不断配备特殊的硬件和软件功能来配合这些红色荧光蛋白的使用。例如,用于深层组织和活体成像的显微镜的进化,是实现双光子激发红色荧光蛋白充分利用的必要条件[106]。
| [1] | Prasher DC, Eckenrode VK, Ward WW, et al. Primary structure of the Aequorea victoria green-fluorescent protein[J]. Gene, 1992, 111 (2): 229–233. DOI:10.1016/0378-1119(92)90691-H |
| [2] | Matz MV, Fradkov AF, Labas YA, et al. Fluorescent proteins from nonbioluminescent Anthozoa species[J]. Nat Biotechnol, 1999, 17 (10): 969–973. DOI:10.1038/13657 |
| [3] | Strack RL, Strongin DE, Bhattacharyya D, et al. A non-cytotoxic DsRed variant for whole-cell labeling[J]. Nature Methods, 2008, 5 (11): 955–957. DOI:10.1038/nmeth.1264 |
| [4] | Tao W, Evans B, Yao J, et al. Enhanced green fluorescent protein is a nearly ideal long-term expression tracer for hematopoietic stem cells, whereas DsRed-express fluorescent protein is not[J]. Stem Cells, 2007, 25 (3): 670–678. |
| [5] | Yang F, Moss LG, Phillips GN Jr. The molecular structure of green fluorescent protein[J]. Nat Biotechnol, 1996, 14 (10): 1246–1251. DOI:10.1038/nbt1096-1246 |
| [6] | Ormö M, Cubitt AB, Kallio K, et al. Crystal structure of the green fluorescent protein[J]. Science, 1996, 273 (5280): 1392–1395. DOI:10.1126/science.273.5280.1392 |
| [7] | Subach OM, Patterson GH, Ting L, et al. A photoswitchable orange-to-far-red fluorescent protein, PSmOrange[J]. Nature Methods, 2011, 8 (9): 771–777. DOI:10.1038/nmeth.1664 |
| [8] | EhrenbergM, 罗文新, 夏宁邵. 绿色荧光蛋白——发现、表达和发展[J]. 生物物理学报, 2008(6): 422–429. |
| [9] | Park N, Song J, Jeong S, et al. Vaccinia-related kinase 3(VRK3) sets the circadian period and amplitude by affecting the subcellular localization of clock proteins in mammalian cells[J]. Biochemical and Biophysical Research Communications, 2017, 487 (2): 320–326. DOI:10.1016/j.bbrc.2017.04.057 |
| [10] | Zhao D, Xue C, Lin S, et al. Notch signaling pathway regulates angiogenesis via endothelial cell in 3D Co-culture model[J]. Journal of Cellular Physiology, 2017, 232 (6): 1548–1558. DOI:10.1002/jcp.25681 |
| [11] | Wachter RM, Watkins JL, Kim H. Mechanistic diversity of red fluorescence acquisition by GFP-like proteins[J]. Biochemistry, 2010, 49 (35): 7417–7427. DOI:10.1021/bi100901h |
| [12] | Strack RL, Bhattacharyya D, Glick BS, et al. Noncytotoxic orange and red/green derivatives of DsRed-Express2 for whole-cell labeling[J]. BMC Biotechnology, 2009, 9 : 32. DOI:10.1186/1472-6750-9-32 |
| [13] | Dash AK, Yende AS, Tyagi RK. Novel Application of red fluorescent protein(DsRed-Express)for the study of functional dynamics of nuclear receptors[J]. Journal of Fluorescence, 2017 : 1–7. |
| [14] | Shaner NC, Lin MZ, Mckeown MR, et al. Improving the photostability of bright monomeric orange and red fluorescent proteins[J]. Nature Methods, 2008, 5 (6): 545–551. DOI:10.1038/nmeth.1209 |
| [15] | Merzlyak EM, Goedhart J, Shcherbo D, et al. Bright monomeric red fluorescent protein with an extended fluorescence lifetime[J]. Nat Methods, 2007, 4 (7): 555–557. DOI:10.1038/nmeth1062 |
| [16] | Sakaue-Sawano A, Kurokawa H, Morimura T, et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression[J]. Cell, 2008, 132 (3): 487–498. DOI:10.1016/j.cell.2007.12.033 |
| [17] | Tsutsui H, Karasawa S, Okamura Y, et al. Improving membrane voltage measurements using FRET with new fluorescent proteins[J]. Nat Methods, 2008, 5 (8): 683–685. DOI:10.1038/nmeth.1235 |
| [18] | Bindels DS, Haarbosch L, van Weeren L, et al. mScarlet:a bright monomeric red fluorescent protein for cellular imaging[J]. Nat Methods, 2017, 14 (1): 53–56. |
| [19] | Pandelieva AT, Baran MJ, Calderini GF, et al. Brighter red fluorescent proteins by rational design of triple-decker motif[J]. ACS Chemical Biology, 2016, 11 (2): 508–517. DOI:10.1021/acschembio.5b00774 |
| [20] | Fan Y, Ai H. Development of redox-sensitive red fluorescent proteins for imaging redox dynamics in cellular compartments[J]. Analytical and Bioanalytical Chemistry, 2016, 408 (11): 2901–2911. DOI:10.1007/s00216-015-9280-3 |
| [21] | Shaner NC, Campbell RE, Steinbach PA, et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein[J]. Nat Biotechnol, 2004, 22 (12): 1567–1572. DOI:10.1038/nbt1037 |
| [22] | Wannier TM, Moore MM, Mou Y, et al. Computational design of the β-sheet surface of a red fluorescent protein allows control of protein oligomerization[J]. PLoS One, 2015, 10 (6): e130582. |
| [23] | Hasegawa J, Ise T, Fujimoto KJ, et al. Excited States of fluorescent proteins, mKO and DsRed:chromophore-protein electrostatic interaction behind the color variations[J]. The Journal of Physical Chemistry B, 2010, 114 (8): 2971–2979. DOI:10.1021/jp9099573 |
| [24] | Bravaya KB, Grigorenko BL, Nemukhin AV, et al. Quantum chemistry behind bioimaging:insights from Ab initio studies of fluorescent proteins and their chromophores[J]. Accounts of Chemical Research, 2012, 45 (2): 265–275. DOI:10.1021/ar2001556 |
| [25] | Ikmi A, Gibson MC. Identification and in vivo characterization of NvFP-7R, a developmentally regulated red fluorescent protein of Nematostella vectensis[J]. PLoS One, 2010, 5 (7): e11807. DOI:10.1371/journal.pone.0011807 |
| [26] | Shcherbo D, Murphy CS, Ermakova GV, et al. Far-red fluorescent tags for protein imaging in living tissues[J]. The Biochemical Journal, 2009, 418 (3): 567–574. DOI:10.1042/BJ20081949 |
| [27] | Kredel S, Nienhaus K, Oswald F, et al. Optimized and far-red-emitting variants of fluorescent protein eqFP611[J]. Chemistry & Biology, 2008, 15 (3): 224–233. |
| [28] | Chica RA, Moore MM, Allen BD, et al. Generation of longer emission wavelength red fluorescent proteins using computationally designed libraries[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107 (47): 20257–20262. DOI:10.1073/pnas.1013910107 |
| [29] | Shkrob MA, Yanushevich YG, Chudakov DM, et al. Far-red fluorescent proteins evolved from a blue chromoprotein from Actinia equina[J]. Biochemical Journal, 2005, 392 (Pt 3): 649–654. |
| [30] | Wannier TM, Mayo SL. The structure of a far-red fluorescent protein, AQ143, shows evidence in support of reported red-shifting chromophore interactions[J]. Protein Science:A Publication of the Protein Society, 2014, 23 (8): 1148–1153. DOI:10.1002/pro.v23.8 |
| [31] | Mcisaac RS, Engqvist MKM, Wannier T, et al. Directed evolution of a far-red fluorescent rhodopsin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111 (36): 13034–13039. DOI:10.1073/pnas.1413987111 |
| [32] | Konold PE, Yoon E, Lee J, et al. Fluorescence from multiple chromophore hydrogen-bonding states in the far-red protein TagRFP675[J]. The Journal of Physical Chemistry Letters, 2016, 7 (15): 3046–3051. DOI:10.1021/acs.jpclett.6b01172 |
| [33] | Hense A, Prunsche B, Gao P, et al. Monomeric Garnet, a far-red fluorescent protein for live-cell STED imaging[J]. Scientific Reports, 2015, 5 : 18006. |
| [34] | Yu D, Dong Z, Gustafson WC, et al. Rational design of a monomeric and photostable far‐red fluorescent protein for fluorescence imaging in vivo[J]. Protein Science:A Publication of the Protein Society, 2015, 25 (2): 308–315. |
| [35] | Bajar BT, Lam AJ, Badiee RK, et al. Fluorescent indicators for simultaneous reporting of all four cell cycle phases[J]. Nat Methods, 2016, 13 (12): 993–996. DOI:10.1038/nmeth.4045 |
| [36] | Li Z, Zhang Z, Bi L, et al. Mutagenesis of mNeptune red-shifts emission spectrum to 681-685 nm[J]. PLoS One, 2016, 11 (4): e148749. |
| [37] | Ren H, Yang B, Ma C, et al. Cysteine sulfoxidation increases the photostability of red fluorescent proteins[J]. ACS Chemical Biology, 2016, 11 (10): 2679–2684. DOI:10.1021/acschembio.6b00579 |
| [38] | Shcherbo D, Merzlyak EM, Chepurnykh TV, et al. Bright far-red fluorescent protein for whole-body imaging[J]. Nat Methods, 2007, 4 (9): 741–746. DOI:10.1038/nmeth1083 |
| [39] | Shcherbo D, Shemiakina Ⅱ, Ryabova AV, et al. Near-infrared fluorescent proteins[J]. Nature Methods, 2010, 7 (10): 827–829. DOI:10.1038/nmeth.1501 |
| [40] | Armengol P, Gelabert R, Moreno M, et al. Chromophore interactions leading to different absorption spectra in mNeptune1 and mCardinal red fluorescent proteins[J]. Physical Chemistry Chemical Physics, 2016, 18 (25): 16964–16976. DOI:10.1039/C6CP01297C |
| [41] | Shu X, Royant A, Lin MZ, et al. Mammalian expression of infrared fluorescent proteins engineered from a bacterial phytochrome[J]. Science(New York, N. Y.), 2009, 324 (5928): 804–807. DOI:10.1126/science.1168683 |
| [42] | Filonov GS, Piatkevich KD, Ting L, et al. Bright and stable near infra-red fluorescent protein for in vivo imaging[J]. Nature Biotechnology, 2011, 29 (8): 757–761. DOI:10.1038/nbt.1918 |
| [43] | Lin L, Wang B, Chen J, et al. mPlum-IFP 1. 4 fluorescent fusion protein may display Förster resonance energy transfer associated properties that can be used for near-infrared based reporter gene imaging[J]. Journal of Biomedical Optics, 2013, 18 (12): 126013. DOI:10.1117/1.JBO.18.12.126013 |
| [44] | Bajar BT, Wang ES, Zhang S, et al. A guide to fluorescent protein FRET pairs[J]. Sensors(Basel, Switzerland), 2016, 16 (9): 1488. DOI:10.3390/s16091488 |
| [45] | Moore MM, Oteng-Pabi SK, Pandelieva AT, et al. Recovery of red fluorescent protein chromophore maturation deficiency through rational design[J]. PLoS One, 2012, 7 (12): e52463. DOI:10.1371/journal.pone.0052463 |
| [46] | Veal EA, Day AM, Morgan BA. Hydrogen peroxide sensing and signaling[J]. Molecular Cell, 2007, 26 (1): 1–14. DOI:10.1016/j.molcel.2007.03.016 |
| [47] | Kumagai A, Ando R, Miyatake H, et al. A bilirubin-inducible fluorescent protein from eel muscle[J]. Cell, 2013, 153 (7): 1602–1611. DOI:10.1016/j.cell.2013.05.038 |
| [48] | Rodriguez EA, Tran GN, Gross LA, et al. A far-red fluorescent protein evolved from a cyanobacterial phycobiliprotein[J]. Nature Methods, 2016, 13 (9): 763–769. DOI:10.1038/nmeth.3935 |
| [49] | Ai H, Hazelwood KL, Davidson MW, et al. Fluorescent protein FRET pairs for ratiometric imaging of dual biosensors[J]. 2008, 5(5):401-403. |
| [50] | Shcherbakova DM, Hink MA, Joosen L, et al. An orange fluorescent protein with a large Stokes shift for single-excitation multicolor FCCS and FRET imaging[J]. Journal of the American Chemical Society, 2012, 134 (18): 7913–7923. DOI:10.1021/ja3018972 |
| [51] | Kogure T, Karasawa S, Araki T, et al. A fluorescent variant of a protein from the stony coral Montipora facilitates dual-color single-laser fluorescence cross-correlation spectroscopy[J]. Nat Biotechnol, 2006, 24 (5): 577–581. DOI:10.1038/nbt1207 |
| [52] | Piatkevich KD, Hulit J, Subach OM, et al. Monomeric red fluorescent proteins with a large Stokes shift[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107 (12): 5369–5374. DOI:10.1073/pnas.0914365107 |
| [53] | Kennis JTM, van Stokkum IHM, Peterson DS, et al. Ultrafast proton shuttling in psammocora cyan fluorescent protein[J]. The Journal of Physical Chemistry B, 2013, 117 (38): 11134–11143. DOI:10.1021/jp401114e |
| [54] | Fron E, De Keersmaecker H, Rocha S, et al. Mechanism behind the apparent large stokes shift in lssmorange investigated by time-resolved spectroscopy[J]. The Journal of Physical Chemistry B, 2015, 119 (47): 14880–14891. DOI:10.1021/acs.jpcb.5b09189 |
| [55] | Piatkevich KD, Malashkevich VN, Almo SC, et al. Engineering ESPT pathways based on structural analysis of LSSmKate red fluorescent proteins with large Stokes shift[J]. Journal of the American Chemical Society, 2010, 132 (31): 10762–10770. DOI:10.1021/ja101974k |
| [56] | Guan Y, Meurer M, Raghavan S, et al. Live-cell multiphoton fluorescence correlation spectroscopy with an improved large Stokes shift fluorescent protein[J]. Molecular Biology of the Cell, 2014, 26 (11): 2054–2066. |
| [57] | Piatkevich KD, English BP, Malashkevich VN, et al. Photoswitchable red fluorescent protein with a large Stokes shift[J]. Chemistry & Biology, 2014, 21 (10): 1402–1414. |
| [58] | Yang J, Wang L, Yang F, et al. mBeRFP, an improved large stokes shift red fluorescent protein[J]. PLoS One, 2013, 8 (6): e64849. DOI:10.1371/journal.pone.0064849 |
| [59] | Pletnev S, Shcherbakova DM, Subach OM, et al. Orange fluorescent proteins:structural studies of LSSmOrange, PSmOrange and PSmOrange2[J]. PLoS One, 2014, 9 (6): e99136. DOI:10.1371/journal.pone.0099136 |
| [60] | Bacia K, Kim SA, Schwille P. Fluorescence cross-correlation spectroscopy in living cells[J]. Nat Methods, 2006, 3 (2): 83–89. DOI:10.1038/nmeth822 |
| [61] | Lindenburg LH, Malisauskas M, Sips T, et al. Quantifying stickiness:thermodynamic characterization of intramolecular domain interactions to guide the design of förster resonance energy transfer sensors[J]. Biochemistry, 2014, 53 (40): 6370–6381. DOI:10.1021/bi500433j |
| [62] | Laviv T, Kim BB, Chu J, et al. Simultaneous dual-color fluorescence lifetime imaging with novel red-shifted fluorescent proteins[J]. 2016, 13(12):989-992. |
| [63] | Zhu X, Zhang L, Kao Y, et al. A tunable fluorescent timer method for imaging spatial-temporal protein dynamics using light-driven photoconvertible protein[J]. Journal of Biophotonics, 2015, 8 (3): 226–232. DOI:10.1002/jbio.201300174 |
| [64] | Takamura A, Hattori M, Yoshimura H, et al. Simultaneous time-lamination imaging of protein association using a split fluorescent timer protein[J]. Analytical Chemistry, 2015, 87 (6): 3366–3372. DOI:10.1021/ac504583t |
| [65] | Tsuboi T, Kitaguchi T, Karasawa S, et al. Age-dependent preferential dense-core vesicle exocytosis in neuroendocrine cells revealed by newly developed monomeric fluorescent timer protein[J]. Molecular Biology of the Cell, 2009, 21 (1): 87–94. |
| [66] | Khmelinskii A, Keller PJ, Bartosik A, et al. Tandem fluorescent protein timers for in vivo analysis of protein dynamics[J]. Nat Biotechnol, 2012, 30 (7): 708–714. DOI:10.1038/nbt.2281 |
| [67] | Barry JD, Donà E, Gilmour D, et al. TimerQuant:a modelling approach to tandem fluorescent timer design and data interpretation for measuring protein turnover in embryos[J]. Development(Cambridge, England), 2015, 143 (1): 174–179. |
| [68] | Khmelinskii A, Knop M. Analysis of protein dynamics with tandem fluorescent protein timers[M]. Exocytosis and Endocytosis, Ivanov AI, New York:Springer New York, 2014, 195-210. |
| [69] | Dona E, Barry JD, Valentin G, et al. Directional tissue migration through a self-generated chemokine gradient[J]. Nature, 2013, 503 (7475): 285–289. |
| [70] | Khmelinskii A, Meurer M, Ho C, et al. Incomplete proteasomal degradation of green fluorescent proteins in the context of tandem fluorescent protein timers[J]. Molecular Biology of the Cell, 2015, 27 (2): 360–370. |
| [71] | Subach FV, Patterson GH, Renz M, et al. Bright monomeric photoactivatable red fluorescent protein for two-color super-resolution sptPALM of live cells[J]. Journal of the American Chemical Society, 2010, 132 (18): 6481–6491. DOI:10.1021/ja100906g |
| [72] | Chudakov DM, Lukyanov S, Lukyanov KA. Tracking intracellular protein movements using photoswitchable fluorescent proteins PS-CFP2 and Dendra2[J]. 2007, 2(8):2024-2032. |
| [73] | Zhang M, Chang H, Zhang Y, et al. Rational design of true monomeric and bright photoactivatable fluorescent proteins[J]. 2012, 9(7):727-729. |
| [74] | Hoi H, Shaner NC, Davidson MW, et al. A monomeric photoconvertible fluorescent protein for imaging of dynamic protein localization[J]. Journal of Molecular Biology, 2010, 401 (5): 776–791. DOI:10.1016/j.jmb.2010.06.056 |
| [75] | Subach OM, Patterson GH, Ting L, et al. A photoswitchable orange-to-far-red fluorescent protein, PSmOrange[J]. Nature Methods, 2011, 8 (9): 771–777. DOI:10.1038/nmeth.1664 |
| [76] | Piatkevich KD, Subach FV, Verkhusha VV. Far-red light photoactivatable near-infrared fluorescent proteins engineered from a bacterial phytochrome[J]. Nature Communications, 2013, 4 : 2153. |
| [77] | Griswold SL, Sajja KC, Jang C, et al. Generation and characterization of iUBC-KikGR photoconvertible transgenic mice for live time lapse imaging during development[J]. Genesis, 2011, 49 (7): 591–598. DOI:10.1002/dvg.20718 |
| [78] | Nickerson A, Huang T, Lin L, et al. Photoactivated localization microscopy with bimolecular fluorescence complementation(BiFC-PALM)[J]. J Vis Exp, 2015 (106): e53154. |
| [79] | Shroff H, White H, Betzig E. Photoactivated localization microscopy(PALM)of adhesion complexes[J]. Current Protocols in Cell Biology, 2013, Chapter 4 : t4–t21. |
| [80] | Brown TA, Tkachuk AN, Shtengel G, et al. Superresolution fluorescence imaging of mitochondrial nucleoids reveals their spatial range, limits, and membrane interaction[J]. Molecular and Cellular Biology, 2011, 31 (24): 4994–5010. DOI:10.1128/MCB.05694-11 |
| [81] | Wang S, Moffitt JR, Dempsey GT, et al. Characterization and development of photoactivatable fluorescent proteins for single-molecule-based superresolution imaging[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111 (23): 8452–8457. DOI:10.1073/pnas.1406593111 |
| [82] | Mckinney SA, Murphy CS, Hazelwood KL, et al. A bright and photostable photoconvertible fluorescent protein for fusion tags[J]. Nature Methods, 2009, 6 (2): 131–133. DOI:10.1038/nmeth.1296 |
| [83] | Fuchs J, Bohme S, Oswald F, et al. A photoactivatable marker protein for pulse-chase imaging with superresolution[J]. 2010, 7(8):627-630. |
| [84] | Subach OM, Entenberg D, Condeelis JS, et al. A FRET-facilitated photoswitching using an orange fluorescent protein with the fast photoconversion kinetics[J]. Journal of the American Chemical Society, 2012, 134 (36): 14789–14799. DOI:10.1021/ja3034137 |
| [85] | Subach FV, Subach OM, Gundorov IS, et al. Monomeric fluorescent timers that change color from blue to red report on cellular trafficking[J]. Nature Chemical Biology, 2009, 5 (2): 118–126. DOI:10.1038/nchembio.138 |
| [86] | Tsuboi T, Kitaguchi T, Karasawa S, et al. Age-dependent preferential dense-core vesicle exocytosis in neuroendocrine cells revealed by newly developed monomeric fluorescent timer protein[J]. Molecular Biology of the Cell, 2009, 21 (1): 87–94. |
| [87] | Griswold SL, Sajja KC, Jang C, et al. Generation and characterization of iUBC-KikGR photoconvertible transgenic mice for live time lapse imaging during development[J]. Genesis, 2011, 49 (7): 591–598. DOI:10.1002/dvg.20718 |
| [88] | Jensen NA, Danzl JG, Willig KI, et al. Coordinate-targeted and coordinate-stochastic super-resolution microscopy with the reversibly switchable fluorescent protein dreiklang[J]. Chemphyschem, 2014, 15 (4): 756–762. DOI:10.1002/cphc.201301034 |
| [89] | Duwé S, Moeyaert B, Dedecker P. Diffraction-unlimited fluorescence microscopy of living biological samples using pcSOFI[J]. Curr Protoc Chem Biol, 2015, 7 : 27–41. |
| [90] | Wang S, Chen X, Chang L, et al. GMars-Q enables long-term live-cell parallelized reversible saturable optical fluorescence transitions nanoscopy[J]. ACS Nano, 2016, 10 (10): 9136–9144. DOI:10.1021/acsnano.6b04254 |
| [91] | Lavoie-Cardinal F, Jensen NA, Westphal V, et al. Two-color RESOLFT nanoscopy with green and red fluorescent photochromic proteins[J]. Chemphyschem, 2014, 15 (4): 655–663. DOI:10.1002/cphc.201301016 |
| [92] | Zhang X, Zhang M, Li D, et al. Highly photostable, reversibly photoswitchable fluorescent protein with high contrast ratio for live-cell superresolution microscopy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113 (37): 10364–10369. DOI:10.1073/pnas.1611038113 |
| [93] | El Khatib M, Martins A, Bourgeois D, et al. Rational design of ultrastable and reversibly photoswitchable fluorescent proteins for super-resolution imaging of the bacterial periplasm[J]. Scientific Reports, 2016, 6 : 18459. DOI:10.1038/srep18459 |
| [94] | Smyrnova D, Zinovjev K, Tuñón I, et al. Thermal isomerization mechanism in dronpa and its mutants[J]. The Journal of Physical Chemistry B, 2016, 120 (50): 12820–12825. DOI:10.1021/acs.jpcb.6b10859 |
| [95] | Zhang X, Chen X, Zeng Z, et al. Development of a reversibly switchable fluorescent protein for super-resolution optical fluctuation imaging(SOFI)[J]. ACS Nano, 2015, 9 (3): 2659–2667. DOI:10.1021/nn5064387 |
| [96] | Morozov D, Groenhof G. Hydrogen bond fluctuations control photochromism in a reversibly photo-switchable fluorescent protein[J]. Angewandte Chemie International Edition, 2016, 55 (2): 576–578. DOI:10.1002/anie.201508452 |
| [97] | Subach OM, Malashkevich VN, Zencheck WD, et al. Structural characterization of acylimine-containing blue and red chromophores in mTagBFP and TagRFP fluorescent proteins[J]. Chemistry & Biology, 2010, 17 (4): 333–341. |
| [98] | Armengol P, Gelabert R, Moreno M, et al. New insights into the structure-spectrum relationship in S65T/H148D and E222Q/H148D green fluorescent protein mutants:a theoretical assessment[J]. Organic & Biomolecular Chemistry, 2014, 12 (48): 9845–9852. |
| [99] | Pletnev S, Subach FV, Dauter Z, et al. Understanding blue-to-red conversion in monomeric fluorescent timers and hydrolytic degradation of their chromophores[J]. Journal of the American Chemical Society, 2010, 132 (7): 2243–2253. DOI:10.1021/ja908418r |
| [100] | Henderson JN, Osborn MF, Koon N, et al. Excited state proton transfer in the red fluorescent protein mKeima[J]. Journal of the American Chemical Society, 2009, 131 (37): 13212–13213. DOI:10.1021/ja904665x |
| [101] | Shu X, Wang L, Colip L, et al. Unique interactions between the chromophore and glutamate 16 lead to far-red emission in a red fluorescent protein[J]. Protein Science:A Publication of the Protein Society, 2009, 18 (2): 460–466. DOI:10.1002/pro.v18:2 |
| [102] | Takemoto K, Matsuda T, Sakai N, et al. SuperNova, a monomeric photosensitizing fluorescent protein for chromophore-assisted light inactivation[J]. Scientific Reports, 2013, 3 : 2629. DOI:10.1038/srep02629 |
| [103] | Pletneva NV, Pletnev VZ, Sarkisyan KS, et al. Crystal structure of phototoxic orange fluorescent proteins with a tryptophan-based chromophore[J]. PLoS One, 2015, 10 (12): e145740. |
| [104] | Subach OM, Cranfill PJ, Davidson MW, et al. An enhanced monomeric blue fluorescent protein with the high chemical stability of the chromophore[J]. PLoS One, 2011, 6 (12): e28674. DOI:10.1371/journal.pone.0028674 |
| [105] | Tsien RY. Rosy dawn for fluorescent proteins[J]. Nat Biotechnol, 1999, 17 (10): 956–957. DOI:10.1038/13648 |
| [106] | Bindels DS, Haarbosch L, van Weeren L, et al. mScarlet:a bright monomeric red fluorescent protein for cellular imaging[J]. 2017, 14(1):53-56. |