肝素和低分子肝素是一大类复杂的黏多糖天然药物,广泛用于临床抗凝血治疗,低分子肝素因其副作用低而应用更为广泛[1-2]。目前,化学裂解法和酶降解法是国内外制备低分子肝素的两种主要方法[3]。化学裂解法的成本较低,目前的大品牌低分子肝素如依诺肝素、达肝素、那曲肝素及帕肝素等都是经化学降解而来。但化学裂解的工艺过程较复杂,往往会产生环保问题,而且低分子肝素生产过程中使用的有毒有害溶剂会对生产人员的健康造成损害。酶降解法反应条件温和、易操作,对环境友好,丹麦利奥公司的汀肝素即由肝素酶裂解肝素而来[4],但这种低分子肝素因为酶切位点不理想而存在收率偏低的问题。
肝素酶是一种肝素类多糖裂解酶,能够将肝素降解为低分子肝素或肝素寡糖,在低分子肝素生产和肝素质量检测等方面具有重要的应用。现阶段,已发现许多种微生物中均存在肝素酶,包括肝素黄杆菌(Flavobacterium heparinum)[5]、解肝素拟杆菌(Prevotella heparinolytica)[6]、棒杆菌(Corynebacterium sp.)[7]、环状芽孢杆菌(Bacillus circulans)[8]、枯草芽孢杆菌(Bacillus subtilis)[9]、鞘胺醇杆菌(Sphingo bacterium sp.)[10]和粪便拟杆菌(Bacteroides stercoris)HJ-15[11],其中,只有肝素黄杆菌中的肝素酶得到商业化应用。
本研究从土壤中筛选分离得到新型产肝素酶菌种食神鞘氨醇杆菌(Sphingo spiritivorum),对其进行微生物发酵和肝素酶分离纯化,得到新型肝素酶SShepⅠ与SShepⅡ,并对两种新型肝素酶进行了一些性质研究。
1 材料与方法 1.1 材料 1.1.1 菌种来源食神鞘氨醇杆菌(Sphingo. spiritivorum),分离自成都市杜甫草堂遗址附近土壤。
1.1.2 试剂肝素(海普瑞公司自产高纯度原料药)、酵母抽提物、蛋白胨(Oxoid Ltd.,Basingstoke,Hampshinre,England),SP Sepharose Fast Flow(GE Healthy)、Q Sepharose Fast Flow(GE Healthy)、Cellufine Sulfate(GE Healthy)、丙烯酰胺(Fluka产品)、甲叉双丙烯酰胺(Fluka产品)、Tris(Amresco进口分装)、低分子量标准蛋白(上海源聚生物科技有限公司)、其他试剂均为国产分析纯。
1.1.3 仪器F6H120717B超净工作台(深圳宝源净化有限公司)、RC12BP低速冷冻离心机(赛默飞世尔科技公司)、AUY220电子天平(日本岛津公司)、AYC-2102C恒温培养振荡器(上海新苗医疗器械制造有限公司)、YXO-LS-75SⅡ高压蒸汽灭菌器(上海博迅实业有限公司)、层析柱、BS-160A自动收集器(上海金鹏分析仪器有限公司)、e2695高效液相色谱仪(美国沃特斯(Waters)公司)、色谱柱、紫外分光光度计(上海元析仪器有限公司)、BCD-208K/A冰箱(海尔集团)。
1.2 方法 1.2.1 菌种筛选成都杜甫草堂遗址附近地表以下10-15 cm处土壤约20 g,与1 g肝素粉末拌匀,室温放置1个月。取少量土样加入无菌水中,30℃摇床震荡(150 r/min)2-3 h。取10 mL上清加入100 mL种子培养基中,如前震荡大约16 h。移取20 mL菌液至200 mL发酵培养基中,再如前震荡培养24 h。离心(4℃,5 000×g,5 min)获得上清液,用天青A法检测肝素浓度,取肝素明显有消耗的菌液继续实验。将菌液稀释涂布于平板培养基上,挑选出单菌落。单菌落在斜面或平板上增殖后接入种子培养基中,培养16 h后,转接发酵培养基中培养约24 h。离心收集细胞,悬浮于25 mmol/L Tris-HCl(含10 mmol/L CaCl2,pH 7.0) 缓冲液,超声破碎,测定肝素酶活性,确认得到的菌株为产肝素酶菌株。
种子培养基:蛋白胨10 g,牛肉膏5 g,酵母粉5 g,NaCl 5 g,加水H2O 1 L搅拌溶解后用1 mol/L NaOH调pH至7.0。1×105 Pa高压蒸汽灭菌20 min,备用。
发酵培养基:肝素8 g,蛋白胨2 g,NaCl 5 g,K2HPO4 2.5 g,(NH4)2SO4 1 g,MgSO4·7H2O 0.5 g,加水H2O 1 L搅拌溶解后用1 mol/L NaOH调pH至7.0。1×105 Pa高压蒸汽灭菌20 min,备用。
固体培养基:技术琼脂粉20 g,肝素8 g,蛋白胨2 g,NaCl 5 g,K2HPO4 2.5 g,(NH4)2SO4 1 g,MgSO4·7H2O 0.5 g,加水H2O 1 L搅拌溶解后用1 mol/L NaOH调pH至7.0。1×105 Pa高压蒸汽灭菌20 min,备用。
1.2.2 菌种鉴定由广东省微生物分析检测中心进行菌种鉴定,通过菌株的镜检观察、理化性质分析和16S rDNA与已知菌株进行基因序列比对确定未知菌株所属的种。
1.2.3 肝素酶的制备和纯化将菌体从平板或斜面上刮取两环接种到种子培养基中,培养16 h后,按15%接种量接入二级液体种子培养基中,培养24 h,再按20%接种量接入2 L发酵培养基中,培养24 h。收集菌液,3 800 r/min、4℃离心45 min,将沉淀悬浮在25 mmol/L Tris-HCl缓冲液(含10 mmol/L CaCl2,pH 7.0) 中,用高压均质机4℃,800 bar,破碎3-4个循环,并离心30 min(12 000 r/min,4℃)。得粗酶液。
将粗酶液在冰浴条件下做硫酸铵沉淀,收取硫酸铵饱和度60%-80%的沉淀并溶解于100 mL的前述Tris-HCl缓冲液中,用4 L同样缓冲液8-10℃过夜透析。将硫酸铵沉淀后的酶上样于SP Sepharose Fast Flow层析柱,用200 mL前述Tris-HCl缓冲液平衡,再用同样缓冲液中0-0.3 mol/L NaCl(各100 mL)梯度洗脱。在上样液和洗脱液中均分布有肝素酶活性的组分,分开收集。
将上述与SP柱结合后被洗脱下来的有酶活性的组分,用2 L前述Tris-HCl缓冲液8-10℃过夜透析,然后上CS(Cellufine Sulfate)柱。用200 mL 0.1 mol/L NaCl(溶于同样缓冲液)平衡,再用同样缓冲液中0.1-1 mol/L NaCl(各100 mL)梯度洗脱,收集洗脱液中有活性的组分。
将上述CS柱后收集到的有酶活性的组分,用2 L前述Tris-HCl缓冲液8-10℃过夜透析,然后再次上SP柱,用200 mL 0.09 mol/L NaCl(溶于同样缓冲液)平衡,再用500 mL 0.105 mol/L NaCl等度洗脱,收集有酶活性组分的洗脱液。
将再次过SP柱后收集到的有酶活性的组分,用等体积的前述Tris-HCl缓冲液混合稀释,再上CS柱。用200 mL 0.1 mol/L NaCl(溶于同样缓冲液)平衡,再用同样缓冲液中0.1-1 mol/L NaCl(各100 mL)梯度洗脱,收集洗脱液中有活性的组分,得到纯的肝素酶,定名SShepⅠ。
将前述SP柱上样流出液有酶活性的组分上样于Q Sepharose Fast Flow层析柱,用200 mL前述Tris-HCl缓冲液平衡,再用同样缓冲液中0-0.5 mol/L NaCl(各100 mL)梯度洗脱,收集有酶活性组分。
将Q柱后收集到的有酶活性组分,用2 L前述Tris-HCl缓冲液8-10℃过夜透析,然后过CS柱。用200 mL的0.1 mol/L NaCl(溶于同样缓冲液)平衡,然后用同样缓冲液中0.1-0.6 mol/L NaCl(各100 mL)梯度洗脱,收集洗脱液中有活性的组分,得到纯的肝素酶,定名SShepⅡ。
1.2.4 肝素酶理化性质考察 1.2.4.1 肝素酶最适pH将肝素底物溶于25 mmol/L Tris-HCl(含10 mmol/L CaCl2)缓冲液中,配制成浓度为1 mg/mL的肝素溶液,分别调至pH为5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5和9.0。
1.2.4.2 肝素酶最适温度将1 mg/mL的肝素溶液置于恒温水浴锅中,分别将温度依次调至25℃、27℃、29℃、31℃、33℃、35℃、37℃、39℃、41℃、43℃、45℃和47℃,在各条件下分别测定肝素酶的活性。
1.2.4.3 金属离子种类对肝素酶活性的影响在肝素底物中分别加入10 mmol/L的CaCl2、NaCl、KCl、MgCl2、CuCl2,以考察金属离子对肝素酶活性的影响。
1.2.4.4 金属离子浓度对肝素酶活性的影响将对酶活有促进作用的几种金属离子分别设置为0、1 mmol/L、10 mmol/L、50 mmol/L、100 mmol/L、500 mmol/L和1 mol/L,测定酶在各种条件下的酶活,考察金属离子浓度对肝素酶活性的影响。
1.2.5 肝素酶酶切位点分析分别用足量肝素黄杆菌肝素酶Ⅰ、Ⅱ、Ⅲ和两种新型肝素酶酶解肝素样品(50 mg/500 µL/3.0 IU),之后经P10层析柱分离得到二糖组分,对二糖的组分进行分析。
1.2.6 检测方法蛋白浓度测定:Folin-酚试剂法(Lowry法)[12],以牛血清白蛋白作标准。酶活力的测定:采用Ma等[5]的方法。SDS-聚丙烯酰胺凝胶电泳参照文献[13]。低分子肝素分子量分布测定参照文献[14]。低分子肝素效价测定参照文献[15]。
2 结果 2.1 菌种的筛选经多轮筛选后,得到1株经验证产肝素酶的细菌,编号为CDF1-2,为白色不透明菌落,发酵后产肝素酶量大约为80 IU/L。经广东省微生物分析检测中心鉴定,为革兰氏阴性、短杆菌,通过16S rDNA与已知菌株进行基因序列比对,确定该菌种属食神鞘氨醇杆菌(Sphingo. spiritivorum)。经检索目前该菌种的相关报道较少,无产肝素酶的报道。
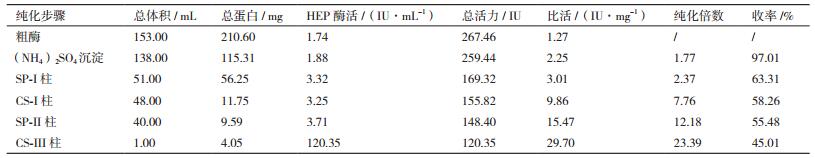
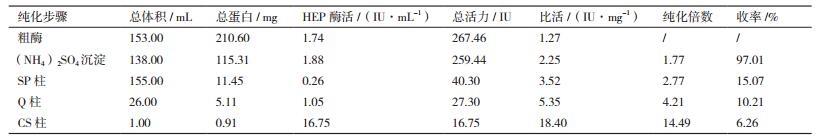
2.2 肝素酶的制备和纯化按照1.2.3中的各纯化步骤实施后,所得酶活、蛋白含量、(比活)纯化倍数、(总活力)产率分别见表 1、表 2所示。纯化后得到了比较纯的酶蛋白,经SDS-PAGE测定,SShepⅠ和SShepⅡ分子量分别约为71 kD和92 kD(图 1)。
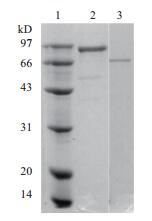
|
| 图 1 肝素酶SShepⅠ(泳道3)和SShepⅡ(泳道2)SDSPAGE电泳图 1:蛋白marker;2:纯化后的SShepⅡ;3:纯化后的SShepⅠ |
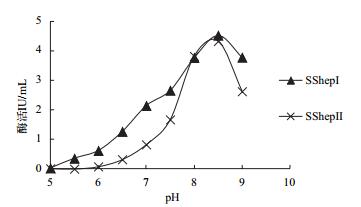
如图 2所示,两种肝素酶以HEP为底物时,两种肝素酶SShepⅠ和SShepⅡ在pH为8.5时酶活达到最高值。
|
| 图 2 肝素酶SShepⅠ和SShepⅡ的最适pH |
如图 3所示,两种酶以HEP为底物时,在温度较低的情况下,酶活比较低,随着温度上升,肝素酶的活性呈现增高的趋势,温度过高两个酶的活性迅速下降。肝素酶SShepⅠ和SShepⅡ的最适温度分别为48℃和46℃。

|
| 图 3 肝素酶SShepⅠ和SShepⅡ的最适温度 |
分别考察了CaCl2、NaCl、KCl、MgCl2和CuCl2等金属离子对SShepⅠ和SShepⅡ两种肝素酶HEP活性的影响,由图 4可知,在空白对照组,上述各酶的活性均较低,而在添加金属离子的情况下,K+、Mg2+、Na+和Ca2+均显示了促进酶活的作用,其中Ca2+的促进作用最强,酶活大幅度提高,其次是Mg2+也具有明显的促进作用,而Cu2+具有抑制酶活的作用,使酶失活。

|
| 图 4 不同离子种类对肝素酶SShepⅠ和SShepⅡ活性的影响 |
鉴于Ca2+、Mg2+对两种肝素酶均有明显的促进作用,进一步考察这两种离子的浓度对肝素酶活性的影响。由图 5可知,不添加Mg2+的情况下,两种酶的活性均较低,而随着Mg2+浓度的升高,两种酶均出现酶活增大的趋势,SShepⅠ和SShepⅡ在100 mmol/L的Mg2+浓度下活性达到最高,而当Mg2+浓度过高时(大于500 mmol/L),酶的活性又降低甚至失活。说明在一定范围的Mg2+浓度下,Mg2+对肝素酶的催化具有促进或者激活作用,使得酶的活性大幅度提高。
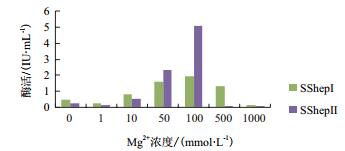
|
| 图 5 Mg2+浓度对肝素酶SShepⅠ和SShepⅡ活性的影响 |
由图 6可知,从10 mmol/L的Ca2+开始,SShepⅠ和SShepⅡ的酶活呈现出明显的提高,两种酶均在100 mmol/L的Ca2+浓度下酶活达到最大,而当Ca2+浓度过高时(大于500 mmol/L),酶的活性又迅速降低,甚至失活。

|
| 图 6 Ca2+浓度对肝素酶SShepⅠ和SShepⅡ活性的影响 |
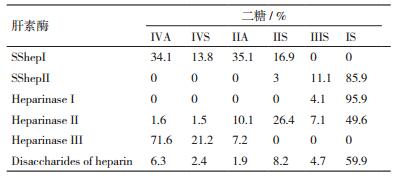
由表 3可知,新型肝素酶SShepⅠ酶解肝素得到的二糖组分中,ⅣA、ⅣS、ⅡA和ⅡS的含量显著高于构成肝素二糖中原始相应二糖的百分含量,ⅢS、IS的含量显著低于构成肝素二糖中原始相应二糖的百分含量;这说明新型肝素酶SShepⅠ对含ⅣA、ⅣS、ⅡA和ⅡS糖链的酶切能力较强,对含ⅢS、IS的糖链不酶切。与已知的3种肝素黄杆菌肝素酶的酶解二糖组分比较发现,新型肝素酶SShepⅠ与肝素黄杆菌肝素酶Ⅲ酶切情况相似,由此可推出,肝素酶SShepⅠ的酶切位点可能为:→4)-α-D-GlcNp2S(or2-Ac)6S(or 6-OH)(1→4)-α-L-IdoAp,→4)-α-D-GlcNp2S(or2-Ac)6S(or 6-OH)(1→4)-β-D-GlcAp。新型肝素酶SShepⅡ酶解肝素得到的二糖组分中,ⅢS、IS的含量显著高于构成肝素二糖中原始相应二糖的百分含量,ⅣA、ⅣS、ⅡA和ⅡS的含量显著低于构成肝素二糖中原始相应二糖的百分含量;这说明新型肝素酶SShepⅡ对含ⅢS、IS的糖链酶切能力较强,对含ⅡS的糖链酶切能力较弱,对含ⅣA、ⅣS、ⅡA的糖链不酶切。与已知的3种肝素黄杆菌肝素酶的酶解二糖组分比较发现,SShepⅡ与肝素黄杆菌肝素酶Ⅰ酶切情况相似,由此可推出,SShepⅡ的酶切位点可能为:→4)-α-D-GlcNp2S6S(or 6-OH)(1→4)-α-L-IdoAp2S,→4)-α-D-GlcNp2S6S(1→4)-α-L-IdoAp,→4)-α-D-GlcNp2S6S(1→4)-β-D-GlcAp。
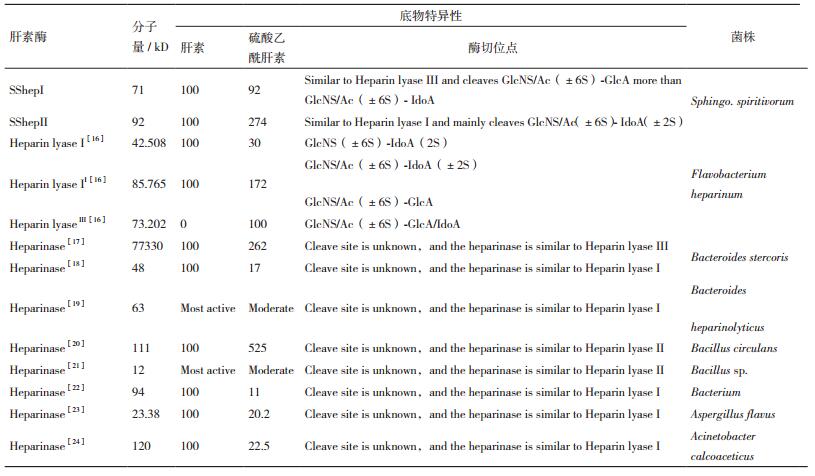
由表 4可以看出,SShepⅠ肝素酶的分子量为71 kD,文献报道的肝素酶分子量大多与之相差较大,较接近的有Heparin lyase Ⅲ、Heparinase in Ref. 17和Heparinase in Ref. 19,但SShepⅠ对肝素和HS的底物特异性与后三者差别明显,因此可认为SShepⅠ与这3种酶不同。SShepⅡ肝素酶的分子量为92 kD,与之接近的有Heparin lyase Ⅱ和Heparinase in Ref. 22,但SShepⅡ对肝素和HS的底物特异性与Heparinase in Ref. 22相差极大,与Heparin lyase Ⅱ也有较大差别。从酶切位点看不具有可比性。因此认为SShepⅡ与已发现的肝素酶均不同。
综上,SShepⅠ和SShepⅡ两种酶的来源、分子量、对肝素和HS降解的特异性以及酶切位点与文献中已报道的肝素酶均有不同。因此,新型肝素酶SShepⅠ和SShepⅡ为两种前未报道过的肝素酶。
2.6 肝素酶降解肝素试验 2.6.1 SShepⅠ和SShepⅡ酶解肝素酶解过程:将1 g肝素用20 mL 50 mmol/L Tris-HCl(含10 mmol/L CaCl2,pH7.4) 缓冲液溶解后加入4 IU纯化后的SShepⅠ或SShepⅡ酶,45℃水浴酶解48 h,水浴100℃煮沸使酶灭活后离心得上清,上清加入氯化钠至终浓度为2.5%,加入1.4倍乙醇(W/W),8-10℃静置24 h后离心,烘干沉淀。分别得到产物0.71 g(SShepⅠ酶解)和0.45 g(SShepⅡ酶解)。
2.6.2 酶解肝素产物分析分析了酶解产物的分子量分布、抗FXa效价和抗FⅡa效价。从分子量分布(图 7)可以看出,两种酶降解肝素均可得到低分子肝素,其中前者的分子量主要分布在2-8 kD,后者分布在0.6-4 kD。经测定效价,SShepⅠ酶解肝素产物抗FXa效价为126 IU/mg,抗FⅡa效价为29 IU/mg;SShepⅡ酶解肝素产物抗FXa效价为135 IU/mg,抗FⅡa效价为8 IU/mg。
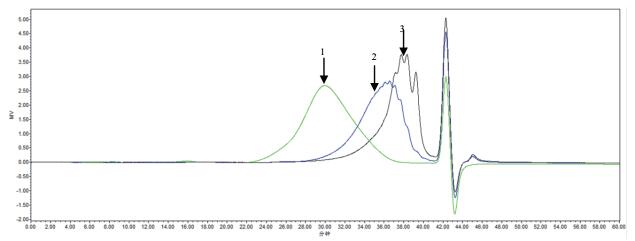
|
| 图 7 新型肝素酶降解肝素分子量分布图 1:未降解的肝素;2:SShepⅠ酶解肝素产物;3:SShe-pII酶解肝素产物 |
随着对低分子肝素的研究不断深入,人们发现,低分子肝素除了具有抗凝活性外,还对肿瘤、炎症、产科妊娠等疾病具有广泛作用[25-27],针对低分子肝素存在多种的药理活性进行新药开发具有广阔的前景。但肝素的结构高度复杂,其糖链上相对于某种药理作用的活性位点在整个混合物中较少,而且容易受其他近似结构干扰,由此对药物开发带来干扰。而肝素酶对肝素的酶切位点比较专一,由此可得到较大量结构相同的片段,有利于药物开发。因此,寻找裂解位点独特的新型肝素酶,具有重要意义。
本研究从土壤中培养分离纯化得到一株食神鞘氨醇杆菌(Sphingo. spiritivorum),发酵后得到粗酶,经多步柱层析纯化后得到新型肝素酶SShepⅠ和SShepⅡ。之后,我们对两种新型肝素酶的理化性质包括最适pH、最适温度、缓冲液离子种类、离子浓度等进行考察,并通过酶解酶解肝素样品,分析二糖组分对两种新型肝素酶的酶切位点进行初步研究。通过相关数据库的检索,未发现利用食神鞘氨醇杆菌生产肝素酶的相关报道和专利,目前,研究人员主要利用食神鞘氨醇杆菌生产酒精脱氢酶,与解淀粉芽孢杆菌、肠系膜明串珠菌混合使用用于处理难降解废水,与荧光假单胞菌形成组合菌处理种子以保护植物免收病原体感染等。另外,通过与文献中已经报道的肝素酶在来源、分子量、对肝素和HS降解的特异性以及酶切位点等方面进行比较后发现,SShepⅠ和SShepⅡ与其他肝素酶不同。证实了肝素酶SShepⅠ和SShepⅡ为两种前未报道过的新型肝素酶。
最后,用得到的新肝素酶试制低分子肝素,显示SShepⅠ酶解产物的分子量分布在2-8 kD,抗FXa效价126 IU/mg,抗FⅡa效价29 IU/mg;SShepⅡ酶解产物的分子量分布在0.6-4 kD,抗FXa效价为135 IU/mg,抗FⅡa效价为8 IU/mg。这两种新型酶制低分子肝素与目前已有的药品低分子肝素如依诺肝素、达肝素、亭扎肝素等性能都有所区别,有望开发成抗凝血效果更好,或具有新临床功能的低分子肝素药物。此外,在后续的研究工作中,我们还需对两种新型肝素酶酶切位点进行深入研究,分析其酶切肝素产物的结构特点,明确其酶切位点。
4 结论本研究采用定向富集方法从土壤中筛选分离得到产肝素酶的新种菌株CDF1-2,属食神鞘氨醇杆菌(Sphingo spiritivorum)。之后利用微生物发酵和蛋白纯化技术制备得到新型肝素酶SShepⅠ和SShepⅡ,并对其理化性质和酶切位点进行了初步研究,最后,用得到的两种新型肝素酶降解肝素得到两种新型酶制低分子肝素。
| [1] | Gray E, Mulloy B, Barrowcliffe TW. Heparin and low-molecular-weight heparin[J]. Thrombo Haemost, 2008, 99 (5): 807–818. |
| [2] | Fareed J, Ma Q, Florian M, et al. Differentiation of low molecular-weight heparins:impact on the future of the management of thrombosis[J]. Semin Thromb Hemost, 2004 (30 Suppl. 1): 89–104. |
| [3] | 杨明康, 李晓燕, 钱捷, 等. 低分子量肝素的研究进展[J]. 齐鲁药事, 2012, 31(11): 663–666. |
| [4] | 李晔, 吴敬君, 叶逢春, 等. 肝素黄杆菌肝素酶Ⅲ基因hepC的可溶表达体系构建及酶学性质研究[J]. 2013, (6): 133-139. |
| [5] | Ma XL, Wang ZS, Li SX, et al. Effect of CaCl2 as activity stabilizer on purification of heparinase Ⅰ from Flavobacterium heparinum[J]. Journal of Chromatography B, 2006 (843): 209–215. |
| [6] | Kazuyuki S. Characterization of heparinase from an oral bacterium Prevotella heparinolytica[J]. J Biochem, 1998, 123 (2): 283–288. DOI:10.1093/oxfordjournals.jbchem.a021934 |
| [7] | 高宁国, 程秀兰, 杨敬, 等. 肝素酶产生菌的筛选及发酵条件[J]. 微生物学报, 1999, 39(1): 64–67. |
| [8] | Yoshida E, Sakai K, Tokuyam S, et al. Purification and characteriz-ation of heparinase that degrades both heparin and heparin sulfate from Bacillus circulans[J]. Biosci Biotechnol Biochem, 2002, 66 (5): 1181–1184. DOI:10.1271/bbb.66.1181 |
| [9] | 罗璠, 王忠彦, 等. 肝素酶产生菌的筛选及其粗酶性质的研究[J]. 四川大学学报:自然科学版, 2002, 39(4): 777–779. |
| [10] | 高宁国, 程秀兰, 杨敬, 等. 鞘胺醇杆菌肝素酶的产生[J]. 微生物学报, 2003, 43(6): 813–816. |
| [11] | Kim DH. Purification and characterization of a novel heparinase from Bacteroides stercoris HJ-15[J]. Journal of Biochemistry, 2000, 128 (2): 323–328. DOI:10.1093/oxfordjournals.jbchem.a022756 |
| [12] | Lowry OH, Roscbrough NJ, Farr AL, et al. Protein measurement with the folin phenol regent[J]. Journal of Biological Chemistry, 1951, 193 (1): 265–275. |
| [13] | 何忠效、张树政. 电泳第2版[M]. 北京: 科学出版社, 1999: 127-130. |
| [14] | The united states Pharmacopeial convention. USP 2003, U. S. A. :United Stated Pharmacopeial Convention, 2003:USP39-NF34. Chemical Test and Assays < 209 > 232-233. |
| [15] | The united states Pharmacopeial convention. USP 2003, U. S. A. :United Stated Pharmacopeial Convention, 2003:USP39-NF34. Chemical Test and Assays < 208 > 230-232. |
| [16] | Daniel LL, Robert JL. Purification and characterizatioonf heparin lyases from Flavobacterium heparinurn[J]. The Journal of Biological Chemistry, 1992, 267 (34): 24347–2435. |
| [17] | Hyun YJ, Lee JH, et al. Cloning, overexpression, and characteriz-ation of recombinant heparinase Ⅲ from Bacteroides stercoris HJ-15[J]. Appli Microbiol Biötechnol, 2010, 86 : 879–890. DOI:10.1007/s00253-009-2327-7 |
| [18] | Kim WS, Kim BT, Kim DH, et al. Purification and characterization of heparin lyase Ⅰ from Bacteroides stercoris HJ-15[J]. Journal of Biochemistry and Molecular Biology, 2004, 37 (6): 684–690. |
| [19] | Nakamura T, Suginaka Y, Takazoe I. Heparinase activity in lesion of periodontal diseases[J]. Bulletin of Tokyo Dental College, 1976, 17 : 147–155. |
| [20] | Yoshida E, Arakawa S, Matsunaga T, et al. Cloning, sequencing, and expression of the gene from Bacillus circulans that codes for a heparinase that degrades both heparin and heparan sulfate[J]. Biosci Biotechnol Biochemi, 2002, 66 : 1873–1879. DOI:10.1271/bbb.66.1873 |
| [21] | Nakamura T, Shibata Y, Fujimura S. Purification and properties of Bacteroides heparinolyticus heparinase(heparin lyase, EC 4. 2. 2. 7)[J]. J Clin Microbiol, 1988 (26): 1070–1071. |
| [22] | Bohmer LH, Pitout MJ, Steyn PL, et al. Purification and characterization of a novel heparinase[J]. Journal of Biological Chemistry, 1990, 265 : 13609–13617. |
| [23] | Banga J, Tripathi CK. Purification and characterization of a novel heparin degrading enzyme from Aspergillus flavus(MTCC-8654)[J]. Appl Environm Microbiol, 2010, 160 : 1004–1016. |
| [24] | Banga J, Tripathi CK. Rapid purification and characterization of a novel heparin degrading enzyme from Acinetobacter calcoaceticus[J]. New Biotechnology, 2009, 26 (11): 100–104. |
| [25] | Maraveyas A, Johnson MJ, et al. Malignant melanoma as a target malignancy for the study of the antimetastatic properties of the heparins[J]. Cancer Metastasis Rev, 2010, 29 (4): 777–784. DOI:10.1007/s10555-010-9263-y |
| [26] | Luo JY, Zhong Y, Cao JC, et al. Efficacy of oral colon-specific delivery capsule of low-molecular-weight heparin on ulcerative colitis[J]. Biomed Pharmacother, 2011, 65 (2): 111–117. DOI:10.1016/j.biopha.2010.12.013 |
| [27] | Kupferminc M, Rimon E, Many A, et al. Low molecular weight heparin versus no treatment in women with previous severe pregnancy complications and placental findings without thrombophilia[J]. Blood Coagul Fibrinolysis, 2011, 22 (2): 123–126. DOI:10.1097/MBC.0b013e328343315c |