2. 山东省医药生物技术研究中心,济南 250062
2. Shandong Medicinal Biotechnology Center, Ji'nan 250062
克拉维酸(Clavulanic acid,CA)是一种高效的β-内酰胺酶抑制剂,广泛应用于临床治疗β-内酰胺类抗生素耐药菌的感染[1]。棒状链霉菌(Streptomyces clavuligerus)是CA的工业生产菌株,但是S. clavuligerus除合成CA外同时也合成头霉素C、全霉素和多种5S克拉维烷类物质,其中克拉维烷和CA具有共同的前期合成途径,与CA竞争前体物质[2],这些副产物不仅影响CA产量的提高同时也增加了纯化工艺的难度。
S. clavuligerus ATCC27064的基因组草图已经完成测序[3, 4],CA的生物合成途径和基因簇也已基本阐明[5-7]。S. clavuligerus中3个基因簇与CA合成有关,分别是CA基因簇(CA gene cluster)、克拉维烷基因簇(Clavam gene cluster)和旁系同源基因簇(Paralogue gene cluster),CA基因簇和克拉维烷基因簇位于基因组,而旁系同源基因簇位于pSCL4质粒[8]。CA基因簇中ceaS2、bls2、pah2、cas2、oat2、oppA1、oppA2、cad、cyp、fd、gcas、orf12和orf16,克拉维烷基因簇中cas1和旁系同源基因簇中ceaS1、bls1、pah1、oat1都参与了CA的合成,cas1、ceaS1、bls1、pah1和oat1是CA基因簇中相应基因的同源基因[9]。
CA合成分为早期途径和晚期途径,早期途径以精氨酸和3-磷酸甘油醛(G3P)为起始原料,在Ceas1/Ceas2、Bls1/Bls2、Pah1/Pah2、Cas1/Cas2等酶的作用下形成中间体克拉维胺酸(Clavaminic acid),晚期途径在Gcas、Cad、Cyp和Fd等酶的作用下将克拉维胺酸转化为CA[9]。克拉维胺酸是CA和克拉维烷合成途径的分支点,部分克拉维胺酸在Gcas的催化下进入CA晚期途径,另一部分克拉维胺酸在Cvm1等克拉维烷合成酶的催化下形成多种5S克拉维烷产物[2]。CA基因簇上游紧挨着头霉素C基因簇,研究显示头霉素C基因簇中ccaR编码一个途径特异性调控因子CcaR,正调控CA合成的早期途径基因ceas2、bls2、pah2和cas2,以及调控基因claR,而claR正调控晚期途径基因oppA1、cad和cyp[10, 11]。
S. clavuligerus F613-1是CA的工业生产菌株,在发酵罐中CA发酵水平达到为4.87 g/L[12]。我们前期完成了F613-1菌株的全基因组测序[13],对全基因组测序数据比较分析显示:与标准菌株ATCC27064相比,F613-1丢失了pSCL1、pSCL2和pSCL3等3个质粒,以及pSCL4质粒的1.1 Mb DNA序列,但是CA基因簇、克拉维烷基因簇和旁系同源基因簇中不存在大规模的基因突变和缺失现象,仅存在数个SNP,这些SNP可能不是F613-1高产CA的主要原因。本研究拟通过转录组测序比较分析F613-1和ATCC27064在转录水平的差异,分析CA合成原料精氨酸及G3P代谢相关基因、CA及其多种副产物合成及调控相关基因,以及ABC转运系统基因的变化情况,以期在转录水平阐明F613-1高产CA的分子机制,为进一步通过基因工程方法提高工业菌株CA产量提供依据。
1 材料与方法 1.1 材料 1.1.1 菌株S. clavuligerus工业生产菌株F613-1和标准菌株ATCC27064。
1.1.2 培养条件固体培养:将孢子涂布于BSCA培养基(含1.5%麦芽提取物、0.3%胰蛋白胨、0.4%葡萄糖、2%琼脂粉,pH7.5) 上,25℃培养10 d收集孢子。液体发酵培养:按106个/mL的接种量将孢子接种于TSB(OXOID)培养基中,25℃、250 r/min培养48 h得到种子液,然后按5%接种量转接至发酵培养基(2.0%大豆超细粉,1.2%玉米淀粉,0.5%酵母提取物,0.08%磷酸氢二钾,pH8.0),25℃、250 r/min。
1.2 方法 1.2.1 固体培养表型比较将F613-1和ATCC27064的孢子分别稀释至106个/mL,各取10 μL孢子悬液划线接种到同一个BSCA平板的两侧,25℃培养,观察生长情况变化。
1.2.2 发酵液总糖、还原糖和生物量测定取培养好的发酵液10 mL,12 000 r/min,4℃,离心10 min,沉淀去除残液后称重计算生物量,上清经0.22 μm过滤后进行糖含量检测。滤液适当稀释后直接进行还原糖含量检测,总糖先经盐酸完全水解,然后用氢氧化钠中和,适当稀释后测定糖含量。糖含量检测采用DNS(二硝基水杨酸)比色法进行。
1.2.3 发酵液的HPLC检测取1.2.2中发酵液离心获得的上清,经0.22 μm过滤后进行HPLC检测。仪器为安捷伦1260,色谱柱为Diamonsil 5 μm C18(250×4.6 mm),柱温40℃,检测波长为230 nm,进样量20 μL。流动相A为0.05 mol/L磷酸二氢钠(磷酸调pH至4.0),流动相B为等体积的甲醇和流动相A混合。洗脱条件:0-4 min为100% A;4-15 min,A由100%逐渐降至50%,B由0%逐渐提高到50%;15-18 min,保持50% A和50% B;流速1 mL/min。
1.2.4 转录组测序与分析链霉菌总RNA采用TRIzol(Ambion)抽提法提取,检验合格后去除rRNA和片段化处理,然后逆转录合成cDNA,连接上接头后进行PCR扩增并纯化,构建PE100文库。PE100文库质检合格后上机测序,高通量测序采用illumina Hiseq2500平台(广州瑞博生物)。原始测序数据先进行去接头序列和低质量序列等处理获得高质量数据,然后用参考基因S. clavuligerus ATCC27064(NZ_CM001015.1) 进行比对。
转录组数据采用Tophat2软件对测序数据覆盖区域及覆盖深度等做综合评估[14];用Audics软件对基因表达进行差异分析[15],通过差异倍数(|log2FoldChange| > 1) 和显著水平(q-value < 0.001) 两个水平挑选出样本间差异表达的基因。KEGG代谢通路分析中差异基因的显著性阈值设为P < 0.05。
2 结果 2.1 F613-1和ATCC27064的固体培养表型比较每隔24 h观察并记录F613-1和ATCC27064在BSCA平板上的生长情况,结果表明与ATCC27064相比,F613-1的生长速度和产孢速度较慢(图 1-A),在BSCA培养上不产色素而ATCC27064合成紫红色的色素(图 1-B)。此外,F613-1气生菌丝较为疏松,产孢(孢子为灰色)数量明显减少(图 1-C和D),在培养后期易与培养基脱离。

|
| 图 1 F613-1与ATCC27064在BSCA培养基上培养表型对比图 A:平板正面;B:平板背面;C,D:分别为F613-1与ATCC27064培养5 d的菌落显微图 |
本研究分别收集F613-1和ATCC27064摇瓶发酵72、96、120和144 h等4个时间点的发酵液,分别进行总糖、还原糖和生物量测定,并用HPLC检测发酵液中CA和非CA物质的合成情况。结果(图 2-A)显示,F613-1与ATCC27064在各时间点的生物量相近(数据未显示),还原糖含量变化趋势也无明显差异,但F613-1的总糖消耗速度明显降低,说明F613-1的糖利用和转化能力可能优于ATCC27064。HPLC分析(图 2-B)显示,F613-1发酵144 h时CA产量达到3.86 g/L,CA峰面积占总峰面积的67.68%,CA产量比ATCC27064提高了约11.7倍,CA峰面积占总峰面积的比例提高了约3.6倍,这表明F613-1的代谢流发生变化,使CA合成量增加而副产物合成量相对减少。

|
| 图 2 F613-1与ATCC27064摇瓶发酵参数对比图 A:发酵时间与总糖及还原糖消耗情况关系;B:发酵时间与CA产量(柱状图)及HPLC检测中CA峰面积占总峰面积百分比(折线图)关系 |
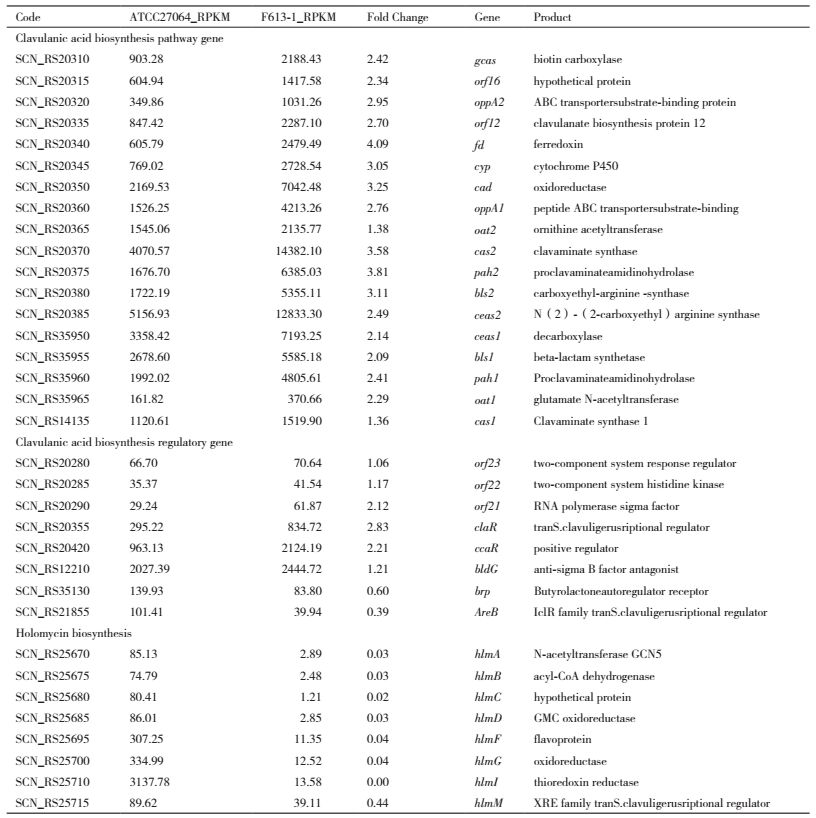
本研究收集了液体发酵72 h(CA合成高峰期)的F613-1和ATCC27064菌丝体,提取总RNA并构建文库,用illuminaHiseq2500测序平台进行测序,测序方式为PE100。测序数据经过质量筛选,F613-1一共得到15 854 608个有效mRNA信息,基因覆盖率为95.26%;ATCC27064一共得到17 798 172个有效mRNA信息,基因覆盖率为96.11%。通过Audics软件分析基因转录水平差异,从样本间基因差异火山图(图 3)可以看出ATCC27064和F613-1之间存在统计意义上的显著差异。与ATCC27064相比较,F613-1菌株共有798个基因的表达量明显发生改变,其中211个基因表达量显著升高,587个基因表达量显著下降,说明F613-1高产CA的变化可能是由部分基因的转录水平变化引起。
|
| 图 3 F613-1和ATCC27064样本间基因差异分析火山图 纵坐标代表显著水平,即log2(P-value);横坐标代表(log ratios),表示log2(Fold Change);红色圆点表示有显著性差异的基因 |
差异分析显示,F613-1中CA基因簇的大部分基因都上调了2-4倍(表 1),其中CA的途径特异性正调控基因claR上调2.83倍,CA早期途径的关键合成基因cas2、pah2和bls2,以及晚期途径的关键合成基因fd、cyp和cad变化最为明显,差异倍数达到3-4倍。此外,CA基因簇上游的头霉素C基因簇同样也整体上调了2-4倍,其中正调控头霉素C和CA合成途径的ccaR基因上调2.21倍(表 1)。与此同时,旁系同源基因簇整体上调2-3倍,其中与CA合成相关的基因ceaS1、pah1、bls1、oat1均上调2-3倍,但克拉维烷正调控基因cvm7P无明显变化,而克拉维烷基因簇整体转录水平也无明显变化,这可能是F613-1大量合成CA而克拉维烷水平较低的原因。全霉素合成基因整体下调4-230倍,个别基因几乎不表达(表 1),已有研究表明全霉素是CA合成的竞争性途径,说明全霉素基因簇沉默是F613-1高产CA的原因之一。总体来看,F613-1菌株中次级代谢产物合成基因簇的基因表达变化使得前体物质更多的流向CA合成途径。
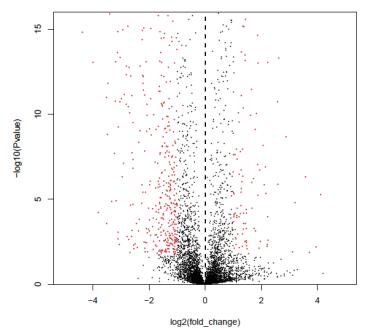
精氨酸和G3P是CA合成途径的两个起始物质。基于KEGG生物学通路数据库的生物通路富集分析发现,精氨酸生物合成通路中多个基因出现连续上调,包括从谷氨酸盐到鸟氨酸(argA、argB、argC/e、argD、argE、argJ),N-乙酰-L瓜氨酸到瓜氨酸(argE),及尿素循环途径中从鸟氨酸到精氨酸(argI/ argF、argG、argA、argH)的基因(图 4-A)。这些基因转录水平的大幅上调可能使F613-1积累更多的精氨酸,促进CA合成。
|
| 图 4 F613-1中精氨酸生物合成途径基因及G3P代谢途径基因表达差异图 红色代表显著上调基因,绿色代表显著下调基因,黑色为无明显差异基因 |
G3P既是CA合成途径的起始物质(图 4-B),同时也是糖酵解途径和磷酸戊糖途径的重要中间产物。分析显示,F613-1菌株中糖酵解途径中果糖1,6-二磷酸酶(fbp)下调2.3倍,减弱了果糖1,6-二磷酸的糖异生反应,有利于G3P合成;同时磷酸丙糖异构酶(tpiA)和G3P脱氢酶(gapA)表达上调,差异倍数分别为2.2倍和2.3倍,有利于甘油代谢产物转化为G3P;此外丙酮酸脱氢酶复合酶系均下调2-4倍,使得糖酵解产物丙酮酸去路受阻(图 4-C);另一方面,柠檬酸循环途径中柠檬酸合酶及亚基分别下调2.4倍和6.1倍,α-酮戊二酸脱氢酶E2组件(sucB)下调4.2倍,进一步减少了丙酮酸的消耗,有利于G3P积累(图 4-D)。
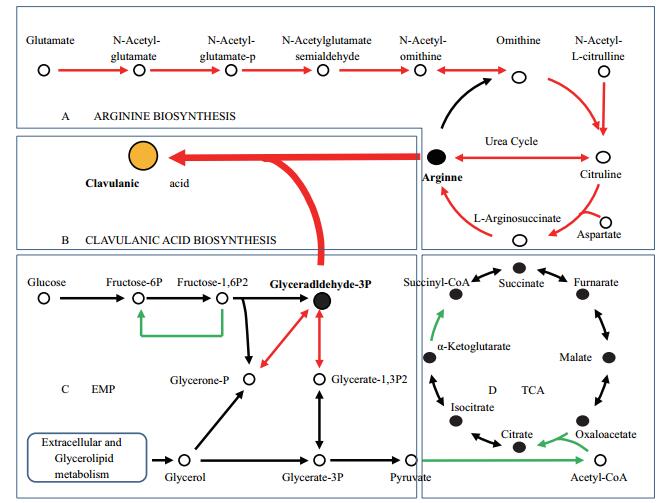
2.6 ABC转运系统基因的表达变化S. clavuligerus中存在众多ABC转运系统基因,分析显示F613-1中有多达80个ABC转运系统相关基因(ABC转运蛋白、ABC转运体透性酶、ABC转运体底物结合蛋白、ABC转运体ATP结合蛋白及ATP酶等)表达水平发生显著变化,占总差异基因的10%,其中42个显著上调,38个显著下调。通过差异基因KEGG代谢通路分析,找到了12个功能明确的ABC转运系统差异表达基因,其中包括负责氨基酸、糖、磷脂和金属离子转运的ABC转运系统(图 5)。
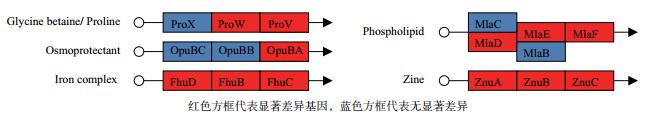
|
| 图 5 F613-1与ATCC27064部分ABC转运系统基因表达变化情况及其功能示意图 红色方框代表显著差异基因,蓝色方框代表无显著差异 |
在S. clavuligerus工业菌株育种的早期,提高CA产量的方法主要以传统的随机诱变筛选为主,但随着CA生物合成途径的逐步阐明,越来越多的基因工程及反向代谢工程育种方法已经应用到S. clavuligerus菌株改造中。超表达CA合成通路基因及调控基因(如claR、ccaR、cas2等)是已被证明有效的提高CA产量的基因工程方法[16]。本研究的转录组数据显示工业菌株F613-1中CA合成基因的表达量虽然整体上调,但是上调幅度并不是很大,特别是合成途径几个关键的基因如ceas1(上调2.14倍)、ceas2(上调2.49倍)和gcas(上调2.42倍)仍然存在进一步提升的空间。其中ceas1/ceas2编码CA合成途径的起始酶,催化精氨酸和G3P合成羧乙酰精氨酸[N2-(2-carboxyethyl)arginine][17],gcas是CA合成途径的关键基因,该基因编码N-甘氨酰-克拉维胺酸合成酶(N-glycyl-clavaminic acid synthetase),是CA前体克拉维胺酸转入CA合成分支途径的关键基因[18]。另一种提高CA产量的策略是阻断副产物的生物合成途径。CA、头霉素C和克拉维烷都属于β-内酰胺类次级代谢产物,三者合成途径属于竞争关系。研究表明敲除头霉素C合成关键基因lat,使CA产量显著提高[19-20]。本研究中F613-1的头霉素C基因簇明显上调,且lat上调幅度接近3倍,不利于CA的合成,因此在基因工程改造中可以敲除F613-1菌株的lat基因。克拉维烷是CA前体的直接竞争者,研究显示调控基因cvm7P正调控克拉维烷的合成基因,对CA合成基因无影响[21],而F613-1中cvm7P转录水平无显著变化,因此敲除F613-1菌株的cvm7P基因可能有助于提高CA产量。此外,研究表明全霉素的合成受精氨酸和CA途径后期中间产物的影响,说明全霉素与CA也可能存在竞争关系[22],因此本研究中F613-1全霉素合成基因的沉默有可能会促进CA的合成。
CA的调控基因中除claR和ccaR变化比较明显外,CA基因簇末端的一个编码σ因子的基因(orf21)也上调了2.12倍(表 1)。文献报道显示,orf21可上调CA早期合成基因ceas2和cas2及调控基因ccaR的转录[23]。文献报道双组分调控系统orf22/orf23可以正调控claR进而提高CA产量[24],但是本研究显示F613-1中orf22/orf23的转录水平无显著变化(表 1)。此外,CA合成途径的正调控因子bldG[25]也在F613-1中无显著变化(表 1)。因此,orf21、orf22/orf23和bldG等调控基因也可以作为提高F613-1 CA产量的改造靶点。
Townsend等[26]用同位素标记法研究了氨基酸对CA合成的影响,发现在CA合成过程中鸟氨酸和精氨酸比其它氨基酸更易被利用,且CA噁唑环结构直接来源于尿素循环。鸟氨酸和精氨酸是尿素循环中两种重要的氨基酸,研究发现鸟氨酸可抑制S. clavuligerus中头霉素C的合成[27]。本研究发现F613-1中由谷氨酸盐起经鸟氨酸到精氨酸代谢途径的基因整体表达上调,明显有利于CA合成。由于谷氨酸成本更加经济,因此在F613-1发酵中可以采用谷氨酸替代鸟氨酸和精氨酸作为CA前体添加剂。
甘油是CA工业发酵常用的添加前体[28],其衍生物G3P是CA合成的限速因素[29-30]。Medema等[31]通过转录组分析另一株CA高产菌株S. clavuligerus DS48802发现,与ATCC27064相比,DS48802中甘油摄取及转化到G3P的基因明显上调,同时三羧酸循环中顺乌头酸酶和柠檬酸合酶基因表达下降,这使得G3P积累并有利于流向CA合成途径。本研究中F613-1的甘油耐受能力显著高于ATCC27064,与G3P合成相关的基因上调而G3P分解基因下调进而积累G3P,与文献报道一致,但是甘油转运及代谢基因(包括甘油转运蛋白基因RS04225/RS03030,甘油激酶基因RS04230/RS03035,α-甘油磷酸脱氢酶基因RS04235/RS21895) 的转录水平无明显变化。此外,F613-1中丙酮酸氧化降解途径受阻虽然有利于G3P积累,但另一方面也减弱了细胞能量供应,影响F613-1的生长速度。因此,通过提高F613-1中甘油转运和G3P合成基因的表达,将有可能改善F613-1生长状况并进一步提高CA产量。
磷酸盐、硫酸盐、脂肪酸、Fe3+等也是发酵过程中影响CA产量的重要因素[32-33],这些物质都是ABC转运系统的底物,F613-1中ABC转运系统基因的表达差异可能影响S. clavuligerus对这些物质的吸收,进而促进CA的合成。此外,本研究还发现S. clavuligerus中唯一的溶菌酶基因(RS20110) 在F613-1中上调了5倍,我们推测过量表达的溶菌酶可能增加细胞壁通透性,有利于细胞摄取营养物质进而促进CA合成。因此,改造工业菌株的营养物质转运系统和细胞膜通透性也可能是提高CA产量的靶点之一。
4 结论CA是S. clavuligerus的次级代谢产物,工业菌株F613-1具有较高的CA合成能力,并且克拉维烷等副产物合成量较低。本研究表明F613-1中CA生物合成途径的合成基因及调控基因的整体上调是其高产CA的主要原因;精氨酸生物合成通路基因的显著上调以及G3P代谢途径基因的表达变化也直接促进了CA生成;全霉素合成基因的大幅下调可能使更多的前体物质流向CA合成途径。此外,大量ABC转运基因差异表达也可能对CA产生起到重要影响。
| [1] | Saudagar PS, Survase SA, Singhal RS. Clavulanic acid:A review[J]. Biotechnol Adv, 2008, 26 (4): 335–351. DOI:10.1016/j.biotechadv.2008.03.002 |
| [2] | Jensen SE. Biosynthesis of clavam metabolites[J]. J Ind Microbiol Biotechnol, 2012, 39 (10): 1407–1419. DOI:10.1007/s10295-012-1191-0 |
| [3] | Medema MH, Trefzer A, Kovalchuk A, et al. The sequence of a 1. 8-mb bacterial linear plasmid reveals a rich evolutionary reservoir of secondary metabolic pathways[J]. Genome Biol Evol,, 2010, 2 (1): 212–224. |
| [4] | Song JY, Jeong H, Yu DS, et al. Draft genome sequence of Streptomyces clavuligerus NRRL 3585, a producer of diverse secondary metabolites[J]. J Bacteriol, 2010, 192 (23): 6317–6318. DOI:10.1128/JB.00859-10 |
| [5] | Thirkettle JE, Baldwin JE, Edwards J, et al. The origin of the β-lactam carbons of clavulanic acid[J]. J Chem Soc, Chem Commun, 1997, 28 (11): 1025–1026. |
| [6] | Miller MT, Bachmann BO, Townsend CA, et al. The catalytic cycle of beta-lactam synthetase observed by x-ray crystallographic snapshots[J]. Proc Natl Acad Sci U S A, 2002, 99 (23): 14752–14757. DOI:10.1073/pnas.232361199 |
| [7] | Caines ME, Sorensen JL, Schofield CJ. Structural and mechanistic studies on N(2)-(2-carboxyethyl)arginine synthase[J]. Biochem Biophys Res Commun, 2009, 385 (4): 512–517. DOI:10.1016/j.bbrc.2009.05.095 |
| [8] | Liras P, Gomez-Escribano JP, Santamarta I. Regulatory mechanisms controlling antibiotic production in Streptomyces clavuligerus[J]. J Ind Microbiol Biotechnol, 2008, 35 (7): 667–676. DOI:10.1007/s10295-008-0351-8 |
| [9] | Song JY, Jensen SE, Lee KJ. Clavulanic acid biosynthesis and genetic manipulation for its overproduction[J]. Appl Microbiol Biotechnol, 2010, 88 (3): 659–669. DOI:10.1007/s00253-010-2801-2 |
| [10] | Paradkar AS, Aidoo KA, Jensen SE. A pathway-specific transcriptional activator regulates late steps of clavulanic acid biosynthesis in Streptomyces clavuligerus[J]. Mol Microbiol, 1998, 27 (4): 831–843. DOI:10.1046/j.1365-2958.1998.00731.x |
| [11] | 朱碧银, 洪文荣. 棒状链霉菌控制抗生素合成的调节机制[J]. 中国抗生素杂志, 国外医药抗生素分册, 2010, 31(6): 271–278. |
| [12] | Jin X, Cao G, Zhang X, et al. Studies on the formation and synthetic mechanism of related substance G in potassium clavulanate production[J]. Braz J Pharm Sci, 2015, 51 (1): 77–83. DOI:10.1590/S1984-82502015000100008 |
| [13] | Cao G, Zhong C, Zong G, et al. Complete genome sequence of Streptomyces clavuligerus F613-1, an industrial producer of clavulanic acid[J]. Genome Announc, 2016, 4 (5): e01020–16. |
| [14] | Trapnell C, Pachter L, Salzberg SL. TopHat:discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25 (9): 1105–1111. DOI:10.1093/bioinformatics/btp120 |
| [15] | Gao D, Kim J, Kim H, et al. A survey of statistical software for analysing RNA-seq data[J]. Hum Genomics, 2010, 5 (1): 56–60. DOI:10.1186/1479-7364-5-1-56 |
| [16] | Hung TV, Malla S, Park BC, et al. Enhancement of clavulanic acid by replicative and integrative expression of ccaR and cas2 in Streptomyces clavuligerus NRRL3585[J]. J Microbiol Biotechnol, 2007, 17 (9): 1538–1545. |
| [17] | Tahlan K, Anders C, Jensen SE. The paralogous pairs of genes involved in clavulanic acid and clavam metabolite biosynthesis are differently regulated in Streptomyces clavuligerus[J]. J Bacteriol, 2004, 186 (18): 6286–6297. DOI:10.1128/JB.186.18.6286-6297.2004 |
| [18] | Jensen SE, Paradkar AS, Mosher RH, et al. Five additional genes are involved in clavulanic acid biosynthesis in Streptomyces clavuligerus[J]. Antimicrob Agents Chemother, 2004, 48 (1): 192–202. DOI:10.1128/AAC.48.1.192-202.2004 |
| [19] | 舒杨, 何建勇, 田威, 等. 棒状链霉菌中lat基因的置换与克拉维酸的选择性生产[J]. 沈阳药科大学学报, 2005, 22(1): 62–66. |
| [20] | Wang YH, Jing CF, Tao MF, et al. Effect of lat disruption on clavulanic acid production[J]. Acta microbiologica Sinica, 2005, 45 (4): 500–503. |
| [21] | Tahlan K, Anders C, Wong A, et al. 5S clavam biosynthetic genes are located in both the clavam and paralog gene clusters in Streptomyces clavuligerus[J]. Chem Biol, 2007, 14 (2): 131–142. DOI:10.1016/j.chembiol.2006.11.012 |
| [22] | De LFA, Lorenzana LM, Martín JF, et al. Mutants of Streptomyces clavuligerus with disruptions in different genes for clavulanic acid biosynthesis produce large amounts of holomycin:possible cross-regulation of two unrelated secondary metabolic pathways[J]. J Bacteriol, 2002, 184 (23): 6559–6565. DOI:10.1128/JB.184.23.6559-6565.2002 |
| [23] | Jnawali HN, Liou K, Sohng JK. Role of σ-factor(orf21)in clavulanic acid production in Streptomyces clavuligerus NRRL3585[J]. Microbiol Res, 2011, 166 (5): 369–379. DOI:10.1016/j.micres.2010.07.005 |
| [24] | Song JY, Kim ES, Kim DW, et al. A gene located downstream of the clavulanic acid gene cluster in Streptomyces clavuligerus ATCC 27064 encodes a putative response regulator that affects clavulanic acid production[J]. J Ind Microbiol Biotechnol, 2009, 36 (2): 301–311. DOI:10.1007/s10295-008-0499-2 |
| [25] | Bignell DR, Tahlan K, Colvin KR, et al. Expression of ccaR, encoding the positive activator of cephamycin C and clavulanic acid production in Streptomyces clavuligerus, is dependent on bldG[J]. Antimicrob Agents Chemother, 2005, 49 (4): 1529–1541. DOI:10.1128/AAC.49.4.1529-1541.2005 |
| [26] | Townsend CA, Ho MF. Biosynthesis of clavulanic acid:origin of the C5 Unit[J]. J Am Chem Soc, 1985, 107 (4): 1065–1066. DOI:10.1021/ja00290a056 |
| [27] | Romero J, Liras P, Martin JF. Utilization of ornithine and arginine as specific precursors of clavulanic acid[J]. Appl Environ Microbiol, 1986, 52 (4): 892–897. |
| [28] | Ser HL, Law JW, Chaiyakunapruk N, et al. Fermentation conditions that affect clavulanic acid production in Streptomyces clavuligerus:a systematic review[J]. Front Microbiol, 2016, 7 (856): 522. |
| [29] | Ives PR, Bushell ME. Manipulation of the physiology of clavulanic acid production in Streptomyces clavuligerus[J]. Microbiology, 1997, 143 (Pt 11): 3573–3579. |
| [30] | Khaleeli N, Li RF, Townsend CA. Origin of the β-Lactam carbons in clavulanic acid from an unusual thiamine pyrophosphate-mediated reaction[J]. J Am Chem Soc, 1999, 121 (39): 9223–9224. DOI:10.1021/ja9923134 |
| [31] | Medema MH, Alam MT, Heijne WH, et al. Genome-wide gene expression changes in an industrial clavulanic acid overproduction strain of Streptomyces clavuligerus[J]. Microb Biotechnol, 2011, 4 (2): 300–305. DOI:10.1111/j.1751-7915.2010.00226.x |
| [32] | Large KP, Ison AP, Williams DJ. The effect of agitation rate on lipid utilization and clavulanic acid production in Streptomyces clavuligerus[J]. J Biotechnol, 1998, 63 (2): 111–119. DOI:10.1016/S0168-1656(98)00082-0 |
| [33] | 蒋顺进, 杨亚勇, 王惠青. 克拉维酸发酵工艺的优化研究[J]. 中国抗生素杂志, 2004, 29(6): 335–337. |