燃料乙醇是可替代化石能源的一种清洁可再生能源,其生产技术研发受到全球关注[1]。木质纤维素原料如农业秸秆等具有廉价、可再生、来源广泛、不存在“与人争粮”等优点,是今后燃料乙醇生产的主要原料。木质纤维素原料经过预处理和水解转化为单糖,主要是葡萄糖和木糖,微生物发酵单糖可产生乙醇。酿酒酵母具有优秀的产乙醇能力和环境耐受力等优点,广泛用于乙醇的生产,但酿酒酵母用于纤维素原料的燃料乙醇生产,面临两个重要问题,一是木糖的发酵,二是对各种抑制物的耐受能力。传统的酿酒酵母不能利用木糖产乙醇,需要外源导入木糖还原酶(Xylose reductase,XR)基因和木糖醇脱氢酶(Xylitol dehydrogenase,XDH)基因或木糖异构酶(Xylose isomerase,Ⅺ)基因,使之具备利用木糖的能力[2]。到目前为止,已构建出较多的优秀木糖发酵菌株[3-5]。但是相比木糖的发酵,菌株是否具有优秀的抑制物耐受能力直接决定了菌株能否应用于实际生产过程。木质纤维素原料在预处理和水解过程中产生各种副产物,主要包括弱酸类(甲酸、乙酸和乙酰丙酸)、呋喃类(糠醛、5-羟甲基糠醛)和酚类(丁香醛、香草醛和苯酚等)[6],它们的存在会严重阻碍酿酒酵母菌株的细胞生长和乙醇发酵[7],因此提高酿酒酵母的抑制物耐受性对燃料乙醇工业化生产尤为重要。
目前有关酿酒酵母抑制物耐受性的研究主要集中在两个方面,一是通过调控某些基因的表达提高菌株的抑制物耐受性;二是通过进化工程提高菌株的抑制物耐受性[8]。基因表达调控的前提是对某种或某类抑制物的抑制机制清楚,涉及的目标基因明确且数量较少,目前在此研究方向有一些研究报道,但多是针对单一抑制物[9, 10],因此基因工程手段对于提升菌株同时对多种抑制物的耐受比较困难。由于目前对酿酒酵母的抑制物耐受机制了解甚少,相比基因工程手段,利用进化工程提升菌株的抑制物耐受性具有一定的优势[11]。已有的一些研究报道,包括针对单一抑制物、混合抑制物、及利用实际糖化液进行驯化,结果都表明进化工程是一个能有效提升抑制物耐受性的手段[12]。但目前相关研究存在以下问题:(1) 针对葡萄糖发酵研究较多,针对木糖发酵的相关研究较少,而木糖发酵受抑制物抑制的程度显著高于葡萄糖发酵[12, 13];(2) 研究多针对单一抑制物,即使是混合抑制物,种类也多局限于两种[14-16];(3) 利用实际糖化液进行驯化,对于获得能耐受某种特定糖化液是有效的,但获得的耐受菌株不一定就能适应其它糖化液的发酵,因为原料、预处理及水解过程会显著影响抑制物组成和分布[17];(4) 绝大部分研究使用的是实验室菌株,其与工业菌株间遗传背景的显著差异使研究成果难以适用于工业菌株。因此,要构建能适用于纤维素燃料乙醇工业化生产的菌株,需要针对工业菌株的木糖发酵过程提升其对各种抑制物的耐受性,使菌株具有一定的普适性。
本课题组以工业酿酒酵母菌株KF-7为出发菌株,通过基因工程手段构建了性能优良的木糖发酵工业菌株[18-20],具备优良木糖发酵性能且对多种抑制物具有较好的耐受能力[18]。但这些菌株利用实际糖化液发酵时木糖的利用被显著抑制,表明需要提升菌株对于多种抑制物共存时的耐受力。本研究以木糖发酵工业菌株KF7-M16为出发菌株[19],通过紫外诱变和驯化工程组合手段,提升其在木糖发酵条件下对3类共9种典型抑制物[21-23]共存时的耐受能力,以期获得具有工业应用潜力的突变菌株。
1 材料与方法 1.1 材料 1.1.1 菌株本研究使用菌株为具有絮凝性的工业酿酒酵母KF7-M16[19],异源表达了毕赤酵母的木糖还原酶(XR)基因XYL1和木糖醇脱氢酶(XDH)基因XYL2以及酿酒酵母的木酮糖激酶(XK)基因XKS1,能利用木糖产生乙醇。
1.1.2 培养基和抑制物表 1和表 2为混合抑制物(Mixed inhibitors,MI)和培养基组成。所有培养基的pH调节至5,在1×105 Pa、121℃灭菌15 min备用。抑制物过滤灭菌后添加至灭菌后的培养基。配制固体培养基时加入2%的琼脂粉。
将出发菌株KF7-M16在2%YPD平板上于30℃下活化24 h,用接种环取一环菌体,接种在5%YPD培养基内于30℃下160 r/min预培养16 h。将培养液稀释,取1 mL加入含有5 mL 0.05 mol/L EDTA液体的培养皿内(为了保证后续驯化初始活菌体数量,紫外诱变时准备多个平行样)。在不断搅拌液体的状态下,紫外灯照射培养皿150 s(细胞存活率约10%),收集液体离心去上清液,将处理后细胞用于驯化过程接种(SUV)。驯化使用的培养基为2.5%YPDX,驯化过程中混合抑制物浓度从40%MI开始逐步提升至100%MI。使用含100 mL培养液的300 mL三角瓶,在温度为30℃,培养液搅拌转速200 r/min,微好氧条件下进行驯化培养。每批传代培养初始接种浓度为OD660 0.5。在每一批的培养过程中定时取样分析细胞浓度(OD660)、葡萄糖、木糖和乙醇浓度。当木糖被大量消耗时进行下一次传代,一直至抑制物浓度达到100%MI。100%MI条件下第3次转接驯化后,一部分细胞用于转接继续驯化,另一部分细胞进行了第2次紫外诱变(DUV),诱变处理后的细胞继续进行了100%MI条件下的驯化。作为对照,同时进行了无紫外处理的驯化(N),驯化过程中的抑制物浓度从40%MI提升至100%MI。
1.2.2 突变菌株分离取100%MI条件下驯化获得的培养液,稀释后涂布于2.5%YPDX+80%MI平板上,于30℃下培养,挑取单菌落,并转移到新的2.5%YPDX+80%MI平板上进行菌落纯化。从无紫外处理驯化体系(N)、一次紫外处理驯化体系(SUV)和二次紫外处理驯化体系(DUV)分别挑取50株突变菌株,分别命名为N01-50,SUV01-50和DUV01-50,用于后续菌株筛选。
1.2.3 突变菌株生长筛选在30℃条件下,将各菌株于2%YPD平板上活化24 h,用牙签挑取少量细胞,接种于含3 mL 2.5%YPDX+80%MI培养基的15 mL试管中,于100 r/min,30℃下培养17 h,测定培养液的细胞浓度(OD660),比较各菌株的生长情况,对菌株进行初次生长筛选。将初次生长筛选中优秀的菌株进行二次生长筛选,方法同上,但培养基中的抑制物浓度为100%MI。
1.2.4 突变菌株木糖利用评价将菌株在2%YPD平板中活化24 h后,用接种环取一环菌体接种于含100 mL 5%YPD液体培养基的500 mL三角瓶中,30℃、160 r/min摇床预培养16 h。取10 mL预培养液离心收集细胞,接种到装有100 mL 2.5%YPDX+100%MI培养基的300 mL三角瓶中,置于30℃恒温水浴锅,200 r/min条件下限氧发酵144 h。取样分析细胞浓度(OD660)、葡萄糖和木糖浓度。
1.2.5 分析方法取5 mL培养液于4℃、12 000×g离心2 min,分别收集菌体和上清液。于所得菌体中加入50 mmol/L EDTA溶液分散细胞,稀释后用UV-VIS分光光度计(JASCO,日本)测定光密度值(OD660)。上清液经0.45 μm滤膜过滤后用于测定葡萄糖、木糖和乙醇浓度。利用高效液相色谱仪(LC-10AD VP,岛津,日本)分析葡萄糖和木糖浓度,检测器为RF-10AXL,炉温为150℃,柱温为65℃[24];利用气相色谱(GC353B,GL Sciences,日本)测定乙醇浓度,异丙醇为内标,检测器为FID,炉温为50℃,注射器和检测器温度均为180℃[24]。
2 结果 2.1 反复批次驯化培养过程中细胞生长、糖消耗及乙醇生成对紫外处理细胞(SUV,DUV)和无紫外处理细胞(N)共进行了16次批次驯化培养,其中40%MI条件下1批次,60%MI条件下4批次,80%MI条件下5批次,100%MI条件下6批次。第1和第2批次每批培养周期为4 d,第3到第16批次每批培养周期为5 d,每批次培养过程中细胞浓度、葡萄糖浓度、木糖浓度和乙醇浓度的变化如图 1所示。

|
| 图 1 驯化过程中细胞浓度(A、B),糖浓度(C)和乙醇浓度(D)的变化 N:无紫外诱变驯化体系;SUV:一次紫外诱变驯化体系;DUV:二次紫外诱变驯化体系 |
如图 1-A所示,驯化启动时(第1批次)的抑制物浓度为40%MI,无论细胞是否进行了紫外诱变,细胞生长都相对比较缓慢,有紫外处理的体系4 d后的细胞浓度(OD660 8.2) 略高于无紫外处理的(OD660 6.42)。从第2批次开始,提高抑制物浓度至60%MI,随转接次数增加细胞生长速率逐渐升高,第5批次时有和无紫外处理体系的细胞浓度分别为15.92和14.48,表明驯化过程有效地提升了细胞在抑制条件下的生长能力。当抑制物浓度提高到80%MI以后,5个批次的转接过程中细胞生长能力没有下降,能维持和60%MI条件下相近的生长能力,有和无紫外处理体系的细胞生长情况相近,没有明显差异。当抑制物浓度提高到100%MI时(第11批次),两体系的细胞生长显著被抑制,培养5 d后细胞浓度只有5左右,但转接后细胞生长迅速恢复,达到80%MI条件下相近水平。后续的几批驯化中,第13和第15批次有紫外处理体系的细胞生长要优于无紫外处理体系,而第14和第16批次中则没有明显差异。通过以上结果可以看出,通过驯化过程,在40%-100%MI抑制物浓度范围内,细胞生长可以保持相对稳定,5 d后的细胞浓度可以达到OD660 15-20。
图 1-B为各批次培养过程中葡萄糖浓度的变化。可以看出,启动的第1批次,两体系中葡萄糖需要2 d才被完全消耗,转接后在60%MI条件下,两体系的葡萄糖在1 d内可被完全消耗。当抑制物浓度提高至80%MI时,两体系的葡萄糖利用速率均下降,需要2 d才能被消耗,但在该抑制物浓度下第4次转接时(第10批次),紫外处理体系的葡萄糖则可在1 d内被完全消耗。当抑制物浓度提升至100%MI后,葡萄糖的消耗速率明显下降,第11批次中,两体系都需要4 d才能被完全消耗,主要可能是由于细胞生长被严重抑制。后续的几个批次中,尽管数据有波动,但可以看出紫外处理体系的葡萄糖利用速率要明显优于无紫外处理体系,第15和16批次中,紫外处理体系中的葡萄糖消耗只需1 d而无紫外处理体系需要3 d。以上结果可以看出,通过驯化过程,在40%-100%MI抑制物浓度范围内,有紫外处理的体系中的葡萄糖可以在1 d内被完全利用,但对于无紫外处理体系,随抑制物浓度升高,葡萄糖的利用速率随之下降,驯化的作用不显著。
图 1-C为各批次培养过程中木糖浓度的变化。可以看出,驯化启动的第1批次,无紫外处理和有紫外处理体系4 d后的木糖消耗率分别只有30%和43.5%,但转接后木糖消耗率逐步提升,60%MI条件下两体系的木糖消耗率能达到80%以上(第4批次)。当抑制物浓度提高至80%MI时,木糖利用略有下降,但通过驯化,两体系的木糖消耗率能达到80%以上。当抑制物浓度提高到100%MI时(第11批次),和葡萄糖的消耗一样,木糖消耗显著被抑制,5 d后的木糖消耗速率只有15%左右,但转接后,木糖利用迅速恢复。尽管后续各批次间有波动,但有紫外处理体系的木糖消耗率可以达到85%,而无紫外处理体系的木糖消耗率则明显低于有紫外处理体系,这与两体系葡萄糖消耗的差异一致。该结果表明,在100%MI抑制物浓度条件下,有紫外处理体系具有更好的糖利用能力,其抑制物耐受能力更强。
图 1-D为各批次培养过程中乙醇浓度的变化。从基于总糖(包括葡萄糖和木糖)消耗计算的乙醇收率可以看出,在40%到80%MI抑制物条件下,两体系的乙醇收率相近,约为0.24-0.26,但当抑制物浓度提高至100%MI后,无紫外处理体系的乙醇收率下降至0.22左右,表明对于无紫外处理体系,高的抑制物浓度不仅抑制了糖的消耗同时也影响乙醇收率,驯化的效果明显不如有紫外处理体系。
100%MI抑制物条件下二次紫外处理再驯化未能有效提升细胞生长和糖消耗,第16批次时的糖消耗情况和一次紫外处理体系接近(图 1)。
以上结果表明,在抑制物条件下进行驯化可以有效的提升细胞的抑制物耐受,提高细胞在高抑制强度下的生长和糖的利用,而驯化前对细胞进行紫外处理可以使细胞获得更好的抑制物耐受能力,显著提高细胞在高抑制条件下的糖利用能力。
2.2 突变菌株分离和高抑制条件下的生长筛选从第16批次3个驯化体系分离单菌株,从各体系随机挑选了50株菌株进行试管培养,在80%MI抑制条件下比较了它们的生长情况。无紫外处理(N)和有紫外处理体系(SUV,DUV)的各50株单菌的生长均好于出发菌株KF7-M16。无紫外处理体系的50株单菌的OD660在0.257-1.041之间(图 2-A),而有紫外处理体系的50株单菌的OD660在0.562-1.654之间(图 2-B),有紫外处理体系单菌株的生长情况总体上优于无紫外处理体系的单菌株。对于二次紫外处理体系的单菌株,OD660在0.076-1.175之间(图 2-C),和无紫外处理和一次紫外处理体系的单菌株相比,菌株间的差异比较大,少量菌株的生长明显比出发菌株弱,而少量菌株的生长明显强于一次紫外处理体系的单菌株,表明二次紫外处理对细胞产生了比较明显的影响。

|
| 图 2 抑制条件下各驯化体系分离菌株的生长评价 |
选择上述三体系中生长较好的菌株(无紫外体系OD660 > 0.6时的17株,一次紫外处理体系OD660 > 0.8时的33株,二次紫外处理体系OD660 > 0.6时的16株)共66株,在100%MI抑制条件下,进行了二次生长筛选,结果如图 3-D所示。66株单菌株的OD660在0.125-0.547之间,显著低于80%MI条件下的生长情况。与出发菌株生长相近的菌株,大部分是二次紫外处理体系的单菌株,而生长比较好的菌株中大部分是一次紫外处理体系的菌株。该结果表明,二次紫外处理可能对细胞造成了比较大的影响,导致部分分离菌株的生长能力下降。
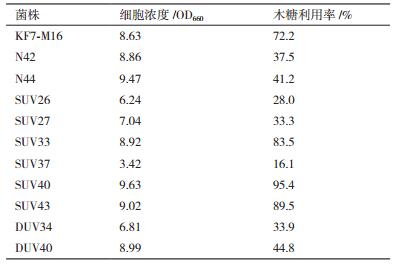
2.3 高抑制条件下突变菌株对木糖的利用对2.2中100%MI条件下前17 h生长最优秀的10株分离菌株(无紫外处理体系2株,一次紫外处理体系6株,二次紫外处理体系2株)进行了发酵实验,以出发菌株KF7-M16为对照,评价它们在100%MI抑制条件下对木糖的利用情况。发酵144 h后细胞浓度以及木糖利用率,如表 3所示。可以看出,只有分离自一次紫外处理体系的3株菌株SUV33,SUV40和SUV43的木糖利用率大于出发菌株,分别为81.4%、95.4%和89.5%,同时这3株菌株的细胞浓度也高于出发菌株。3株突变菌株的基于消耗糖的乙醇收率(0.23-0.24) 和出发菌株(0.22) 相比略有提升。从表 3还可以看出,高的细胞浓度并不一定就有高的木糖利用率,但低细胞浓度会导致低的木糖消耗。有4株菌株的细胞浓度明显低于出发菌株,表明2.2中使用短时间培养(17 h)进行菌株筛选可能会造成一些后期生长有优势的菌株丢失。
抑制物耐受性能优良的木糖发酵菌株是纤维素燃料乙醇生产所必需的。纤维素原料水解液中同时含多种抑制物,而这些抑制物如何影响酿酒酵母生长和发酵的机制尚不清楚,因此相比基因工程手段,驯化工程手段更适合用于抑制物耐受菌株的育种。本研究比较了直接驯化和紫外处理后,驯化对提升工业酿酒酵母菌株木糖发酵时的抑制物耐受能力的效果,结果表明紫外处理后驯化具有更好的效果,其中一个原因可能是紫外处理过程导致的突变使得细胞具有遗传多样性,在后续的驯化过程中获得目标突变菌株的几率得到提高。有研究也报道了在驯化前对细胞进行突变处理可以有效的提升驯化效果[25]。本研究使用的出发菌株本身就具有较好的抑制物耐受性,要在此基础上再进一步提高其抑制物耐受性相对比较困难;同时本研究考虑的抑制物种类多,它们同时存在时抑制效应严重。也导致提升抑制物耐受很困难,这些也是在本研究中紫外处理对提升抑制物耐受性具有明显贡献的原因。但从二次紫外处理驯化体系的结果可以看出,一次以上的紫外处理对抑制物耐受提升没有贡献,反而会导致生长和发酵能力下降,抑制物耐受能力降低。显然,重复的紫外处理导致基因组突变范围扩大,细胞的基础代谢受到影响,从而难以获得保持良好生长和发酵性能的抑制物耐受菌株。
本研究使用了含9种抑制物的抑制物混合物进行菌株的驯化,这9种抑制物包含了纤维素糖化液中典型的抑制物(弱酸类、呋喃醛),以及文献中报道的在糖化液中检测到的4种酚类等化合物,目的是希望获得的突变菌株能够具有更好的普适性。这9种抑制物的存在显著抑制了出发菌株的木糖消耗,发酵周期延长,木糖消耗缓慢。通过驯化,细胞生长和木糖利用能力得到了比较明显的提升。目前尚没有可在同时含这9种抑制物的条件下(如100%MI)能够进行木糖发酵的同类菌株的报道。目前有关提升木糖发酵菌株的抑制物耐受性的研究中所涉及的抑制物种类都比较单一,如乙酸[14]、甲酸[26]、呋喃醛[10]、丁香醛[27, 28]、香草醛[29]及松柏醛[30]。这些研究也表明,要使木糖发酵菌株具有能同时耐受多种抑制物的能力,难度非常大。优良的出发菌株、合理的研究设计和有效的技术手段是可能获得目标菌株的关键。
本研究最后分离筛选获得三株抑制物耐受高于出发菌株的突变菌株,但这些菌株的发酵性能和原驯化体系的发酵性能(图 1)相比并没有提高,且各突变菌株细胞浓度显著低于驯化体系。之所以在驯化后进行突变菌株的分离和筛选是基于以下考虑:原驯化体系中存在各种随机突变细胞,细胞间在抑制物耐受以及生长和发酵性能上会有很大差异,驯化体系的发酵结果是这些细胞群体表现,从中可以获得比群体表现更优秀的突变菌株。但突变菌株的分离和筛选并没有获得预期结果,可能有以下两个方面的原因:(1) 驯化体系中细胞可能具备不同的抗抑制机制,它们同时存在,在功能上相互补充,使细胞群体比单一突变菌株表现出更好的生长和发酵性能;(2) 突变菌株的筛选是基于培养初期的细胞生长,而初期生长最有优势的菌株反而发酵能力弱。因此,基于培养初期细胞生长情况进行突变菌株的筛选会丢失一些后期生长有优势,且发酵能力强的突变菌株。基于以上考虑,后续将进一步扩大突变菌株的筛选范围以期获得更多性能优良的突变菌株,然后利用细胞融合、基因组改组(Genome shuffling)等手段将各突变菌株的优良特性进行整合,构建出更优秀的抑制物耐受菌株,为纤维素燃料乙醇的工业化生产提供优良菌株来源。
4 结论本研究比较了9种抑制物同时共存胁迫条件下,直接驯化和紫外处理结合驯化的手段对提升木糖发酵工业菌株KF7-M16的抑制物耐受性的效果,结果表明紫外处理结合驯化比直接驯化能更有效的提升细胞对高浓度混合抑制物的耐受能力。通过突变菌株分离和筛选,获得3株抑制物耐受能力高于出发菌株的突变菌株,它们在抑制物浓度100%MI条件下的木糖利用率比出发菌株高11.3%-23.2%。
| [1] | Cardona CA, Quintero JA, Paz IC. Production of bioethanol from sugarcane bagasse:status and perspectives[J]. Bioresource Technology, 2010, 101 (13): 4754–4766. DOI:10.1016/j.biortech.2009.10.097 |
| [2] | Chu BCH, Lee H. Genetic improvement of Saccharomyces cerevisiae for xylose fermentation[J]. Biotechnology Advances, 2007, 25 (5): 425–441. DOI:10.1016/j.biotechadv.2007.04.001 |
| [3] | Wahlbom CF, van Zyl WH, Jönsson LJ, et al. Generation of the improved recombinant xylose-utilizing Saccharomyces cerevisiae TMB3400 by random mutagenesis and physiological comparison with Pichia stipitis CBS6054[J]. FEMS Yeast Research, 2003, 3 (3): 319–326. DOI:10.1016/S1567-1356(02)00206-4 |
| [4] | Brat D, Boles E, Wiedemann B. Functional expression of a bacterial xylose isomerase in Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2009, 75 (8): 2304–2311. DOI:10.1128/AEM.02522-08 |
| [5] | Zhou H, Cheng J, Wang BL, et al. Xylose isomerase overexpression along with engineering of the pentose phosphate pathway and evolutionary engineering enable rapid xylose utilization and ethanol production by Saccharomyces cerevisiae[J]. Metabolic Engineering, 2012, 14 (6): 611–622. DOI:10.1016/j.ymben.2012.07.011 |
| [6] | Palmqvist E, Hahn-Hägerdal B. Fermentation of lignocellulosic hydrolysates. Ⅱ:inhibitors and mechanisms of inhibition[J]. Bioresource Technology, 2000, 74 (1): 25–33. DOI:10.1016/S0960-8524(99)00161-3 |
| [7] | Palmqvist E, Hahn-Hägerdal B. Fermentation of lignocellulosic hydrolysates. Ⅰ:inhibition and detoxification[J]. Bioresource Technology, 2000, 74 (1): 17–24. DOI:10.1016/S0960-8524(99)00160-1 |
| [8] | Moysés DN, Reis VCB, Almeida JRM, et al. Xylose fermentation by Saccharomyces cerevisiae:challenges and prospects[J]. International Journal of Molecular Sciences, 2016, 17 (3): 207. DOI:10.3390/ijms17030207 |
| [9] | Demeke MM, Dumortier F, Li Y, et al. Combining inhibitor tolerance and D-xylose fermentation in industrial Saccharomyces cerevisiae for efficient lignocellulose-based bioethanol production[J]. Biotechnology for Biofuels, 2013, 6 (1): 120. DOI:10.1186/1754-6834-6-120 |
| [10] | Ask M, Bettiga M, Duraiswamy VR, et al. Pulsed addition of HMF and furfural to batch-grown xylose-utilizing Saccharomyces cerevisiae results in different physiological responses in glucose and xylose consumption phase[J]. Biotechnology for Biofuels, 2013, 6 (1): 181. DOI:10.1186/1754-6834-6-181 |
| [11] | Nevoigt E. Progress in metabolic engineering of Saccharomyces cerevisiae[J]. Microbiology and Molecular Biology Reviews, 2008, 72 (3): 379–412. DOI:10.1128/MMBR.00025-07 |
| [12] | Novy V, Krahulec S, Wegleiter M, et al. Process intensification through microbial strain evolution:mixed glucose-xylose fermentation in wheat straw hydrolyzates by three generations of recombinant Saccharomyces cerevisiae[J]. Biotechnology for Biofuels, 2014, 7 (1): 49. DOI:10.1186/1754-6834-7-49 |
| [13] | Casey E, Sedlak M, Ho NWY, et al. Effect of acetic acid and pH on the cofermentation of glucose and xylose to ethanol by a genetically engineered strain of Saccharomyces cerevisiae[J]. FEMS Yeast Research, 2010, 10 (4): 385–393. DOI:10.1111/fyr.2010.10.issue-4 |
| [14] | Sànchezi Nogué V, Narayanan V, Gorwa-Grauslund MF. Short-term adaptation improves the fermentation performance of Saccharomyces cerevisiae in the presence of acetic acid at low pH[J]. Applied Microbiology and Biotechnology, 2013, 97 (16): 7517–7525. DOI:10.1007/s00253-013-5093-5 |
| [15] | Peng LU, Chen L, Li G, et al. Influence of furfural concentration on growth and ethanol yield of Saccharomyces kluyveri[J]. Journal of Environmental Sciences, 2007, 19 (12): 1528–1532. DOI:10.1016/S1001-0742(07)60249-6 |
| [16] | Oliva JM, Negro MJ, Saez F, et al. Effects of acetic acid, furfural and catechol combinations on ethanol fermentation of Kluyveromyces marxianus[J]. Process Biochemistry, 2006, 41 (5): 1223–1228. DOI:10.1016/j.procbio.2005.12.003 |
| [17] | Ballesteros M, Oliva JM, Negro MJ, et al. Ethanol from lignocellulosic materials by a simultaneous saccharification and fermentation process(SFS)with Kluyveromyces marxianus CECT10875[J]. Process Biochemistry, 2004, 39 (12): 1843–1848. DOI:10.1016/j.procbio.2003.09.011 |
| [18] | Li YC, Mitsumasu K, Gou ZX, et al. Xylose fermentation efficiency and inhibitor tolerance of the recombinant industrial Saccharomyces cerevisiae strain NAPX37[J]. Applied Microbiology and Biotechnology, 2016, 100 (3): 1531–1542. DOI:10.1007/s00253-015-7167-z |
| [19] | Zeng WY, Tang YQ, Gou M, et al. Transcriptomes of a xylose-utilizing industrial flocculating Saccharomyces cerevisiae strain cultured in media containing different sugar sources[J]. AMB Express, 2016, 6 (1): 51. DOI:10.1186/s13568-016-0223-y |
| [20] | Zeng WY, Tang YQ, Gou M, et al. Comparative transcriptomes reveal novel evolutionary strategies adopted by Saccharomyces cerevisiae with improved xylose utilization capability[J]. Applied Microbiology and Biotechnology, 2017, 101 : 1753–1767. DOI:10.1007/s00253-016-8046-y |
| [21] | Landaeta R, Aroca G, Acevedo F, et al. Adaptation of a flocculent Saccharomyces cerevisiae strain to lignocellulosic inhibitors by cell recycle batch fermentation[J]. Applied Energy, 2013, 102 : 124–130. DOI:10.1016/j.apenergy.2012.06.048 |
| [22] | Koppram R, Albers E, Olsson L. Evolutionary engineering strategies to enhance tolerance of xylose utilizing recombinant yeast to inhibitors derived from spruce biomass[J]. Biotechnology for Biofuels, 2012, 5 (1): 32. DOI:10.1186/1754-6834-5-32 |
| [23] | 苟梓希, 李云成, 谢采芸, 等. 工业酿酒酵母菌株KF-7对发酵抑制物的耐受性[J]. 应用与环境生物学报, 2015, 21(2): 248–255. |
| [24] | Tang YQ, Koike Y, Liu K, et al. Ethanol production from kitchen waste using the flocculating yeast Saccharomyces cerevisiae strain KF-7[J]. Biomass and Bioenergy, 2008, 32 (11): 1037–1045. DOI:10.1016/j.biombioe.2008.01.027 |
| [25] | 郭雪娇, 查健, 姚坤, 等. 选育耐受复合抑制剂酿酒酵母提高乙醇产量[J]. 中国生物工程杂志, 2016, 26(5): 97–105. |
| [26] | Sanda T, Hasunuma T, Matsuda F, et al. Repeated-batch fermentation of lignocellulosic hydrolysate to ethanol using a hybrid Saccharomyces cerevisiae strain metabolically engineered for tolerance to acetic and formic acids[J]. Bioresource Technology, 2011, 102 (17): 7917–7924. DOI:10.1016/j.biortech.2011.06.028 |
| [27] | Zaldivar J, Martinez A, Ingram LO. Effect of selected aldehydes on the growth and fermentation of ethanologenic Escherichia coli[J]. Biotechnology and Bioengineering, 1999, 65 (1): 24–33. DOI:10.1002/(ISSN)1097-0290 |
| [28] | 林贝, 赵心清, 葛旭萌, 等. 玉米秸秆酸解副产物对重组酿酒酵母6508-127发酵的影响[J]. 中国生物工程杂志, 2007, 27(7): 61–67. |
| [29] | Endo A, Nakamura T, Ando A, et al. Genome-wide screening of the genes required for tolerance to vanillin, which is a potential inhibitor of bioethanol fermentation, in Saccharomyces cerevisiae[J]. Biotechnology for Biofuels, 2008, 1 (1): 3. DOI:10.1186/1754-6834-1-3 |
| [30] | Yi X, Gu H, Gao Q, et al. Transcriptome analysis of Zymomonas mobilis ZM4 reveals mechanisms of tolerance and detoxification of phenolic aldehyde inhibitors from lignocellulose pretreatment[J]. Biotechnology for Biofuels, 2015, 8 (1): 153. DOI:10.1186/s13068-015-0333-9 |