2. 中国科学院天津工业生物技术研究所,天津 300308
2. Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308
3-(4-羟基苯基)丙酸,英文名称3-(4-Hydr-oxyphenyl)propionic acid(HPPA),是药物(如盐酸西曲酸酯、盐酸艾司洛尔)[1, 2],天然产物(如杨梅醇、根皮素)的重要中间体[3, 4],也是一些生物降解材料的基本骨架[5],存在于铁皮石斛、小叶榕等植物中[6, 7]。目前,HPPA主要通过化学方法合成,如以苯酚为起始原料,在催化剂三氯化铝的作用下,与丙烯腈反应生成3-(4-)羟基苯丙腈,再通过水解得到化合物HPPA[8]。然而,化学合成方法产率低,成本高,反应条件复杂且难于控制,容易造成环境污染。利用微生物合成HPPA将会有效的避免化学合成中复杂条件的控制以及环境污染等问题[9]。之前已经有报道在大肠杆菌中合成HPPA[10]。然而在酵母中合成HPPA尚未见报道。酵母菌作为公认的食品安全菌株,具有遗传背景清晰,遗传操作技术简单,生长迅速,培养成本低,易于大规模化液体发酵生产等优势,被广泛地应用于生物合成研究中[11]。
对香豆酸(p-coumaric acid),是苯丙氨酸次级代谢途径中的重要中间体。在植物中,主要通过苯丙氨酸氨基转移酶(PAL)、肉桂酸羟化酶(C4H)催化苯丙氨酸生成对香豆酸[12]。在微生物大肠杆菌、酿酒酵母中,主要通过异源表达酪氨酸氨基转移酶(TAL),催化酪氨酸转化生成对香豆酸[13, 14]。本研究以酿酒酵母BY4742为出发菌株,引入约氏黄杆菌来源的酪氨酸转移酶(FjTAL)[15]、拟南芥来源的对香豆酰辅酶A连接酶(At4CL)[13]以及酿酒酵母自身来源的超长链烯酰辅酶A还原酶(ScTSC-13)[16, 17]、硫酯水解酶(Acyl-CoA thioesterase),以实现HPPA在酿酒酵母中的从头合成,为生物合成高价值的HPPA衍生物奠定基础。本研究所构建的合成途径如图 1所示。
|
| 图 1 3-(4-羟基苯基)丙酸的生物合成途径 p-Coumaroyl-CoA:对香豆酰辅酶A;Dihydro-p-coumroyl-CoA:3-(4-羟基苯基)丙酰辅酶A;HPPA:3-(4-羟基苯基)丙酸 |
大肠杆菌DH5α和酿酒酵母BY4742均由本实验室保存,DH5α用于质粒扩增和基因克隆,酿酒酵母BY4742用于蛋白的过表达及HPPA的合成。pESC-LEU,pESC-URA是酵母表达载体,分别具有2μ复制起始点,半乳糖诱导的GAL1/10双向诱导型启动子,购于Novagen公司。
1.1.2 试剂及培养基Phusion超保真DNA聚合酶购自New England Biolabs(NEB)公司;限制性内切酶Sal Ⅰ、Hind Ⅲ、BamH1、Xho Ⅰ以及T4连接酶均购于Fermentas公司;对香豆酸、咖啡酸、3-(4-羟基苯基)丙酸(HPPA)、3,4-二羟基苯丙酸(DHPPA)的标准品购于南京广润生物化学有限公司。
YPD培养基:1%酵母膏(Yeast Extract),2%蛋白胨(Peptone),2%葡萄糖(Glucose),若制固体培养基,加入2%琼脂粉(Agarpower);SC-1培养基:0.67%无氨基酵母氮源(YNB,Yeast Nitrogen Base),0.2% Dropout mix,2%葡萄糖(Glucose);SC-2培养基:0.67%无氨基酵母氮源(YNB,Yeast Nitrogen Base),0.2% Dropout mix,2%半乳糖(Galactose)。
1.2 方法 1.2.1 基因扩增At4CL(GenBank:KX817185.1) 来自植物拟南芥(Arabidopsis thaliana),以拟南芥全长cDNA为模板,PCR引物为At4CL-5FP-BamHI:CGCGGATCCATGGCGCCACAAGCAGTT(下划线为BamHI酶切位点),At4CL-3RP-xhoⅠ:CCGCTCGAGTCACAATCCATTTGCTAG(下划线为XhoⅠ酶切位点),PCR扩增At4CL基因片段被克隆到载体pESC-URA质粒上,得到质粒pESC-URA-At4CL。PCR参数:预变性95℃ 3 min;95℃ 30 s,58℃ 30 s,72℃ 1.5 min共30个循环;最后延伸72℃ 10 min;凝胶电泳回收目的片段。
1.2.2 基因扩增FjTAL(Genpet:WP_012023194.1) 来自约氏黄杆菌(Flavobacterium johnsoniae),由上海捷瑞公司合成,为了在酿酒酵母使用对FjTAL基因进行了密码子优化,命名为FjTALsyn来表示。合成的基因片段被克隆到pUC19载体上,由捷瑞公司构建成载体pUC19-FjTALsyn。用此质粒作PCR模板;PCR引物为FjTALsyn-5FP-SalⅠ:GCGGTCGACATGAA-TACTATTAATGAA(下划线为SalⅠ酶切位点),FjT-ALsyn-3RP-HindⅢ:CCCAAGCTTTTAATTATTAATC-AAATG(下划线为HindⅢ酶切位点)。PCR程序:预变性95℃ 3 min;95℃ 30 s,58℃ 30 s,72℃ 1.5 min共30个循环;最后延伸72℃ 10 min;凝胶电泳回收目的片段。
1.2.3 质粒及重组菌构建以限制性内切酶BamH1和XhoⅠ对质粒pESC-URA和At4CL分别进行酶切;通过凝胶电泳回收载体及基因片段,以载体与基因片段摩尔数比为1:3进行连接,转化大肠杆菌DH5α,获得质粒pESC-URA-At4CL。以限制性内切酶SalⅠ和Hind Ⅲ对质粒pESC-LEU和FjTALsyn分别进行酶切;凝胶电泳回收载体及基因片段后再进行连接,转化大肠杆菌DH5α,获得质粒pESCLEU-FjTALsyn。利用PEG/LiAc法转化酵母[18],分别转化质粒pESC-LEU,pESCLEU-FjTALsyn,pESC-URA,pESC-URA-At4CL,pESC-LEU & pESC-URA,pESCLEU-FjTALsyn & pESC-URA-At4CL至酿酒酵母菌BY4742(BL100),获得重组菌BL101,BL102,BL103,BL104,BL105,BL106。
1.2.4 重组菌发酵培养挑取重组菌株单克隆于3 mL的液体SC-1培养基中(含葡萄糖2%),30℃培养16 h;按体积比1:100转接至50 mL的液体SC-1培养基中,30℃培养48 h;3 000 r/min离心5 min,收集菌体,加入50 mL SC-2发酵培养基(含半乳糖2%)进行重悬,于30℃继续培养72 h并在此培养过程中进行取样分析。
1.2.5 对香豆酸及其衍生物的LC-MS分析鉴定收集SC-2发酵液1 mL,12000 r/min,离心10 min取上清进行检测。LC-MS检测系统配有紫外检测器的安捷伦1260系统和配有ESI例子源探针的bruker microQ-TOFⅡ质谱仪。液相色谱柱为Agela Innoval MP C18柱(4.6×250 mm);UV检测波长为201 nm;流动相A= H2O(含0.1%甲酸),B=100 %甲醇;流速= 1 mL/min,检测条件为:0-5 min 5% B,40 min 5% B到100 % B(线性梯度);进样量20 μL;ESI负离子源,分子量扫描范围50-800。
2 结果 2.1 酪氨酸氨基转移酶FjTAL在重组菌BL102中的生物转化活性研究对重组菌BL102进行液体发酵培养,添加4 mmol/L外源酪氨酸,取72 h的发酵液进行HPLC检测。结果(图 2-A)显示,在29.3 min处有新化合物的产生,与对香豆酸标准品的峰保留时间(Rt)一致。对香豆酸的分子量为164,LC-MS检测结果(图 2-B)显示29.3 min峰的[M+H]+ = 165,确定该峰对应的化合物为对香豆酸(1)。在发酵24 h、48 h、72 h进行取样,测定产物对香豆酸生成量。结果(图 2-C)显示随着发酵时间的延长,对香豆酸产量逐步增加,72 h时产量达到237.5±3.3 mg/L,酪氨酸转化率达到36.2±0.5%。

|
| 图 2 酵母菌BL101,BL102发酵液HPLC检测及LC-MS结果 A(a:对照BL101发酵72 h发酵液HPLC图谱;b:酵母菌BL102发酵72 h时发酵液HPLC图谱);B:酵母菌BL101生成的对香豆酸的LC-MS图谱;C:发酵时间对BL102菌株的对香豆酸产量、酪氨酸转化率的影响 |
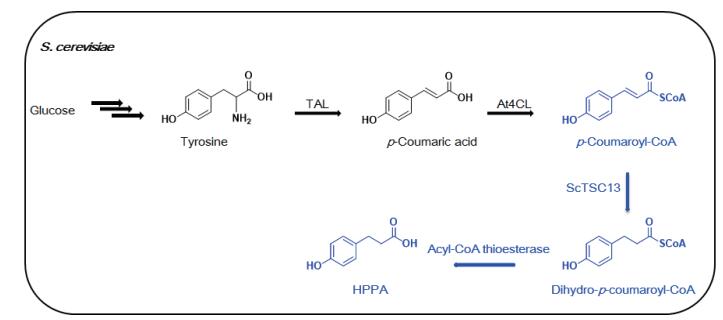
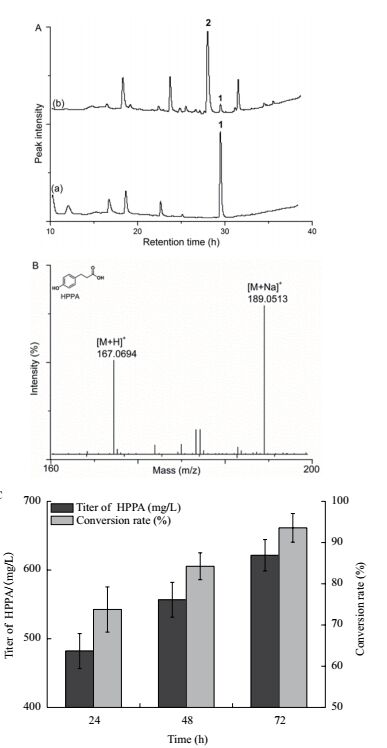
对重组菌BL104进行液体发酵培养,添加4 mmol/L外源对香豆酸,取72 h的发酵液进行HPLC检测。结果(图 3-A)显示,在29.3 min处的对香豆酸(1) 基本全部被转化,而在27.5 min处有新化合物的产生,与HPPA的标准品的峰保留时间(Rt)一致。HPPA的分子量为166,LC-MS检测结果(图 3-B)显示27.5 min峰的[M+H]+ = 167,确定该峰对应的化合物为HPPA(2)。在发酵24 h、48 h、72 h进行取样,测定产物HPPA生成量。结果(图 3-C)显示随着发酵时间的延长,HPPA产量逐步增加,72 h时产量达到624.8±7.9 mg/L,对香豆酸转化率达到94.1±1.2%。以同样的方法培养菌株BL104,添加4 mmol/L咖啡酸(3),取72 h的发酵液进行HPLC检测。结果(图 4)显示,72 h时DHPPA产量达到665.4±8.7 mg/L,咖啡酸转化率达到91.4±1.2%。
|
| 图 3 酵母菌BL103,BL104发酵液(添加4 mmol/L对香豆酸)HPLC检测及LC-MS结果 A(a:对照BL103发酵72 h时发酵液HPLC图谱;b:酵母菌BL104发酵72 h时发酵液HPLC图谱);B:酵母菌BL104生成的HPPA的LC-MS图谱;C:发酵时间对BL104菌株的HPPA产量、对香豆酸转化率的影响 |
|
| 图 4 酵母菌BL103,BL104发酵液HPLC检测及LC-MS结果 A(a:对照BL103发酵72 h时发酵液HPLC图谱;b:酵母菌BL104发酵72 h时发酵液HPLC图谱);B:酵母菌BL104产生的DHPPA的LC-MS图谱;C:发酵时间对BL104菌株的的DHPPA的产量、咖啡酸转化率的影响 |
对重组菌BL106进行发酵培养,取72 h发酵液进行HPLC检测。结果(图 5-A)显示,在29.3 min、27.5 min处有两个新峰,分别与对香豆酸(1)、HPPA(2) 的标准品保留时间(Rt)一致,LC-MS检测结果显示29.3 min峰的[M+H]+ = 165,27.5 min峰的[M+H]+ = 167,确定两个峰对应的化合物分别为对香豆酸(1)、HPPA(2)。在发酵12、24、36、48、60和72 h进行取样,测定产物HPPA生成量。结果(图 5-B)显示,随着发酵时间的延长,HPPA产量逐步增加,72 h时产量达到70.1±0.2 mg/L。按照以上条件发酵培养BL106菌株,同时添加4 mmol/L酪氨酸前体,在发酵12、24、36、48、60和72 h进行取样,测定产物HPPA生成量。结果(图 5-C)显示,HPPA产量随发酵时间的延长而逐渐增加,72 h后产量达到155.3±2.3 mg/L。

|
| 图 5 酵母菌BL106发酵液HPLC检测与分析 A(a:对照BL105发酵72 h时发酵液HPLC图谱;b:酵母菌BL106发酵72 h时发酵液HPLC图谱;c:酵母菌BL106发酵72 h(添加4 mmol/L外源酪氨酸)后发酵液HPLC);B:发酵时间对菌株BL106的HPPA产量的影响;C:发酵时间对菌株BL106(添加4 mmol/L外源酪氨酸)的HPPA产量的影响 |
本研究在酿酒酵母BY4742中表达了来自拟南芥的对香豆酸辅酶A连接酶(At4CL)基因,约氏黄杆菌的酪氨酸转氨酶(FjTAL)基因,同时利用酿酒酵母内源酶,首次实现了在酿酒酵母中从头合成HPPA,产量达到70 mg/L。在酿酒酵母中,HPPA的合成路径及其相关的酶尚未鉴定清楚[19],推测HPPA是由对香豆酸转化生成的。
近年来,Michael Eichenberger等[16, 17]在酿酒酵母中合成二氢查耳酮类化合物的研究中,利用酵母内源ScTSC13,植物来源的二氢查耳酮合酶(CHS)合成根皮素。其中,ScTSC13是酵母细胞膜依赖的脂肪酸链延长系统中催化反式-2-稀酰-辅酶A双键还原的酶,对香豆酰辅酶A含有a,β-稀酰-辅酶A基团,由此推测ScTSC13催化对香豆酰辅酶A生成3-(4-羟基苯基)丙酰辅酶A。以对香豆酸为前体,在酿酒酵母中异源表达4CL,CHS合酶基因,实现了根皮素的生物合成,并初步确定了ScTSC13对对香豆酰辅酶A的催化活性。
本研究在酿酒酵母中异源表达At4CL的条件下,添加4 mmol/L外源对香豆酸,发酵结果显示,对香豆酸几乎全部转化成产物HPPA。推测其合成路径是:(1) 对香豆酸在At4CL的作用下生成对香豆酰辅酶A;(2) 对香豆酰辅酶A利用ScTSC13还原生成3-(4-羟基苯基)丙酰辅酶A;(3)3-(4-羟基苯基)丙酰辅酶A在内源硫酯酶的作用下水解生成HPPA与游离的辅酶A。除了对香豆酸之外,咖啡酸在At4CL存在的条件下,也能被酵母菌转化生成DHPPA,转化效率达到90%以上,为后续研究ScTSC13的底物宽泛性提供了良好的基础。目前,途径中的内源硫酯酶还尚未鉴定。在酵母中存在多种酰基辅酶A硫酯酶,包括脂肪酸代谢途径中已鉴定的YJR019C[20]、Pte1p[21],均有可能水解3-(4-羟基苯基)丙酰辅酶A生成HPPA与游离的辅酶A,有待进一步的探索与研究。
4 结论本研究在酿酒酵母中表达了约氏黄杆菌及拟南芥来源的两个外源基因,同时利用酵母内源超长链烯酰辅酶A还原酶、硫酯水解酶,首次实现了在酿酒酵母中从头合成HPPA。通过前体添加实验,对HPPA生物合成途径中关键酶的生物转化效率进行了分析,HPPA产量得到显著提高。
| [1] | Araki H, Kawabata A, Kuroda R, et al. Compositions for preventing and treating digestive organs diseases:US, US7550437[P]. 2009. |
| [2] | Tang YH, Wang JY, Hu HH, et al. Analysis of species-dependent hydrolysis and protein binding of esmolol enantiomers[J]. Journal of Pharmaceutical Analysis, 2012, 2 (3): 220–225. DOI:10.1016/j.jpha.2012.01.007 |
| [3] | Kawai S, Nakata K, Ichizawa H, et al. 3-(4-Hydroxyphenyl)propionic acid is involved in the biosynthesis of myricanol in Myrica rubra[J]. Journal of Wood Science, 2012, 56 (2): 148–153. |
| [4] | Yahyaa M, Davidovich-Rikanati R, Eyal Y, et al. Identification and characterization of UDP-glucose:Phloretin 4' -O-glycosyltransferase from Malus x domestica Borkh[J]. Phytochemistry, 2016, 130 : 47–55. DOI:10.1016/j.phytochem.2016.06.004 |
| [5] | Croitoru R, Fiţigău F, Broek LAMVD, et al. Biocatalytic acylation of sugar alcohols by 3-(4-hydroxyphenyl)propionic acid[J]. Process Biochemistry, 2012, 47 (12): 1894–1902. DOI:10.1016/j.procbio.2012.06.015 |
| [6] | 管惠娟, 张雪, 屠凤娟, 等. 铁皮石斛化学成分的研究[J]. 中草药, 2009, 40(12): 1873–1876. |
| [7] | 黄洋, 邵慧凯, 李康, 等. 小叶榕叶抗炎成分分析及活性评价[J]. 中成药, 2014, 36(6): 1227–1233. |
| [8] | 张明星, 盛喆. 对羟基苯丙酸的制备与应用[J]. 精细与专用化学品, 2010, 18(9): 48–49. |
| [9] | 李晓林, 周威, 庄以彬, 等. 3, 4-二羟基扁桃酸在大肠杆菌中的生物合成[J]. 生物技术通报, 2017, 33(1): 135–140. |
| [10] | Jing S, Lin Y, Shen X, et al. Aerobic biosynthesis of hydrocinnamic acids in Escherichia coli, with a strictly oxygen-sensitive enoate reductase[J]. Metabolic Engineering, 2016, 35 : 75–82. DOI:10.1016/j.ymben.2016.02.002 |
| [11] | Hong KK, Nielsen J. Metabolic engineering of Saccharomyces cerevisiae:a key cell factory platform for future biorefneries[J]. Cellular & Molecular Life Sciences Cmls, 2012, 69 (16): 2671–2690. |
| [12] | Gosch C, Halbwirth H, Stich K. Phloridzin:biosynthesis, distribution and physiological relevance in plants[J]. Phytochemistry, 2010, 71 (8-9): 838–843. DOI:10.1016/j.phytochem.2010.03.003 |
| [13] | Jiang J, Bi H, Zhuang Y, et al. Engineered synthesis of rosmarinic acid in Escherichia coli, resulting production of a new intermediate, caffeoyl-phenyllactate[J]. Biotechnology Letters, 2016, 38 (1): 81–88. DOI:10.1007/s10529-015-1945-7 |
| [14] | Jendresen CB, Stahlhut SG, Li M, et al. Novel highly active and specific tyrosine ammonia-lyases from diverse origins enable enhanced production of aromatic compounds in bacteria and yeast[J]. Applied & Environmental Microbiology, 2015, 81 (13): 4458–76. |
| [15] | Rodriguez A, Kildegaard KR, Li M, et al. Establishment of a yeast platform strain for production of p-coumaric acid through metabolic engineering of aromatic amino acid biosynthesis[J]. Metabolic Engineering, 2015, 31 : 181. DOI:10.1016/j.ymben.2015.08.003 |
| [16] | Sepp D. Kohlwein, Sandra E, et al. Tsc13p is required for fatty acid elongation and localizes to a novel structure at the nuclear-vacuolar interface in Saccharomyces cerevisiae[J]. 2001, 21(1):109-125. |
| [17] | Eichenberger M, Lehka BJ, Folly C, et al. Metabolic engineering of Saccharomyces cerevisiae for de novo production of dihydrochalc-ones with known antioxidant, antidiabetic, and sweet tasting prope-rties[J]. Metabolic Engineering, 2016 . |
| [18] | Gietz RD, Woods RA. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method[J]. Methods in enzymology, 2002, 350 (350): 87–96. |
| [19] | Beekwilder J, Wolswinkel R, Jonker H, et al. Production of resveratrol in recombinant microorganisms[J]. Applied & Environmental Microbiology, 2006, 72 (8): 5670–5672. |
| [20] | Kal AJ, Hettema EH, Van dBM, et al. In silicio search for genes encoding peroxisomal proteins in Saccharomyces cerevisiae[J]. Cell biochemistry and biophysics, 2000, 32 (1): 1–8. |
| [21] | Maeda I, Delessert S, Hasegawa S, et al. The peroxisomal Acyl-CoA thioesterase Pte1p from Saccharomyces cerevisiae is required for efficient degradation of short straight chain and branched chain fatty acids[J]. Journal of Biological Chemistry, 2006, 281 (17): 11729–11735. DOI:10.1074/jbc.M511762200 |




