2. 广东省微生物研究所 省部共建华南应用微生物国家重点实验室 广东省菌种保藏与应用重点实验室 广东省微生物应用新技术公共实验室, 广州 510070;
3. 中国科学院大学, 北京 100049;
4. 广东省科技图书馆, 广州 510070
2. State Key Laboratory of Applied Microbiology Southern China, Guangdong Provincial Key Laboratory of Microbial Culture Collection and Application, Guangdong Open Laboratory of Applied Microbiology, Guangdong Institute of Microbiology, Guangzhou 510070;
3. University of Chinese Academy of Sciences, Beijing 100049;
4. Guangdong Science and Technology Library, Guangzhou 510070
多重耐药菌的广泛传播以及超级耐药细菌的出现, 使“无药可用”的隐忧从学界蔓延到普通大众。世界经济论坛就在《全球风险报告2013》中发出预警:“由于细菌的耐药, 在不久的将来抗生素可能不再唾手可得, 我们有回到前抗生素时代的危险, 轻微的擦伤都可能致命”。天然产物在现代药物发现和发展过程中扮演着极其重要的角色[1], 是药物创新的重要途径和来源[2]。放线菌是重要的活性天然产物产生菌, 但在历经数十年的挖掘之后, 高频率的重复发现严重影响了活性天然产物的筛选效率。利用传统方法从放线菌等药源微生物中筛选新的活性天然产物, 特别是作用机制新颖的活性天然产物变得越来越困难, 国际制药公司于20世纪末纷纷放弃传统天然产物的筛选工作[3]。而随着天蓝色链霉菌(Streptomyces coelicolor)A3(2)[4]等代表性菌株基因组测序的完成, 人们发现链霉菌基因组中存在众多的次级代谢产物生物合成基因簇, 潜在的次级代谢产物远远多于目前已发现的数量, 这意味着放线菌中仍有许多新的活性次级代谢产物有待被发现, 为人们进一步从中挖掘新的活性天然产物提供了理论依据。在常规实验室培养条件下, 不表达或表达活性极低的生物合成基因簇被称为隐性或沉默生物合成基因簇。新一代测序技术的出现使基因组测序在技术、费用、时间等方面的门槛大大降低[5], 大量隐性生物合成基因簇的累积, 为天然产物的挖掘开启了第二次黄金时代[6]。
目前激活隐性生物合成基因簇的主要方法包括“一菌多素”(One strain-many compounds, OSMAC)[7]、共培养[8]、核糖体工程[9]、生物信息学预测以及基于遗传修饰的异源表达、启动子改造等, 国际国内已有多篇综述总结和梳理了相关方法[10-12]。尽管方法多样, 但总体上, 如何高效激活这些大量的隐性生物合成基因簇仍是当前面临的重要挑战。另一方面, 放线菌特别是链霉菌在常规实验室培养条件下产生的一至数种主要次级代谢产物容易掩盖潜在的新产物, 在一定程度上阻碍了潜在新活性产物的检出。利用适当的策略, 避免或减少主要次级代谢产物的干扰, 同时对现有激活方法进行组合, 可能能够提高新活性次级代谢产物的挖掘效率。例如, 天蓝色链霉菌所产天蓝霉素(Coelimycin)P1, 就是通过失活放线紫红素(Actinorhodin)、十一烷基灵菌红素(Metacycloprodigiosin)和CDA(Calcium-Dependent Antibiotic)等主要产物的基因簇后, 对突变株进一步实施核糖体工程后获得高产突变株, 从而鉴定其结构的[13]。本文对该策略进行了系统地阐述, 并称之为“移树养苗”(Tree-removal)策略。
粤蓝链霉菌(Streptomyces vietnamensis)是本课题组分离得到的一株链霉菌新种[14]。前期研究表明, 其主导次级代谢产物是榴菌素(Granaticin)[15]。全基因组测序分析发现[16], 除榴菌素基因簇外, 还存在至少28个潜在的次级代谢产物生物合成基因簇, 其中包括与Kinamycin生物合成基因簇等存在较高相似性的基因簇, 值得进一步挖掘。由于主导产物榴菌素存在抗菌和抗肿瘤活性, 在活性筛选时对其它可能的活性代谢产物的检出造成干扰。本研究利用“移树养苗”策略挖掘粤蓝链霉菌隐性活性次级代谢产物的初步结果。
1 材料与方法 1.1 材料 1.1.1 菌株来源粤蓝链霉菌DMR1(Δgra-orf1-3::aac(3)-IV)为本实验室前期构建的突变株, 不能产生该菌主要次级代谢产物榴菌素(Granaticin)[17]。抗菌实验标准菌株大肠杆菌(Escherichia coli)ATCC8739、枯草芽孢杆菌(Bacillus subtilis)ATCC6633、金黄色葡萄球菌(Staphylococcus aureus)ATCC6538、白色念珠菌(Candida albicans)ATCC10231和耐药金黄色葡萄球菌ATCC43300(耐甲氧西林、苯唑西林)均来源于广东省微生物菌种保藏中心。其它耐药菌:大肠杆菌460(耐环丙沙星、左氧氟沙星、氨苄西林、头孢他啶、头孢噻肟、头孢西丁、四环素、氯霉素、卡那霉素、庆大霉素、链霉素等11种抗生素)、大肠杆菌480(耐药性同大肠杆菌460)、金黄色葡萄球菌206(耐环丙沙星、氨苄西林、头孢他啶、头孢噻肟、四环素、氯霉素、头孢曲松、青霉素、苯唑西林、红霉素、替卡西林、磺胺甲恶唑、阿米卡星等13种抗生素)、沙门氏菌(Salmonella sp.)31(耐恩诺沙星、萘啶酮酸、氯霉素、氟苯尼考、乙酰甲喹等5种抗生素)和沙门氏菌47(耐药谱同沙门氏菌31), 由华南农业大学蒋红霞教授惠赠。
1.1.2 培养基(1) YEME培养基:酵母提取物3 g、蛋白胨5 g、麦芽提取物3 g、葡萄糖10 g, 补水至1 L, pH7.2, 灭菌后加入2.5 mol/L MgCl2 2 mL;(2) 高氏一号培养基:可溶性淀粉20 g、KNO3 1 g、K2HPO4 0.5 g、MgSO4·7H2O 0.5 g、NaCl 0.5 g、FeSO4 0.01 g, 补水至1 L, pH7.2;(3) YD培养基:酵母提取物4 g、麦芽提取物10 g、葡萄糖4 g、MgCl2 2 g、CaCl2 1.5 g, 补水至1 L, pH7.2;(4) 氯化钙培养基(CL):CaCl2 45 g、酵母提取物40 g、葡萄糖5 g, 补水至1 L, pH7.8;(5) 蔗糖察氏培养基:NaNO3 2 g、K2HPO4 1 g、MgSO4·7H2O 0.5 g、KCl 0.5 g、FeSO4·7H2O 0.01 g、蔗糖30 g, 补水至1 L, pH7.2;(6) 马铃薯葡萄糖培养基(PDA):去皮马铃薯200 g, 去皮, 切成小块, 放于锅中加水煮沸0.5 h, 用双层纱布过滤, 取其滤液加葡萄糖20 g, 补水至1 L;(7) 燕麦培养基(ISP3):燕麦20 g, 研磨, 煮沸0.5 h, 用双层纱布过滤, 取其滤液加微量元素溶液1 mL, 补水至1 L, pH7.2, (微量元素溶液:FeSO4·7H2O 0.1 g、MnCl2·4H2O 0.1 g、ZnSO4·7H2O 0.1 g溶于100 mL ddH2O中);(8) 营养肉汤培养基(NA):蛋白胨10 g、牛肉膏3 g、NaCl 5 g, 补水至1 L, pH7.0-7.2。(9) 麦芽汁培养基:麦芽提取物130 g, 补水至1 L, pH5.6±0.2。
1.2 方法 1.2.1 发酵和提取将不产榴菌素的粤蓝链霉菌突变株DMR1甘油保存种划线于YEME平板上, 30℃培养36 h, 挑取单菌落接种于YEME培养基中, 30℃、180 r/min培养36 h, 即为种子液。按5%接种于100 mL发酵培养基中, 30℃、180 r/min, 振动培养7 d, 发酵液经离心(6 000×g、20 min)或用4层纱布过滤, 使菌液分离。发酵上清液用等体积的乙酸乙酯萃取4次, 40℃减压浓缩成粗提物, 用甲醇溶解定容至1 mL。每个处理重复3次。
1.2.2 抗菌活性测定采用琼脂糖扩散法[18]。冷却至45℃的培养基, 加入终浓度约0.5-3×106 cfu/mL的菌体, 混匀后倒入预先放牛津杯培养皿中, 凝固后取出牛津杯, 在孔内加入待测样品。细菌用NA培养基, 白色念珠菌用麦芽汁培养基, 各自适合的培养温度37℃或28℃培养24-48 h, 测定抑菌圈的直径。每个处理重复3次。
1.2.3 TLC-生物显影取粗提物溶解在甲醇中, 毛细管上样, 在氯仿/甲醇(10:1) 中展开, 展层完毕的TLC板, 与含有枯草芽孢杆菌(约2×106 CFU/mL)的NA平板接触一段时间后, 移除TLC板, 含菌平板于37℃培养24 h, 进行生物自显影。
1.2.4 热激及添加诱导物将突变菌株DMR1种子液接种于100 mL高氏一号, 接种量5%, 30℃、180 r/min培养6 h后, 转入42℃培养1 h, 或添加DMSO至终浓度3%, 于30℃继续培养至7 d。
1.2.5 粗提物HPLC分析HPLC仪为岛津LC-20A, 色谱柱为Welch AQ-C18柱(3.9×150 mm, 5 μm)。粗提物用适量甲醇溶解, 0.22 μm滤膜过滤后上样, 进样量5 μL, 流速1 mL/min, 柱温为室温, 流动相为甲醇/水, 使用梯度洗脱, 甲醇浓度随时间的变化为:0-30 min:5%→25%, 30-60 min:25%→50%, 60-80 min:50%→80%, 80-100 min:80%→100%, 100-110 min:100%。
2 结果 2.1 “移树养苗”(Tree-removal)策略在常规培养条件下, 以链霉菌为代表的放线菌通常能产一至数种主要的次级代谢产物, 而其它次级代谢产物生物合成途径处于沉默状态。在这种情形下, 沉默途径即使被特定条件在一定程度上激活, 也可能会被原主要次级代谢产物所掩盖, 而在生物活性检测或化学分析时不易被检出。通过失活主要次级代谢产物的生物合成基因簇, 简化次级代谢产物背景, 可能能够提高新活性次级代谢产物的挖掘效率。我们将这种策略称为“移树养苗”(Tree-removal)策略:即将菌株的主要次级代谢产物的生物合成基因簇失活, 获得不产相应主导产物的突变株, 然后通过OSMAC、核糖体工程和共培养等非特异性激活方法, 使原本不表达或低量表达的产物产量得到一定程度的提高, 在目标产物结构及其生物合成基因簇鉴定后, 通过定向或理性遗传改造等方法, 使其产量进一步提高而成为新的主导产物, 依次循环直至发现若干新产物。该策略的核心是失活主要次级代谢产物的生物合成基因簇, 最重要的作用就是排除已知的主要次级代谢产物的干扰, 简化产物背景, 利于新产物的检出。在实际应用时, 目标菌主要次级代谢产物的生物合成基因簇失活后, 可根据具体情况灵活选择一种或多种激活方法的组合。
2.2 利用“移树养苗”策略挖掘粤蓝链霉菌隐性次级代谢产物 2.2.1 改变培养基成分改变培养基成分也是OSMAC最常用的手段。首先考察高氏一号等6种不同培养基对粤蓝链霉菌突变株DMR1次级代谢的影响。抑菌活性测试发现, 高氏一号、PDA和ISP3培养基发酵粗提物对枯草芽孢杆菌和金黄色葡萄球菌具有显著的抑菌活性, 其余培养基发酵粗提物对所试菌株无明显活性(表 1)。TLC分析发现, 其余不同培养基的发酵产物存在一定的差别, 表现出抑菌活性的3种培养基较为类似, 其中高氏一号发酵产物最为丰富。进一步对高氏一号、PDA和ISP3发酵粗提物进行TLC-生物显影, 发现3种发酵粗提物均在含菌平板上相近的区域出现了抑菌透明圈, 对应TLC的Rf值约为0.7(图 1)。由于高氏一号和PDA在产物类型上更为接近, 因此, 选择高氏一号和ISP3进行后续研究。

|
| 图 1 粤蓝链霉菌突变株DMR1不同培养基发酵粗提物生物显影 A、B、C分别是在可见光、254 nm和365 nm下的薄层层析结果, D为在含有枯草芽孢杆菌ATCC6633的NA平板上的显影, 层析板与NA平板的接触时间为5 min;1-3:分别为高氏1号培养基、PDA培养基和ISP3培养基处理 |
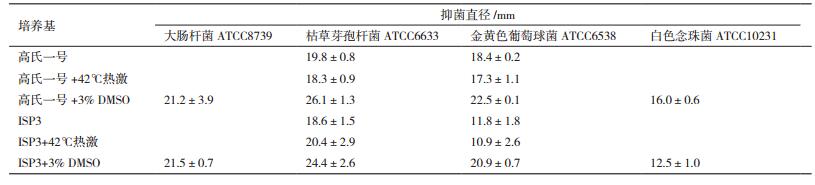
微生物对不同的胁迫条件有特定的应激机制, 从而可能触发特定隐性基因簇的表达。结果(表 2)显示, 42℃热激1 h处理与对照相比, 高氏一号和ISP3发酵粗提物的抑菌活性无显著变化;而添加3% DMSO后, 高氏一号和ISP3发酵粗提物不仅对枯草芽孢杆菌ATCC6633和金黄色葡萄球菌ATCC6538的抑菌活性显著增强, 而且对大肠杆菌ATCC8739和白色念珠菌ATCC10231也出现了较好抑制作用。将各粗提物进行HPLC, 发现在高氏一号和ISP3两种培养基中, 热激处理与未诱导对照的次级代谢产物特征几乎相同, 而DMSO诱导后, 两种培养基的次级代谢产物特征与对照相比存在少量的差异峰(图 2), 这与活性测试的结果相吻合。以枯草芽孢杆菌作为指示菌, 利用微孔板进一步追踪抑菌活性部位, 发现高氏一号和ISP3两种培养基活性部位的保留时间均在58-62 min之间(图 2, 微孔板抑菌活性测试结果未显示)。

|
| 图 2 粤蓝链霉菌突变株DMR1热激和DMSO诱导后发酵粗提物HPLC分析 A、B, 分别为粤蓝链霉菌突变株DMR1在高氏1号培养基和ISP3培养基中培养后粗提物的液相分析结果;GS-CK、GS-42℃、GS-DMSO分别表示在高氏1号培养基中未诱导、热激和添加DMSO的处理;IPS3-CK、IPS3-42℃、IPS3-DMSO分别表示在ISP3培养基中未诱导、热激和添加DMSO的处理;竖虚线提示了各处理间的差异峰, 灰色区域是保留时间在58-62 min的馏分, 对枯草芽孢杆菌具有抑制作用 |
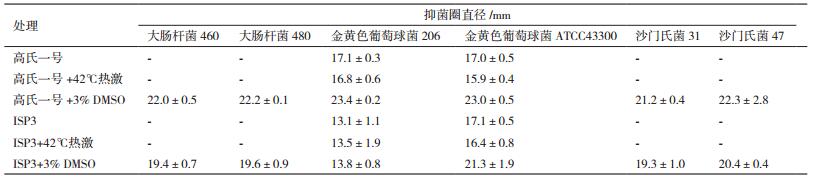
进一步对发酵粗提物对耐药菌的抑菌活性进行分析发现, 高氏一号、ISP3的对照组和热激组发酵粗提物对耐药菌金黄色葡萄球菌206和金黄色葡萄球菌ATCC43300有显著的抑制活性;而3% DMSO处理组粗提物对所有受试大肠杆菌、金黄色葡萄球菌和沙门氏菌的耐药菌株均有显著的抑制活性(表 3), 提示该活性物质具有广谱抗耐药菌的活性。图 4展示了ISP3发酵粗提物对各耐药菌所形成的抑菌圈。

|
| 图 4 突变株DMR1的ISP3发酵粗提物对耐药菌形成的抑菌圈 A-F:分别是含有耐药菌大肠杆菌(460、480)、金黄色葡萄球菌(206、ATCC43300)、沙门氏菌(31、47) 的营养琼脂平板;孔1-3, 分别是在ISP3培养基正常条件培养、添加3% DMSO和42℃热激1 h处理的粗提物, CK为甲醇, Amp为50 μg/mL的氨苄西林, 所有样品添加量为60 μL/孔 |
当前, 放线菌天然产物研究面临的一大挑战是:高频率重复发现的同时, 缺乏挖掘隐性次级代谢产物的高效、普适的手段。这主要是由于现有实验室培养方法的模式化和缺乏对产生菌的代谢调控网络的深入认识。以链霉菌为例, 该类菌有着复杂的形态发育分化和丰富的次级代谢, 且二者之间具有密切的联系, 而所有这些过程都被细微和精确的调控系统所控制。相应地, 其基因组上存在至多可达约12%的调控基因(超过1 000个)[19], 这些基因共同组成一个复杂的调控网络, 使人们难以在短期内从总体上认识和把握该网络的作用机制。在打开这一“暗箱”以前, 我们必须通过各种方法和策略来提高隐性次级代谢产物的挖掘效率。“移树养苗”策略, 将产生菌主导产物的生物合成基因簇失活, 使产生菌不再产生原主导产物后, 再对突变株进行深度挖掘, 其有益效果在于:(1) 可改变产生菌代谢通路的流向, 使前体物质能够流向处于弱势的次级代谢途径;(2) 代谢产物背景更为简单, 排除了原主导产物的干扰, 使隐性次级代谢产物更容易被生物活性测试、化学分析等方法检出。
“移树养苗”策略的有效性此前已被证实。前文提到的天蓝霉素(Coelimycin)P1, 虽然最初是通过敲除调控基因scbR2后发现天蓝色链霉菌能够在部分培养基中产生一种黄色色素[20], 但最终确认和解析其结构时就利用了该策略[13]。本文的研究对象粤蓝链霉菌的主导产物是榴菌素。由于榴菌素存在抗菌和抗肿瘤活性, 在活性筛选时对其它可能的活性代谢产物的检出造成干扰。本研究利用“移树养苗”策略, 对榴菌素基因簇失活后的突变株利用“一菌多素”的方法进行进一步挖掘, 新发现了该菌能产生具有广谱和抗多种耐药菌活性的次级代谢产物, 也表明该策略能够提高隐性活性次级代谢产物的挖掘效率。
热激是常用的激活方法, 它可能通过微生物应激机制而影响细胞的生理代谢过程。然而在本研究中, 从抗菌活性和HPLC分析结果来看, 42℃热激1 h并未对粤蓝链霉菌产生显著的影响, 表明热激对不同菌株的效果存在差异。添加DMSO后, 粤蓝链霉菌高氏一号培养基的活性部位(保留时间58-62 min)在280 nm下的吸收峰增加不明显, 而在520 nm下有一定程度增强;ISP3培养基则相反, 甚至DMSO诱导后在520 nm下几乎检测不到任何峰, 这提示高氏一号和ISP3培养基所产生的活性物质可能为不同物质, 也说明同一诱导物对同一菌株在不同培养基上的诱导效应也可能存在差别。
耐药菌已成为严重和日益增长的威胁, 挖掘和发现新型抗生素对人类的健康极为关键[21]。放线菌作为传统重要的药源微生物, 虽受重复发现等问题的困扰, 但仍是重要新型抗生素的来源。只要采取一些适当方法和策略, 仍可不断从中发现新的抗生素, 如Lassomycin[22], 天赐霉素(Tiancimycin)[23]等。由于除榴菌素外, 粤蓝链霉菌基因组中其它潜在的次级代谢产物生物合成基因簇均与已知的抗生素基因簇不同, 因此, 本研究所发现的广谱抗耐药菌活性次级代谢产物, 很可能是一种新型的抗生素。目前, 我们已进行了发酵条件的优化, 进一步提高了活性产物的产率, 分离纯化和结构鉴定正在进行中。
4 结论本研究系统总结概况了“移树养苗”策略, 并将之应用于粤蓝链霉菌隐性活性次级代谢产物的挖掘, 从中新发现了具有广谱抗耐药菌的活性物质, 证明了该策略能够提高隐性活性次级代谢产物的挖掘效率。
| [1] | Butler MS, Robertson AAB, Cooper MA. Natural product and natural product derived drugs in clinical trials[J]. Natural Product Reports, 2014, 31 (11): 1612–1661. DOI:10.1039/C4NP00064A |
| [2] | Newman DJ, Cragg GM. Natural products as sources of new drugs over the 30 years from 1981 to 2010[J]. Journal of Natural Products, 2012, 75 (3): 311–335. DOI:10.1021/np200906s |
| [3] | Li JWH, Vederas JC. Drug discovery and natural products: End of an era or an endless frontier?[J]. Science, 2009, 325 (5937): 161–165. DOI:10.1126/science.1168243 |
| [4] | Bentley SD, Chater KF, Cerdeno-Tarraga AM, et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2)[J]. Nature, 2002, 417 (6885): 141–147. DOI:10.1038/417141a |
| [5] | Mardis ER. The impact of next-generation sequencing technology on genetics[J]. Trends in Genetics, 2008, 24 (3): 133–141. DOI:10.1016/j.tig.2007.12.007 |
| [6] | Shen B. A new golden age of natural products drug discovery[J]. Cell, 2015, 163 (6): 1297–1300. DOI:10.1016/j.cell.2015.11.031 |
| [7] | Bode HB, Bethe B, H?fs R, et al. Big effects from small changes: Possible ways to explore nature's chemical diversity[J]. Chembiochem, 2002, 3 (7): 619–627. DOI:10.1002/1439-7633(20020703)3:7<619::AID-CBIC619>3.0.CO;2-9 |
| [8] | Mearns-Spragg A, Bregu M, Boyd KG, et al. Cross-species induction and enhancement of antimicrobial activity produced by epibiotic bacteria from marine algae and invertebrates, after exposure to terrestrial bacteria[J]. Letters in Applied Microbiology, 1998, 27 (3): 142–146. DOI:10.1046/j.1472-765X.1998.00416.x |
| [9] | Shima J, Hesketh A, Okamoto S, et al. Induction of actinorhodin production by rpsL(encoding ribosomal protein S12) mutations that confer streptomycin resistance in Streptomyces lividans and Streptomyces coelicolor A3(2)[J]. Journal of Bacteriology, 1996, 178 (24): 7276–7284. DOI:10.1128/jb.178.24.7276-7284.1996 |
| [10] | Zarins-Tutt JS, Barberi TT, Gao H, et al. Prospecting for new bacterial metabolites: a glossary of approaches for inducing, activating and upregulating the biosynthesis of bacterial cryptic or silent natural products[J]. Natural Product Reports, 2016, 33 (1): 54–72. DOI:10.1039/C5NP00111K |
| [11] | Rutledge PJ, Challis GL. Discovery of microbial natural products by activation of silent biosynthetic gene clusters[J]. Nature Reviews Microbiology, 2015, 13 (8): 509–523. DOI:10.1038/nrmicro3496 |
| [12] | 齐志, 孙东昌, 裘娟萍. 微生物沉默基因簇激活方法的研究进展[J]. 科技通报, 2016, 32(2): 82–86. |
| [13] | Gomez-Escribano JP, Song L, Fox DJ, et al. Structure and biosynthesis of the unusual polyketide alkaloid coelimycin P1, a metabolic product of the cpk gene cluster of Streptomyces coelicolor M145[J]. Chemical Science, 2012, 3 (9): 2716–2720. DOI:10.1039/c2sc20410j |
| [14] | Zhu HH, Guo J, Yao Q, et al. Streptomyces vietnamensis sp. nov., a streptomycete with violet blue diffusible pigment isolated from soil in Vietnam[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57 (8): 1770–1774. DOI:10.1099/ijs.0.64774-0 |
| [15] | 邓名荣, 朱春华, 郭俊, 等. 粤蓝链霉菌中紫罗兰蓝色素成分的分离与鉴定[J]. 华南理工大学学报:自然科学版, 2011, 39(5): 132–137. |
| [16] | Deng MR, Guo J, Ma LY, et al. Complete genome sequence of Streptomyces vietnamensis GIMV4. 0001T, a genetically manipulable producer of the benzoisochromanequinone antibiotic granaticin[J]. Journal of Biotechnology, 2015, 200 : 6–7. DOI:10.1016/j.jbiotec.2015.02.009 |
| [17] | Deng MR, Guo J, Zhu HH. Streptomyces vietnamensis GIMV4. 0001: a granaticin-producing strain that can be readily genetically manipulated[J]. Journal of Antibiotics, 2011, 64 (4): 345–347. DOI:10.1038/ja.2011.3 |
| [18] | 邓名荣, 郭俊, 朱红惠. 粤蓝链霉菌代谢产物的抗菌抗肿瘤活性及相关基因的初步研究[J]. 天然产物研究与开发, 2010, 22(3): 367–372. |
| [19] | Romero-Rodriguez A, Robledo-Casados I, Sanchez S. An overview on transcriptional regulators in Streptomyces[J]. Biochimica Et Biophysica Acta-Gene Regulatory Mechanisms, 2015, 1849 (8): 1017–1039. DOI:10.1016/j.bbagrm.2015.06.007 |
| [20] | Gottelt M, Kol S, Gomezescribano JP, et al. Deletion of a regulatory gene within the cpk gene cluster reveals novel antibacterial activity in Streptomyces coelicolor A3(2)[J]. Microbiology, 2010, 156 (8): 2343–2353. DOI:10.1099/mic.0.038281-0 |
| [21] | Butler MS, Blaskovich MAT, Owen JG, et al. Old dogs and new tricks in antimicrobial discovery[J]. Current Opinion in Microbiology, 2016, 33 : 25–34. DOI:10.1016/j.mib.2016.05.011 |
| [22] | Gavrish E, Sit Clarissa S, Ca oS, et al. Lassomycin, a ribosomally synthesized cyclic peptide, kills Mycobacterium tuberculosis by targeting the ATP-dependent protease ClpC1P1P2[J]. Chemistry & Biology, 2014, 21 (4): 509–518. |
| [23] | Yan X, Ge H, Huang T, et al. Strain prioritization and genome mining for enediyne natural products[J]. mBio, 2016, 7 (6): e02104–16. |