2. 甘肃农业大学生命科学技术学院,兰州 730070
2. College of Life Science and Technology, Gansu Agricultural University, Lanzhou 730070
基因编辑技术在构建动物模型及研究基因功能等方面发挥重要作用。传统生产重组蛋白是通过基因打靶技术将外源基因导入动物原核期受精卵获得转基因动物, 使重组蛋白在动物乳腺中特异表达, 国内外学者分别通过制备转基因小鼠、奶山羊、奶牛等动物乳腺生物反应器获得人溶菌酶(Human Lysozyme, HLZ)[1]、人抗胰蛋白酶(Human a1-antitrypsin, al-AT)[2]、人凝血Ⅸ因子(Human clotting factor Ⅸ, hFⅨ)[3]及人血清白蛋白(Human Serum Albumin, HSA)[4]等重组蛋白。
然而, 利用乳腺生物反应器生产外源蛋白难以实现产业化发展, 其中, 重组蛋白表达量低是主要限制因素, 究其原因可能是外源基因在动物基因组中的随机整合, 受到邻近染色体影响[5]或插入位点受到内源性基因抑制引起[6, 7]。而利用基因编辑技术将外源基因定点插入到乳蛋白启动子之后, 利用乳蛋白上下游调控序列指导外源蛋白表达, 成为制备高效稳定动物乳腺生物反应器的新方向[8]。Wright等[2]将外源基因定点整合到乳蛋白启动子之后, 获得转基因绵羊乳汁中人抗胰蛋白酶表达量高达35 g/L。而Naruse等[9]将人类Ⅱ型胶原蛋白基因插入到牛β-酪蛋白启动子表达载体后, 构建小鼠乳腺生物反应器模型, 结果发现小鼠在泌乳期外源蛋白表达量仅在10-50 µL/mL。Marcelo等[10]研究发现乳蛋白启动子在转基因动物中的基因拷贝数对外源蛋白的表达也有很大影响, 研究表明含有6-10个基因拷贝数的小鼠中, 其乳汁中重组人GH蛋白的表达量达2.63 mg/mL。而较低拷贝数(2-3) 的小鼠的表达量仅为1 mg/mL;然而, 虽然可以将外源基因定点整合到乳蛋白启动子之后, 但不同动物外源蛋白表达量相差较大, 且大多处于较低水平。
近年来, 一种成簇规律间隔回文段重复序列及邻近相关基因(CRISPR/Cas9), 在细菌[11]、动物[12]、植物[13]以及人类细胞[14]上都表现出强大的基因编辑能力。同时, 有研究表明敲除α-乳蛋白基因的小鼠乳汁变得黏稠, 甚至无法泌乳哺乳后代, 而利用基因打靶技术置换人α-乳蛋白基因后, 小鼠泌乳机能恢复正常, 人α-乳蛋白也有表达[15]。而在敲除小鼠β-酪蛋白基因后, 小鼠泌乳及繁殖均未受到影响, 只是乳汁中酪蛋白胶粒直径减小[16], 小鼠与大动物之间毕竟存在较大差异, 因此有必要探究β-酪蛋白对奶山羊泌乳性能的影响。为此, 本实验利用CRISPR/Cas9系统对奶山羊胎儿成纤维细胞的β-酪蛋白基因进行敲除, 筛选高效敲除效率sgRNA, 并获得敲除CSN2基因的突变细胞, 为制备CSN2基因敲除奶山羊提供核供体材料, 为研究β-酪蛋白对动物泌乳性能影响奠定基础。
1 材料与方法 1.1 材料 1.1.1 仪器HSW24型恒温水浴锅、THZ-82A型恒温摇床、电子天平PCR仪(Bio-Rad)、凝胶成像仪(Bio-Rad Universal Hood Ⅱ)、荧光显微镜、CO2培养箱、高压蒸汽锅、涡旋振荡仪。
1.1.2 实验材料奶山羊胎儿成纤维细胞F2代(西北农林科技大学动物医学院惠赠), 限制性内切酶BbsⅠ、T4连接酶(M0202L)均购自美国NEB公司;一抗(Rabbit Anti-Beta casein)及二抗(Mouse Anti-rabbit-Lg G)均购自北京博奥森生物技术有限公司, G418(新霉素)和胰蛋白酶购自GBICO公司;脂质体(LipofectamineTM2000) 购自Invitrogen公司。
1.2 方法 1.2.1 靶位点sgRNA的选择参照奶山羊β-酪蛋白基因(ID:100860784), 按照“20N+NGG”原则在第二、八外显子处设计5对特异识别靶序列DNA的sgRNA寡聚核苷酸链, 分别在编码链5'端添加CACC, 非编码链3'端添加AAAC, 由上海金唯智公司合成, sgRNA序列见表 1。
sgRNA寡聚核苷酸链经程序性退火(95℃ 5 min自然降至室温), 连入含Bbs I酶切位点的胶回收产物PX330-eGFP(本实验室保存)载体中, 通过测序验证正确连入后, 分别命名为PX330-eGFP-sgRNA T1、PX330-eGFP-sgRNA T2、PX330-eGFP-sgRNA T3。
1.2.3 敲除效率鉴定培养奶山羊胎儿成纤维细胞并将其分为PX330(阴性对照组)、PX330-eGFP-sgRNA T1/T2/T3和PX330-eGFP-sgRNA E1/E2, 按照5 µg DNA与10 µL脂质体转染细胞, 48 h后经流式分选获绿色荧光细胞, 扩大培养后提取细胞基因组DNA, 扩增各靶点片段上下游共计685 bp(扩增引物F:5'-TCAAAGACCCACTGAATA-3';R:5'-ATGGCTGAGGTAGGTTTA-3')及819 bp(扩增引物F:5'-CTTTCTCCAACCGTCAT-3';R:5'-TGCCTCCAATCTCCTAA-3'), 经凝胶成像鉴定条带单一, PCR纯化后, 使用T7E I酶切检测敲除效率, 反应体系:目的片段200 ng;NEB buffer 1 μL;95℃, 5 min;95-85℃(-2℃/s);85-25℃(-0.1℃/s);12℃保存, 然后加入T7E I酶1 μL, 37℃反应20 min, 2%琼脂糖凝胶电泳20 min。突变条带百分比用Gel-Pro analyzer凝胶成像软件分析灰度值。
1.2.4 Western blot检测提取PX330-eGFP-sgRNA T质粒转染后细胞总蛋白, 经SDS-PAGE电泳并转移至PVDF膜后, 用5%脱脂奶粉封闭30 min, 分别加入1:300比例稀释的一抗(Rabbit Anti-Beta casein)和1:500比例稀释的鼠抗兔抗体(Mouse Anti-rabbit-Lg G), 室温孵育2 h, TBST洗膜后, 使用DAB显色并拍照。
1.2.5 PX330-Neo-sgRNA质粒构建及共转染选择第八外显子处具高效敲除效率sgRNA, 构建PX330-Neo-sgRNA E1敲除载体, 测序鉴定正确连入后, 按照如下组合转染细胞:
A方案:PX330-eGFP-sgRNA T和PX330-eGFP-sgRNA E;B方案:PX330-eGFP-sgRNA T和PX330-Neo-sgRNA E。
其中, A方案阳性细胞基因组, 按B方案转染细胞48 h后, 经流式分选后继续培养细胞, 加入800 µg/mL G418进行药物筛选, 当发现细胞大量死亡时, 降低G418浓度至200 µg/mL, 直至出现单克隆细胞, 停药继续扩大培养, 提取细胞基因组, 扩增靶点上下游8 288 bp片段(扩增引物F:5'-AAAGACCCACTGAATACTAAAGAGACCTCATTG-3';R:5'-TGCCTCCAATCCTAAAGCTTACCAAAAG-3'), 使用T7E I酶切鉴定大片段敲除效率。
1.2.6 荧光定量PCR在CSN2第七号外显子设计荧光定量PCR检测引物(F:5'-ACAGCCTCCCACA-AAA-CATC;R:5'-AAGACTGGACCAGAGGCAGA-3'), 分析不同敲除载体组合转染组细胞CSN2 mRNA表达情况, 反应体系为20.0 μL:上下游引物各0.5 μL、cDNA模板2.0 μL、2×QuantiNovaTM SYBR® Green PCR Mix 10.0 μL和ddH2O 7.5 μL。反应程序如下:95℃ 2 min;95℃ 5 s, 60℃ 10 s, 72℃ 15 s, 45个循环;60℃-94℃熔解曲线。
1.2.7 细胞免疫荧光染色接种细胞于24孔板中, 待其汇合度达30%-40%, 经固定及透析处理后, 使用5%脱脂奶粉封闭30 min, 加入1:300稀释一抗(Rabbit Anti-Beta casein), 4℃过夜孵育, 以1:500稀释二抗(Mouse Anti-rabbit-Lg G), 避光作用2 h, DAPI染核并于荧光显微镜下观察、拍照, 使用Image J对荧光图片进行自动荧光定量分析。
2 结果 2.1 PX330-eGFP-sgRNA载体构建使用各Oligo F与PX330-检(R:5'-CGGGTAC-CTCTAGAGCCATT-3')进行菌液PCR, 得一条大小为196 bp的特异性条带, 与预期结果相符, 将正确菌落送测序, 测序结果与sgRNA序列完全匹配, 表明sgRNA打靶载体构建成功(图 1)。

|
| 图 1 Oligo退火产物连接PX330-eGFP菌落PCR电泳图 M:DL2000 DNA Marker;1:PX330-eGFP-sgRNA T1;2:PX330-eGFP-sgRNA T2;3:PX330-eGFP-sgRNA T3;4:PX330-eGFP-sgRNA E1;5:PX330-eGFP-sgRNA E2 |
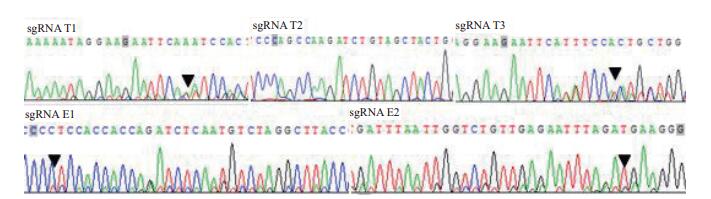
按5 µg DNA与10 µL脂质体混合转染细胞, 48 h后经流式分选仪分选细胞, 提取细胞基因组DNA, PCR扩增靶点基因后送测序, 使用Chromas观察靶点处图谱, 结果(图 2)表明sgRNA T1、sgRNA T3及sgRNA E1、sgRNA E2均发挥出切割作用, 即峰图靶点处出现不同程度的叠峰, sgRNA T2作用靶点图谱未出现任何叠峰, 可能是未发挥作用或发挥作用后经过了正确的修复。
|
| 图 2 奶山羊胎儿成纤维细胞CSN2靶基因PCR产物测序图谱 注:箭头所指位置为突变位点 |
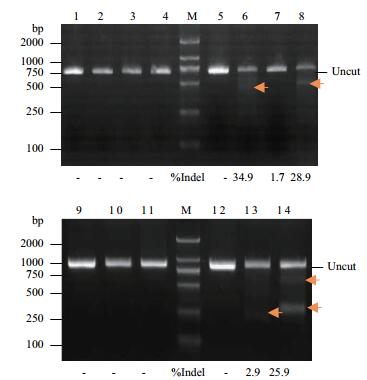
使用T7E I酶切鉴定各sgRNA敲除效率, 突变条带所占百分比用Gel-Pro analyzer凝胶成像软件分析灰度值, 切出多条条带可大致反映突变效率, 其sgRNA敲除效率分别为34.9%、1.7%、28.9%、2.9%和25.9%(图 3)。
|
| 图 3 T7E I酶切测效图 M:DL2000 DNA Marker;1-4:分别为未经过T7E I酶切的空白、sgRNA T1、sgRNA T2及sgRNA T3 PCR产物;5-8:分别为T7E I酶切的空白、sgRNA T1、sgRNA T2及sgRNA T3 PCR产物;9-11:分别为未经过T7E I酶切的空白、sgRNA E1及sgRNA E2 PCR产物;12-14:分别为T7E I酶切的空白、sgRNA E1及sgRNA E2 PCR产物 |
单克隆测序结果与PCR产物测序结果相吻合, 其中碱基突变范围在2-31 bp之间, 结果(表 2)表明sgRNA T1和sgRNA E2具有较高敲除效率。
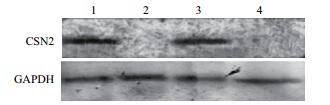
分别提取PX330-eGFP-sgRNA T转染细胞总蛋白, 经SDS-PAGE检测奶山羊胎儿成纤维细胞CSN2表达情况, 结果(图 4)显示sgRNA T2质粒转染组细胞CSN2有表达, 与测序结果吻合, 表明sgRNA T2无效, 而sgRNA T1与sgRNA T3质粒转染细胞CSN2未表达, 表明sgRNA T1与sgRNA T3能有效敲除奶山羊胎儿成纤维细胞CSN2基因。
|
| 图 4 Western blot鉴定CSN2 1:GFFs细胞总蛋白;2-4:分别为PX330-eGFP-sgRNA T1、PX330-eGFP-sgRNA T2及PX330-eGFP-sgRNA T3转染细胞总蛋白 |
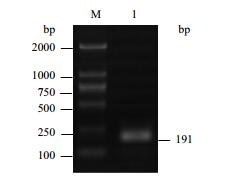
使用Oligo-CSN2-E8-F1与PX330-检-R进行菌液PCR, 得一条大小为191 bp特异性条带(图 5), 与预期结果一致, 经测序验证成功连入后, 命名为PX330-Neo-sgRNA E1。
|
| 图 5 Oligo退火产物连接PX330菌落PCR电泳图 M:DL2000 DNA Marker;1:PX330-Neo-sgRNA E1 |
使用PX330-eGFP-sgRNA T1与PX330-Neo-sgR-NA E1及PX330-eGFP-sgRNA E1两两组合(eGFP+ Neo, eGFP+ eGFP)转染细胞(图 6)。使用T7E I酶切鉴定混合转染sgRNA敲除效率, 突变条带所占百分比用Gel-Pro analyzer凝胶成像软件分析灰度值, 其敲除效率分别为:16.2%和13%, 扩增目的基因后凝胶成像检测(图 7)发现, 出现一条非特异性条带, 说明双载体转染奶山羊胎儿成纤维细胞, 能有效对CSN2基因大片段(最长7 760 bp)进行敲除(图 8)。
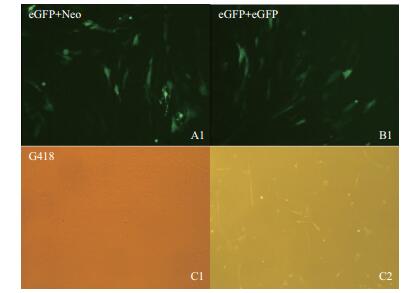
|
| 图 6 转染及药筛细胞图(100×) A1:eGFP+ eGFP转染48 h后细胞绿色荧光表达情况;B1:eGFP+Neo转染48 h后细胞绿色荧光表达情况;C1:G418处理前细胞形态, C2:G418处理第6天细胞形态 |

|
| 图 7 T7E I酶切测效图 M:1kb DNA Marker;1-3:分别是未经过T7E I酶切对照组、eGFP+ Neo组, eGFP+eGFP组PCR产物;4-6:分别是T7E I酶切对照组、eGFP+ Neo组, eGFP+eGFP组PCR产物;箭头所指为非特异性条带 |

|
| 图 8 Cas9重组蛋白介导的GFFs大片段CSN2基因敲除 PAM基序表示为红色;靶点蓝色表示;突变绿色表示;N#为单克隆序号 |
使用CSN2-mRNA-F/R引物进行定量PCR检测发现, 两种不同组合方式获得的细胞CSN2在mRNA水平上的表达存在明显差异(图 9)。其中eGFP+Neo组合获得的细胞CSN2在mRNA水平表达显著低于对照组(P < 0.05), 说明eGFP+Neo组比eGFP+eGFP组能获得更高的阳性细胞率。
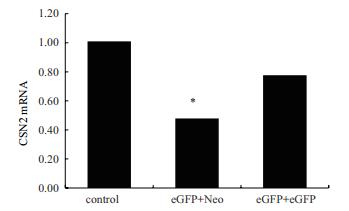
|
| 图 9 各组细胞CSN2 mRNA表达比较 *:P < 0.05, 与对照组比较 |
采用免疫荧光法对双载体转染细胞进行染色(图 10), 使用Image J图像分析软件对细胞CSN2表达情况进行评估, 结果(图 11)表明eGFP+Neo组合细胞CSN2表达明显少于eGFP+eGFP组(P < 0.05)。说明eGFP+Neo组合能提高突变细胞在正常细胞中的比例。
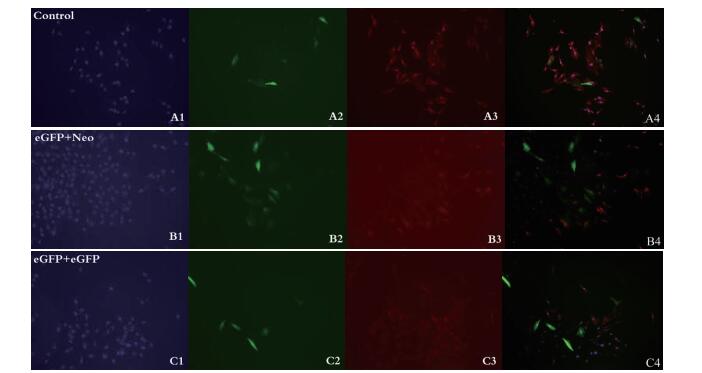
|
| 图 10 细胞荧光染色图(100×) A1-C1:分别为对照组、eGFP+Neo组、eGFP+eGFP组细胞核DAPI染色图;A2-C2:分别为对照组、eGFP+Neo组、eGFP+eGFP组绿色荧光蛋白表达图;A3-C3:分别为对照组、eGFP+Neo组、eGFP+eGFP组细胞CSN2荧光染色图;A4-C4:分别为对照组、eGFP+Neo组、eGFP+eGFP组细胞核、绿色荧光蛋白及CSN2合并图 |
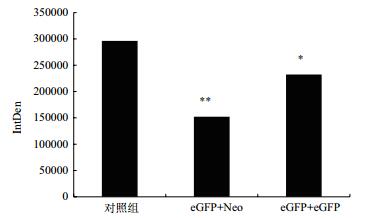
|
| 图 11 细胞CSN2表达统计图 *:P < 0.05, 与对照组比较 |
CSN2是酪蛋白家族中的主要成员之一, 约占动物乳汁酪蛋白总量的80%, 该蛋白能与其他酪蛋白结构形成胶团, 影响动物乳汁物理性质[17], 对其在乳汁中高效表达的特性进行深入研究, 将有助于改善重组蛋白在动物乳汁中高效特异性表达等问题。早期研究基因功能的主要方法有同源重组(Homologous recombination, HR)、锌指核酸酶(Zinc-finger nucleases, ZFNs)及转录激活因子样效应物核酸酶(Transcription activator-like effector nucleases, TALEN)技术等, 因其存在诸多问题, 如打靶效率低、技术难度大及载体构建复杂等, 难以大范围推广使用。新型基因编辑技术CRISPR/Cas9系统借助在靶点处产生DNA双链断裂(Doublestrand breaks, DSB), 通过非同源末端连接(Non-homologous end joining, NHEJ)修复机制修复DSBs, 引发切割位点DNA碱基插入或缺失, 从而实现基因敲除的目的, 该技术因简易、高效、低成本等优势[18, 19], 已被成功应用于小鼠、大鼠、斑马鱼、果蝇等多种模式动物, 包括基因功能[20]、遗传疾病治疗[21]等研究。
本研究利用CRISPR/Cas9基因编辑技术在奶山羊胎儿成纤维细胞CSN2基因靶位点处引入突变, 发现该系统发挥基因编辑的效率与靶序列sgRNA构成有关[22, 23]。本实验分别选择5'-TN(19) NGG与5'-CN(19) NGG两种sgRNA类型, 通过T7E I酶切实验分析发现sgRNA T1与sgRNA E1具有较高基因编辑效率, 分别为34.9%和25.9%, 该sgRNA类型为5'-TN(19) NGG, 而sgRNA T2未表现出基因编辑作用, 实验结果表明5'-TN(19) NGG类型sgRNA具有较高基因编辑效率, 与前人报道类似[24]。相同类型sgRNA表现出不同的基因编辑效率, 可能与sgRNA对靶序列的结合能力有关。
CRISPR/Cas9系统对靶位点编辑会引起多种形式突变, 如碱基缺失、碱基增加或替换等[25, 26], 本研究靶点突变类型主要为碱基缺失, 部分为碱基替换。研究发现各sgRNA引起靶点部分碱基缺失数均为3的非整数倍, 造成蛋白无法正常被翻译, 通过移码突变实现CSN2基因真正意义的敲除。通常CRISPR/Cas9系统造成DSBs所引起的NHEJ, 使我们无法获得全部基因功能, 例如, 长链非编码区RNA(Long non-coding RNAs, lncRNA)功能[27]、框内突变[28]等。本实验利用双载体转染方案, 首次验证CRISPR/Cas9系统在奶山羊细胞水平长片段基因编辑的有效性, 敲除长度长达7 760 bp, 通过T7E I酶切实验分析发现eGFP+Neo组与eGFP+ eGFP组基因突变率分别为16.2%和13%, 突变率处于较低水平, 造成这一现象的原因有待进一步研究, 不过该方法有效降低假阳性细胞, 为全面深入研究CSN2基因对奶山羊泌乳性能研究提供物质基础。
在长片段基因修饰转基因动物制备过程中, 通常使用打靶早期胚胎获得转基因动物, 经过复杂繁程序筛选突变个体, 而本实验利用载体筛选特异性, 将eGFP基因与含Neo基因的敲除载体结合, 转染奶山羊胎儿成纤维细胞, 挑阳性单克隆细胞进行培养, 解决了制备转基因动物周期长, 筛选阳性动物繁琐等问题, 本实验获得敲除CSN2基因奶山羊胎儿成纤维细胞, 为制备敲除CSN2奶山羊提供材料。
4 结论实验成功利用CRISPR/Cas9系统在奶山羊胎儿成纤维细胞CSN2基因靶位点处引入突变, 其突变率最高可达34.9%, 首次在奶山羊胎儿成纤维细胞上实现7 760 bp基因长片段的敲除, 验证该系统对奶山羊基因编辑的有效性, 并优化大片段阳性细胞制备方案, 获得CSN2基因敲除的细胞为制备CSN2基因敲除奶山羊提供了核供体。
| [1] | Maga EA, Shoemaker CF, Rowe JD, et al. Production and processing of milk from transgenic goats expressing human lysozyme in the mammary gland[J]. J Dairy Sci, 2006, 89 (2): 518–524. DOI:10.3168/jds.S0022-0302(06)72114-2 |
| [2] | Wright G, Carver A, Cottom D, et al. High Level Expression of Active Human Alpha-1-Antitrypsin in the Milk of Transgenic Sheep[J]. Nature Biotechnology, 1991, 9 (9): 830–834. DOI:10.1038/nbt0991-830 |
| [3] | Clark AJ, Bessos H, Bishop JO, et al. Expression of Human Anti-Hemophilic Factor Ⅸ in the Milk of Transgenic Sheep[J]. Nature Biotechnology, 1989, 7 (5): 487–492. DOI:10.1038/nbt0589-487 |
| [4] | Luo Y, Wang Y, et al. Production of transgenic cattle highly expres-sing human serum albumin in milk by phiC31 integrase-mediated gene delivery[J]. Transgenic Res, 2015, 24 (5): 875–883. DOI:10.1007/s11248-015-9898-0 |
| [5] | Clark AJ, Bissinger P, Bullock DW, et al. Chromosomal position effects and the modulation of transgene expression[J]. Reprod Fertil Dev, 1994, 6 (5): 589–598. DOI:10.1071/RD9940589 |
| [6] | Dorer DR. Do transgene arrays form heterochromatin in vertebrates?[J]. Transgenic Research, 1997, 6 (1): 3–10. DOI:10.1023/A:1018460413680 |
| [7] | Garrick D, Fiering S, Martin DI, et al. Repeat-induced gene silencing in mammals[J]. Nature Genetics, 1998, 18 (1): 56–59. DOI:10.1038/ng0198-56 |
| [8] | Hennighausen L. The prospects for domesticating milk protein genes[J]. J Cell Biochem, 1992, 49 (4): 325–332. DOI:10.1002/(ISSN)1097-4644 |
| [9] | Naruse K, Yoo SK, Sun MK, et al. Analysis of tissue-specific expression of Human Type Ⅱ Collagen cDNA driven by different sizes of the upstream region of the β-Casein promoter[J]. Biosci Biotechnol Biochem, 2006, 70 (1): 93–98. DOI:10.1271/bbb.70.93 |
| [10] | Cerdán MG, Young JI, Zino E, et al. Accurate spatial and temporal transgene expression driven by a 3. 8-kilobase promoter of the bovine beta-casein gene in the lactating mouse mammary gland[J]. Mol Reprod Dev, 1998, 49 (3): 236. DOI:10.1002/(ISSN)1098-2795 |
| [11] | Charpentier E. Programmable Dual-RNA-Guided DNA endonucl-ease in adaptive bacterial immunity[J]. Ieice Technical Report Electron Devices, 1995, 95 : 55–60. |
| [12] | Hwang WY, Fu Y, Reyon D, et al. Heritable and precise zebrafish genome editing using a CRISPR-Cas system[J]. PLoS One, 2013, 8 (7): e68708. DOI:10.1371/journal.pone.0068708 |
| [13] | Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31 (8): 686. DOI:10.1038/nbt.2650 |
| [14] | Ho TT, Zhou N, Huang J, et al. Targeting non-coding RNAs with the CRISPR/Cas9 system in human cell lines[J]. Nucleic Acids Research, 2015, 43 (3): e17. DOI:10.1093/nar/gku1198 |
| [15] | Stinnakre MG, Vilotte JL, Soulier S, et al. Creation and phenotypic analysis of α-Lactalbumin-Deficient mice[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91 (14): 6544–6548. DOI:10.1073/pnas.91.14.6544 |
| [16] | Kumar S, Clarke AR, Hooper ML, et al. Milk composition and lactation of beta-casein-deficient mice[J]. Proc Natl Acad Sci USA, 1994, 91 (13): 6138–6142. DOI:10.1073/pnas.91.13.6138 |
| [17] | Brophy B, Smolenski G, Wheeler T, et al. Cloned transgenic cattle produce milk with higher levels of beta-casein and kappa-casein[J]. Nature Biotechnology, 2003, 21 (2): 157–162. DOI:10.1038/nbt783 |
| [18] | Belhaj K, Chaparrogarcia A, Kamoun S, et al. Editing plant genomes with CRISPR/Cas9[J]. Current Opinion in Biotechnology, 2015, 32 : 76–84. DOI:10.1016/j.copbio.2014.11.007 |
| [19] | Baltes NJ, Voytas DF. Enabling plant synthetic biology through genome engineering[J]. Trends Biotechnol, 2015, 33 (2): 120–131. DOI:10.1016/j.tibtech.2014.11.008 |
| [20] | Lu JX, Xue YH, Ke PZ, et al. CRISPR-Cas9: a new and promising player in gene therapy[J]. J Medi Genet, 2015, 52 (5): 289. DOI:10.1136/jmedgenet-2014-102968 |
| [21] | Feng W, Dai Y, et al. The potential of the combination of CRISPR/Cas9 and pluripotent stem cells to provide human organs from Chimaeric Pigs[J]. Int J Mol Sci, 2015, 16 (3): 6545–6556. DOI:10.3390/ijms16036545 |
| [22] | Xie K, Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system[J]. Mol Plant, 2013, 6 (6): 1975–1983. DOI:10.1093/mp/sst119 |
| [23] | Doench JG, Hartenian E, Graham DB, et al. Rational design of hi-ghly active sgRNAs for CRISPR-Cas9-mediated geneinactivation[J]. Nat Biotechnol, 2014, 32 (12): 1262–1267. DOI:10.1038/nbt.3026 |
| [24] | Ma X, Zhang Q, Zhu Q, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant, 2015, 8 (8): 1274–1284. DOI:10.1016/j.molp.2015.04.007 |
| [25] | Nekrasov V, Staskawicz B, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonucl-ease. Nat Biotechnol[J]. . Nat Biotechnol, 2013, 31 (8): 691. DOI:10.1038/nbt.2655 |
| [26] | Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013, 31 (8): 688–691. DOI:10.1038/nbt.2654 |
| [27] | Nelson CE, Hakim CH, Ousterout DG, et al. In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy[J]. Science, 2015, 351 (6271): 403. |
| [28] | Latteyer S, Klein-Hitpass L, Khandanpour C, et al. A 6 bp in frame germline deletion in exon 7 of RET leads to increased RET phosphorylation, ERK activation and MEN2A[J]. Experimental & Clinical Endocrinology & Diabetes, 2015, 122 (3): 41–52. |






