我国各类盐碱地面积约0.99亿hm2, 盐渍化土壤面积约0.369亿hm2, 残余盐渍化土壤面积约0.448亿hm2, 潜在盐渍土化土壤为0.173亿hm2[1]。利用盐生植物改良修复盐碱土具有投资少, 见效快的特点[2]。但盐碱胁迫能造成植物体离子失衡和渗透胁迫, 研究植物耐盐机制, 筛选和培育抗盐植物品种, 提高植物本身的耐盐能力, 是改良和开发利用盐碱地经济而有效的方法[3]。土壤中过高浓度的Na+会造成植物的生理干旱, 扰乱细胞的离子平衡, 导致膜功能失调和代谢活动的减弱, 从而抑制生长并最终导致细胞死亡[4]。植物细胞抵御Na+毒害的主要策略有Na+外排和Na+区域化[5], 其中Na+外排过程中, 质膜Na+/H+逆向转运蛋白依靠质膜H+-ATPase形成的H+跨膜电化学势梯度为其提供驱动力, 将细胞质中过多的Na+外排, 从而减轻了Na+对细胞质内的各类代谢酶的伤害[6][7]。
SOS(Salt overly sensitive)信号转导途径是植物耐盐性相关的重要信号转导途径之一。目前, 在拟南芥(Arabidopsis thaliana)[6]、互花米草(Spartina alterniflora)[8]、胡杨(Populus euphratica)[9]、海滨锦葵(Kosteletzkya virginica)[10]、獐茅(Aeluropus littoralis)[11]、大叶补血草(Limonium gmelinii)[12]、甘蔗(Saccharum hybrid)[13]等植物上已成功克隆到SOS1基因, 并进行了耐盐功能分析。在拟南芥中, AtSOS1基因编码一个质膜Na+/H+逆向转运蛋白, C-末端为一长亲水性尾链, 位于胞质中, 作为感受器感知胞质中Na+浓度变化, 引发胞质Ca2+信号的产生, 随后Ca2+信号刺激SOS3-SOS2复合物激活SOS1, 将Na+排出胞外, 从而提高植物的耐盐性[14-16]。拟南芥SOS1突变体对盐非常敏感, 过表达SOS1基因能提高其耐盐性, 盐胁迫下积累更少的Na+[17], 表明SOS1基因在拟南芥耐盐性中发挥着重要作用。Yue等[18]在烟草(Nicotiana tabacum)中过表达拟南芥SOS1基因发现可提高耐盐性, 且转基因植株保持较高的K+/Na+。权庚等[8]研究发现过表达互花米草SaSOS1基因的水稻(Oryza sativa)对盐胁迫具有较强的适应性, 表明转SaSOS1基因能提高水稻耐盐性。Wu等[9]在胡杨中通过设计简并引物RACE技术克隆得到PeSOS1基因, PeSOS1在叶片中的表达明显受高盐胁迫(200 mmol/L NaCl)诱导, 并定位在细胞膜。但国内外尚未见黄花草木樨(Melilotus officinalis(L.)Lam.)MoSOS1基因的克隆和表达模式分析的报道。黄花草木樨为豆科草本植物具有抗旱、耐寒、耐盐碱、耐土壤贫瘠等的特性[19], 适应性较广, 防风挡沙, 保持水土[20, 21], 还是一种优良牧草和优质绿肥[22, 23]。本实验采用RT-PCR及RACE方法克隆黄花草木樨MoSOS1基因, 并分析其结构特征与表达模式, 为深入研究MoSOS1的功能特征奠定科学基础。
1 材料与方法 1.1 材料 1.1.1 实验材料黄花草木樨种子由中国农业科学院北京畜牧兽医研究所提供。
1.1.2 主要试剂TaKaRa MiniBEST Plant RNA提取试剂盒、Prime Script RTase第一链cDNA合成试剂盒、克隆载体pMD19-TVector、DNA marker、TaKaRa LA Taq试剂盒、大肠杆菌菌株Escherichia coli DH5α等购自大连宝生物工程有限公司, 其它生化试剂均为进口或国产分析纯。
1.1.3 实验材料培养用5% NaClO溶液将草木樨种子消毒5 min后, 于培养皿中发芽。待幼苗种子萌发后移至水培盒上, 于1×Hoagland营养液中生长, 间隔7 d更换一次Hoagland营养液, 光照周期为白天16 h/夜晚8 h, 昼夜温度分别为25℃和18℃, 光照强度为600 μmol·m-2·s-1空气相对湿度为60%左右。在人工气候箱中培育至6周龄后进行200 mmol/L NaCl盐胁迫处理。
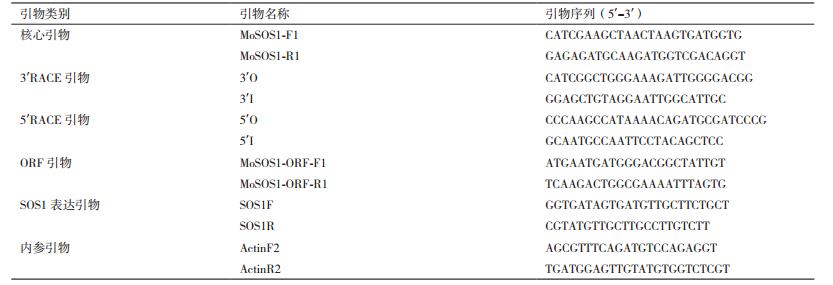
1.1.4 引物设计和合成使用Primer 5.0软件设计核心引物, DNAMAN 6.0软件进行3'及5'RACE引物设计、序列拼接和分析测序得到的基因序列, 引物由上海生工合成(表 1)。
取6周龄的黄花草木樨幼苗, 用200 mmol/L NaCl溶液胁迫48 h, 诱导相关耐盐基因的表达, 采集植物鲜叶, 经无菌水冲洗数次至无盐分残留, 消毒滤纸吸干植物叶片表面水分, 称取100 mg装于1.5 mL RNase-free管中, 用宝生物MiniBEST RNA植物RNA提取试剂盒提取植物总RNA, 利用PrimeScriptTM RTase第一链cDNA试剂盒合成cDNA(大连TaKaRa公司)。
MoSOS1完整编码区的cDNA扩增, 先比较Gen Bank数据库中拟南芥SOS1基因序列(NM_126259.4)、大豆SOS1基因(NM_001258010.1) 及绿豆SOS1基因序列(KC855193.1) 同源保守序列, 根据保守同源序列设计简并引物:MoSOS1-F1/MoSOS1-R1。PCR扩增出MoSOS1的保守核心片段。扩增的保守核心片段cDNA插入到pMD19-T载体(大连TaKaRA公司), 由北京擎科生物技术公司进行测序。MoSOS1完整编码区cDNA的获得采用cDNA末端快速扩增法(Rapid amplification of cDNA ends, RACE), PCR扩增得到的目的基因片段, 通过1.2%的琼脂糖凝胶电泳进行检测, 并用TaKaRa MiniBEST DNA Fragment Purification Kit Ver 4.0纯化试剂盒纯化回收DNA目的片段, 将回收产物连接到pMD19-T载体上后转化克隆并进行阳性菌株鉴定, 由北京擎科生物技术公司测序。
1.2.2 实时荧光定量RT-PCR采用实时荧光定量PCR方法, 分析不同盐浓度(0、50、100和200 mmol/L NaCl)处理48 h后黄花草木樨地上部和根中MoSOS1基因的表达模式。盐处理下黄花草木樨地上部和根中的总RNA提取参照宝生物TaKaRa MiniBEST Plant RNA试剂盒说明书进行。cDNA第一链的合成按照Prime ScriptTM RT reagent Kit with gDNA Eraser试剂盒说明书进行(宝生物工程有限公司)。黄花草木樨MoSOS1基因实时荧光定量RT-PCR引物SOS1F/SOS1R, PCR产物长度为168 bp, 其内参基因Actin实时荧光定量PCR引物ActinF2/ ActinR2, PCR产物长度为80 bp。
参照考宝生物SYBR® Premix Ex Taq Ⅱ试剂盒说明书的方法, 在Step One Plus仪器上进行实时荧光定量PCR实验。反应体系:cDNA 1 µL, SYBR® Premix Ex Taq Ⅱ 10 µL, ROX Reference Dye 0.4 µL, 正、反向引物分别为1 µL, 加水补至20 µL, 40个循环。采用2-△△CT方法计算黄花草木樨基因MoSOS1在不同盐浓度胁迫下的相对表达量, 设置3次重复。
1.2.3 生物信息学分析生物信息学分析方法如下。利用ORF finder在线软件, 对黄花草木樨MoSOS1基因进行开放阅读框分析并翻译;利用ExPASy中ProtParam pI/Mw(http://web.expasy.org/protparam/)在线工具对黄花草木樨MoSOS1基因所编码蛋白一级结构进行预测;利用PRABI(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_hnn.pl)进行黄花草木樨MoSOS1二级结构预测;利用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/Signal P/)进行黄花草木樨MoSOS1蛋白信号肽分析;用NCBI上Blast工具进行同源氨基酸序列查找, 并利用DNAMAN对所获得同源氨基酸序列以及鹰嘴豆SOS1、大豆SOS1等蛋白序列进行序列比对, 最后在MEGA6.0中采用NJ(Neighbor-Joining)法(Boot Strap 1000) 构建进化树。氨基酸序列跨膜预测在TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)网站上进行。利用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行MoSOS1保守区段预测。
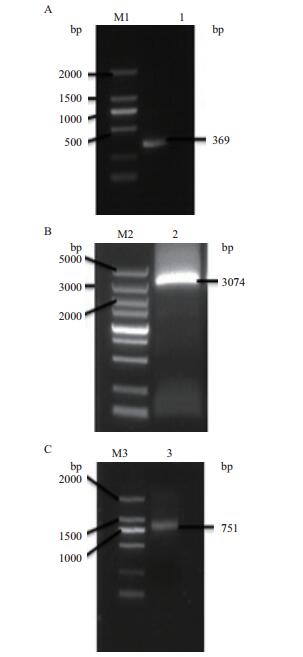
2 结果 2.1 黄花草木樨MoSOS1基因的克隆根据大豆(Glycine max)、绿豆(Vigna radiat)、拟南芥SOS1蛋白基因保守同源序列设计简并引物(MoSOS1-F1、MoSOS1-R1), 以黄花草木樨叶片总RNA反转录合成的cDNA为模板, PCR扩增出黄花草木樨MoSOS1保守核心cDNA片段, 经测序发现核心片段为369 bp(图 1-A)。用MoSOS1保守核心片段的已知序列, 设计引物进行3'RACE和5'RACE。3'RACE扩增产物经测序为3 074 bp(图 1-B), 5'RACE扩增产物经DNA测序为751 bp(图 1-C)。经DNAMAN软件序列拼接得到黄花草木樨MoSOS1的全长cDNA序列, 长度为3 931 bp(图 2)。根据MoSOS1拼接序列设计MoSOS1阅读框引物(MoSOS1-ORF-F1、MoSOS1-ORF-R1), PCR扩增出一条约3 000 bp的特异目的条带(图 3), 经克隆测序得到了黄花草木樨MoSOS1的开放阅读框(ORF)序列2 874 bp, 可编码957个氨基酸, 推测等电点为5.31, 分子量为112.8 kD。
|
| 图 1 MoSOS1核心区基因(A)、3' RACE(B)及5' RACE(C)PCR扩增 M1:DL2000 marker;M2:DL5000 marker;M3:DL2000 marker |

|
| 图 2 全长基因序列及其推导的编码氨基酸序列 黄色区域代表 3'及5'非编码区, 绿色区为起始密码子和终止密码子 |

|
| 图 3 MoSOS1 PCR扩增产物 |
通过在线工具ExPASy中ProtParam pI/Mw, 预测黄花草木樨MoSOS1基因编码蛋白质的一级结构显示, 该蛋白分子量为112.8 kD, 包含957个氨基酸残基。MoSOS1蛋白等电点(pI)为5.31, 有负电荷残基(Asp+Glu)102个, 正电荷残基(Arg+Lys)88个, 蛋白质三维结构不稳定系数(Ⅱ)为45.32, 平均疏水性(GRAVY)-0.000, 脂肪系数(AI)98.91。蛋白质不稳定系数大40, 蛋白质表现为不稳定状态。因此, 推测黄花草木樨MoSOS1基因编码的蛋白质为不稳定的酸性蛋白质。
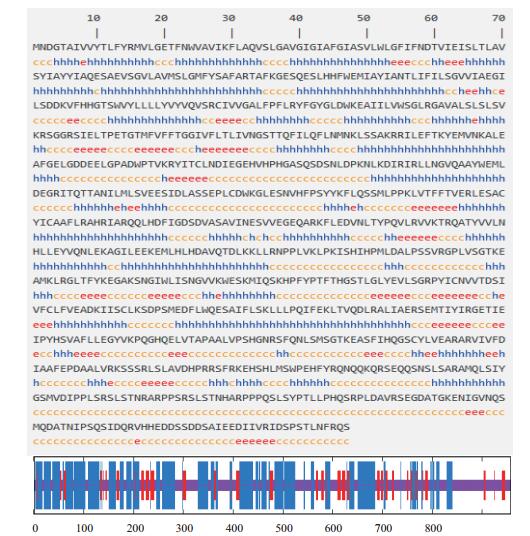
MoSOS1蛋白二级结构预测在SOPMA在线(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_hnn.pl)预测, MoSOS1蛋白质二级结构主要由α-螺旋和无规卷曲结构交错构成, 其中含α-螺旋43.68%, 延伸链12.96%, 无规卷曲43.36%(图 4)。
|
| 图 4 MoSOS1二级结构结构预测 ɑ螺旋(h)、延伸链(e)、转角(t)和无规则卷曲(c)分别由蓝、红、绿和黄色线条表示 |
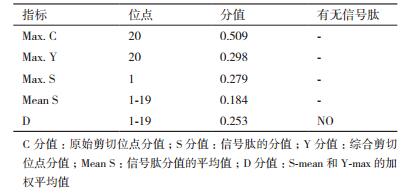
根据SignalP 4.1 Server软件预测可知, 第20位谷氨酸残基具有较高的原始剪切分值0.509和最高的综合剪切位点分值0.298, 第1位甲硫氨酸残基具有最高的信号肽分值0.279。由于最后获得氨基酸残基的加权平均值较小, 为0.253, 推测MoSOS1基因所编码的蛋白不存在信号肽, 即为非分泌型蛋白, 说明该蛋白在细胞质合成后不能被转运(表 2)。
NCBI保守结构预测表明, 黄花草木樨MoSOS1蛋白包含Na+/H+ Exchanger superfamily和一个cNMP(Cyclic nucleotide-monophosphate)结合位点及一个CAP_ED(Catabolite gene activator protein-effector domain)superfamily(图 5-A)。运用在线分析软件TMHMM Server 2.0(http://www.cbs.dtu.dk/service/TMHMM/)分析目的蛋白的跨膜结构;发现MoSOS1含有8个跨膜区域, N、C端位于细胞外。多重序列比较表明, MoSOS1跨膜区氨基酸序列位置分别为5-27, 34-56, 61-78, 85-104, 119-141, 153-175, 190-208, 228-250(图 5-B)。

|
| 图 5 黄花草木樨MoSOS1蛋白的保守结构(A)及跨膜结构(B)预测 |
将推测的MoSOS1氨基酸序列与其它植物SOS1的氨基酸序列进行比较发现, 它与鹰嘴豆(Cicer arietinum)、大豆(Glycine max)、绿豆(Vigna radiat)的同源性分别达到89%、84%和68%(图 6)。系统进化树分析表明, 黄花草木樨MoSOS1与鹰嘴豆和大豆亲缘关系最近, 而与山萮菜(Eutrema salsugineum)的亲缘关系较远(图 7)。由此表明MoSOS1是一类Na+/H+逆向转运蛋白, 与鹰嘴豆、大豆等的质膜SOS1具有相同功能。

|
| 图 6 黄花草木樨MoSOS1蛋白与其它植物蛋白的氨基酸序列多重比对 |

|
| 图 7 不同物种SOS1蛋白的系统进化分析 AsSOS1(FN356230.1, Aegilops speltoides拟斯卑尔脱山羊草);TtSOS1(EU552490.1, Triticum turgidum subsp. durum圆锥小麦);TaSOS1(FN356232.1, Triticum aestivum面包小麦);AtaSOS1(FN356231.1, Aegilops tauschii节节麦);LpSOS1(AY987046.1, Lolium perenne黑麦草);DsSOS1(GU480079.1, Distichlis spicata盐草);OsSOS1(AY785147.1, Oryza sativa水稻);McSOS1(EF207776.1, Mesembryanthemum crystalline冰叶日中花);SbSOS1(EU879059.1, Salicornia brachiat海蓬子);MoSOS1(Melilotus officinalis黄花草木樨);CaSOS1(XM_012716988.1, Cicer arietinum鹰嘴豆);GmSOS1(NM_001258010.1, Glycine max大豆);VrSOS1(KC855193.1, Vigna radiat绿豆);VvSOS1(NM_001281211.1, Vitis vinifera葡萄);CsSOS1(NM_001305732.1, Cucumis sativus黄瓜);NtSOS1(KC292267.1, Nitraria tangutorum番茄);KvSOS1(KJ577576.1, Kosteletzkya virginica海滨锦葵);AthSOS1(NM_126259.4, Arabidopsis thaliana拟南芥);EsSOS1(KF671959.1, Eutrema salsugineum山萮菜) |
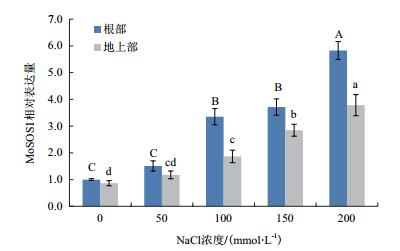
通过实时荧光定量PCR检测6周龄黄花草木樨幼苗盐胁迫48 h后MoSOS1基因的表达模式, 发现MoSOS1基因在根和地上部均有表达, 且表达量随着盐胁迫浓度的增加而有增加趋势。在100、150及200 mmol/L NaCl胁迫下, MoSOS1基因表达量要显著高于对照(P < 0.05), 且根部MoSOS1表达水平分别为地上部1.8、1.3和1.5倍(图 8)。由此表明MoSOS1的表达受盐胁迫诱导和调节, 且具有根部表达的特异性。
|
| 图 8 不同浓度NaCl处理下黄花草木樨地上部和根中MoSOS1基因的表达分析 不同大写字母和不同小写字母分别代表根部和地上部MoSOS1基因在不同盐胁迫下显著性差异(P < 0.05) |
本文以RT-PCR及RACE方法, 成功克隆到黄花草木樨SOS1转运蛋白基因的cDNA, 其编码957个氨基酸, 同源比对结果显示MoSOS1蛋白与鹰嘴豆Na+/H+逆向转运蛋白的同源性最高, 达89%, 与大豆SOS1和绿豆SOS1的同源性分别达84%和68%, 说明SOS1蛋白在不同物种间具有较高的保守性。拟南芥SOS1蛋白C末端是调节活性的区域, 高度亲水, 其结构含有多个蛋白激酶作用位点, 参与钙调素的结合以及多种信号的启动反应[24]。贾圆圆等[25]研究发现刚毛柽柳(Tamarix hispida)ThSOS1蛋白C端具有一个较长的亲水性尾巴, 能够使SOS1与逆境相关调控因子发生互作, 从而适应盐渍环境。本实验也发现黄花草木樨MoSOS1蛋白C末端含有一个长的亲水性尾巴, 推测其能够与逆境相关调控因子发生互作, 从而调控细胞内的Na+含量。
Shi等[6]研究发现拟南芥SOS1蛋白跨膜区与细菌、真菌质膜Na+/H+逆向转运蛋白相似性极高, SOS1突变体对盐胁迫敏感, 系统发育分析表明SOS1是一类质膜Na+/H+逆向转运蛋白。Shi等[17]经过酵母功能互补试验及SOS1-GFP定位证明拟南芥SOS1是质膜Na+/H+逆向转运蛋白。王澍等[26]成功克隆到黄瓜(Cucumis sativus)SOS1基因全长cDNA, 亚细胞定位表明该蛋白存在于细胞膜上, 酵母功能互补试验显示CsSOS1参与Na+/H+的转运。系统发育分析显示MoSOS1氨基酸序列与高等植物中已知序列质膜型Na+/H+逆向转运蛋白基因(SOS1)的同源性较高, 表明黄花草木樨MoSOS1是一类质膜Na+/H+逆向转运蛋白。
刘峰等[13]分析甘蔗SOS1蛋白二级结构发现该蛋白无规则卷曲所占比例最高, 为51.3%, 其次是α-螺旋占32.86%, 延伸链占15.84%, β-螺旋为0。郭庆水等[27]分析木榄(Bruguiera gymnorrhiza)质膜型Na+/H+逆向转运蛋白二级结构发现其α-螺旋占50.13%, 无规则卷曲所占30.70%, 延伸链占13.96%, β-螺旋为5.2%。本实验分析表明黄花草木樨MoSOS1蛋白质二级结构与甘蔗SOS1及木榄SOS1蛋白类似, 主要由α-螺旋和无规卷曲结构交错构成, 其中含α-螺旋43.68%, 无规卷曲43.36%, 延伸链12.96%, β-螺旋为0%。此外, 刘峰等[13]分析甘蔗ScSOS1蛋白包含CAP_ED(catabolite gene activator protein-effector domain)superfamily和一个Crp domain(cAMP-receptor proteins), 含有一个ligand结合位点和一个cNMP(Cyclic nucleotide-monophosphate)结合位点。王艳红等[10]分析海滨锦葵耐盐基因KvSOS1蛋白发现其N-末端具有NhaP、Na+/H+ Exchanger、b_cpa1等保守结构域, C -末端有cNMP、Crp等保守结构域, 表明该基因属于Na+/H+转运蛋白家族成员。NCBI保守结构预测表明MoSOS1蛋白包含Na+/H+ Exchanger superfamily, 一个cNMP(Cyclic nucleotide-monophosphate)结合位点以及一个CAP_ED(Catabolite gene activator protein-effector domain)superfamily, 说明MoSOS1与甘蔗ScSOS1和海滨锦葵KvSOS1蛋白具有相似结构, 属于Na+/H+转运蛋白家族成员, 参与细胞内Na+的调控转运。
程玉祥[28]研究星星草(Puccinellia tenuiflora)质膜型Na+/H+逆向转运蛋白(SOS1) 基因的克隆, 半定量RT-PCR分析表明PtSOS1基因表达明显受盐胁迫上调。郑琳琳等[29]研究发现盐胁迫能诱导唐古特白刺(Nitraria tangutorum)质膜Na+/H+逆向转运蛋白NtSOS1基因的表达。本实验发现黄花草木樨幼苗盐胁迫48 h后, MoSOS1基因在根和地上部均有表达, 且表达量随着盐胁迫浓度的增加而有增加趋势, 表明盐胁迫能上调MoSOS1基因的表达。Raquel等[30]研究发现马铃薯(Solanum tuberosum)SOS1蛋白不仅具有Na+/H+逆向转运功能, 而且能调节植物不同器官间Na+分布。Yang等[31]在拟南芥中过表达AtSOS1基因, 转基因植株能耐受200 mmol/L的NaCl处理, 且转基因植物木质部的Na+含量要明显低于非转基因植株, 说明SOS1蛋白在长距离的Na+运输中发挥作用。Shi等[17]在拟南芥中过表达AtSOS1基因, 转基因植株上调SOS1基因的表达, 且盐处理下, 过表达SOS1基因植株在木质部蒸腾流及地上部积累更少的Na+, 且转基因植株提高耐盐性是通过限制Na+在细胞内积累, 从木质部吸收Na+, 从而降低地上部Na+含量。Guo等[32]研究盐胁迫下野生型拟南芥和SOS突变体SOS1活性, 通过幼苗成像分析和电生理的研究, 表明SOS1具有在根部分生组织和茎维管束薄壁细胞表达的特异性, SOS1参与盐胁迫下Na+外排作用。本实验发现MoSOS1具有根部表达的特异性, 根部MoSOS1表达水平高于地上部。这可能是由于根部分生组织没有液泡, 盐胁迫下不能进行Na+区隔化[33], 根部组织只有通过上调MoSOS1基因的表达, 将进入根系的Na+及时排除体外, 从而维持植株根系较低的Na+含量;另一方面, 有利于将木质部蒸腾流中的Na+及时吸收以减少地上部Na+含量, 从而维持植株地上部较低的Na+含量。
4 结论黄花草木樨具有较强的抗旱耐盐性。本实验从黄花草木樨中成功克隆到MoSOS1基因, ORF 2 874 bp, 编码957个氨基酸。MoSOS1蛋白与鹰嘴豆Na+/H+逆向转运蛋白的同源性高达89%, 与大豆SOS1和绿豆SOS1的同源性分别达84%和68%。实验结果还表明在NaCl胁迫下, MoSOS1基因表达具有组织特异性, 根中表达量大于地上部, 在黄花草木樨耐盐分子机制中发挥作用。
| [1] | 李彬, 王志春, 孙志高, 等. 中国盐碱地资源与可持续利用研究[J]. 干旱地区农业研究, 2005, 23(2): 154–157. |
| [2] | 王苗, 齐树, 葛美丽. 盐生植物对滨海盐泽土生物改良的研究进展[J]. 安徽农业科学, 2008, 36(7): 2898–2899. |
| [3] | 张璐, 孙向阳, 尚成海, 田赞. 天津滨海地区盐碱地改良现状及展望[J]. 中国农学通报, 2010, 26(18): 180–185. |
| [4] | Zhu JK. Plant solt tolerance[J]. Trends in Plant Science, 2001, 6 (2): 66–71. DOI:10.1016/S1360-1385(00)01838-0 |
| [5] | Guo Q, Meng L, Mao PC, Tian XX. Role of silicon in alleviating salt-induced toxicity in white clover[J]. Bulletin of Environmental Contamination and Toxicology, 2013, 91 (2): 213–216. DOI:10.1007/s00128-013-1034-3 |
| [6] | Shi HZ, Ishitani M, Kim C, Zhu JK. The Arabidopsis thaliana salt tolerance gene sos1 encodes a putative Na+/H+ antiporter[J]. PNAS, 2000, 97 (12): 6896–6901. DOI:10.1073/pnas.120170197 |
| [7] | 张宏飞, 王锁民. 高等植物Na+吸收、转运及细胞内Na+稳态平衡研究进展[J]. 植物学通报, 2007, 24(5): 561–571. |
| [8] | 权庚, 张侠, 尹海波, 郭善利. 过量表SaSOS1水稻的幼苗鉴定及生理特性分析[J]. 河南农业科学, 2015, 44(3): 14–18. |
| [9] | Wu YX, Ding N, Zhao X, et al. Molecular characterization of PeSOS1: the putative Na+/H+ antiporter of Populus euphratica[J]. Plant Molecular Biology, 2007, 65 (1): 1–10. |
| [10] | 王艳红, 王鸿磊. 海滨锦葵耐盐基因KvSOS1的克隆与表达载体构建[J]. 江苏农业科学, 2016, 44(10): 76–80. |
| [11] | 王鹤, 张高华, 王旭达, 等. 獐茅耐盐基因SOS1的克隆及序列分析[J]. 中国农业大学学报, 2012, 17(3): 28–33. |
| [12] | 周玲玲, 祝建波, 曹连莆. 大叶补血草Na+/H+逆向转运蛋白基因(SOS1)的克隆与序列分析[J]. 园艺学报, 2009, 36(9): 1353–1358. |
| [13] | 刘峰, 苏炳华, 黄珑, 等. 甘蔗Na+/H+逆转运蛋白基因的克隆与表达分析[J]. 作物学报, 2016, 42(4): 501–512. DOI:10.7606/j.issn.1009-1041.2016.04.16 |
| [14] | Yamaguchi T, Apse MP, Shi HZ, Blumwald E. Topological analysis of a plant vacuolar Na+/H+ antiporter reveals a luminal C terminus that regulates antiporter cation selectivity[J]. PNAS, 2003, 100 (21): 12510–12515. DOI:10.1073/pnas.2034966100 |
| [15] | Zhu JK. Regulation of ion homeostasis under salt stress[J]. Current Opinion in Plant Biology, 2003, 6 (5): 441–445. DOI:10.1016/S1369-5266(03)00085-2 |
| [16] | Zhang JZ, Creelman RA, Zhu JK. Using information from Arabidopsis to engineer salt, cold, and drought tolerance in crops[J]. Plant Physiology, 2004, 135 (2): 615–621. DOI:10.1104/pp.104.040295 |
| [17] | Shi HZ, Lee BH, Wu SJ, Zhu JK. Overexpression of a plasma membrane Na+/H+ antiporter gene improves salt tolerance in Arabidopsis thaliana[J]. Nature Biotechnology, 2003, 21 : 81–85. |
| [18] | Yue YS, Zhang MC, Zhang JC, et al. SOS1 gene overexpression increased salt tolerance in transgenic tobacco by maintaining a hig-her K+/Na+ ratio[J]. Journal of Plant Physiology, 2012, 169 (3): 255–261. DOI:10.1016/j.jplph.2011.10.007 |
| [19] | 汪永平, 骆凯, 胡小文, 等. PEG和NaCl胁迫对草木樨种子萌发和幼苗生长的影响[J]. 草业科学, 2016, 33(6): 1174–1176. DOI:10.11829/j.issn.1001-0629.2015-0534 |
| [20] | 何冬梅. 白花草木樨的栽培技术[M]. 呼和浩特: 内蒙古畜牧业杂志社, 2005: 76-77. |
| [21] | 马丽. 浅谈草木樨的综合利用[M]. 乌鲁木齐: 新疆畜牧业, 2005: 56-57. |
| [22] | Van Riper LC, Larson DL. Role of invasive Melilotus officinalis in two native plant communities[J]. Plant Ecology, 2009, 200 (1): 129–139. DOI:10.1007/s11258-008-9438-6 |
| [23] | 景春梅, 刘慧, 席琳乔, 马春晖. 优质牧草、绿肥草木樨的研究进展[J]. 草业科学, 2014, 31(12): 2308–2315. DOI:10.11829/j.issn.1001-0629.2014-0084 |
| [24] | 马风勇, 石晓霞, 许兴, 等. 拟南芥SOS基因家族与植物耐盐性研究进展[J]. 中国农学通报, 2013, 29(21): 121–125. DOI:10.11924/j.issn.1000-6850.2012-3194 |
| [25] | 贾圆圆, 张春蕊, 王玉成, 等. 刚毛柽柳Na+/H+逆向转运蛋白基因的克隆与表达分析[J]. 植物研究, 2016, 36(3): 380–387. |
| [26] | 王澍, 任晴雯, 樊国盛. 黄瓜质膜型Na+/H+逆向转运蛋白基因(SOS1)的克隆与序列分析[J]. 上海交通大学学报:农业科学版, 2012, 30(3): 6–10. |
| [27] | 郭庆水, 于伟, 徐立新, 袁潜华. 木榄质膜型Na+/H+逆向转运蛋白的基因克隆与序列分析[J]. 热带作物学报, 2012, 33(10): 1800–1807. DOI:10.3969/j.issn.1000-2561.2012.10.016 |
| [28] | 程玉祥. 星星草质膜型Na+/H+逆向转运蛋白基因的克隆和特性分析[J]. 植物生理学通讯, 2008, 44(1): 59–64. |
| [29] | 郑琳琳, 张慧荣, 贺龙梅, 王迎春. 唐古特白刺质膜Na+/H+逆向转运蛋白基因的克隆与表达分析[J]. 草业学报, 2013, 22(4): 179–186. DOI:10.11686/cyxb20130422 |
| [30] | Olias, R Eljakaoui Z, Li J, et al. The plasma membrane Na+/H+ antiporter SOS1 is essential for salt tolerance in tomato and affects the partitioning of Na+ between plant organs[J]. Plant, Cell & Environment, 2009, 32 (7): 904–910. |
| [31] | Yang Q, Chen ZZ, Zhou XF, et al. Overexpression of SOS1(Salt Overly Sensitive)genes increases salt tolerance in transgenic Arabidopsis[J]. Molecular Plant, 2009, 2 (1): 22–31. DOI:10.1093/mp/ssn058 |
| [32] | Guo KM, Olga Babourina, Zed Rengel. Na+/H+ antiporter activity of the SOS1 gene: lifetime imaging analysis and electrophysiological studies on Arabidopsis seedlings[J]. Physiologia Plantarum, 2009, 137 (2): 155–158. DOI:10.1111/ppl.2009.137.issue-2 |
| [33] | Pardo J, Cubero B, Leidi E, Quintero F. Alkali cation exchangers: roles in cellular homeostasis and stress tolerance[J]. Journal of Experimental Botany, 2006, 57 (5): 1181–1199. DOI:10.1093/jxb/erj114 |