水稻是禾本科的模式作物, 也是我国主要粮食作物。随着耕地面积的减少、人口的增加、农村劳动力的缩减、农村城镇化的加剧, 对水稻总产量的需求越来越大, 而水稻理想株型改良对提高产量具有至关重要的作用。水稻理想株型主要由分蘖、株高、植株形态等农艺性状决定。植株形态特别注重叶片的直立性和叶片的适度卷曲。水稻叶片适度卷曲能保持叶片直立不披垂, 改善群体中后期基部的受光条件, 提高光能利用率, 从而有利于水稻产量的提高, 同时由于冠层基部光量的增加还能增强根系活力, 进而提高群体的抗倒性, 其重要作用已在超高产育种中得到充分体现[1, 2]。
水稻作为单子叶模式植物, 主要通过叶长、叶宽、披垂度和卷曲度等形态因子来界定叶片形态及空间伸展姿态。水稻叶片的细胞结构主要包括上下表皮细胞、叶肉细胞、薄壁细胞、泡状细胞、木质部、韧皮部和维管束等, 每一个组织都发挥着不可或缺的作用, 其中叶片表皮中的泡状细胞发育形态往往与叶片形态密切相关。泡状细胞是水稻的动力细胞[3]。在单子叶植物中, 泡状细胞是一类壁薄, 高度液泡化的大细胞, 禾本科植物叶片中的泡状细胞在两维管束之间成束出现[4]。Linsbsuer[5]研究认为泡状细胞主要起着水分储存的作用。然而更多的研究认为[1.2], 泡状细胞可能还具有另外一个重要的作用, 即通过其内含的水分得失控制叶片的伸展和卷曲运动, 从而达到影响叶片形态和光能作用的目的。
水稻叶片可以通过控制泡状细胞数量的多少、体积的大小以及泡状细胞中所含水分的多少来控制叶片的卷曲。一般情况下, 泡状细胞失水能够使叶片形成向近轴面方向的一个作用力, 从而使叶片发生正卷。泡状细胞吸水可以形成向远轴面方向的作用力, 又可以使卷叶慢慢平展。目前对控制泡状细胞的基因功能研究也有了一些进展。Hong等[6]报道BRD1基因在控制水稻泡状细胞个体数量方面起着重要作用。突变体brd1叶片的泡状细胞数量比野生型多, 但该突变体叶片并没有发生反卷, 因为该基因除了影响泡状细胞的数量外, 还影响叶片的其它细胞结构, 该突变体植株表现为矮化、叶片严重扭曲。Dai等[7]报道水稻YAB1基因不仅参与了GA生物合成的反馈调控, 而且在YAB1基因干涉株系中, 检测到泡状细胞数量增加且叶片卷曲, 对侧生器官的腹-背轴极性的建立却并没有产生影响, 这不同于在拟南芥中报道的所有的YABBY成员在侧生器官发育中决定腹-背轴的极性建立[8, 9], 进一步揭示YABBY家族基因在双子叶植物和单子叶植物在进化上的功能差异。Hibara等[10]报道ADL1基因, ADLl基因编码一个类钙调素半胱氨酸蛋白酶, 该突变体ADL1叶片背腹的表皮细胞形态改变, 形态类似泡状细胞, 导致叶片支持力减小, 造成了叶片的背腹卷曲, 该基因在控制泡状细胞的分化和发育、叶原基腹-背轴极性的正确建立方面起着关键作用。Li等[11]报道, 从T-DNA突变体库中克隆了ACL1基因, ACL1基因编码116个氨基酸, 在BY240(ACL1过表达突变体)中, 叶片的泡状细胞个数增加、体积变大, 叶片的表皮细胞也增大, 它的高度同源基因ACL2的过量表达也可使泡状细胞变大, 数目增多, 导致叶片外卷。Zhao等[12]报道控制叶夹角的基因LC2, 其编码VIN3样蛋白, LC2发生突变能够改变叶表皮细胞的分裂的数量, 叶片中泡状细胞的数量增加, 同时LC2在叶发育过程中受到脱落酸、赤霉素、生长素以及油菜素类脂的诱导表达。Xiang等[13]报道半显性卷叶基因SRL1, SRL1编码糖基磷脂酰肌醇锚定蛋白, 定位于质膜上。SRL1通过负向调节编码液泡H+-ATPase亚基和H+-焦磷酸酶基因的表达, 抑制泡状细胞形成, 进而调控叶片卷曲。上述基因主要是涉及泡状细胞的数量、体积、泡状细胞的分化及排列模式等。本研究主要目的是获取光合效率提高、农艺性状优异的突变体, 并研究其细胞学成因及克隆该基因, 旨为该基因的功能研究提供遗传原材料。
1 材料与方法 1.1 材料 1.1.1 水稻材料水稻卷叶突变体clf1来源于本实验室构建的T-DNA插入突变体库, 遗传背景为粳稻品种日本晴。经多代自交和选择, 是遗传稳定的材料。
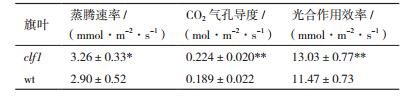
1.1.2 农艺性状统计及光合参数测定使用美国LI-COR LI-6400XT便携式光合仪对完全抽穗期的野生型和突变体的光合效率、蒸腾速率、CO2气孔导度进行测定。
1.2 方法 1.2.1 叶片的组织学石蜡切片分析在分蘖盛期分别取突变体clf1与日本晴的新鲜叶片, 分别用FAA固定液(70%酒精90 mL, 40%甲醛5 mL, 冰醋酸5 mL)固定, 乙醇脱水, 石蜡包埋。取8 μm的横切片, 用苏木精染色, 在蔡司荧光显微镜A1下观察、拍照, 观察其显微结构。
1.2.2 突变体clf1的遗传规律分析将突变体clf1与日本晴正反交, 统计其F1、F2代遗传分离比。同时将突变体clf1与籼稻品种Dular杂交, 收获其F1、F2代, 构建遗传定位群体, 用于基因的初定位和精细定位。
1.2.3 基因的初定位与精细定位根据F2代植株表型, 选取隐型表型单株10株, 分别剪取等量叶片, 采用改进过的CTAB法[13]提取隐性表型植株DNA。精细定位时用同样方法提取用于连锁分析的F2代单株DNA。本实验室所用Indel引物由北京擎科新业生物技术有限公司合成。利用网上所公布的粳稻亚种日本晴和籼稻品种9311的全基因组序列进行比对, 利用在籼稻粳稻之间片段大小差异设计引物, 引物设计软件为DNAMAN version4.0, 用设计好的引物在日本晴、Dular亲本之间检测多态性, 然后利用多态性引物对前述F2代隐性单株表型进行初定位, 再用与连锁的初定位标记对隐性F2代全部单株进行连锁分析, 确定引物与F2代隐性表型群体的连锁关系。反应在PCR扩增仪(Bio-Rad S1000 Thermal Cycler)中进行。反应产物在4%琼脂糖凝胶上电泳, 电泳电压180 V, 时间20 min。电泳完成后在胶片观察灯下观察结果并照相。将电泳图谱进行数值转换:与隐性亲本带型一致的记为1型带, 与显性亲本带型一致的记为2型带, 同时具有双亲本带型的的记为3型带。对获得的数据进行分析, 并进行遗传作图。
1.2.4 候选基因的克隆及分析通过生物信息学分析, 筛选定位区间内与叶片发育相关的基因, 设计引物并进行测序, 根据测序结果分析突变原因。
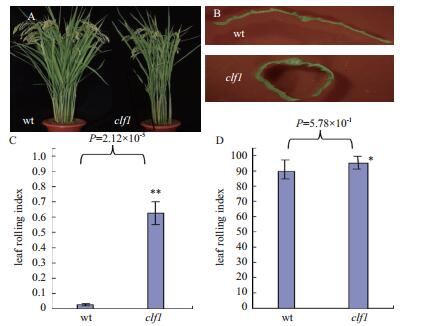
2 结果 2.1 突变体clf1的形态学特征突变体clf1在叶片生长至第10叶后, 叶片表现向远轴面卷曲, 随着植株的生长发育, 突变体叶片的卷曲趋势越来越显著, 至植株完全成熟时, 叶片呈半卷曲成筒状(图 1-B)。把叶片的横切面置于体视镜下观察, 可以清晰的看到野生型叶片平展, 而突变体叶片两侧明显外卷(图 1-B)。与此相对应的是, 突变体clf1的卷曲度(LRIs)在生长后期在0.57-0.73之间变动;而野生型在生长发育的整个过程中, 叶片都是表现为平展状态, LRIs一直都在0左右(图 1-C)。clf1叶片的直立度(LEIs)也比野生型的高, 并且这种趋势从第10叶一直保持到花期(图 1-D)。突变体clf1与野生型相比, 在株高、粒长、粒宽、千粒重等均无显著差异。适度卷曲能增加叶片的直立度, 突变体clf1叶片的直立显著地提高植株的光合效率(表 1)。
|
| 图 1 成熟期野生型与clf1的植株形态比较图 A:日本晴与clf1植株对比图;B:日本晴与clf1叶片横切面比较图;C.日本晴与clf1叶片卷曲度比较图;D:日本晴与clf1叶片直立度比较图 |
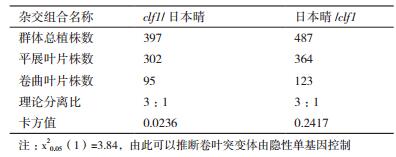
对clf1、与日本晴杂交的F1、F2代叶片卷曲性状进行调查统计。F1代叶片与野生型相似, 较平展, 表明卷叶相对于平展叶片为隐性性状。F2代, 叶片出现明显的分离, 平展叶与卷曲叶的分离比为3:1, 符合卡平方比(表 2)。由此可见, 控制突变体clf1的卷曲表型受一对单隐性核基因控制。
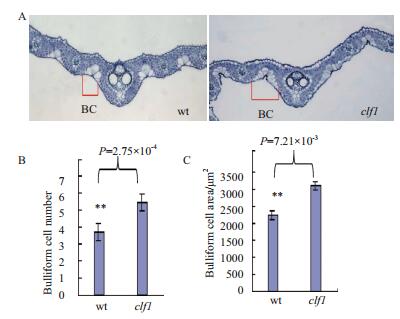
水稻叶片主要包括上下表皮、薄壁细胞、厚壁细胞、叶肉细胞、维管束等。两个维管束之间有泡状细胞, 叶片水分减少, 泡状细胞就失去膨压而收缩, 稻叶失水而卷曲。水稻叶片各组织的改变都可能引起叶片的卷曲。为了从细胞学结构上更深入地揭示该卷叶性状形成的原因, 我们对日本晴和突变体clf1的叶片进行石蜡切片分析, 以揭示其显微结构(图 2-A), 结果发现突变体clf1上表皮的泡状细胞数量和体积明显变大、变多。
|
| 图 2 野生型与clf1的石蜡切片显微结构比较图 A:日本晴(左侧)和clfl(右侧)显微结构对比图;B:日本晴与clf1泡状细胞数目统计图;C:日本晴与clf1泡状细胞体积统计图 |
为了对泡状细胞大小和面积进行定量, 利用image J软件定量分析日本晴和突变体clf1泡状细胞的面积和数量(图 2-B、C)。统计数据结果显示, 突变体clf1的泡状细胞面积要比野生型大13.6%, 突变体泡状细胞的数量平均比野生型多2-3个, 由于泡状细胞是储存水分的重要器官, 突变体clf1中泡状细胞数量变大和体积变大, 因此叶片水分含水量增加, 导致叶片的膨压增强而反卷。以上数据表明突变体clf1叶片卷曲是由于泡状细胞的体积和数量变化引起的。
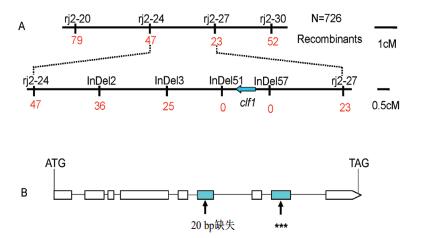
2.4 CLF1基因的初步定位和精细定位用均匀分布的589对Indel引物对亲本Dular和日本晴进行多态性分析, 有150对引物在Dular和日本晴之间有多态性。用150对引物对clf1/Dular F2代群体隐性表型植株进行分析, 发现10株突变体表型单株的DNA与rj2-24和rj2-27完全连锁, 取20株突变体表型单株以及10株野生型表型单株验证, 证明rj2-24和rj2-27与clf1位点连锁, 在rj2-24和rj2-27之间合成InDel引物, 其中有20对在亲本间具有多态性, 其中多态性较好的标记是InDel2、InDel3、InDel51、InDel57(表 3)等, 利用66个突变体表型单株将CLF1定位在InDel3与rj2-27之间, 进一步利用350株突变表型(隐性单株)群体和新开发的分子标记最终把CLF1基因定位在InDel51与InDel57之间, 物理距离约250 kb(图 3-A)。
|
| 图 3 CLF1基因在第2号染色体上的定位 A:CLF1的定位情况;B:CLF1基因结构图 |
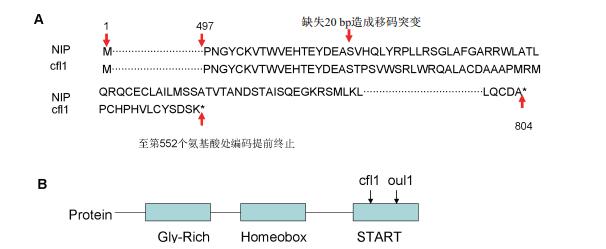
CLF1基因定位在InDel51与InDel57分子标记之间, 物理距离250 kb(图 3-A)。在250 kb中, 约44个基因, 通过生物信息学分析, 确定了LOC_OS02G45250为CLF1的候选基因。对LOC_OS02G45250基因进行测序, 在clf1突变体中, LOC_OS02G45250基因的第6外显子缺失20 bp(图 3-B), 造成编码产物至552处提前终止(图 4-A)。该基因与已报道的水稻卷叶基因Roc5(Rice outermost cell-specific gene5) 为等位基因, Roc5基因是由于T-DNA插入至第六内含子造成功能缺失导致, 其编码一个具有GL2类同源异型结构域的转录因子, clf1与oul1有相同结构域START的突变(图 4-B)。
|
| 图 4 CLF1氨基酸和蛋白功能结构域差异比较 A:突变体clf1和野生型之间CLF1氨基酸序列差异比较;B:来源于不同突变体的CLF1突变位点差异比较 |
表明了START结构域对LOC_OS02G45250基因的功能发挥具体重要作用。START结构域被确认是一种功能宽泛的脂类结合结构域, 可以参与防止蛋白酶水解, 有的参与脂质转运, 信号转导及转录调控等过程, 也能参与植物胁迫与病原菌反应, START结构域是如何调控泡状细胞的发育还需进一步研究。
3 讨论叶片的适度卷曲是重要的水稻遗传资源, 在水稻高产株型改良中具有重要的利用价值。但是到目前为止, 叶片的适度卷曲的克隆基因较少。主要原因有:叶片的适度卷曲在杂交群体F2或后代的表型易受环境因素影响, 经常会干扰了定位的结果, 如Roc5基因最早定位开始于1995年, 但是直到2011年才通过T-DNA的方法克隆, 表明了适度卷叶基因的定位的复杂性。到目前为止, 适度卷曲的基因均通过T-DNA方法克隆, 如OsAGO7和Roc5等, 表明T-DNA方法或其它方法比图位克隆法更适合适度卷曲类型突变体的基因克隆。本实验中, 由于适度卷叶基因的定位的复杂性, 采用单株定位的方法而非传统的混池定位法, 这样避免了混池法定位的不准确性。
经过复杂的图位克隆过程, 克隆了控制叶片卷曲的基因CLF1, 该蛋白与Roc5同, 但相应突变体是等位突变体。oul1是鉴定Roc5基因的完全失活突变体, 证明前期Roc5基因的正确性。本文中CLF1鉴定是用图位克隆获得, 尽管其来源于T-DNA插入突变体库, 但实际位点的突变是由于碱基缺失。因此, 大多数情况下, 插入缺失造成的indel可能比T-DNA插入造成的突变更多些。同时, 我们比较了突变体clf1与oul1(Roc5基因突变)的表型异同点。在2个突变体中, 叶片均向远轴面卷曲, 叶片上表皮的泡状细胞数量和大小均比野生型多。但是, 突变体oul1育性降低, 而突变体clf1育性好, 突变体clf1的实际穗粒数优于突变体oul1, 在生产上, 突变体clf1具有较大的生产应用潜力。由于CLF1基因具有较大的生产潜力, 是否可以在已测序3 000份水稻种质中获取优异的等位变异是下一步的重点。
在clf1突变体中, LOC_OS02G45250基因的第六外显子缺失20 bp, 造成编码产物提前终止, 突变位于基因的START结构域, 前期Roc5基因由T-DNA插入START结构域而导致失活, 表明了START结构域对LOC_OS02G45250基因的功能发挥具体重要作用。START结构域被确认是一种功能宽泛的脂类结合结构域, 可以参与防止蛋白酶水解, 有的参与脂质转运, 信号转导及转录调控等过程, 也能参与植物胁迫与病原菌反应, START结构域是如何调控泡状细胞的发挥还需进一步研究。
4 结论本研究通过筛选水稻T-DNA突变体库, 获得高光效突变体clf1, 并经过图位克隆, 克隆了该基因, 且位于ROC5基因。为进一步研究该基因的功能, 提供重要的遗传资料。
| [1] | Zhang GH, Xu Q, Zhu XD, et al. SHALLOT-LIKE1 is a KANADI transcription factor that modulates rice leaf rolling by regulating leaf abaxial cell development[J]. Plant Cell, 2009, 21 (3): 719. DOI:10.1105/tpc.108.061457 |
| [2] | Zou LP, Sun XH, Zhang ZG, et al. Leaf Rolling Controlled by the Homeodomain Leucine Zipper Class Ⅳ Gene Roc5 in Rice[J]. Plant Physiology, 2011, 156 (3): 1589. DOI:10.1104/pp.111.176016 |
| [3] | J. B. F. Physiological Plant Anatomy[J]. Philosophical Transactions of the Royal Society of London, 1914, 364 (1536): 3711–3735. |
| [4] | Jane W N, Chiang SHT. Morphology and development of bulliform cells in Arundo formosana Hack[J]. 国立台湾大学理学院植物学系研究报告, 1991, 36 (1): 85–97. |
| [5] | Linsbauer L. FIFTY YEARS OF PLANT PHYSIOLOGY IN AUSTRIA[J]. Plant Physiology, 1937, 12 (3): 565. DOI:10.1104/pp.12.3.565 |
| [6] | Hong Z, Ueguchi-Tanaka M, Shimizu-Sato S, et al. Loss-of-function of a rice brassinosteroid biosynthetic enzyme, C-6 oxidase, prevents the organized arrangement and polar elongation of cells in the leaves and stem[J]. Plant Journal, 2002, 32 (4): 495–508. DOI:10.1046/j.1365-313X.2002.01438.x |
| [7] | Dai M, Zhao Y, Ma Q, et al. The rice YABBY1 gene is involved in the feedback regulation of gibberellin metabolism[J]. Plant Physiology, 2007, 144 (1): 121. DOI:10.1104/pp.107.096586 |
| [8] | Bowman JL. The YABBY gene family and abaxial cell fate[J]. Current Opinion in Plant Biology, 2000, 3 (1): 17–22. DOI:10.1016/S1369-5266(99)00035-7 |
| [9] | Dai M, Zhao Y, Ma Q, et al. The rice YABBY1 gene is involved in the feedback regulation of gibberellin metabolism[J]. Plant Physiology, 2007, 144 (1): 121. DOI:10.1104/pp.107.096586 |
| [10] | Hibara K I, Obara M, Hayashida E, et al. The ADAXIALIZED LEAF1, gene functions in leaf and embryonic pattern formation in rice[J]. Developmental Biology, 2009, 334 (2): 345–354. DOI:10.1016/j.ydbio.2009.07.042 |
| [11] | Li L, Shi ZY, Li L, et al. Overexpression of ACL 1(abaxially curled leaf 1) increased bulliform cells and induced abaxial curling of leaf blades in rice[J]. Molecular Plant, 2010, 3 (5): 807–817. DOI:10.1093/mp/ssq022 |
| [12] | Zhao SQ, Hu J, Guo LB, et al. Rice leaf inclination2, a VIN3-1ike protein, regulates leaf angle through modulating cell division of the collar[J]. Cell Res, 2010, 20 (8): 935–947. DOI:10.1038/cr.2010.109 |
| [13] | Xiang JJ, Zhang GH, Qian Q, et al. SEMI-ROLLED LEAF1 encodes a putative glycosylphosphatidylinositol-anchored protein and modulates rice leaf rolling by regulating the formation of bulliform cells[J]. Plant Physiology, 2012, 159 (4): 1488–1500. DOI:10.1104/pp.112.199968 |