线粒体是一种含有线粒体DNA的半自主复制细胞器,能够编码构成呼吸链复合体的部分亚基。粟酒裂殖酵母线粒体DNA可以编码24种tRNA、8种mRNA、2种rRNA及1个rnpB[1]。线粒体通过对糖、脂肪、氨基酸等氧化(放能)和ADP磷酸化(储能)的偶联反应完成能量转换,达到合成ATP的目的,所形成的ATP直接为细胞生命活动提供95%以上的能量[2-4]。线粒体有自己的遗传体系,能够独立进行DNA的复制、转录及蛋白质的翻译,同时在线粒体的不同部位分布着大量的酶和辅酶,它们主要参与三羧酸循环、电子传递、氨基酸代谢、能量转换、脂肪酸分解、DNA复制和RNA合成过程[5, 6]。线粒体的机能与人类健康密切相关,线粒体功能异常可引起许多人类疾病,如神经退行性疾病,代谢综合征、糖尿病,癌症[7]、心脏病等。有关线粒体疾病在2012年发表于Nature和The LANCET的综述中有详细介绍[8, 9],有关线粒体功能的研究继续成为热点。
通过pombase数据库分析,粟酒裂殖酵母中Mef2(SPBC660.10)被预测是一个线粒体蛋白,并且可能参与线粒体的翻译过程。它具有两个同源蛋白,分别是芽殖酵母中的Mef2和人体中的EFG2。在芽殖酵母,人们发现Mef2是一个线粒体翻译延伸因子[10],它参与线粒体的代谢过程,与线粒体的有氧呼吸密切相关,并且对维持线粒体基因组很重要[11-13]。研究表明,在人体中EFG2既是一个线粒体翻译延伸因子[14],也是一个线粒体核糖体循环因子[15, 16]。EFG2基因缺失会诱发一系列由线粒体功能异常引起的疾病,如肌肉收缩异常等。过表达EFG2基因时,可以抑制由A3243G tRNALeu(UUR)MELAS引起的乳酸中毒和卒中样发作的线粒体脑病[17]。由此可见,EFG2与线粒体功能有着密切的联系。
虽然芽殖酵母中对mef2基因的研究已经较为成熟,但粟酒裂殖酵母中mef2基因还没有被报道,它的生物学功能尚不清楚。因此,本研究旨在探究Mef2蛋白在裂殖酵母中参与线粒体功能的机制,为研究粟酒裂殖酵母线粒体蛋白提供一定的理论依据。
1.1 材料与方法 1.1 材料 1.1.1 菌株与质粒粟酒裂殖酵母单倍体菌株yAS56、pFA6a-KanMX6、FA6a-KanMX6-GFP为本实验室保存。
1.1.2 培养基YES培养基(per 100 mL):3 g葡萄糖,0.5 g酵母粉,20 mg亮氨酸(L-leucine),20 mg尿嘧啶(Uracil),固体培养基加2.0 g的琼脂粉。YES+6%甘油:将YES培养基中的葡萄糖成分替换为6%的甘油。
YES+G418培养基:在YES培养基的基础上,补加KanMX浓度至100 mg/mL。KanMX是一种氨基苷类抗菌素衍生物,对真核细胞均有毒性作用,能阻断细胞内蛋白质合成。KanMX用在酵母体系中,可扩大筛选使用菌株的范围,便于提高外源基因的表达量。
1.1.3 仪器与试剂PrimeSTAR DNA Polymerase和蛋白marker购自TaKaRa公司;PCR引物由上海生工生物工程技术服务有限公司合成;SDS、琼脂粉、HEPES、Sorbitol、Tris、Mops、30%丙烯酰胺和EDTA购自索莱宝公司;鲑鱼精和氨苄霉素购自Sigma公司;尼龙膜、DIG标记及检测Kit Ⅱ购买于罗氏公司;甲醛、2×RNA Loading Buffer、DEPC水、DEPC为生工产品。
1.2 方法 1.2.1 生物信息学分析粟酒裂殖酵母mef2基因序列来源于酵母基因组数据(S.pombe_GeneDB,http://www.pombase.org/);粟酒裂殖酵母Mef2p及其人的同源蛋白EFG2的序列查找使用NCBI数据库(http://www.ncbi.nlm.nih.gov/),芽殖酵母同源蛋白Mef2的查找使用SGD数据库(http://www.yeastgenome.org/);利用NCBI中的blastp进行序列同源性比对;线粒体信号肽预测分析采用MitoProt Ⅱ(http://ihg.gsf.de/ihg/mitoprot.html)。
1.2.2 构建菌种通过融合PCR的方法构建Δmef2突变体:在mef2的编码区前后分别选择约300 bp碱基作为上下游同源臂,以野生型基因组为模板扩增上下游同源臂,再以pFA6a-KanMX6(携带一个抗G418的kanR基因)质粒为模板,扩增KanMX筛选标记片段,最后用三段融合的方法扩增上游同源臂+KanMX+下游同源臂片段。醋酸锂转化方法将三段融合片段导入yAS56菌株中,利用G418进行筛选。另外,实施融合PCR实验在mef2基因组上添加一个GFP标签,构建出Mef2-GFP菌株。
1.2.3 点圈实验将活化好的yAS56和Δmef2单菌落分别接种于10 mL的YES液体中,30℃培养过夜,作为种子液。将各个菌的种子液转接至新鲜YES液体培养基中至OD600为0.2。于30℃,220 r/min,培养12 h;收集菌液,调整初始OD600为3左右,以10倍差异进行梯度稀释,不同浓度各取3 μL点圈于YES和YES+6%甘油培养基上;在30℃恒温培养箱培养5 d,拍照。
1.2.4 GFP荧光定位把已活化的Mef2-GFP菌接入YES液体中,30℃过夜培养,转接至起始OD600约0.2,培养6-8 h后,OD600约0.8-1之间,取1 mL菌液,用PBS将细胞清洗一次,最终悬浮于200 μL PBS中,加入1 L Mitotracker Red染料,30℃,染色2 min,5 000×g离心1 min,弃上清,加入100 µL PBS重悬菌体,取3 µL悬浊液制成装片,用于荧光显微镜观察。制片后,利用Zeiss Axio imager A1 microscope(Zeiss,Jena,Germany)进行荧光观察。在63倍油镜下找到细胞,并在DIC通道拍照,然后转换显微镜至GFP通道(GFP滤光片为Catalog No.41017),激发波长为488 nm,GFP信号通过Chroma(Brattleboro,VT)获得,观察荧光信号,并拍照,最后转换显微镜至DsRed通道观察红色荧光信号,并拍照[18, 19]。相机系统为Sensicam QE cooled digital camera system(Cooke Corp);图像整合系统为MetaMorph/MetaFluor combination package analysis software(Uniwersal Imaging,West Chester,PA);图像后期处理使用Adobe Photoshop CS5(Adobe,San Jose,CA)。
1.2.5 Northern blotting将yAS56和Δmef2菌株落接入YES液体培养基中,收集对数期的菌液,采用热酚法抽提总RNA,将RNA混入乙醇中于-20℃保藏;取出适量的RNA进行处理,最后溶于DEPC水中,补充2×RNA Loading于65℃中孵育15 min,马上放置冰上冰浴10 min,待电泳。对于RNA > 500 bp(rps3、atp6、cox1、cox2、cox3、cob1、rns、rnl)进行1%的琼脂糖甲醛变性胶电泳,而 < 500 bp的RNA(atp9、atp8、rnpB)使用6%的尿素变性胶进行电泳。紫外交联完之后,在65℃中进行预杂交30 min,接着在42℃下杂交过夜,再用地高辛试剂盒进行检测,最后采用图像系统曝光5-20 min。线粒体基因探针采用带有DIG标记的随机引物进行PCR扩增或者引物5' 端直接用DIG标记进行修饰。
1.2.6 线粒体纯化和Western blotting抽提Δmef2和yAS56菌株的线粒体[20, 21],利用Western blotting进行线粒体相关蛋白质的检测。处理线粒体的粗提液:当检测Cox2、Atp6、Cox4和内参Hsp60时,将粗提液与蛋白质Loading混匀放入100℃中水浴10 min;当检测Cox1、Cob1和Cox3时,则将粗提液与蛋白质Loading混合物置于45℃中孵育3 min。通过Western blotting检测,用ODDESSY进行扫描显色。本研究选择线粒体基质的蛋白HSP60作为内参,检测线粒体相关蛋白Cox1、Cox2、Cox3、Cob1、Atp6及Cox4的表达量,抗体及稀释比例分别为Anti-HSP60(1:1 000),Anti-Atp6(1:1 000),Anti-Cox1(1:1 000),Anti-Cox2(1:500),Anti-Cox3(1:500),Anti-Cox4(1:1 000)和Anti-Cob1(1:500)。
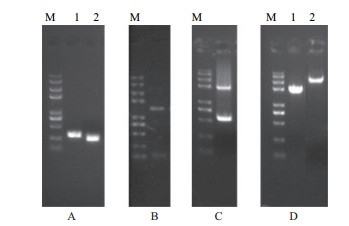
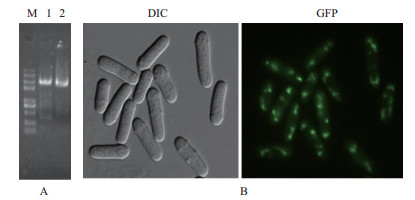
2 结果 2.1 Δmef2突变体和Mef2-GFP菌株的构建及鉴定选择mef2编码区前面300 bp作为上游同源臂,编码区后面250 bp作为下游同源臂,经PCR扩增的产物如图 1-A所示;以pFA6a-KanMX6质粒为模板扩增出KanMX标签的大小是1 357 bp(图 1-B);上游同源臂+KanMX+下游同源臂的融合片段的大小是1 907 bp(图 1-C)。将融合片段导入到yAS56菌株里,挑选转化子进行PCR验证,结果如图 1-D所示,Δmef2突变体扩增出的目的基因大小是1 907 bp,野生型菌株扩增出的目的基因大小是3 107 bp,这说明Δmef2突变体构建成功。另外,运用融合PCR技术构建上游同源臂+GFP+KanMX+下游同源臂片段(图 2-A),大小2 694 bp。挑取转化子用荧光显微镜进行检测,可以观察到绿色荧光(图 2-B),说明菌株Mef2-GFP构建成功。
|
| 图 1 Δmef2突变体的构建及验证 M:DNA分子量标准(100 bp -1 kb);A(1:上游同源臂的PCR扩增片段,2:下游同源臂的PCR扩增片段);B:筛选标记KanMX的PCR扩增片段;C:上游同源臂+KanMX+下游同源臂的融合片段;D(1:敲除菌的PCR验证产物,2:野生型的PCR验证产物) |
|
| 图 2 Mef2-GFP菌株的构建及验证 M:DNA分子量标准(100 bp -1 kb);A(1,2:Mef2-GFP中上游同源臂+GFP+KanMX+下游同源臂的融合PCR扩增片段;B:通过荧光显微镜观察鉴定菌株Mef2-GFP) |
为了研究Mef2在粟酒裂殖酵母中的生理功能,构建了Δmef2菌株。采用线粒体功能缺陷菌表型生长的普遍研究方,即用以甘油为唯一碳源的非发酵培养基来培养yAS56和Δmef2菌株。实验结果如图 3所示,与野生型菌株相比,在发酵型培养基上,mef2基因缺失突变株生长状态与野生型一样;而在非发酵型培养基上,Δmef2菌株生长趋势明显弱于野生型,表现出显著的呼吸受损现象。这个实验结果表明,Δmef2菌株是呼吸缺陷型菌株,mef2基因的缺失会导致粟酒裂殖酵母线粒体功能异常。

|
| 图 3 Δmef2在非发酵培养基上的生长情况 |
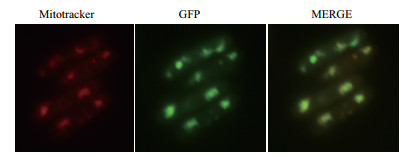
mef2基因的敲除会影响线粒体功能,这暗示着Mef2蛋白极有可能是进入线粒体中发挥功能。经过生物信息学分析发现Mef2的N端具有一段包含31个氨基酸的线粒体定位序列(MLRIVWKPLKIRLPVWRRYQSNISINSIRN),其可能性高达93.17%。因此,本研究采用线粒体染料示踪法来确定Mef2的定位。以Mitotracker Red染活细胞线粒体作为线粒体marker,在荧光显微镜下发红色荧光区域的就是线粒体;而Mef2-GFP菌株因为带有绿色荧光标签,则会在荧光显微镜下会呈现绿色荧光。如图 4所示,发现同一视野中绿色荧光和红色荧光可以重叠,展现出Mef2与线粒体具有共定位现象,这说明Mef2定位于线粒体中。
|
| 图 4 Mef2-GFP的荧光显微镜观察 Mitotracker:线粒体红色荧光探针染色后的观察结果;GFP:绿色荧光蛋白示踪结果;Merge:将Mitotracker Red染料和GFP观察结果融合 |
由前期实验结果可知,mef2缺失菌株在非发酵型培养基上生长明显受损,影响粟酒裂殖酵母线粒体功能的正常发挥。因此推测mef2的缺失对于这些线粒体编码蛋白的含量造成了影响,进而导致线粒体呼吸链功能的缺失。为了探究这种受损的原因,在实验室已有实验条件的前提下,我们从线粒体基因组编码蛋白的合成途径来进行研究。先利用Northern blotting实验检测线粒体基因组编码蛋白的转录水平。
在真核生物进行有氧呼吸是通过线粒体中的电子传递链和氧化磷酸化的过程来实现的,电子传递链中的4个复合体是由核基因组编码的蛋白和线粒体基因组编码的蛋白共同组成的,其中Cox1、Cox2和Cox3参与组成呼吸链复合体Ⅳ,Cob1则参与组成呼吸链复合体Ⅲ,Atp6参与ATPase的合成,它们都是线粒体自身编码的蛋白。
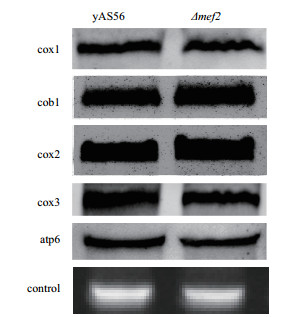
通过Northern blotting实验来检测cox1、cox2、cox3、cob1和atp6的mRNA水平。实验结果(图 5)显示,Δmef2菌株中cox1、cox2、cox3、cob1和atp6的mRNA水平和野生型yAS56相比,都没有出现显著性差异。这个结果说明Mef2不影响cox1、cox2、cox3、cob1和atp6 等的mRNA水平。
|
| 图 5 Northern blotting检测Δmef2菌中线粒体基因组编码的mRNAs水平 |
Northern blotting实验表明mef2的缺失不会影响cox1、cox2、cox3、cob1和atp6 等的mRNA水平,说明Mef2可能与线粒体基因组编码的蛋白翻译有关。因此本研究利用Western blotting技术来分析线粒体基因组编码的Cox1、Cox2、Cox3、Atp6和Cob1的表达量,进一步探究引起Δmef2菌株出现呼吸缺陷型生长表型原因。在本实验中用Cox4作为对照以及用Hsp60作为上样量控制,其中Cox4是一个由核基因组编码的线粒体呼吸链相关蛋白。
与野生型菌株yAS56相比,Δmef2菌株中Cox3、Atp6和Cob1蛋白在线粒体中的表达量显著降低,Cox1蛋白的表达量也出现一定程度的降低(图 6),这说明mef2基因的缺失会导致线粒体呼吸链相关蛋白表达量出现不同程度的降低。线粒体呼吸链上的复合体不能正常组装,细胞不能正常进行氧化磷酸化及利用非发酵型碳源,导致了粟酒裂殖酵母细胞在非发酵碳源培养基上呈现出呼吸缺陷型生长状态。因此,mef2基因对线粒体呼吸链相关蛋白Cox3、Atp6和Cob1的表达非常重要,它的缺失会阻碍线粒体呼吸链复合物的合成,导致线粒体呼吸链无法发挥其正常功能。

|
| 图 6 Western blotting检测Δmef2菌中线粒体基因组编码的蛋白质表达量 |
在芽殖酵母中Δmef2菌在以甘油为唯一碳源的非发酵培养基上也会表现出呼吸缺陷型生长;经研究发现芽殖酵母中mef2基因对维持线粒体DNA有着非常重要的作用[14],另外,芽殖酵母中Mef2蛋白不仅参与线粒体编码蛋白质翻译中的延伸过程[22],还涉及翻译中核糖体的循环利用过程。而在本研究中发现粟酒裂殖酵母中mef2基因的缺失也会在非发酵型培养基引起生长缓慢。这说明mef2基因与线粒体功能存在密切的联系。从生物信息学分析中可以得出:粟酒裂殖酵母中Mef2与芽殖酵母中的同源蛋白Mef2序列相似性大约是35%,与在人体中的同源蛋白EFG2的同源性达到41%。所以这3个蛋白存在着一定的同源性。因此,猜测粟酒裂殖酵母中的Mef2和其在芽殖酵母中的同源蛋白Mef2具有类似的功能。实验结果已经证明,粟酒裂殖酵母中mef2基因的缺失不影响线粒体相关蛋白质的mRNA水平,却引起这类蛋白质的表达量锐减,证明它的确是参与线粒体相关蛋白质的翻译过程。但是蛋白质的翻译过程要经过氨基酸的活化、肽链的起始、肽链的延伸、肽链的终止以及多肽链的折叠或加工。经过NCBI Blast中的蛋白质分析结果中发现粟酒裂殖酵母中的Mef2存在推定的保守结构域,如翻译延伸因子G结构域、翻译延伸因子EFTu/EF1A结构域和小GTP结合蛋白结构域。这些保守结构域都表明Mef2很有可能参与蛋白质翻译过程中肽链的延伸。而粟酒裂殖酵母中的Mef2是否也参与肽链的延伸还无法确定,需要后续用更多的实验去验证。
蛋白质的定位往往会与其功能相关。在芽殖酵母中Mef2已经被证实定位于线粒体内膜上,虽然本研究已经证实粟酒裂殖酵母中的Mef2定位于线粒体中,但是它在线粒体中的具体定位还不清楚,所以接下来可以设计更多的实验来研究其具体定位,可以帮助我们更多的了解Mef2p在线粒体中所发挥的功能。
4 结论本研究通过同源重组的方式敲除mef2获得Δmef2突变体,发现Δmef2突变体是一种呼吸缺陷型菌。利用GFP荧光定位实验证明了Mef2定位于线粒体中。运用Northern blotting技术得出mef2的缺失不影响cox1、cox2、cox3、cob1和atp6等的mRNA水平。Western blotting实验结果说明mef2的缺失会导致线粒体基因组编码的Cox1、Cox3、Atp6和Cob1蛋白表达量下降。综上所述,这些结果都阐明Mef2参与线粒体基因组编码的Cox3、Cox1、Atp6和Cob1的翻译。
| [1] | Kühl I, Dujeancourt L, Gaisne M, et al. A genome wide study in fission yeast reveals nine PPR proteins that regulate mitochondrial gene expression[J]. Nucleic Acids Research, 2011, 39 (18): 8029. DOI:10.1093/nar/gkr511 |
| [2] | Lemaire C, Dujardin G. Preparation of respiratory chain complexes from Saccharomyces cerevisiae wild-type and mutant mitochondria[M]. Humana Press: 2008: 65-81. |
| [3] | Højlund K, Wrzesinski K, Larsen PM, et al. Proteome analysis reveals phosphorylation of ATP synthase beta-subunit in human skeletal muscle and proteins with potential roles in type 2 diabetes[J]. Journal of Biological Chemistry, 2003, 278 (12): 10436–10442. DOI:10.1074/jbc.M212881200 |
| [4] | Moss DA, Bendall DS. Cyclic electron transport in chloroplasts. The Q-cycle and the site of action of antimycin[J]. Biochimica et Biophysica Acta(BBA)-Bioenergetics, 1984, 767 (3): 389–395. DOI:10.1016/0005-2728(84)90036-7 |
| [5] | Sullivan LB, GUI DY, Hosios AM, et al. Supporting aspartate biosynthesis is an essential function of respiration in proliferating cells[J]. Cell, 2015, 162 (3): 552–63. DOI:10.1016/j.cell.2015.07.017 |
| [6] | Zhao X, León IR, Bak S, et al. Phosphoproteome analysis of functional mitochondria isolated from resting human muscle reveals extensive phosphorylation of inner membrane protein complexes and enzymes[J]. Molecular & Cellular Proteomics, 2011, 10 (1): M110. 000299. |
| [7] | 马丽晶, 徐勉. 线粒体基因突变所致糖尿病发病机制及治疗进展[J]. 医学综述, 2010, 16(2): 275–277. |
| [8] | Scipioni L, Thompson W, Sijbrandij S, et al. An essential role of the mitochondrial electron transport chain in cell proliferation is to enable aspartate synthesis[J]. Cell, 2015, 162 (3): 540–551. DOI:10.1016/j.cell.2015.07.016 |
| [9] | Neufeldcohen A, Robles MS, Aviram R, et al. Circadian control of oscillations in mitochondrial rate-limiting enzymes and nutrient utilization by PERIOD proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113 (12): 201519650. |
| [10] | Kai SD, Fritz S, Fuchs F, et al. Genetic basis of mitochondrial function and morphology in Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2002, 13 (3): 847–853. DOI:10.1091/mbc.01-12-0588 |
| [11] | Maheshwari KK, Marzuki S. Defective assembly of the mitochondrial ribosomes in yeast cells grown in the presence of mitochondrial protein synthesis inhibitors[J]. Biochimica et Biophysica Acta, 1985, 824 (4): 273–283. DOI:10.1016/0167-4781(85)90033-8 |
| [12] | Merz S, Westermann B. Genome-wide deletion mutant analysis reveals genes required for respiratory growth, mitochondrial genome maintenance and mitochondrial protein synthesis in Saccharomyces cerevisiae[J]. Genome Biology, 2009, 10 (9): 1–18. |
| [13] | Rasmussen SW. A 37. 5 kb region of yeast chromosome X includes the SME1, MEF2, GSH1 and CSD3 genes, a TCP-1-related gene, an open reading frame similar to the DAL80 gene, and a tRNA(Arg)[J]. Yeast, 1995, 11 (9): 873–883. DOI:10.1002/(ISSN)1097-0061 |
| [14] | Hammarsund M, Wilson W, Corcoran M, et al. Identification and characterization of two novel human mitochondrial elongation factor genes, hEFG2, and hEFG1, phylogenetically conserved through evolution[J]. Human Genetics, 2001, 109 (5): 542–550. DOI:10.1007/s00439-001-0610-5 |
| [15] | Tsuboi M, Morita H, Nozaki Y, et al. EF-G2mt is an exclusive recycling factor in mammalian mitochondrial protein synthesis[J]. Molecular Cell, 2010, 82 (9): 825–31. |
| [16] | Valente L, Tiranti V, Marsano RM, et al. Infantile encephalopathy and defective mitochondrial DNA translation in patients with mutations of mitochondrial elongation factors EFG1 and EFTu[J]. American Journal of Human Genetics, 2007, 80 (1): 44. DOI:10.1086/510559 |
| [17] | Sasarman F, Antonicka H, Shoubridge EA. The A3243G tRNALeu(UUR)MELAS mutation causes amino acid misincorporation and a combined respiratory chain assembly defect partially suppressed by overexpression of EFTu and EFG2[J]. Human Molecular Genetics, 2008, 17 (23): 3697–3707. DOI:10.1093/hmg/ddn265 |
| [18] | Greenberg RA, Sobhian B, Pathania S, et al. Multifactorial contributions to an acute DNA damage response by BRCA1/BARD1-containing complexes[J]. Genes & Development, 2006, 20 (1): 34. |
| [19] | Chalfie M, Tu Y, Euskirchen G, et al. Green fluorescent protein as a marker for gene expression[J]. Science, 1994, 263 (5148): 802. DOI:10.1126/science.8303295 |
| [20] | Christian B, Haque E, Spremulli L. Ribosome shifting or splitting: It is all up to the EF-G[J]. Molecular Cell, 2009, 35 (4): 400–402. DOI:10.1016/j.molcel.2009.08.003 |
| [21] | Maheshwari KK, Marzuki S. Defective assembly of the mitochondrial ribosomes in yeast cells grown in the presence of mitochondrial protein synthesis inhibitors[J]. Biochimica et Biophysica Acta, 1985, 824 (4): 273–283. DOI:10.1016/0167-4781(85)90033-8 |
| [22] | Callegari S, Gregory PA, Sykes MJ, et al. Polymorphisms in the mitochondrial ribosome recycling factor EF-G2mt/MEF2 compromise cell respiratory function and increase atorvastatin toxicity[J]. PLoS Genetics, 2011, 8 (6): e1002755. |




