2. 青藏高原动物遗传资源保护与利用四川省重点实验室,成都 610041
2. Key Laboratory of Sichuan Province for Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Exploitation, Chengdu 610041
Krüppel样转录因子(Krüppel-like factors,KLF)是一类转录调节蛋白,其广泛参与细胞增殖、分化、凋亡以及胚胎发育等一系列生理过程的调控。目前,哺乳动物KLFs家族已有17个成员被确认,分别是KLF1-KLF17[1, 2]。KLF3作为KLFs家族重要成员之一,因其在N端具有较多的碱性氨基酸,又被称为碱性KLF因子(Basic krüppel-like factor,BKLF),目前已证实KLF3在脂肪生成[3]、B细胞发育[4]、红细胞生成[5]和心肌细胞分化[6]等过程中发挥重要调控作用。
研究发现KLF3基因敲除小鼠相对于正常小鼠具有较少的白色脂肪组织,且脂肪垫的细胞数量更少,体积更小[3]。3T3-L1前体脂肪细胞系研究显示,脂肪细胞分化过程中KLF3 mRNA和蛋白的表达水平伴随脂肪细胞分化过程呈下降的趋势,在KLF3基因过表达情况下发现3T3-L1前体脂肪细胞分化被抑制,且进一步的研究指出KLF3可通过结合C/EBPα的启动子来抑制脂肪细胞分化。Zhang等[7, 8]研究表明KLF3可能通过促进脂肪酸β-氧化来调节脂质代谢,还可通过克氏病影响动物正常生殖能力及行为,同时KLF3在调控脂蛋白组装和分泌过程中也扮演重要角色。此外,KLF3还与其他KLFs相互协调在脂肪生成中发挥重要作用,在3T3-L1脂肪细胞分化过程中,KLF3基因的表达水平随KLF2表达水平的下降而降低,KLF3与KLF2都可抑制C/EBPα的表达[9],说明两者可能构成一个协同调控网络。同时也有研究指出KLF1、KLF3和KLF8也可共同组成一个调控网络来发挥作用[10],但这个调控网络是否影响脂肪细胞分化尚还不清楚。
目前,国内外鲜有对高原动物KLF3基因的实验研究,而牦牛KLF3基因的研究尚属空白。因此本实验利用RT-PCR方法克隆获得牦牛KLF3基因序列,并进行序列分析,利用荧光定量PCR技术检测该基因在不同组织中的表达特性,旨在阐明牦牛KLF3基因的结构和功能,为后续研究牦牛KLF3基因的作用提供参考。
1 材料与方法 1.1 材料 1.1.1 实验动物与组织的采集选取四川省阿坝藏族羌族自治州小金县的4-6岁健康的麦洼公牦牛6头,清晨空腹屠宰后,迅速采集其脾脏、肺脏、肾脏、皮下脂肪、背最长肌等组织,用DEPC水清洗后,剪切成2 g左右的组织块,置于无RNA酶的细胞冻存管中,于液氮中冻存后带回实验室备用。
1.1.2 主要试剂Trizol试剂、SYBR®Premix Ex TaqTM(2×)与pMD-19T载体购自大连TaKaRa公司,2×Long Taq PCR Master Mix、感受态细胞DH5α和DNA纯化回收试剂盒(Universal DNA Purification Kit)均购自天根生化科技有限公司,RevertAid First Strand cDNA Synthesis Kit购自Thermo公司。苯酚、氯仿、异丙醇、酒精等其他试剂均为国产生化试剂。
1.2 方法 1.2.1 牦牛KLF3基因克隆按照Trizol试剂盒说明书提取麦洼牦牛皮下脂肪组织的总RNA,并用1%的琼脂糖凝胶电泳检测RNA完整性。利用紫外分光光度计检测总RNA浓度和质量,利用RevertAid First Strand cDNA Synthesis Kit合成cDNA第一链,用1%的琼脂糖凝胶电泳检测cDNA的质量。根据GenBank上牛KLF3基因序列(XM_005207873),利用Primer Premier 5.0软件设计PCR克隆引物(表 1)。PCR反应总体系为:2×Long Taq PCR Master Mix 12.5 μL,模板cDNA 1 μL,10 μmol/L上、下游引物各1 μL,最后加ddH2O至25 μL。扩增条件:预变性(94℃ 4 min),变性(94℃ 30 s),退火(56℃ 30 s),延伸(72℃ 90 s),38个循环,延伸(72℃ 10 min)。然后1%琼脂糖凝胶电泳检测扩增产物,胶回收试剂盒纯化目的片段,再将产物与pMD-19T载体连接,并转化至感受态细胞DH5α中,Amp+平板挑取阳性菌落,经菌液PCR鉴定后送成都擎科梓熙生物技术有限公司测序。
利用NCBI工具ORF Finder对获得的cDNA序列进行开放阅读框预测,并翻译为氨基酸序列;ExPASy ProtParam分析蛋白理化性质(http://web.expasy.org/protparam/);NetPhos 3.1、NetOGlyc 1.0和NetNGlyc 3.1分析预测磷酸化位点、O糖基化位点、N糖基化位点;利用SignalP 4.1 Server进行信号肽分析(http://www.cbs.dtu.dk/services/SignalP/),TMHMM预测跨膜结构域(http://www.cbs.dtu.dk /services/TMHMM/),PSORT Ⅱ进行亚细胞定位(http://www.genscript.com/psort.html);利用NCBI的Conserved Domain程序预测结构域(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi);STRING交互式数据库进行蛋白相互作用分析(http://string-db.org/);利用Hopfield预测蛋白质二级结构(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html),PHYRE2和Raswin软件对蛋白三级结构进行预测比对(http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id =index);Protfun 2.2预测分析生物学功能(http://www.cbs.dtu. dk/services/ProtFun/);NCBI中Blast进行同源性分析,clustalx1.83和MEGA5.0构建进化树。
1.2.3 牦牛KLF3基因组织表达差异分析利用Primer Premier 5.0软件,根据获得的麦洼牦牛KLF3序列和牦牛GAPDH基因序列(XM_014482068)设计PCR特异引物(表 1),采用荧光定量PCR检测KLF3基因在各组织中的表达差异。PCR反应体系为20 μL:10 μmol/L上下游引物各1 μL,模板cDNA 1 μL,SYBR® Premix Ex Taq TM(2×)PCR 10 μL,ddH2O 7 μL。PCR反应条件:预变性(95℃ 3 min),变性(95℃ 10 s),退火(60℃10 s),延伸(72℃ 15 s),38个循环。每个待测样本设3个重复,并设置3个无cDNA模板的样本为阴性对照,用2-ΔΔCt对实时定量PCR数据进行均一化处理,利用SPSS 18.0软件进行方差分析,采用Duncan法对各组织间mRNA表达进行多重比较。
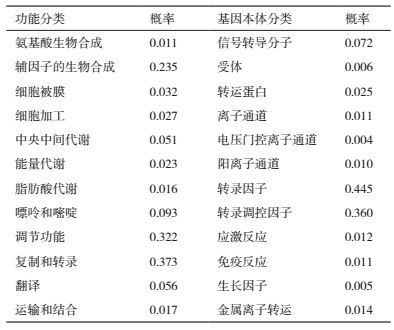
2 结果 2.1 牦牛KLF3基因克隆与理化性质分析以麦洼牦牛皮下脂肪组织cDNA为模板经PCR扩增后获得KLF3基因序列全长1 137 bp,其中包含CDS区1 041 bp,5' UTR序列32 bp和3' UTR序列64 bp,其终止密码子为TGA,编码346个氨基酸残基(图 1)。提交GenBank获得登录号为:KX964630。
|
| 图 1 牦牛与普通牛、山羊、人、小鼠KLF3氨基酸序列同源性比对 黑色部分表示同源性为100%;粉色部分表示同源性大于75%;蓝色部分表示同源性大于50%;黄色部分表示大于33%;白色部分表示同源性低于33%;•表示氨基酸的缺失;方框表示锌指结构域 |
ExPASy在线工具对牦牛KLF3氨基酸序列的理化性质进行分析,结果显示,该蛋白分子式为C1724H2714N484O501S22,分子质量为38.942 kD,理论等电点为9.44,不稳定指数为86.84,亲水性总平均值为-0.654,说明为不稳定亲水碱性蛋白。氨基酸组成结果(图 1)显示脯氨酸(P)所占的比例最高为13.6%(47个氨基酸),同时带负电荷的氨基酸残基(Asp+Glu)总数为30,带正电荷的氨基酸残基(Arg+Lys)总数为43。通过软件比对发现麦洼牦牛KLF3与普通牛(XP_010820342.1)的核苷酸同源性为99.82%,氨基酸同源性为100%,共检测到2个碱基突变,分别导致密码子ACG→ACA、GGG→GGC,但均属同义突变,牦牛KLF3的氨基酸序列与普通牛相比并没有发生改变。
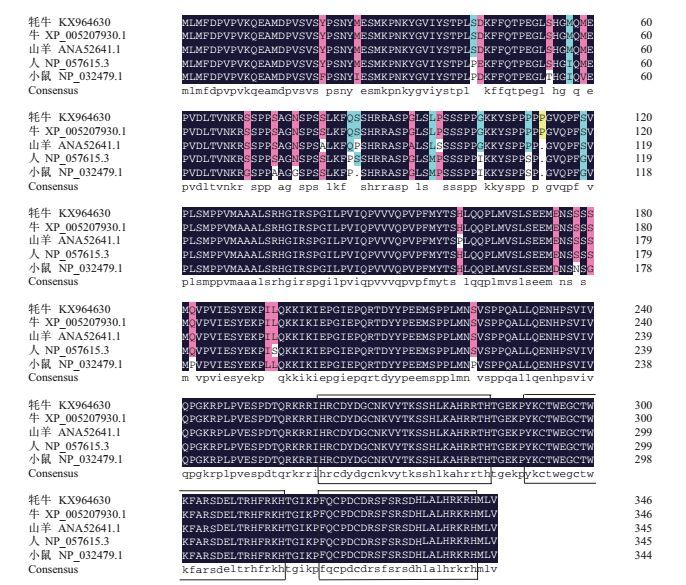
2.2 牦牛KLF3基因序列分析磷酸化位点预测结果显示,牦牛KLF3蛋白有34个丝氨酸(Ser)磷酸化位点,10个苏氨酸(Thr)磷酸化位点,3个酪氨酸(Tyr)磷酸化位点;糖基化位点预测结果发现在176位存在1个N-糖基化位点,无O-糖基化位点;SignalP和TMHMM预测KLF3蛋白无信号肽剪切位点和跨膜结构域,PSORT Ⅱ进行亚细胞定位,发现其主要在核(95.7%)和细胞质(4.3%)中发挥生物学作用;生物学功能(图 2)预测显示KLF3具有典型的3个连续的锌指结构C2H2(261-285、291-315、321-343位氨基酸)。STRING交互式数据库进行蛋白相互作用分析,结果(图 3)表明KLF3蛋白可能和CTBP1、CTBP2、WEE1、WEE2和XRN1等蛋白存在相互作用,构建蛋白相互作用网络;KLF3蛋白二级结构预测结果显示59(17.05%)个氨基酸可能形成α-螺旋,50(14.45%)个氨基酸可能形成β-折叠,237(68.5%)个氨基酸可能形成无规则卷曲;三级结构分析结果显示KLF3蛋白具有3个锌指结构,与预测一致。Protfun预测KLF3功能分类,结果(表 2)显示该蛋白在复制与转录、调节功能以及辅因子的生物合成过程中发挥作用的可能性为0.373、0.322和0.235,作为转录因子、转录调控因子以及信号转导分子发挥作用的可能性为0.445、0.360和0.072,明显高于其他功能。
|
| 图 2 KLF3氨基酸序列生物学功能预测 |

|
| 图 3 牦牛KLF3蛋白相互作用分析 |
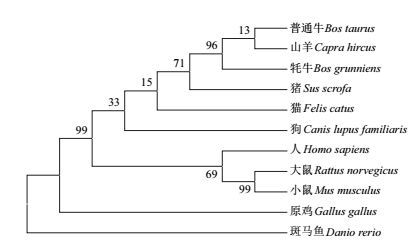
通过NCBI中Blast进行氨基酸同源性比对(图 4)发现,牦牛KLF3氨基酸序列与普通牛(XP_010820342.1)、猪(NP_001177179.1)、山羊(ANU06073.1)、人(NP_009181.2)、大鼠(NP_001099212.1)、小鼠(NP_776141.1)、猫(XP_006931231.1)、狗(XP_545954.3)、原鸡(XP_015141159.1)和斑马鱼(NP_571934.3)的同源性依次是100%、98%、98%、97%、94%、94%、98%、97%、81%和41%。系统进化树分析表明麦洼牦牛与普通牛和山羊的亲缘关系最近,符合分类学和物种进化规律。
|
| 图 4 NJ法构建牦牛KLF3氨基酸序列系统进化树 |
通过qPCR方法检测KLF3 mRNA在牦牛脾脏、肺脏、肾脏、脂肪和背最长肌中的相对表达量(图 5),发现KLF3 mRNA在肺脏和肝脏中的表达水平最高,极显著高于其他组织(P < 0.01)。

|
| 图 5 牦牛KLF3基因组织表达谱 GAPDH作为内参基因;n=6;不同大写字母表示差异极显著(P < 0.01) |
牦牛是我国青藏高原的特有畜种,是高原地区优良的乳肉兼用型品种。牦牛肉因其独特的风味和营养价值丰富等特点而备受消费者的青睐,但其肉品质相比于普通牛较差[11-12],而肌内脂肪含量是影响动物肉质的重要因素之一[13],过量的脂肪沉积会影响动物产品的风味和品质,适量的肌内脂肪含量可调节肉的嫩度、风味与多汁性[14],因此从分子育种的角度来提高牦牛肉品质特性,对于开扩牦牛市场,推动高原地区牦牛产业的发展具有现实和深远的意义。
本实验利用RT-PCR技术成功克隆获得牦牛KLF3基因序列1 137 bp,其中包含CDS区1 041 bp,编码346个氨基酸,与普通牛(XP_010820342.1)的核苷酸同源性为99.82%,氨基酸同源性为100%,表明两者之间可能并不存在生物学功能上的差异。生物信息学分析预测结果显示,牦牛KLF3蛋白有34个丝氨酸磷酸化位点,10个苏氨酸磷酸化位点,3个酪氨酸磷酸化位点,无信号肽剪切位点和跨膜结构域,主要在细胞核中发挥生物学作用;生物学功能预测显示KLF3具有3个连续的锌指结构,哺乳动物KLF3序列具有高度保守性,锌指结构域的同源性几乎达100%,这些结构域可以有效地与DNA分子上富含GC的序列(GC盒或CACCC元件)结合[1],也可以与蛋白质互作从而起转录调节作用[15]。研究表明KLF3可通过招募CtBP1、CtBP2及wee1等辅阻遏物抑制其转录[3, 16, 17],与本研究的蛋白相互作用分析结果一致。蛋白质三级结构对比显示不同物种间的KLF3蛋白具有较高的相似性,但并不完全一致,表明其具有物种特异性。Protfun预测KLF3功能分类,考虑到KLF3编码产物蛋白所属家族因素,推测KLF3可能在调节功能和作为转录因子发挥信号转导和转录调控等作用。
研究表明KLF3基因在不同动物的不同组织中的表达存在较大差异,王茫桔等[18]通过Northern印迹发现KLF3在人组织中广泛表达,且在肝脏组织中的表达水平最高,表达量随肝脏发育成熟而下降。Crossley等[19]同样通过Northern印迹发现KLF3在成年小鼠的肝、肺和脾等组织中的表达水平较高。Zhang等[20]的研究也表明KLF3在鸡的各组织中也均存在表达,但低表达于肌肉组织,高水平表达胰腺。本实验通过qPCR方法检测KLF3在牦牛5个组织中的表达情况,结果表明KLF3在肺和肝脏组织中的表达水平较高,与上述报道存在相似及不同之处,进一步证实了该基因的组织表达具有物种特异性,这也提示KLF3可能是一种在体内广泛发挥作用的转录因子。研究指出,随脂肪细胞的分化,KLF3表达逐渐降低,显示KLF3是脂肪细胞分化的负调控因子,但敲除KLF3基因后小鼠的脂肪组织减少[3],意味着KLF3可能在脂肪组织生长发育过程中具有多个调控功能[21]。在3T3-L1细胞中过表达KLF3,前体脂肪细胞分化被抑制[3],而RNA干扰KLF3可增加酰基乙酰辅酶A合成酶、酰基乙酰辅酶A氧化酶,进而引起动物肠道脂肪积累[7]。上述研究结果表明KLF3在脂肪细胞分化过程中发挥重要的调控作用,但其在牦牛成脂分化过程中的表达模式与调控作用是否与已有的报道一致这将是本实验室下一步的研究内容,有待于干扰和过表达等实验的进一步分析和验证。
4 结论本研究成功克隆获得牦牛KLF3基因序列1 137 bp,编码346个氨基酸残基,为不稳定亲水碱性蛋白,无信号肽结构和跨膜结构域,主要在细胞核中发挥作用。组织表达谱图显示KLF3基因在牦牛的肺和肝脏组织中的表达水平较高。
| [1] | Pearson R, Fleetwood J, Eaton S, et al. Krüppel-like transcription factors:a functional family[J]. The International Journal of Biochemistry & Cell Biology, 2008, 40 (10): 1996–2001. |
| [2] | Chen Z, Lei T, Chen X, et al. Porcine KLF gene family:Structure, mapping, and phylogenetic analysis[J]. Genomics, 2010, 95 (2): 111–119. DOI:10.1016/j.ygeno.2009.11.001 |
| [3] | Sue N, Jack BH, Eaton SA, et al. Targeted disruption of the basic Krüppel-like factor gene(Klf3)reveals a roal in adipogenesis[J]. Molecular and Cellular Biology, 2008, 28 (12): 3967–3978. DOI:10.1128/MCB.01942-07 |
| [4] | Turchinovich G, Vu TT, Frommer F, et al. Programming of marginal zone B-cell fate by basic Kruppel-like factor(BKLF/KLF3)[J]. Blood, 2011, 117 (14): 529–537. |
| [5] | Funnell AP, Maloney CA, Thompson LJ, at al. Erythroid Krüppel-like factor directly activates the basic Krüppel-like factor gene in erythroid cells[J]. Molecular and Cellular Biology, 2007, 27 (7): 2777–2790. DOI:10.1128/MCB.01658-06 |
| [6] | Himeda CL, Ranish JA, Pearson RC, at al. KLF3 regulates muscle-specific gene expression and synergizes with serum response factor on KLF binding sites[J]. Molecular and Cellular Biology, 2010, 30 (14): 3430–3443. DOI:10.1128/MCB.00302-10 |
| [7] | Zhang J, Bakheet R, Parhar RS, et al. Regulation of fat storage and reproduction by Krüppel-like transcription factor KLF3 and fat-associated genes in Caenorhabditis elegans[J]. Journal of Molecular Biology, 2011, 411 (3): 537–553. DOI:10.1016/j.jmb.2011.06.011 |
| [8] | Zhang J, Hashmi S, Cheema F, et al. Regulation of lipoprotein assembly, secretion and fatty acid β-oxidation by Krüppel-like transcription factor, klf3[J]. Journal of Molecular Biology, 2013, 425 (15): 2641–2655. DOI:10.1016/j.jmb.2013.04.020 |
| [9] | Wu J, Srinivasan SV, Neumann JC, et al. The KLF2 transcription factor does not affect the formation of preadipocytes but inhibits their differentiation into adipocytes[J]. Biochemistry, 2005, 44 (33): 11098–11105. DOI:10.1021/bi050166i |
| [10] | Eaton SA, Funnell AP, Sue N, et al. A network of Kruppel-like factors(Klfs), Klf8 is repressed by Klf3 and activated by Klf1 in vivo[J]. The Journal of Biological Chemistry, 2008, 283 (40): 26937–26947. DOI:10.1074/jbc.M804831200 |
| [11] | 万红玲, 雒林通, 吴建平. 牦牛肉品质特性研究进展[J]. 畜牧兽医杂志, 2012, 31(1): 36–40. |
| [12] | 拜彬强, 郝力壮, 柴沙驼, 等. 牦牛肉品质特性研究进展[J]. 食品科学, 2014, 35(17): 290–296. DOI:10.7506/spkx1002-6630-201417055 |
| [13] | 朱武政, 林亚秋, 江明锋, 等. 肉用山羊脂代谢相关基因与肌内脂肪含量的相关分析[J]. 畜牧兽医学报, 2016, 47(7): 1333–1341. |
| [14] | Wang YH, Byrne KA, Reverter A, et al. Transcriptional profiling of skeletal muscle tissue from two breeds of cattle[J]. Mamm Genome, 2005, 16 (3): 201–210. DOI:10.1007/s00335-004-2419-8 |
| [15] | Zhang W, Kadam S, Emerson BM, et al. Site-specific acetylation by p300 or CREB binding protein regulates erythroid Kruppel-like factor transcriptional activity via its inetraction with the SWI-SNF complex[J]. Molecular and Cellular Biology, 2001, 21 (7): 2431–2422. |
| [16] | Kitamura T, Suzuki H, Tamura TA. Mouse Wee1 gene is repressed by Krüppel-like factor 3(KLF3) via interaction with multiple upstreamelements[J]. Gene, 2012, 492 (2): 361–367. DOI:10.1016/j.gene.2011.11.016 |
| [17] | Dewi V, Kwok A, Lee S, et al. Phosphorylation of Krüppel-like factor 3(KLF3/BKLF)and C-terminal binding protein 2(CtBP2) by homeodomain-interacting protein kinase 2(HIPK2) modulates KLF3 DNA binding and activity[J]. The Journal of Biological Chemistry, 2015, 290 (13): 8591–8605. DOI:10.1074/jbc.M115.638338 |
| [18] | 王茫桔, 瞿祥虎, 王立升, 等. 人类新型Krǜppel类转录因子hBKLF的cDNA克隆、亚细胞定位及表达特征[J]. 遗传学报, 2003, 30(1): 1–9. |
| [19] | Crossley M, Whitelaw E, Perkins A, et al. Isolation and characterization of the cDNA encoding BKLF/TEF-2, a major CACCC-box-binding protein in erythroid cells and selected other cells[J]. Molecular and Cellular Biology, 1996, 16 (4): 1695–1705. DOI:10.1128/MCB.16.4.1695 |
| [20] | Zhang ZW, Wu CY, Li H, et al. Expression and functional analyses of Krüppel-like factor 3 in chicken adipose tissue[J]. Bioscience Biotechnolgy Biochemistry, 2014, 78 (4): 614–623. DOI:10.1080/09168451.2014.896735 |
| [21] | 张志威, 李辉, 王宁. KLF转录因子家族与脂肪细胞分化[J]. 中国生物化学与分子生物学报, 2009, 25(11): 983–990. |