2. 福建农林大学艺术学院园林学院,福州 350002
2. College of Art & Landscape Architecture, Fujian Agriculture and Forestry University, Fuzhou 350002
绿竹(Dendrocalamopsis oldhami(Munro))属禾本科(Gramineae)竹亚科(Bambusoideae)绿竹属(Dendrocalamopsis)丛生竹种,是绿竹属中分布最广、栽培最多的竹种[1]。绿竹具有发笋期长、生长迅速、产量丰富、笋味鲜美,质地柔软的优点,是我国南方优良速生的笋材两用丛生竹种之一,具有较高的商品效益,是主产地的经济来源之一[2]。绿竹原产我国台湾省淡水,我国主要分布在浙江南部、福建、台湾、广东、广西和海南等省区[3]。
目前,国内关于绿竹的研究报道,主要集中在良种繁育技术研究、生物量研究、生理特性研究,蛋白组学研究等方面[4-10],而关于分子遗传学研究[11-13]较少,且绿竹分子标记的研究中主要是运用随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)技术,余学军等[11]利用RAPD标记对22个不同栽培类型的绿竹进行多态性分析。吴益民等[12]构建孝顺竹、凤尾竹、绿竹、白绿竹4个竹种RAPD指纹图谱。但是RAPD在实验过程中容易产生假带、共迁移等现象,所以结果不够稳定,重复性较低。简单重复序列区间(Inter simpce sequence repeat,ISSR)技术是1994年Zietkiewicz等[14]在简单重复序列(Simple sequence repeat,SS-R)分子标记技术的基础上发展起来的一种新的分子标记方法,具有简单快捷易操作,DNA用量较少,试验成本低等优点,由于其多态性好、稳定性高和重复性高的特性被广泛应用于多种物种的品种鉴定和遗传多样性分析等方面[15, 16]。而利用ISSR分子标记技术对绿竹ISSR-PCR体系建立尚未有报道。本实验采用正交试验设计的方法对dNTPs浓度、Mg2+浓度、Taq DNA聚合酶浓度、引物浓度和模板DNA浓度设计5因素4水平试验,建立绿竹ISSR-PCR最佳反应体系,并优化扩增程序、筛选出高多态性引物,旨在为绿竹的种质资源的鉴定、基因定位、遗传多样性分析、分子育种、DNA指纹图谱构建等研究奠定了基础。
1 材料与方法 1.1 材料供试的材料于2016年8月采自于福建省东山国有赤山林场的绿竹地理种源试验样地。对不同的植株随机选取3-5片嫩叶,放置于塑料自封袋中,并用变色硅胶进行迅速干燥,将干燥的材料速带回实验室,于-80℃超低温冰箱保存备用。
1.2 方法 1.2.1 绿竹基因组总DNA的提取与检测绿竹总DNA的提取采用的是杭州博日公司Biospin植物基因组DNA提取试剂盒(Cat#BSC13S1)法,并对该种方法略作改良。提取完后用微量紫外分光光度计测定DNA的浓度和纯度。并用1%的琼脂糖凝胶电泳检测DNA的完整性与清晰性,看是否有降解或断裂现象,将满足试验条件的DNA稀释到20 ng/μL,并置于-20℃冰箱保存。
1.2.2 绿竹ISSR-PCR扩增反应体系的优化用L16(45)正交试验设计方法对dNTPs浓度、Mg2+浓度、Taq DNA聚合酶浓度、引物浓度和模板DNA浓度设计5因素4水平试验(表 1),共16个处理,每个处理2个重复,来研究各因素对ISSR-PCR扩增的影响。反应体系的总体积为20 μL,引物选择UBC815。扩增程序为:94℃预变性5 min;94℃变性45 s,(根据引物的退火温度)复性30 s,72℃延伸90 s,循环35次,72℃延伸10 min,4℃保存。
将PCR产物于1.5%的琼脂糖凝胶上,电泳45 min,电压设为5 v/cm。并用紫外凝胶成像分析仪拍照记录电泳情况。利用直观分析法对每个处理结果进行评分,一共划分为16个等级,以条带的清晰度、明亮度、特异性高低为评分标准,最高的记为16分,最低的记为1分[17]。评分结果用DPS7.05统计软件进行方差分析。
1.2.3 绿竹ISSR-PCR扩增反应程序的优化在最优反应体系的基础上,采用单因素试验的方法对退火温度和循环次数进行筛选,根据引物U815的Tm值52.9℃,在45-60℃之间设置了12个温度梯度,系统自动生成的12个温度梯度分别为44.9℃,45.2℃,46.1℃,47.5℃,49.3℃,51.2℃,53.3℃,55.2℃,57.1℃,58.5℃,59.6℃,60.1℃。对循环次数设置了30次、32次、35次、38次、40次5个梯度,以筛选出最佳的ISSR-PCR扩增反应程序。
1.2.4 绿竹ISSR扩增体系稳定性验证以不同种源的绿竹的基因组总DNA为模板,在最佳反应体系和最佳扩增程序下,用引物UBC815进行ISSR-PCR扩增,检验建立的扩增体系的稳定性。
1.2.5 ISSR有效引物的筛选以优化的反应体系及扩增程序体系为基础进行引物筛选,在100条ISSR引物中筛选出14条扩增条带清晰、多态性较高、重复性好的引物。
2 结果 2.1 绿竹基因组总DNA的提取结果部分绿竹基因组总DNA凝胶电泳结果如图 1所示,我们可以看出DNA条带清晰明亮,无明显拖尾现象,说明所提取的DNA基因组完整性较好,无明显降解现象。利用微量紫外分光光度计测定DNA的A260/A280的比值均在1.8-2.0之间,说明DNA的纯度较高。

|
| 图 1 绿竹基因组DNA电泳图 |
绿竹正交试验的电泳结果如图 2所示,在16个处理组合中,由于dNTPs、Mg2+、Taq DNA聚合酶、引物和模板DNA浓度5个影响因素各水平组合的不同,扩增结果存在明显的差异。对正交试验的各处理的扩增结果进行评分,评分结果见表 1。根据评分计算每个因素同一水平下的总和T和平均值X,以及极差R,计算结果见表 2。

|
| 图 2 正交设计ISSR-PCR反应体系扩增结果 M:200 bp Marker;1-16:正交试验的16个处理 |
极差R值越大,说明该因素对绿竹ISSR-PCR扩增的影响越大。由表 2可知,各因素对ISSR-PCR扩增结果的影响从大到小依次为:Mg2+ > dNTPs > 模板DNA > Taq DNA聚合酶 > 引物。平均值X最大值所对应的水平即为该因素的最佳浓度。所以dNTPs水平3、Mg2+水平1、Taq DNA聚合酶水平3、引物水平2、模板DNA水平4最好。因此绿竹ISSR-PCR正交试验得出的最佳水平为:dNTPs浓度为0.2 mmol/L,Mg2+浓度为2.0 mmol/L,TaqDNA聚合酶浓度为1.5 U,引物浓度为0.5 μmol/L,DNA浓度为为60 ng。
为了进一步判断各个因素对绿竹ISSR-PCR扩增影响的显著程度,对正交试验的结果进行方差分析,结果见表 3。其中F值越大,说明该因素对绿竹ISSR-PCR扩增的影响越大。由表 3可知,各因素对ISSR-PCR扩增结果的影响从大到小依次为:Mg2+ > dNTPs > 模板DNA > Taq DNA聚合酶 > 引物。与极差分析的结果一致。其中dNTPs、Mg2+和模板DNA对绿竹ISSR-PCR扩增影响达到极显著的水平(P < 0.01)。Taq DNA聚合酶对绿竹ISSR-PCR扩增影响达到显著的水平(P < 0.05)。引物浓度对绿竹ISSR-PCR扩增影响不显著。
为了进一步确定每个因素的最适宜的浓度水平,对5个因素进行Duncan多重比较,结果见表 4。由之前的极差分析和方差分析可知,dNTPs对绿竹ISSR-PCR扩增的影响仅次于Mg2+,从多重比较可以看出,dNTPs水平1、水平2、水平3和水平4差异显著,而水平1、水平2、水平3之间的差异并不显著,从经济角度最终选择dNTPs最佳浓度为0.2 mmol/L。Mg2+对绿竹ISSR-PCR扩增的影响最大,Mg2+水平1、水平2和水平3、水平4差异显著,水平1和水平2两者差异不显著,综合而言,最终选择Mg2+最佳浓度为2.0 mmol/L。当TaqDNA聚合酶浓度为1.5 U时,极差分析的均值最大,且与其他3个水平差异显著,所以最终选择TaqDNA聚合酶最佳浓度为1.5 U。在本试验中,引物浓度对绿竹ISSR-PCR扩增的影响最小,且4个水平间差异不显著,从经济角度最终选择引物最佳浓度为0.4 μmol/L。当模板DNA浓度为60 ng,极差分析的均值最大,且与其他3个水平差异显著,所以最终选模板DNA最佳用量为60 ng。
综上所述,最终确定的绿竹ISSR-PCR扩增最佳反应体积为:dNTPs浓度为0.2 mmol/L,Mg2+浓度为2.0 mmol/L,TaqDNA聚合酶浓度为1.5 U,引物浓度为0.4 μmol/L,DNA浓度为60 ng。
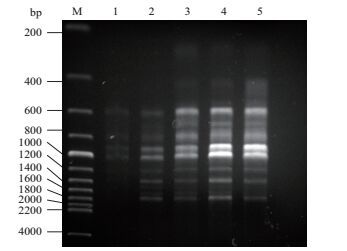
2.3 绿竹ISSR-PCR扩增反应程序的优化结果不同的循环次数对ISSR-PCR扩增有一定的影响。本实验对循环次数设置了30次、32次、35次、38次和40次5个梯度,实验结果如图 3所示。当循环次数为38次时,扩增条带最为清晰明亮且稳定。所以,选择38次循环为ISSR-PCR反应的最佳循环次数。
|
| 图 3 循环次数对ISSR-PCR的影响 M:200 bp Marker;1:30 cycles,2:32 cycles,3:35 cycles,4:38 cycles,5:40 cycles |
不同的退火温度对ISSR-PCR扩增也有一定的影响。根据引物U815的Tm值52.9℃,在45-60℃之间设置了12个温度梯度,结果(图 4)显示退火温度对扩增影响明显,当退火温度为47.5℃时,扩增的条带数最为清晰稳定,当退火温度大于47.5℃时,高分子量区域和中等分子量区域条带的扩增效率下降,直至无扩增条带,且在低分子量区域也逐渐出现弥散现象,整体电泳条带数逐渐减少,清晰度也逐渐减弱,因此,最终确定引物U815的最佳退火温度为47.5℃。
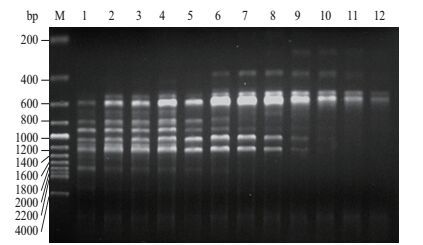
|
| 图 4 退火温度对ISSR-PCR的影响 M:200 bp Marker;1-12:44.9℃,45.2℃,46.1℃,47.5℃,49.3℃,51.2℃,53.3℃,55.2℃,57.1℃,58.5℃,59.6℃,60.1℃ |
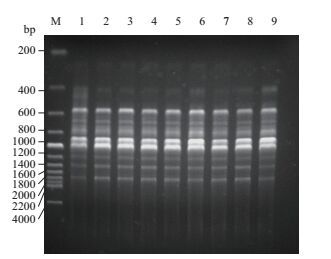
利用引物UBC818对不同种源的绿竹个体的基因组总DNA样品进行进行ISSR-PCR扩增,扩增结果见图 5。由图 5可知,在该体系下均能扩增出条带清晰、稳定,多态性丰富的条带。说明该体系稳定可靠,适合绿竹ISSR-PCR扩增反应。
|
| 图 5 不同种源的绿竹个体扩增结果 M:200 bp Marker;1-9:福州、闽清1、闽清2、闽侯1、闽侯2、福清1、福清2、东山1、东山2 |
根据绿竹优化后的最佳反应体系和扩增程序,对100条随机引物进行扩增。部分引物筛选结果如图 6所示。结果显示利用建立的体系筛选不同的引物时,大部分引物都能扩增出清晰明亮的条带,只有少部分引物扩增出的条带数较少或不能扩增出条带。说明本实验建立的绿竹最佳反应体系具有较高的重复性和稳定性。最终在100条引物中筛选出14条扩增条带清晰不拖尾,稳定性较好的引物(表 5)。

|
| 图 6 ISSR有效引物的筛选 M:200 bp Marker;1-24依次为:UBC849、UBC850、UBC851、UBC852、UBC853、UBC854、UBC855、UBC856、UBC857、UBC858、UBC859、UBC860、UBC861、UBC862、UBC863、UBC864、UBC865、UBC866、UBC867、UBC868、UBC869、UBC870、UBC871、UBC872 |
ISSR分子标记技术已经广泛应用于种质资源的鉴定、基因定位、遗传多样性分析、指纹图谱的构建等方面的研究,是目前使用最广泛的分子标记技术之一[18-20]。ISSR-PCR扩增结果受到物种类型、反应体系、扩增程序、引物等的综合影响。本试验采用正交试验设计的方法对dNTPs浓度、Mg2+浓度、Taq DNA聚合酶浓度、引物浓度、模板DNA用量设计5因素4水平试验,并对扩增程序进行优化,利用优化后的体系对100条ISSR引物进行筛选。正交试验设计可以克服单因素试验不能考虑到各影响因素之间的相互作用的缺点,还可以解决完全组合设计需要大量试验、耗时耗费的不足,具有设计均衡分散、齐整可比、效应明显、节约时间和成本等优点[17],能够通过较少的处理组合快速找到最佳的ISSR-PCR反应体系,并能分析出不同因素对扩增结果影响的显著性。但是正交试验直观分析又有一定的主观随机性,所以需要设置重复试验来减少打分误差,增加数据的可靠性[21]。
在本实验中,不同的处理组合对绿竹ISSR-PCR扩增结果的影响差异明显。极差分析和方差分析的结果均表明各因素对绿竹ISSR-PCR扩增结果的影响从大到小依次为:Mg2+ > dNTPs > 模板DNA > Taq DNA聚合酶 > 引物。Mg2+是影响绿竹ISSR-PCR扩增最显著的因素,Mg2+浓度变化直接影响Taq DNA聚合酶的活性,并间接影响引物与模板DNA的双联杂交体的解链温度、引物的退火温度、扩增产物的特异性以及引物二聚体的生成[22-23]。靳晓丽等[24-26]研究均显示Mg2+对ISSR-PCR扩增影响最大,所以正确的把握好Mg2+的用量很关键。dNTPs对绿竹ISSR-PCR扩增的影响仅次于Mg2+,dNTPs是PCR扩增的底物,dNTPs浓度过低会降低DNA的合成速率,导致扩增产量的下降;dNTPs浓度过高又会与Mg2+相鳌合从而间接影响Taq DNA聚合酶的活性,导致碱基的错误渗入和非特异性扩增[27, 28]。模板DNA浓度对绿竹ISSR-PCR扩增的影响也是极显著的,在本实验中DNA模板浓度越高,扩增的条带越亮产量越多。Taq DNA聚合酶对绿竹ISSR-PCR扩增的影响是显著的,Taq DNA聚合酶浓度过低会导致扩增效率较低,条带较弱;浓度过高又会产生非特异性条带[29]。在本实验中引物浓度对绿竹ISSR-PCR扩增的影响不显著,这可能与本实验中引物浓度设置的水平范围有关,在本实验中,引物浓度在0.4-0.7 μmol/L之间设置了4个水平,4个水平间差异不显著,与包蕊等[30]研究的在ISSR-PCR反应中引物浓度影响最小的研究结果一致。
循环次数和退火温度对绿竹ISSR-PCR扩增也有一定影响,循环次数较少,扩增的条带数也较少且清晰度较低;循环次数过多又会增加非特异性扩增。退火温度较低会导致模板DNA与引物的非特异性结合且背景深;退火温度较高又会使得模板DNA与引物的结合率下降,从而使得扩增出来的条带数较少[31]。在一定的温度范围内,随着退火温度的增加,扩增出的产物的特异性越好,一般最佳的退火温度低于引物的真实Tm值5℃左右[32]。
4 结论本研究采用L16(45)正交试验设计方法对dNTPs浓度、Mg2+浓度、Taq DNA聚合酶浓度、引物浓度和模板DNA浓度设计5因素4水平试验,并通过直观分析和正交极差分析对比,确定了这5个因素对ISSR-PCR扩增的影响力大小以及最佳用量。最后确定适合绿竹ISSR-PCR的最佳反应体系为:dNTPs浓度为0.2 mmol/L,Mg2+浓度为2.0 mmol/L,TaqDNA聚合酶浓度为1.5 U,引物浓度为0.4 μmol/L,DNA浓度为60 ng。验证实验表明,该体系稳定可靠,适合于绿竹ISSR-PCR扩增。
| [1] | 郑蓉, 方伟, 郑维鹏, 等. 绿竹研究[J]. 竹子研究汇刊, 2007, 01: 20–26. DOI:10.3969/j.issn.1000-6567.2007.01.005 |
| [2] | 李谦, 周本智, 安艳飞, 等. 绿竹根系分布及生物力学性质[J]. 应用生态学报, 2014(5): 1319–1326. |
| [3] | 中国科学院中国植物志编辑委员会. 中国植物志第九卷(第一分册)[M]. 北京: 科学出版社, 1996: 141. |
| [4] | Sirikuly AN, Sagwansupyakorn C, Luangprasert N, et al. Propagation of Lu Chu bamboo(Bambusa oldhamii Munro)by culm cutting[J]. Natural Sciences, 1997, 31 (4): 393–398. |
| [5] | 姚旺. 两种丛生竹的侧枝扦插技术及生根机理研究[D]. 福州: 福建农林大学, 2016. |
| [6] | 夏海涛, 王月英, 李效文, 等. 1~2年生浙南绿竹地上生物量分配与模型构建[J]. 林业实用技术, 2014(10): 3–5. |
| [7] | 李凯. 海盐胁迫下绿竹光合生理生化响应的研究[D]. 临安: 浙江农林大学, 2013. |
| [8] | 叶晶, 陶立华, 柯和佳, 等. 绿竹地上部营养元素的吸收、积累和分配特性[J]. 浙江农林大学学报, 2015(4): 545–550. DOI:10.11833/j.issn.2095-0756.2015.04.008 |
| [9] | 丁鹏. 不同盐度胁迫下绿竹差异蛋白质组学研究[D]. 福州: 福建农林大学, 2015. |
| [10] | 周佳平. 绿竹及拟南芥SEP3蛋白原核表达、纯化及寡聚体分析[D]. 临安: 浙江农林大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10341-1014101629.htm |
| [11] | 余学军, 张立钦, 方伟, 等. 绿竹不同栽培类型RAPD分子标记的研究[J]. 西南林学院学报, 2005, 4: 98–101. DOI:10.3969/j.issn.2095-1914.2005.04.021 |
| [12] | 吴益民, 黄纯农, 王君晖. 四种竹子的RAPD指纹图谱的初步研究[J]. 竹子研究汇刊, 1998, 3: 10–14. |
| [13] | 郑蓉. 绿竹遗传多样性与优良地理种源选择的研究[D]. 北京: 北京林业大学, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10022-2008085022.htm |
| [14] | Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 1994, 20 (2): 176–183. DOI:10.1006/geno.1994.1151 |
| [15] | Arcade A, Ansclin F, Faivare RP, et al. Application of AFLP, RA-PD and ISSR markers to genetic mapping of European and Japanses larch[J]. Theoretical and Applied Genetics, 2000, 100 (2): 299–307. DOI:10.1007/s001220050039 |
| [16] | Casasoh M, Mattioni C, Cherubina, et al. A genetic linkage map of European chestnut(Castanea sativa Mill.)based on RAPD, ISSR and isozyne markers[J]. Theoretical and Applied Genetics, 2001, 102 : 1190–1199. DOI:10.1007/s00122-001-0553-1 |
| [17] | 何正文, 刘运生, 陈立华, 等. 正交设计直观分析法优化PCR条件[J]. 湖南医科大学学报, 1998(4): 76–77. |
| [18] | 张旻桓, 金晓玲, 成仿云, 等. 湖南产牡丹遗传多样性的ISSR分析[J]. 中草药, 2016(7): 1193–1198. |
| [19] | 李绍臣, 李凤明, 张立民, 等. 吉林省天然黄檗种群遗传多样性ISSR分析[J]. 生态学报, 2016(13): 4006–4012. |
| [20] | 葛亚英, 张飞, 沈晓岚, 等. 丽穗凤梨ISSR遗传多样性分析与指纹图谱构建[J]. 中国农业科学, 2012(4): 726–733. |
| [21] | 唐辉, 陈宗游, 史艳财, 等. 正交设计优化地枫皮ISSR-PCR反应体系[J]. 中草药, 2013(5): 610–615. |
| [22] | 邱帅, 丁信誉, 席梦利, 等. 东方百合SRAP体系优化及引物筛选[J]. 南京林业大学学报:自然科学版, 2013, 03: 6–10. |
| [23] | 张玲玲. 南方红豆杉DNA指纹图谱技术研究[D]. 福州: 福建农林大学, 2009. http://cdmd.cnki.com.cn/Article/CDMD-10389-2009170281.htm |
| [24] | 靳晓丽, 田新会, 杜文华. 鹰嘴豆ISSR反应体系的正交优化[J]. 草地学报, 2015(6): 1303–1309. DOI:10.11733/j.issn.1007-0435.2015.06.025 |
| [25] | 钱志瑶, 周道堂, 黄秀平, 等. 黔产宽叶缬草ISSR反应体系的建立与优化[J]. 生物技术通报, 2015(7): 69–75. |
| [26] | 原勤勤, 文亚峰, 何钢, 等. 用正交设计法优化枣树ISSR-PCR反应体系[J]. 中南林业科技大学学报, 2010(2): 70–74. |
| [27] | 郑志雷. 厚朴遗传多样性研究及指纹图谱的构建[D]. 福州: 福建农林大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10389-2010181435.htm |
| [28] | 邱帅, 丁信誉, 席梦利, 等. 东方百合SRAP体系优化及引物筛选[J]. 南京林业大学学报:自然科学版, 2013, 3: 6–10. |
| [29] | 胡延萍, 王莉, 包蕊, 等. 正交设计优化黑褐苔草ISSR-PCR反应体系[J]. 草地学报, 2014, 4: 911–914. DOI:10.11733/j.issn.1007-0435.2014.04.037 |
| [30] | 包蕊, 胡延萍, 王莉, 等. 华扁穗草ISSR-PCR反应体系优化及引物筛选[J]. 中国草地学报, 2014(6): 46–52. |
| [31] | 王芳, 廖柏勇, 李培, 等. 苦楝SSR-PCR反应体系优化及引物筛选[J]. 林业科学研究, 2016, 2: 167–175. |
| [32] | Henegariu O, Heerema NA, Dlouhy SR, et al. Multiplex PCR:critical parameters and step-by-step protocol[J]. BioTechniques, 1997, 23 : 504–511. |