2. 中南大学生物冶金教育部重点实验室,长沙 410083
2. Key Laboratory of Biometallurgy, Ministry of Education, Central South University, Changsha 410083
生物浸出技术是低成本、环境友好地开发矿产资源的高新技术,对保证我国矿产资源可持续发展具有重要意义。
对于生物浸出反应机制,大家普遍接受“非接触浸出”和“接触浸出”的联合机制[1, 2]。“非接触浸出”机制是指浸出溶液中的Fe2+离子被溶液中悬浮细菌氧化成Fe3+离子,Fe3+与矿物表面接触氧化硫化矿物,而Fe3+被还原成Fe2+,依次循环;“接触浸出”机制是指生物浸出反应过程中,微生物分泌胞外多聚物(Extracellular polymeric substances,EPS),吸附至矿物表面,细菌群体生长并形成微菌落,营造出不连续的、局部的、三维网状聚集的、微米量级厚度的生物膜,营造出细菌生存和矿物溶解的微观环境。生物膜作为矿物、细菌和浸出溶液间的中间媒介,是细菌生存和矿物溶解的共同场所,因此,生物膜在生物浸出体系中具有非常重要的作用,已成为目前生物浸出反应理论研究的热点。
生物膜EPS的主要成分是多糖、蛋白质及脂肪酸等。在生物浸矿领域,对EPS功能及其作用,目前研究最多的是胞外多糖,而蛋白质作为EPS的关键组分之一,其功能作用和机理研究较少;报道的胞外蛋白的分离提取方法也多种多样,而胞外蛋白关键成分的结构、功能及其作用的研究更少,不利于基于EPS的生物膜在生物冶金过程中作用机制的阐明。本文综述了胞外蛋白特别是多糖结合型胞外蛋白的研究进展、胞外蛋白的现有研究方法及其对冶金微生物胞外蛋白研究的适用性,展望了浸矿微生物胞外蛋白研究的前景,旨为生物冶金领域研究EPS关键组分胞外蛋白结构及其作用提供重要的理论和方法支撑。
1 胞外蛋白对生物浸出的影响目前,越来越多的人认可胞外多聚物(EPS)关键组分胞外蛋白在生物膜形成、结构稳定和硫化矿溶解等方面起到关键作用[3]。Sampson等[4]归纳了包括微生物表面EPS黏液层的分泌,蛋白结合受体分泌,多糖和蛋白复合物的分泌等在内的浸矿生物吸附到矿物表面的作用方式。Zeng等[5]使用GC-MS联用技术分析了吸附到矿物表面的微生物产生胞外多聚物的成分,主要包括蛋白质、糖类和脂肪酸等。Zhang等[6]通过对硫营养条件下Acidithiobacillus ferrooxidans的EPS中蛋白质成分的研究认为,部分蛋白质通过Pr-SH与元素硫结合,进而直接参与硫的代谢活动。Zhang等[7]研究了极端嗜酸古菌Ferroplasma acidiphilum在矿物表面的聚集及生物膜的形成情况发现,其在黄铁矿表面形成单层生物膜,且其最先吸附到黄铜矿表面的裂缝处。Guo等[8]通过比较基因组学分析揭示了Sulfobacillus thermosulfidooxidans ST的代谢多样性及其环境适应机制,研究发现至少230个假定的转运蛋白,其中结构元件大约有90个转运系统和50个代表性运输家族。Morales[9]通过研究S. thermosulfidooxidans在黄铁矿表面生成EPS的情况发现,EPS矩阵在整个浸矿过程中不断变化,其中胞外蛋白的含量受介质中氮源含量变化的影响。此外,胞外蛋白的含量也与生物膜的形成阶段有关。
胞外蛋白可以与金属离子发生螯合或键合作用,促进微生物细胞之间的聚集;微生物也可通过形成多糖-蛋白质复合物吸附在矿物表面,组成生物膜的网状骨架等。因此,在浸矿领域深入研究浸矿菌EPS关键组分胞外蛋白在生物浸出过程中的含量变化、基因表达以及在矿物表面的分布情况对微生物吸附、细菌的生长、生物膜形成和稳定等的影响,可为阐明胞外蛋白在生物浸出过程中的作用机理提供理论依据,对深入理解EPS的作用机理和实际应用都有重要意义。
2 胞外蛋白的作用及其机理通常胞外蛋白根据存在形式分为结合型胞外蛋白和溶解型胞外蛋白。从生物降解角度来说,胞外蛋白分为可降解的蛋白和不可降解的蛋白;根据其在生物膜形成过程中的作用又可分为两类:一类是胞外结构蛋白,主要形成生物膜的骨架,促进生物膜的形成等;一类是胞外非结构蛋白,大部分是一些酶类,主要用于生物膜的降解、防止膜污染等[10, 11]。
2.1 胞外结构蛋白胞外结构蛋白在生物膜中所占比例较高,可分为多糖结合型胞外蛋白(Polyasccharides binding extracellular proteins)、细胞结合型胞外蛋白(Cell-binding extracellular proteins)、生物膜结合型胞外蛋白(Biofilm-associated protein)、核酸结合型胞外蛋白(Nucleic acid binding proteins)。
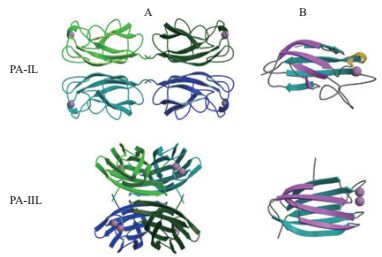
2.1.1 多糖结合型蛋白多糖结合型蛋白是生物膜形成初期的重要组成部分。如Branda等[12]通过研究革兰氏阳性菌Bacillus subtilis生物膜的形成发现,胞外蛋白TasA与多糖结合生成网状生物膜骨架,若这个组分缺失,则不能形成成熟的生物膜。Gilboa-Garber等[13, 14]对Pseudomonas aeruginosa 胞外蛋白中多糖结合型胞外蛋白LecA(PA-IL)、LecB(PA-IIL)凝集素的提取方法、相关组分定性定量及功能等方面的研究,结果表明外源LecA(PA-IL)凝集素受到了群体感应系统(Quorum-sensing,QS)的调控,lecA表达过少,导致生物膜的厚度及覆盖面积减少,lecA过表达,则会促进生物膜的形成;而LecB(PA-IIL)凝集素与Ca2+等多价阳离子键合形成具有高亲和力的多价配体可抑制该菌生物膜的形成,甚至使已形成生物膜的完全解体。此外,在P. aeruginosa中还发现LecA、LecB凝集素分别对半乳糖和海藻糖有较高的亲和力,是P. aeruginosa膜形成初期不可或缺的组分,LecA与LecB的多糖复合物晶体结构,如图 1所示[15, 16]。
2.1.2 细胞结合型胞外蛋白细胞型结合蛋白能促进细胞和细胞外多糖的识别聚集,促进生物膜的形成和稳定。Branda等[12]在研究Bacillus subtilis生物膜形成时发现CdrA蛋白可促进细胞表面蛋白与胞外多糖的结合,从而加剧更多细胞与生物膜的结合,促进生物膜的形成与稳定。Park等[17]通过蛋白质组学技术发现活性污泥的EPS中富含根枯蛋白和鞭毛蛋白等多种蛋白,这些蛋白与细菌细胞的聚集、吸附及生物膜的形成有关。Boretska等[18]采用CLSM技术发现硫细菌Thiobacillus thioparus DSM 505的细胞结合型胞外蛋白在生物腐蚀过程中起重要作用。
2.1.3 生物膜结合型胞外蛋白生物膜结合型蛋白绝大部分分布在细胞表面,主要用于维持生物膜结构的致密性和稳定性。Lasa等[10]研究了Bap蛋白在不同菌种形成生物膜中的结构与功能发现,Bap蛋白主要分布在细胞表面,且促进前期细胞间的粘附及非生物表面的吸附。Rendueles等[19]研究细菌在生物膜系统中的代谢与细胞间的交流时发现,生物膜结合型蛋白能够有效促进生物膜结合大肠杆菌素,从而适应外界环境的变化。
2.1.4 核酸结合型胞外蛋白核酸结合型蛋白一般与胞外DNA紧密结合,维持生物膜结构稳定。此外,当细胞发生裂解、自溶时,胞内DNA释放出来后可以与核酸结合型胞外蛋白结合,从而防止细胞遗传物质的丢失[2]。Wolffe[20]研究发现Y-box蛋白有固定的中心位点与核酸结合,从而实现信号的传导或监管相关物质的转录过程。Harami等[21]发现有翼的螺旋域(Winged helix domain,WHD)广泛存在于核酸中,这个螺旋域主要与蛋白结合形成核酸结合蛋白(Nucleic acid binding proteins),胞内的核酸结合蛋白主要行使转录监管机构的功能,分泌到胞外的核酸结合蛋白行使信号传导的功能或形成生物膜的基本骨架,促进生物膜的形成与结构稳定。
2.2 胞外非结构蛋白胞外非结构蛋白主要包括各种细胞的胞外酶,是胞外蛋白的另一个主要存在形式。很多胞外酶蛋白都是在生物膜中被发现的,与生物膜的降解有关。胞外酶蛋白通过多种方式间接影响生物膜的形成和结构稳定,如在细胞生长的衰亡期,胞外酶蛋白降解EPS中的部分多糖类物质,一方面为细胞生长提供碳源和能源;另一方面促进细胞从生物膜中脱落和释放出来;但胞外大分子聚合物的过多水解又会造成生物膜结构的失稳和破坏,如Li等[22]在研究好氧污泥颗粒在饥饿阶段EPS产生情况时发现,蛋白质和碳水化合物的浓度分别从18.0 mg/g、7.1 mg/g降到了7.0 mg/g、2.9 mg/g,从而改变EPS的成分和细胞表面的疏水性。Lasa和Tielen等[10, 23]研究了胞外酶对P. aeruginosa生物膜形成的影响发现,其分泌多种水解酶水解大分子物质,这些水解酶的过度水解会影响EPS的组成成分、细胞表面性质及细胞的能动性,进而影响生物膜的形成和结构稳定。
3 研究胞外蛋白的关键技术与手段 3.1 胞外蛋白的提取及组分分析方法由胞外蛋白组分分析的技术路线(图 2)可知,EPS提取是实验的第一步,EPS的提取方法直接影响胞外蛋白的提取量,选取适宜的提取方法就显得非常重要。Higgins和Novak[24]采用NaOH提取方法从活性污泥中提取EPS,再与其他技术结合获得LecA凝集素等不同类型胞外蛋白。张丽丽等[25]通过比较加热、超声、离心和加碱等方法提取好氧颗粒污泥的EPS发现,用热碱法提取的EPS中蛋白质的含量较高。张瑞永[26]用水浴-离心、超声波-离心、热碱法-离心3种方法提取A. ferrooxidans的胞外蛋白,实验结果表明水浴加热-离心的方法所得蛋白质的含量较高。

|
| 图 2 胞外蛋白组分分析的技术路线图 |
现在已有很多技术手段对胞外蛋白组分进行定性或定量的分析。单独使用某一方法或技术存在一些不足之处,但通过对这些技术的联用,我们可以得到很多关于蛋白的含量、空间分布、官能团和二级结构等信息。Higgins[24]从活性污泥中提取EPS,通过SDS-PAGE电泳得到不同分子量的胞外蛋白,然后用高压液相色谱(HPLC)、蛋白质测序仪对其进一步分析,获得LecA凝集素等不同类型胞外蛋白。Yang等[27]用同位素标记相对和绝对定量(iTRAQ)与2-DLC-MS/MS联用的方法,分析了Cronobacter sp. 中与生物膜形成有关的几种关键蛋白,为研究生物膜的形成机制提供了重要信息。Zeng[28]采用酶解法和加热法提取生物浸出过程中黄铜矿表面的EPS,并通过LC-MS等方法检测,初步得到关于EPS裂解后一些蛋白质、糖类和脂肪酸的成分及含量。
目前,在生物浸出过程中常用振荡、水浴与离心相结合的方法提取胞外蛋白,采用双向电泳与质谱的联用的技术对纯培养条件下微生物体内或体外的一些功能蛋白进行鉴定。由于胞外蛋白的提取与分离方法的复杂性,目前关于胞外蛋白的分离纯化及鉴定还没有统一的规定,选择适宜的方法分离和纯化胞外蛋白是今后生物冶金领域胞外蛋白研究工作的重点。
3.2 胞外蛋白功能基因的定量研究方法胞外蛋白功能基因定量的方法有很多种,参考相关文献,将常用于基因定量的研究方法汇总(表 1),以期从中选择合适的方法技术用于胞外蛋白功能基因的定量研究。由表 1可知,不同方法有其各自的优缺点,比较前沿的技术有核酸酶保护法、核酸依赖的扩增技术、数字PCR技术等。
在生物浸出过程中,实时荧光定量PCR技术因其操作简便、灵敏度高、重复性好等优点,被广泛地用于基因的定量分析。刘晶等[39]用RT-PCR技术检测黄铜矿浸出过程中游离菌数量与吸附菌数量在不同pH条件下的动态变化,阐明游离菌与吸附菌相互促进生长,引起黄铜矿的不断溶解。张瑞永[26]从A. ferrooxidans中挑选10个可能与硫活化相关的基因,采用RT-PCR方法对这多个可能与硫活化相关的基因进行转录水平表达差异研究发现,在元素硫中生长的细菌的硫活化相关基因的相对表达量明显上调,证实这些基因与元素硫的活化氧化相关联。在此研究背景下,RT-PCR是对生物浸出中胞外蛋白功能基因进行定量研究的有效技术。但在具体实验中要根据实验的目的、实验对象及其实验室设备等选择相应的实验方法。
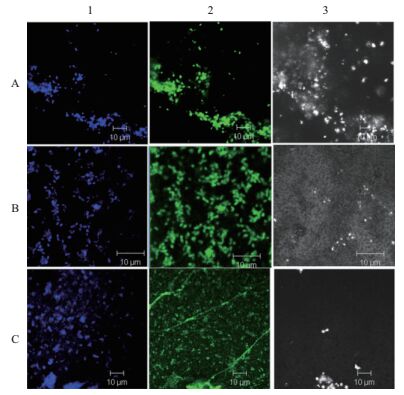
3.3 胞外蛋白原位观察手段及方法国内外主要采用多种电镜组合技术和光谱学方法对生物膜和EPS进行进一步研究[40, 41],如激光共聚焦显微镜(CLSM)、三维荧光显微镜(3D-EEM)、原子力显微镜(AFM)、场发射扫描电镜(FE-SEM)、核磁共振光谱技术(NMR)及傅立叶变换红外光谱仪(FTIR)等。Mcswain等[41]采用CLSM观察了废水生物处理过程中厌氧颗粒的表面形态,并采用多种荧光染料对预处理后的厌氧颗粒染色。Garny等[42]将共聚焦激光扫描电镜技术与核磁共振光谱技术结合起来,用于研究生物膜的结构、组成和分子流动性。Zhang等[43]采用CLSM、AFM等研究了生物浸出过程中极端嗜酸古菌F. acidiphilum在矿物表面的聚集现象和生物膜形成情况发现,其首先吸附到黄铁矿的裂缝中,并在黄铁矿表面形成单层生物膜。Boretska等[18]研究了硫细菌Thiobacillus thioparus DSM 505在钢铁表面产生EPS的情况,并采用CLSM技术观察4,6-二脒基-2-苯基吲哚(DAPI)特异性标记核酸及PWM凝集素特异性标记多聚乙酰葡萄糖胺(PNAG)在生物腐蚀过程中的变化,研究发现生物膜在金属表面建立相关的界面空间以促进生物腐蚀反应的发生(图 3)。
在今后对生物浸出过程中胞外蛋白的研究,通过对CLSM与3D-EEM、FTIR等电镜技术的联用,可以较为准确、特异性地分析生物浸出过程中胞外蛋白的生成及含量变化,从而为实时定量分析胞外蛋白在细菌与矿物界面中的作用及进一步探究浸矿菌的作用机理奠定理论基础。
4 展望近年来,随着对不同领域胞外蛋白研究的深入,关于胞外蛋白的研究越来越多,提取胞外蛋白的方法及研究胞外蛋白的技术也都趋于成熟。但在生物浸矿领域,对胞外多聚物关键组分胞外蛋白的研究存在很多困难,主要集中在以下几个方面:
(1)目前对EPS的提取方法还没形成统一的标准。这就导致了用不同方法提取的胞外蛋白的量及性质各不相同,从而造成对胞外蛋白研究结果的差异;(2)EPS中的关键组分胞外蛋白的分离纯化方法。在生物浸矿领域,提取浸矿菌EPS的量本身较少,从提取的EPS中分离纯化出胞外蛋白的量就更少,若胞外蛋白分离纯化的方法不当,会严重影响后面对胞外蛋白的研究,因此选择适当的胞外蛋白分离纯化方法也是本课题的一大挑战;(3)胞外蛋白功能特性方面的研究。对蛋白质功能特性的研究主要是通过使用现代仪器与基因组学、蛋白质组学、转录组学等结合的方法。但这些方法操作较为繁琐,技术还不是很成熟,要对浸矿菌EPS中的胞外蛋白进行深入研究还需要进一步的探究。
虽然对矿物表面胞外多聚物关键组分胞外蛋白的提取方法和技术需要完善,但通过实验的探究、现代技术与方法的联用,对浸矿菌胞外蛋白的研究存在很大的发展空间。此外,通过对浸矿菌EPS胞外蛋白关键组分胞外蛋白的深入研究可以深层次观察和分析生物浸矿过程中矿物-溶液-微生物的多相界面反应,揭示和阐明尚存争议的生物浸矿机理,为生物浸矿工业的快速发展奠定一定的理论基础。
| [1] | Boukahil I, Czuprynski CJ. Characterization of Mannheimia haemolytica biofilm formation in vitro[J]. Veterinary Microbiology, 2015, 175(1): 114–122. DOI:10.1016/j.vetmic.2014.11.012 |
| [2] | Flemming HC, Wingender J. The biofilm matrix[J]. Nature Reviews Microbiology, 2010, 8(9): 623–633. |
| [3] | Garcia-Meza J, Fernandez J, Lara R, et al. Changes in biofilm structure during the colonization of chalcopyrite by Acidithiobacillus thiooxidans[J]. Appl Microbiol Biotechnol, 2013, 97(13): 6065–6075. DOI:10.1007/s00253-012-4420-6 |
| [4] | Sampson M, Phillips C, Blake R. Influence of the attachment of acidophilic bacteria during the oxidation of mineral sulfides[J]. Minerals Engineering, 2000, 13(4): 373–389. DOI:10.1016/S0892-6875(00)00020-0 |
| [5] | Zeng W, Qiu G, Zhou H, et al. Community structure and dynamics of the free and attached microorganisms during moderately thermophilic bioleaching of chalcopyrite concentrate[J]. Bioresource Technology, 2010, 101(18): 7068–7075. DOI:10.1016/j.biortech.2010.04.003 |
| [6] | Zhang CG, Zhang RY, Xia JL, et al. Sulfur activation-related extracellular proteins of Acidithiobacillus ferrooxidans[J]. Transactions of Nonferrous Metals Society of China, 2008, 18(6): 1398–1402. DOI:10.1016/S1003-6326(09)60015-7 |
| [7] | Zhang R, Bellenberg S, Castro L, et al. Colonization and biofilm formation of the extremely acidophilic archaeon Ferroplasma acidiphilum[J]. Hydrometallurgy, 2014, 150: 245–252. DOI:10.1016/j.hydromet.2014.07.001 |
| [8] | Guo X, Yin H, Liang Y, et al. Comparative genome analysis reveals metabolic versatility and environmental adaptations of Sulfobacillus thermosulfidooxidans Strain ST[J]. PLoS One, 2014, 9(6): e99417–e99417. DOI:10.1371/journal.pone.0099417 |
| [9] | Morales MA. Extracellular polymeric substances(EPS)production in Sulfobacillus thermosulfidooxidans and its relevance on attachment to metal sulfides[D]. New York:Universidad Nacional de Colombia, 2012. |
| [10] | Lasa I, Penades JR. Bap:a family of surface proteins involved in biofilm formation[J]. Res Microbiol, 2006, 157(2): 99–107. DOI:10.1016/j.resmic.2005.11.003 |
| [11] | Yu GH, He PJ, Shao LM, et al. Extracellular polymeric substances(EPS)and extracellular enzymes in aerobic granules[J]. Drying Technology, 2010, 28(7): 910–915. DOI:10.1080/07373937.2010.490766 |
| [12] | Branda SS, Chu F, Kearns DB, et al. A major protein component of the Bacillus subtilis biofilm matrix[J]. Molecular Microbiology, 2006, 59(4): 1229–1238. DOI:10.1111/mmi.2006.59.issue-4 |
| [13] | Gilboa-Garber N, Mizrahi L, Garber N. Purification of the galactose-binding hemagglutinin of Pseudomonas aeruginosa by affinity column chromatography using sepharose[J]. FEBS letters, 1972, 28(1): 93–95. DOI:10.1016/0014-5793(72)80685-9 |
| [14] | Gilboa-Garber N, Katcoff DJ, Garber NC. Identification and characterization of Pseudomonas aeruginosa PA-IIL lectin gene and protein compare d to PA-IL[J]. FEMS Immunology & Medical Microbiology, 2000, 29(1): 53–57. |
| [15] | Imberty A, Wimmerova M, Mitchell EP, et al. Structures of the lectins from Pseudomonas aeruginosa:insights into the molecular basis for host glycan recognition[J]. Microbes and Infection, 2004, 6(2): 221–228. DOI:10.1016/j.micinf.2003.10.016 |
| [16] | Diggle SP, Stacey RE, Dodd C, et al. The galactophilic lectin, LecA, contributes to biofilm development in Pseudomonas aeruginosa[J]. Environ Microbiol, 2006, 6: 1095–1104. |
| [17] | Park C, Novak JT, Helm RF, et al. Evaluation of the extracellular proteins in full-scale activated sludges[J]. Water Research, 2008, 42(14): 3879–89. DOI:10.1016/j.watres.2008.05.014 |
| [18] | Boretska M, Bellenberg S, Moshynets O, et al. Change of extracell-ular polymeric substances composition of Thiobacillus thioparus in presence of sulfur and steel[J]. Microbial & Biothemical Technolog, 2013, 5(3): 068–073. |
| [19] | Rendueles O, Beloin C, Latourlambert P, et al. A new biofilm associated colicin with increased efficiency against biofilm bacteria[J]. Isme Journal, 2014, 8(6): 1275–1288. DOI:10.1038/ismej.2013.238 |
| [20] | Wolffe AP. Structural and functional properties of the evolutionarily ancient Y-box family of nucleic acid binding proteins[J]. Bioessays, 1994, 16(4): 245–251. DOI:10.1002/(ISSN)1521-1878 |
| [21] | Harami GM, Gyimesi M, Kovacs M. From keys to bulldozers:expanding roles for winged helix domains in nucleic-acid-binding proteins[J]. Trends Biochem Sci, 2013, 38(7): 364–371. DOI:10.1016/j.tibs.2013.04.006 |
| [22] | Li ZH, Kuba T, Kusuda T. The influence of starvation phase on the properties and the development of aerobic granules[J]. Enzyme & Microbial Technology, 2006, 38(5): 670–674. |
| [23] | Tielen P, Rosenau F, Wilhelm S, et al. Extracellular enzymes affect biofilm formation of mucoid Pseudomonas aeruginosa[J]. Microbiology, 2010, 156(7): 2239–2252. DOI:10.1099/mic.0.037036-0 |
| [24] | Higgins MJ, Novak JT. Characterization of exocellular protein and its role in bioflocculation[J]. Journal of Environmental Engineering, 1997, 123(5): 479–485. DOI:10.1061/(ASCE)0733-9372(1997)123:5(479) |
| [25] | 张丽丽.姜理英, 方芳, 等. 好氧颗粒污泥胞外多聚物的提取及成分分析[J]. 环境工程学报, 2007, 1(4): 127–130. |
| [26] | 张瑞永. Acidithiobacillus ferrooxidans ATCC 23270硫活化相关胞外蛋白质研究[D]. 长沙: 中南大学, 2009. |
| [27] | Yang J, He Y, Jiang J, et al. Comparative proteomic analysis by iTRAQ-2DLC-MS/MS provides insight into the key proteins involved in Cronobacter sp. biofilm formation[J]. Food Control, 2016, 63: 93–100. DOI:10.1016/j.foodcont.2015.11.029 |
| [28] | Zeng W, Qiu G, Zhou H, et al. Characterization of extracellular polymeric substances extracted during the bioleaching of chalcopyrite concentrate[J]. Hydrometallurgy, 2010, 100(3): 177–180. |
| [29] | Felkin LE, Taegtmeyer AB, Barton PJ. Real-time quan-titative polymerase chain reaction in cardiac transplant research[J]. Methods Mol Biol, 2006, 333: 305–330. |
| [30] | 任广睦, 刘季, 王英元. 实时荧光定量PCR技术应用于核酸定量检测的研究进展及展望[J]. 山西医科大学学报, 2007, 37: 973–976. |
| [31] | 余润兰, 刘亚楠, 周丹, 等. 生物浸出过程中的藻酸盐作用及其机理的研究进展[J]. 中国有色金属学报, 2015, 25(6): 1687–1693. |
| [32] | Berk AJ, Sharp PA. Sizing and mapping of early adenovirus mRNAs by gel electrophoresis of S1 endonuclease-digested hybrids[J]. Cell, 1977, 12(3): 721–732. DOI:10.1016/0092-8674(77)90272-0 |
| [33] | 孔毅, 吴如金, 吴梧桐. 高效毛细管电泳及其在蛋白质、多肽分析中的应用[J]. 药学进展, 2000, 24(4): 204–208. |
| [34] | Green CD, Simons JF, Taillon BE, Lewin DA. Open systems:panoramic views of gene expression[J]. J Immunol Methods, 2001, 250: 67–79. DOI:10.1016/S0022-1759(01)00306-4 |
| [35] | Deiman B, Aarle P, Sillekens P. Characteristics and applications of nucleic acid sequence-based amplification(NASBA)[J]. Molecular Biotechnology, 2002, 20(1): 160–179. |
| [36] | Weusten JAM, Carpay WM, Tom AM, et al. Principles of quantitation of viral loads using nucleic acid sequence-based amplification in combination with homogenous detection using molecular beacons[J]. Nucleic Acids Res, 2002, 30(6): e26. DOI:10.1093/nar/30.6.e26 |
| [37] | Ma J, Li N, Guarnera M, et al. Quantification of plasma miRNAs by Digital PCR for cancer diagnosis[J]. Biomark Insights, 2013, 8: 127–136. |
| [38] | Sanders R, Huggett JF, Bushell CA, et al. Evaluation of digital PCR for absolute DNA quantification[J]. Anal Chem, 2011, 83: 6474–648. DOI:10.1021/ac103230c |
| [39] | 刘晶. pH对嗜酸氧化亚铁硫杆菌分泌胞外多聚物及其吸附性能的影响[D]. 长沙: 中南大学, 2013. |
| [40] | 游雪娇, 李良秋, 马连营, 等. 激光共聚焦扫描显微镜在抗菌机理研究中的应用[J]. 微生物学通报, 2015, 4(6): 1108–1121. |
| [41] | Mcswain B, Irvine R, Hausner M, et al. Composition and distribution of extracellular polymeric substances in aerobic flocs and granular sludge[J]. Applied and Environmental Microbiology, 2005, 71(2): 1051–1057. DOI:10.1128/AEM.71.2.1051-1057.2005 |
| [42] | Garny K, Neu T, Horn H, et al. Combined application of 13 C NMR spectroscopy and confocal laser scanning microscopy-Investigation on biofilm structure and physico-chemical properties[J]. Chemical Engineering Science, 2010, 65(16): 4691–4700. DOI:10.1016/j.ces.2010.05.013 |
| [43] | Marabita F, Candia P, Torri A, et al. Normalization of circulating microRNA expression data obtained by quantitative real-time RT-PCR[J]. Briefings in Bioinformatics, 2015, 5(3): 68–73. |