原发性肝癌的发病率居全球第5位,致死率排第3位,其中以肝细胞癌(Hepatocellular carcinoma,HCC)最为常见[1]。MicroRNA是一类长度为18-24个核苷酸的小分子非编码RNA,从线虫到人类的多种生物种均有发现。它们可通过结合于靶mRNA非翻译区(UTR),从而调控mRNA的翻译和降解。大量研究表明,肝细胞中miRNA表达异常或功能失调与其细胞增殖、细胞周期、分化、病毒复制、肝细胞癌变的发生等生理与病理学过程密切相关。p53(Tumor protein 53,TP53)作为一种肿瘤抑制转录因子参与调控绝大多数肿瘤的发生与发展。前期研究证实,在抗肿瘤药物阿霉素(Doxorubicin,Dox)诱导下可激活p53从而调控包括microRNA在内的下游靶基因的转录调控[2]。本课题组前期通过Dox诱导肝癌HepG2细胞后进行小RNA测序(small RNA sequencing)分析发现了一系列受p53调控的miRNA[3]。通过qRT-PCR实验证实,Dox处理HepG2细胞24 h后,miR-3661表达量上调10.17倍[3],通过文献搜索并未有相关报道。基于上述研究以及前期分析,本研究通过生物信息学分析与分子生物学实验手段对miR-3661在肝癌中的调控作用进行系统的分析,并从系统生物学角度发掘miR-3661与其他相关基因形成的调控网络,旨在通过实验证明miR-3661对肝癌细胞的抑制作用,为进一步探明miRNA与肝肿瘤的发病机理奠定了理论与实验基础。
1 材料与方法 1.1 材料Hsa-miR-3661序列及结构下载自miRbase(版本21)(http://www.mirbase.org/)[4]和UCSC(https://genome.ucsc.edu/),人类蛋白相互作用数据共268组下载自BioGRID3.4数据库(https://thebiogrid.org/)[5]。
人肝癌细胞HepG2由本实验室传代保管;FBS购自法国Biowest公司;DMEM培养基购自美国Gibco公司;hsa-miR-3661 mimic、miRNA mimic control和miRNA negative control(5Cy3)购自广州锐博;酶标仪购自美国BioTek公司;Cell Counting Kit-8试剂盒购自美国Sigma公司;培养箱购自日本Panasonic公司。
1.2 方法 1.2.1 miR-3661的基因预测与功能富集分析使用miRanda(版本Aug.2010)(http://www.microrna.org/)[6]对miR-3661靶基因进行预测,实验证实的靶基因获取自miRTarBase(6.0版本)数据库。使用DAVID(6.7版本)[7]对miR-3661靶基因进行GO[8]与KEGG通路富集分析,以P值 < 0.05作为显著性阈值。
1.2.2 miR-3661的p53结合位点筛选与p53-miR-3661调控网络构建设定miRNA的TSS上游10 kb和下游1 kb作为miRNA启动子区域。将基于p53 ChIP-seq数据[9]获取到的p53结合位点与miR-3661的启动子区进行比对确定miR-3661的p53结合位点,整合miR-3661靶基因与BioGRID蛋白相互作用数据,使用Cytoscape(版本3.3.0)[10]软件构建调控网络。
1.2.3 miR-3661对HepG2细胞增殖的影响设置HepG2细胞hsa-miR-3661 mimic转染和HepG2细胞荧光阴性对照mimic转染,细胞处理72 h后进行CCK-8细胞增殖检测,计算生长抑制率:生长抑制率(%)=(1-实验组平均OD值/对照组平均OD值)×100%。
2 结果 2.1 hsa-miR-3661基本信息通过Dox诱导肝癌HepG2细胞,进行小RNA测序(small RNA sequencing)(两次生物学重复)后发现了33条受p53调控的显著差异表达miRNA。筛选标准:(1)在两组样品中表达倍数变化均满足|log2(fold change)|> 1;(2)至少在一个样品中reads数大于10;(3)至少在一个样品中满足P < 0.01。我们发现hsa-miR-1277、hsa-miR-146b、hsa-miR-3661、hsa-miR-3662、hsa-miR-371a、hsa-miR-4485和hsa-miR-4521这7条miRNA在先前的肝癌研究中并未有相关报道。通过qRT-PCR证实发现,在Dox处理HepG2细胞24 h后,miR-3661表达量上调显著(上调10.17倍),因此推测miR-3661在肝癌细胞HepG2的调控中可能发挥重要作用。为此选取miR-3661对其进行了一系列的分析与验证。首先我们对miR-3661的基本信息进行了分析,miR-3661位于人类的第5号染色体,前体长96 bp,具有典型的茎环结构,染色体定位:chr5:134225757-134225852[+],成熟序列:UGACCUGGGACUCGGACAGCUG。在UCSC基因组浏览器中显示miR-3661具有高度的保守性(图 1)。
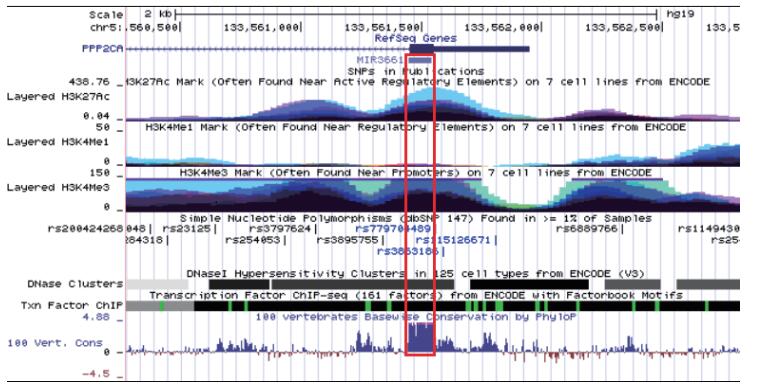
|
| 图 1 hsa-miR-3661组蛋白修饰、临近基因、保守性 |
MiRanda软件预测得到靶基因1 009个,miRTarBase实验证实靶基因30个。通过DAVID对miR-3661的靶基因进行功能富集分析表明,hsa-miR-3661的靶基因显著富集于p53调控肿瘤相关生物学进程,如细胞凋亡、细胞周期阻滞、细胞迁移、细胞生长、细胞黏附、细胞分化和增殖(图 2)。其中SMAD3、GSK3B、MSX1、THBS1、TGFBR1、PTEN、WNT4、CDKN1A及TFAP4参与了上述绝大多数的生物学进程。

|
| 图 2 369个靶基因参与20个癌症相关生物学功能热图 |
通过对KEGG通路进行富集分析表明,hsa-miR-3661的靶基因显著富集于癌症信号通路、MAPK信号通路和ErbB信号通路肿瘤相关通路(图 3)。如miRNA-3661富集到pathway in cancer通路的有34个靶基因:CYCS、akt2、TGFB3、bcr、RXRA、wnt5b、TRAF5、AXIN2、ARNT、NFKB2、FGF2、Egf、PTENP1、PPARG、tgfbr、TGFA、GSTP1、egfr、Epas1、Stat5b、NFKB1、rassf5、WNT4、Skp2、sufU、ARNT2、AXIN1、SMAD3、PTK2、IGF1R、HIF1A、CDKN1A、GSK3B和DAPK1。其中AXIN1、SMAD3、PTK2、IGF1R、HIF1A、CDKN1A、GSK3B和DAPK1这8个基因同时也是p53的靶基因。
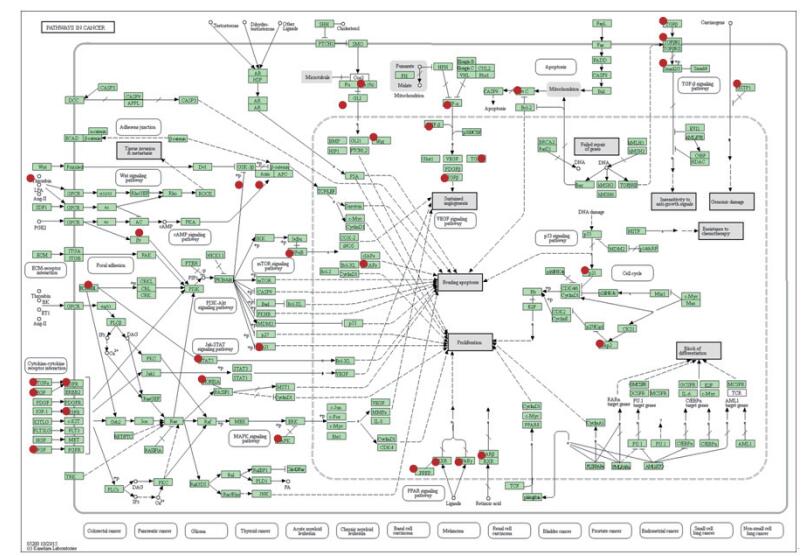
|
| 图 3 癌症中的信号通路 红色点代表含有靶基因的进程 |
将5个p53 ChIP-seq的p53转录位点(TFBS)数据与miR-3661的启动子区域对比后发现在miR-3661的启动子区存在一个p53结合位点,在5号染色体的133561448-133561543区域,位于TSS下游79 kb处(图 4),p53-DNA结合序列为:GCTGCAGCTGCTTGTGG。这一结果说明p53直接相互作用于miR-3661,并且诱导miR-3661表达上调从而调控下游靶基因功能。
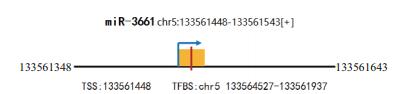
|
| 图 4 miR-3661转录启动位点与p53结合位点 miR-3661标为橙色,p53结合位点标为红色 |
MiR-3661靶基因参与的网络复杂,为了提取和p53以及肿瘤相关的子网络,我们对参与p53关键生物进程(细胞周期、细胞凋亡与细胞增殖)的miR-3661的143个靶基因进行了调控网络图绘制(图 5)。从BioGRID获取了这143个靶基因的268组人蛋白相互作用数据,使用Cytoscape软件对p53、miR-3661及参与细胞周期、细胞凋亡与细胞增殖的靶基因进行了网络构建。从图中可以看到,MSX1、HIF1A、MELK、SOD2、HIPX2、CYP1B1和CLU同时参与细胞凋亡和细胞增殖的调控;BTG2、TFAP4、THBS1、ING4、E4F1、MDM4、GFBR1和SMAD3同时参与细胞周期与细胞增殖的调控;MYBBP1A参与细胞周期与细胞凋亡的调控;而ERN1和CDKN1A同时参与细胞周期、细胞凋亡与细胞增殖的调控。

|
| 图 5 p53-miRNA-mRNA调控网络图 红色三角代表p53,紫色长方形代表miR-3661,粉色六边形为3种癌症相关生物学功能,其余为靶基因,其中绿色基因参与2种功能的调控,黄色基因参与3种 |
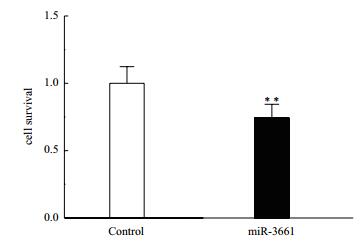
通过转染miR-3661来检验其是否对肝肿瘤细胞HepG2有明显抑制作用。首先通过荧光显微镜观察转染效率(图 6),发现荧光阴性对照mimic成功转入至HepG2细胞内,在绿色荧光的激发下呈红色,转染效率较高,可达80%以上。检测结果(图 7)表明,在HepG2中过表达miR-3661显著抑制了细胞的增殖过程,抑制率为25.70%,P-value = 0.001 460 497,差异显著。
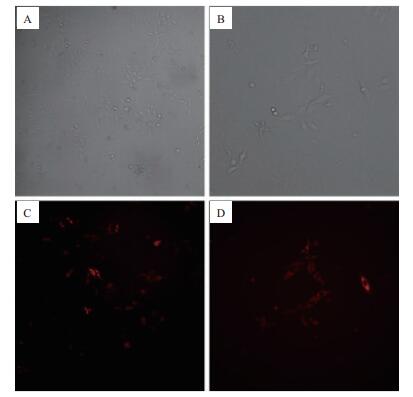
|
| 图 6 HepG2细胞转染效率 A、B为普通光源,C、D是绿光荧光视野;A、C为同一个视野(100×),B、D为同一个视野(200×) |
|
| 图 7 hsa-miR-3661抑制HepG2细胞增殖 |
本文从生物信息学与分子生物学实验两个角度证实hsa-miR-3661作为p53调控因子参与了肝癌细胞HepG2的肿瘤相关进程并抑制其增殖。MiRNA-3661位于人类的5号染色体上,发现其前体RNA(pre-miRNA)具有典型的茎环结构,经过加工,形成22 nt的单链RNA,这一长度与大多数的miRNA的长度一致。通过miRanda预测得到1 009个miRNA靶基因,其中有5条已被实验证实。对靶基因进行GO功能富集分析表明,369个miR-3661的靶基因显著富集于细胞周期、细胞凋亡、癌症通路、细胞增殖等多种与肿瘤相关的生物学进程中。这些参与生物进程的基因中,有研究表明TGF-β1会通过p53的表达上调来促进细胞周期蛋白激酶CDK的活性来调控细胞周期,而SMAD3会终止这一过程[11];THBS1作为肿瘤特异性的细胞外基质蛋白,已发现在口腔鳞状细胞癌中促进癌细胞迁移[12];而TFAP2C可通过上调TGFBR1进而激活PAK1信号从而促进肿瘤发展[13];PTEN作为公认的抑癌基因,在多种癌症中表现出对癌细胞增殖的负调控;WNT4属于WNT蛋白,在动物发育中起到重要作用,表达异常可能引起肿瘤;TFAP4参与调节众多细胞增殖和基因表达,在结肠直肠癌和其他一些人类恶性肿瘤中表达上调,与有丝分裂的保真性密切相关[14]。同样是在结肠直肠癌,研究发现GSK3B驱动了结肠直肠癌向IV期的发展[15]。使用KEGG通路分析发现miR-3661靶基因参与3种肿瘤相关通路。癌症通路共富集34个靶基因,P值为0.000 48;ErbB信号通路共富集21个靶基因,P值为0.00 79;MAPK信号通路共富集23个靶基因,P值为0.037。在参与的通路中,已知AXIN1是抑癌基因,在Wnt通路中可作为抑制剂来下调信号通路中主要的效应分子[16];PTK2的高表达与肿瘤复发相关[17];IGF1R的表达上调会促进肿瘤的发生[18];而在对胃癌的相关报道中,HIF1A的高表达会促进胃癌细胞血管的再生,导致预后差[19];DAPK1(死亡相关蛋白激酶1)具有肿瘤抑制功能,并介导多种细胞过程,包括细胞凋亡和自噬[20]。
结合p53ChIP-seq数据进行分析,我们发现miRNA-3661的假定启动子区存在一个p53结合位点,位于5号染色体的133561448-133561543区域,TSS下游79 kb处,且处于miR-3661转录本的茎环结构域,这种情况在研究人类基因保守的TFBS时曾被报道过:TFBS可位于miRNA的pre-miRNA区域[21]。为了从系统生物学角度阐明p53、miR-3661以及其靶基因之间的调控关系,我们构建了三者的调控网络图。从图中我们发现有两个基因:ERN1和CDKN1A,它们可同时被p53与miRNA-3661调节并可作用于p53关键的生物学进程:细胞周期、细胞凋亡和细胞增殖。已在乳腺癌的研究中证实,使用激酶抑制剂抑制ERN1后,会使乳腺癌细胞致瘤性下降,与Strietz等人发现的在乳腺癌中ERN1能够抑制潜在的肿瘤启始细胞现象一致[22]。同时CDKN1A(p21)也已经有实验证实可以作为抑制前列腺癌增长的药物靶向位点[23]。
前期我们通过realtimePCR实验,证实经阿霉素处理24 h后HepG2细胞中miR-3661表达量相比对照组上调了10.17倍,这与我们前期的小RNA测序结果一致。p53作为肿瘤抑制转录因子其正调控的靶基因在p53表达上调后应当呈现与p53一致的生物学功能,如参与抑制细胞增殖。miR-3661作为p53的正调控靶基因,其生物学功能也应当具备抑制细胞增殖的能力,为此通过CCK-8细胞增殖实验证实在HepG2中过表达miR-3661显著抑制了细胞的增殖过程,抑制率为25.70%,这与预期的miR-3661功能相一致。
4 结论MiRNA-3661序列保守并存在p53结合位点,显著参与肿瘤相关细胞周期调控、细胞增殖、细胞凋亡等肿瘤相关生物学过程,同时也显著参与癌症信号通路、MAPK信号通路与ErbB信号通路。p53、hsa-miR-3661和靶基因的调控网络从系统生物学角度阐述了参与肿瘤生物进程的关键靶基因。实验证实过表达miR-3661可以显著抑制肝癌细胞HepG2的增殖过程(P-value= 0.00146)。
| [1] | Yang JD, Roberts LR. Epidemiology and management of hepatocellular carcinoma[J]. Infectious Disease Clinics of North America, 2010, 24(4): 899–919. DOI:10.1016/j.idc.2010.07.004 |
| [2] | Liu J, Zhang C, Feng Z. Tumor suppressor p53 and its gain-of-function mutants in cancer[J]. Acta Biochimica et Biophysica Sinica, 2014, 46(3): 170–179. DOI:10.1093/abbs/gmt144 |
| [3] | Yang Y, Liu W, Ding R, et al. Comprehensive expression profiling and functional network analysis of p53-regulated MicroRNAs in HepG2 Cells treated with doxorubicin[J]. PLoS One, 2016, 11(2): e0149227. DOI:10.1371/journal.pone.0149227 |
| [4] | Agarwal V, Bell GW, Nam JW, Bartell DP. Predicting effective microRNA target sites in mammalian mRNAs[J]. Elife, 2015, 4: e05005. |
| [5] | Chatr-Aryamontri A, Breitkreutz BJ, Oughtred R, et al. The BioGRID interaction database:2015 update[J]. Nucleic Acids Res, 2015, 43(Database issue): D470–478. |
| [6] | Betel D, Koppal A, Agius P, et al. Comprehensive modeling of microRNA targets predicts functional non-conserved and non-canonical sites[J]. Genome Biology, 2010, 11(8): R90. DOI:10.1186/gb-2010-11-8-r90 |
| [7] | Huang DW, Sherman BT, Lempicki RA. Bioinformatics enrichment tools:paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Research, 2009, 37(1): 1–13. DOI:10.1093/nar/gkn923 |
| [8] | Vlachos IS, Paraskevopoulou MD, Karagkouni D, et al. DIANA-TarBase v7. 0:indexing more than half a million experimentally supported miRNA:mRNA interactions[J]. Nucleic Acids Research, 2015, 43(D1): D153–D159. DOI:10.1093/nar/gku1215 |
| [9] | Zeron-Medina J, Wang X, Repapi E, et al. A polymorphic p53 response element in KIT ligand influences cancer risk and hsa undergone natural selection[J]. Cell, 2013, 155(2): 410–422. DOI:10.1016/j.cell.2013.09.017 |
| [10] | Shannon P, Markiel A, Ozier O, et al. Cytoscape:a software environment for integrated models of biomolecular interaction networks[J]. Genome Research, 2003, 13(11): 2498–2504. DOI:10.1101/gr.1239303 |
| [11] | Park SJ, Yang SW, Kim BC. Transforming growth factor-beta1 induces cell cycle arrest by activating atypical cyclin-dependent kinase 5 through up-regulation of Smad3-dependent p35 expression in human MCF10A mammary epithelial cells[J]. Biochem Biophys Res Commun, 2016, 472(3): 502–507. DOI:10.1016/j.bbrc.2016.02.121 |
| [12] | Pal SK, Nguyen CT, Morita KI, et al. THBS1 is induced by TGFB1 in the cancer stroma and promotes invasion of oral squamous cell carcinoma[J]. J Oral Pathol Med, 2016, 45(10): 730–739. DOI:10.1111/jop.2016.45.issue-10 |
| [13] | Kim W, Kim E, Lee S, et al. TFAP2C-mediated upregulation of TGFBR1 promotes lung tumorigenesis and epithelial-mesenchymal transition[J]. Exp Mol Med, 2016, 48(11): e273. DOI:10.1038/emm.2016.125 |
| [14] | D'Annibale S, Kim J, Magliozzi R, et al. Proteasome-dependent degradation of transcription factor activating enhancer-binding protein 4(TFAP4) controls mitotic division[J]. J Biol Chem, 2014, 289(11): 7730–7737. DOI:10.1074/jbc.M114.549535 |
| [15] | Palaniappan A, Ramar K, Ramalingam S. Computational identification of novel stage-specific biomarkers in colorectal cancer progression[J]. PLoS One, 2016, 11(5): e0156665. DOI:10.1371/journal.pone.0156665 |
| [16] | Pećina-Šlaus N, Kafka A, Vladušić T, et al. AXIN1 expression and localization in meningiomas and association to changes of APC and e-cadherin[J]. Anticancer Res, 2016, 36(9): 4583–4594. DOI:10.21873/anticanres |
| [17] | Sethuraman A, Brown M, Seagroves TN, et al. SMARCE1 regulates metastatic potential of breast cancer cells through the HIF1A/PTK2 pathway[J]. Breast Cancer Res, 2016, 18(1): 81. DOI:10.1186/s13058-016-0738-9 |
| [18] | Yasumoto M, Sakamoto E, Ogasawara S, et al. Muscle RAS oncogene homolog(MRAS)recurrent mutation in Borrmann type IV gastric cancer[J]. Cancer Med, 2017, 6(1): 235–244. DOI:10.1002/cam4.2017.6.issue-1 |
| [19] | Wu Y, Yun D, Zhao Y, et al. Down regulation of RNA binding motif, single-stranded interacting protein 3, along with up regulation of nuclear HIF1A correlates with poor prognosis in patients with gastric cancer[J]. Oncotarget, 2017, 8(1): 1262–1277. |
| [20] | Singh P, Ravanan P, Talwar P. Death associated protein kinase 1(DAPK1):a regulator of apoptosis and autophagy[J]. Front Mol Neurosci, 2016, 9: 46. |
| [21] | Piriyapongsa J, Jordan IK, Conley AB, et al. Transcription factor binding sites are highly enriched within microRNA precursor sequences[J]. Biology Direct, 2011, 6: 61. DOI:10.1186/1745-6150-6-61 |
| [22] | Strietz J, Stepputtis SS, Precac BT, et al. ERN1 and ALPK1 inhibit differentiation of bi-potential tumor-initiating cells in human breast cancer[J]. Oncotarget, 2016, 7(50): 83278–83293. |
| [23] | Guo H, Xu Y, Fu Q. Curcumin inhibits growth of prostate carcinoma via miR-208-mediated CDKN1A activation[J]. Tumor Biology, 2015, 36(11): 8511–8517. DOI:10.1007/s13277-015-3592-y |




