桂花 (Osmanthus fragrans Lour.) 是我国十大传统名花之一,也是我国重要的园林植物之一。根据花期桂花可分为秋桂和四季桂两大类,秋桂又可根据花色分为金桂 (黄色至金黄色系)、银桂 (黄白色至黄色系) 和丹桂 (橙色至橙红色系) 三大品种群[1]。前人通过对秋桂不同花色品种花瓣呈色物质成分进行分析发现,类胡萝卜素 (Carotenoid) 的种类及其含量是影响秋桂花色呈现的最主要因素[2, 3]。其中,叶黄素 (Lutein)、α-胡萝卜素 (α-carotene) 和β-胡萝卜素 (β-carotene) 这3种类胡萝卜素的含量与秋桂花瓣颜色的呈现紧密相关[3]。
类胡萝卜素属于萜烯类化合物,呈现黄色、橙红色和红色,是观赏植物花瓣的重要呈色物质之一[4]。根据类胡萝卜素的化学结构,主要分为胡萝卜素和叶黄素类化合物两大类[5]。到目前为止,通过各种生物学方法和手段已经分离和鉴定了参与植物类胡萝卜素生物合成的全部基因[4, 6]。
类胡萝卜素异构酶 (Carotenoid isomerase,CRT-ISO) 能催化前番茄红素 (7,9,7',9'-四顺式-番茄红素,7,9,7',9'-tetra-cis-lycopene) 生成全反式番茄红素 (All-trans-lycopene)[7, 8]。植物CRTISO基因首先在拟南芥 (Arabidopsis thaliana)[9]和番茄 (Lycopersicon esculentum)[10]中被分离和鉴定。Park等[9]在拟南芥中定位并克隆了CRTISO基因,将其转化大肠杆菌,证实CRTISO具有催化多顺式类胡萝卜素向全反式类胡萝卜素异构化的功能。相对于野生型番茄,Isaacson等[10]发现CRTISO缺失突变体tangerinemic和CRTISO低丰度表达突变体tangerine3183果实中积累大量前番茄红素和ζ-胡萝卜素,而番茄红素和β-胡萝卜素含量较低。
此后,除了模式植物和园艺作物[11-13]以外,许多观赏植物的CRTISO基因也相继被分离,如菊花 (Chrysanthemum morifolium)[14]、百合 (Lilium spp.)[15]、金盏菊 (Calendula officinalis)[16]和香石竹 (Dianthus caryophyllus)[17]。通过对菊花的研究发现,富含叶黄素的黄花品种的CRTISO基因表达量显著高于叶黄素含量低的白花品种[14]。另有研究发现,不同牵牛属 (Ipomoea) 植物和百合不同品种番茄红素下游类胡萝卜素的含量与CRTISO基因表达量紧密相关[15, 18]。
本研究利用已构建的桂花‘堰虹桂’花瓣转录组数据库[19]中Unigene序列信息,结合RT-PCR技术克隆得到桂花OfCRTISO基因cDNA全长,对其基因序列及其编码氨基酸序列进行分析,并采用实时荧光定量PCR技术对该基因在‘堰虹桂’不同发育阶段花序和营养器官中以及不同花色品种花瓣中的表达模式进行分析,旨在为进一步研究桂花OfCRTISO基因的功能,探讨桂花花色形成的分子机制提供理论依据。
1 材料与方法 1.1 材料以浙江农林大学桂花资源圃地栽桂花丹桂品种‘堰虹桂’、银桂品种‘小叶苏桂’和金桂品种‘金球桂’为试验材料。
不同发育阶段花序 (图 1) 取样:2014年8月9日、8月15日、8月22日、8月29日、9月4日、9月10日、9月17日、9月23日、9月24日和9月25日对‘堰虹桂’不同发育状态花序进行取样。其中,8月9日至9月17日花序状态为花蕾期 (Bud stage),简写为B1-B7;9月23日花序状态为铃梗期 (Linggeng stage,L);9月24日花序状态为初开期 (Initial flowering stage,IF);9月25日花序状态为盛开期 (Full flowering stage,FF)。取样时间均为上午10时,花序样品离体后速冻于液氮中,于-80℃储藏备用。

|
| 图 1 桂花‘堰虹桂’不同发育阶段花序 B:花蕾期;L:铃梗期;IF:初开期;FF:盛开期 |
营养器官取样:2014年9月25日对‘堰虹桂’一年生茎 (当年生枝条先端第2至第3节之间的茎)、嫩叶 (当年生枝条先端第1对叶片) 和成熟叶 (当年生枝条先端第3对叶片) 进行取样,液氮速冻后,于-80℃储藏备用。
不同花色品种花瓣取样:2014年9月下旬对‘堰虹桂’、‘小叶苏桂’和‘金球桂’铃梗期、初开期和盛开期花序的花瓣进行取样,取样时间均为上午10时,离体后速冻于液氮中,于-80℃储藏备用。
1.2 方法 1.2.1 总RNA提取与cDNA的第一链的合成采用RNAprep pure Plant Kit (天根,北京) 按照说明书步骤提取各样品的总RNA。紫外分光光度计和1%琼脂糖凝胶电泳检测总RNA浓度和质量。采用Reverse Transcriptase M-MLV (TaKaRa,大连) 按照说明书合成cDNA的第一链后,储存于-20℃备用。
1.2.2 OfCRTISO基因的克隆根据前期课题组已构建的桂花‘堰虹桂’花瓣转录组数据库[19],从中筛选出与CRTISO基因相关序列Locus_7474,设计特异性引物F1(5'-TTACTTTTACTTGTTAACCACCG-3') 和R1(5'-GCAATCACTATTTCTAATCCTCT-3'),以‘堰虹桂’盛开期花瓣RNA反转的cDNA为模板,对桂花OfCRTISO基因最大阅读框序列进行PCR扩增。PCR反应体系:10×Buffer 2.5 μL,25 mmol/L MgCl2 1.5 μL,10 mmol/L dNTP 0.5 μL,10 μmol/L上、下游引物各0.5 μL,Taq DNA聚合酶1.25 U,cDNA 2 μL,加去离子水至25 μL。PCR反应条件:94℃预变性5 min,94℃变性30 s、60℃退火30 s、72℃延伸2.5 min,35个循环,最后72℃保温10 min,4℃结束反应。
PCR反应产物经1%琼脂糖凝胶电泳检测后回收、纯化,与pMD18-T载体 (TaKaRa) 连接,转化大肠杆菌DH5α感受态细胞,蓝白斑筛选阳性克隆,经PCR鉴定后送上海生工生物工程技术服务有限公司测序。
1.2.3 序列分析测序获得的序列通过NCBI提供的ORF Finder进行ORF查找,序列翻译、氨基酸序列比对采用DNAMAN 6.0软件进行。通过ExPASy (http://www.expasy.org/tools/protparam.html) 提供的Prot-Param在线软件对蛋白质的分子量和等电点进行分析。通过NetPhos 2.0(http://www.cbs.dtu.dk/services/NetPhos/) 对蛋白磷酸化位点进行预测;使用Plant-PLoc软件 (http://www.csbio.sjtu.edu.cn/bioinf/plant/) 对编码蛋白的亚细胞定位进行预测;使用MEGA 5.05软件中的Neighbor-Joining (邻位相连法,NJ) 法建立系统进化树。
1.2.4 实时荧光定量PCR按照Takara公司的SYBR® Premix Ex TaqTM Ⅱ(Tli RNaseH Plus) 试剂盒的说明书,采用7300 real-time PCR仪 (Applied Biosystems) 检测桂花OfCRTISO基因的相对表达量。OfCRTISO基因表达引物为F2(5'-GAAAAACAAG-GGATTCTCGGA-3') 和R2(5'-GCAGATAGTAGG-CAAGGGTCAA-3'),内参基因OfACT表达引物为F3(5'-CCCAAGGCAAACAGAGAAAAAAT-3') 和R3(5'-ACCCCATCACCAGAATCAAGAA-3')[20]。所用反应体系和程序如下,PCR反应体系为20 μL,其中2×SYBR Premix Ex Taq Ⅱ(Tli RNaseH Plus)10 μL,10 μmol/L上下游定量引物各0.8 μL,模板cDNA 2 μL,50×ROX Reference Dye 0.4 μL,用灭菌双蒸水补齐至20 μL。荧光定量PCR扩增采用两步法,即在95℃预变性30 s之后,先运行40个循环的95℃ 5 s,60℃ 31 s,再运行熔解曲线阶段的95℃15 s,60℃ 1 min,95℃ 30 s,60℃ 15 s。反应后根据熔解曲线分析PCR产物的特异性,分别导出目的基因和内参基因的Ct值,利用参照基因的ΔCt法计算目的基因的相对表达量。
2 结果 2.1 桂花OfCRTISO基因的克隆与序列分析提取所有样品总RNA均有28S和18S两条清晰条带,RNA无降解,部分样品总RNA经1%琼脂糖凝胶电泳检测如图 2所示。紫外分光光度计检测OD260/OD280均在1.9-2.0之间,浓度为210-420 ng/μL之间。以‘堰虹桂’盛开期花瓣总RNA反转的cDNA为模板,扩增出一条约2 200 bp的单一特异性条带 (图 3)。经测序获得一条长度为2 241 bp的cDNA序列 (图 4),Blast比对发现该序列与CRTISO基因同源,命名为OfCRTISO。

|
| 图 2 桂花部分样品总RNA电泳图 |

|
| 图 3 桂花OfCRTISO基因的PCR扩增电泳图 M:分子量标准;1:OfCRTISO基因片段 |

|
| 图 4 桂花OfCRTISO基因的核苷酸序列及推测的氨基酸序列 起始密码子和终止密码子用下划线表示,翻译终止以*标注。丝氨酸激酶磷酸化位点用阴影表示,苏氨酸激酶磷酸化位点用方框表示,酪氨酸激酶磷酸化位点用双横线表示 |
利用NCBI提供的ORF Finder进行分析发现,OfCRTISO包含一个长度为1 842 bp的开放读码框,5'非翻译区长50 bp,3'非翻译区长349 bp。其开放读码框编码一个含有613个氨基酸残基的蛋白质 (图 4)。
2.2 桂花OfCRTISO基因编码氨基酸的基本特征利用NCBI Conserved Domain Search进行保守域分析表明,OfCRTISO编码的蛋白含有1个carot_isom保守域 (第96-590位氨基酸残基)。利用Prot-Param软件分析桂花OfCRTISO氨基酸序列的基本特征发现,该蛋白的分子式为C3079H4818N816O875S24,相对分子量为68.04 kD,理论等电点 (pI) 为8.58。该蛋白中亮氨酸 (Leu) 最多占10.4%;色氨酸 (Trp) 含量最低,只占0.8%。
利用NetPhos 2.0软件对OfCRTISO蛋白进行磷酸化位点的分析 (图 4) 发现,该蛋白含有14个丝氨酸激酶磷酸化位点 (分别为第72、85、91、149、155、200、238、282、363、442、473、481、498和531位氨基酸),5个苏氨酸激酶磷酸化位点 (分别为第299、382、425、519和522位氨基酸) 和7个酪氨酸激酶磷酸化位点 (分别为第86、127、244、343、403、485和534位氨基酸)。经Plant-PLoc软件分析,推测OfCRTISO蛋白定位于叶绿体中。
2.3 桂花OfCRTISO基因编码氨基酸的同源性比较利用DNAMAN软件对桂花OfCRTISO及其他11个物种的CRTISO氨基酸序列进行了多重比对,发现桂花OfCRTISO与已知胡麻科和茄科植物的CRTISO氨基酸序列有很高的同源性 (图 5),其中与芝麻 (Sesamum indicum) CRTISO序列 (XP_011077785) 相似性最高,序列一致性为81%。

|
| 图 5 桂花OfCRTISO与其他物种CRTISO氨基酸序列对比 |
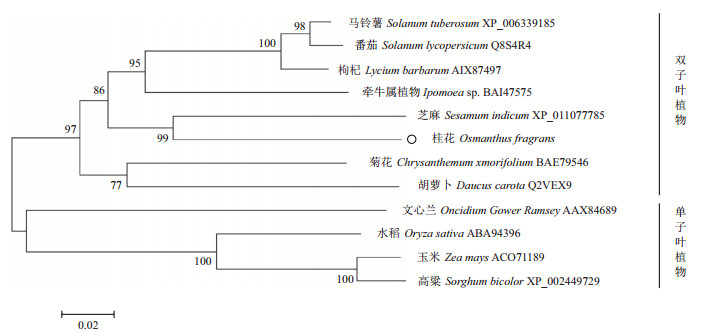
为进一步了解OfCRTISO与其他植物CRTISO之间的进化关系,用上述11个物种的CRTISO氨基酸序列与桂花OfCRTISO构建了系统进化树 (图 6) 发现,桂花OfCRTISO与其他双子叶植物CRTISO聚为一大支,而水稻 (Oryza sativa) 和玉米 (Zea mays) 等单子叶植物则聚在另一大支。马铃薯 (Solanum tuberosum)、番茄 (Solanum lycopersicum) 和枸杞 (Lycium barbarum) 等茄科植物聚为一小支,表明CRTISO氨基酸的进化与植物的进化基本一致。桂花OfCRTISO与芝麻CRTISO聚为同一小支,表明其具有更近的亲缘关系 (图 6)。
|
| 图 6 桂花OfCRTISO与其他物种CRTISO氨基酸序列的进化树分析 |
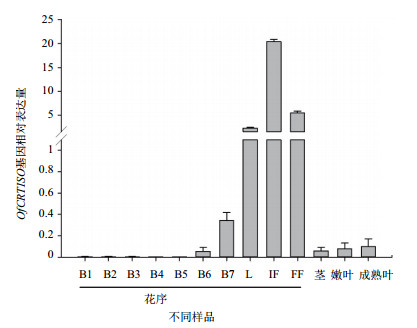
桂花OfCRTISO基因在‘堰虹桂’营养器官 (一年生茎、嫩叶和成熟叶) 和不同发育阶段花序中的表达模式如图 7所示,结果表明,OfCRTISO基因在花蕾前期 (B1-B5) 花序中的表达量极低,花蕾后期 (B6-B7) 时其表达量逐渐增加。随着花瓣展开,花序中OfCRTISO基因的表达量急剧增大,于初开期达到最大值;而当花序开放至盛开期时,其表达量显著下降。OfCRTISO基因在一年生茎、嫩叶和成熟叶片中的表达量无显著差异,与B6时期花序中表达量相近。
|
| 图 7 OfCRTISO基因在‘堰虹桂’营养器官和不同发育阶段花序中的表达量 B:花蕾期;L:铃梗期;IF:初开期;FF:盛开期 |
随着花序开放过程 (铃梗期至盛开期),OfCRTISO基因在银桂品种‘小叶苏桂’花瓣中的表达量不变,在金桂品种‘金球桂’花瓣中表达量逐渐增加,而在丹桂品种‘堰虹桂’花瓣中的表达量呈现先增加再减少的趋势 (图 8)。

|
| 图 8 OfCRTISO基因在不同花色桂花品种花瓣中的表达量 L:铃梗期;IF:初开期;FF:盛开期 |
OfCRTISO基因在铃梗期不同花色桂花品种花瓣中的表达量相近;初开期时,‘堰虹桂’花瓣中OfCRTISO基因的表达量显著高于‘金球桂’和‘小叶苏桂’,‘金球桂’花瓣中的OfCRTISO基因的表达量显著高于‘小叶苏桂’;盛开期‘金球桂’和‘堰虹桂’花瓣中OfCRTISO基因的表达量相近,均显著高于‘小叶苏桂’(图 8)。
3 讨论对于桂花而言,虽然已明确类胡萝卜素的种类与含量是影响其花色呈现的最主要因素[21],但是,桂花类胡萝卜素合成的分子机制仍不清晰[2, 3, 19]。本研究从桂花中获得了CRTISO基因cDNA全长序列,并分析该基因在‘堰虹桂’不同发育阶段花序和营养器官中以及不同花色品种花瓣中的表达模式,为探讨桂花花色形成的分子机制奠定基础。
前人研究证实CRTISO基因在拟南芥和番茄基因组中均以单拷贝形式存在[9, 10],本研究利用已构建的桂花转录组数据库只获得1个桂花CRTISO基因序列,推测CRTISO基因在桂花基因组中也可能以单拷贝形式存在。氨基酸序列对比和系统进化树分析表明桂花OfCRTISO与芝麻CRTISO (XP_011077785) 序列相似性最高,两者亲缘关系最近 (图 6)。
CRTISO可以催化前番茄红素生成全反式番茄红素[7, 8],是植物类胡萝卜素生物合成过程中一个重要的催化酶。观赏植物花瓣中类胡萝卜素合成与CRTISO基因的表达量紧密相关[14, 15, 18]。与菊花品种‘Yellow Paragon’[14]和百合品种‘Connecticut King’[15]相似,桂花‘堰虹桂’花序发育过程中,类胡萝卜素总量逐渐增加,至盛开期时达到最大[19]。本研究发现,‘堰虹桂’花序和花瓣中OfCRTISO基因的表达量均于初开期达到最大,但是盛开期时,其表达量显著下降。这与百合品种‘Connecticut King’花蕾发育过程中LhCRTISO基因的表达模式[15]一致,推测桂花‘堰虹桂’初开期OfCRTISO基因的高丰度表达对盛开期类胡萝卜素的大量积累至关重要。
百合、香石竹和菊花不同花色品种开花早期CRTISO基因的表达量均无差异[6, 14, 15],这与本研究结果相似,桂花不同花色品种铃梗期时花瓣中OfCRTISO基因的表达量也相同。而开花中期,菊花黄色花品种‘Yellow Paragon’类胡萝卜素总量和CRTISO基因的表达量则显著高于白色花品种‘Paragon’[14]。同样的,百合品种‘Connecticut king’ LhCRTISO基因的表达量于开花中期显著高于百合品种‘Montreux’,而在该时期‘Connecticut king’类胡萝卜素总量也显著高于‘Montreux’[15],表明CRTISO基因的表达量与类胡萝卜素的积累紧密相关。在本研究中,初开期时‘堰虹桂’和‘金球桂’花瓣中OfCRTISO基因的表达量急剧上升,均显著高于‘小叶苏桂’中的表达量,而‘堰虹桂’中OfCRTISO基因的表达量上升更为显著。侯丹测定‘堰虹桂’、‘金球桂’和‘小叶苏桂’花瓣中类胡萝卜素含量发现,‘堰虹桂’花瓣中类胡萝卜素总量最高,‘金球桂’次之,而‘小叶苏桂’最低[21],这可能与3个品种初开期OfCRTISO基因的表达量正相关。盛开期时,‘堰虹桂’和‘金球桂’花瓣中OfCRTISO基因的表达量相近,而类胡萝卜素含量存在差异,表明桂花初开期OfCRTISO基因的表达高低对盛开期花瓣中类胡萝卜素积累的影响更大。
4 结论桂花OfCRTISO基因含有长为1 842 bp的开放读码框,编码613个氨基酸残基。桂花OfCRTISO与已知胡麻科和茄科植物的CRTISO序列相似性最高。OfCRTISO基因在桂花‘堰虹桂’初开期表达量最高,初开期OfCRTISO基因的表达量影响盛开期各品种花瓣中类胡萝卜素的积累。
| [1] | 向其柏, 刘玉莲. 中国桂花品种图志[M]. 杭州: 浙江科学技术出版社, 2008. |
| [2] | Han Y, Li L, Dong M, et al. cDNA cloning of the phytoene synthase (PSY) and expression analysis of PSY and carotenoid cleavage dioxygenase genes in Osmanthus fragrans[J]. Biologia, 2013, 68 (2): 258–263. |
| [3] | Han Y, Wang X, Chen W, et al. Differential expression of carotenoid-related genes determines diversified carotenoid coloration in flower petal of Osmanthus fragrans[J]. Tree Genetics & Genomes, 2014, 10 (2): 329–338. |
| [4] | Zhu C, Bai C, Sanahuja G, et al. The regulation of carotenoid pigmentation in flowers[J]. Archives of Biochemistry and Biophysics, 2010, 504 (1): 132–141. DOI:10.1016/j.abb.2010.07.028 |
| [5] | Tanaka Y, Ohmiya A. Seeing is believing: engineering anthocyanin and carotenoid biosynthetic pathways[J]. Current Opinion in Biotechnology, 2008, 19 (2): 190–197. DOI:10.1016/j.copbio.2008.02.015 |
| [6] | Ohmiya A. Qualitative and quantitative control of carotenoid accumulation in flower petals[J]. Scientia Horticulturae, 2013, 163 (6): 10–19. |
| [7] | Breitenbach J, Vioque A, Sandmann G. Gene sll0033 from Synechocystis 6803 encodes a carotene isomerase involved in the biosynthesis of all-E Lycopene[J]. Zeitschrift für Naturforschung Section C-A Journal of Biosciences, 2001, 56 (9-10): 915–917. |
| [8] | Masamoto K, Wada H, Kaneko T, et al. Identification of a gene required for cis-to-trans carotene isomerization in carotenogenesis of the cyanobacterium Synechocystis sp. PCC 6803[J]. Plant & Cell Physiology, 2001, 42 (12): 1398–1402. |
| [9] | Park H, Kreunen SS, Cuttriss AJ, et al. Identification of the carotenoid isomerase provides insight into carotenoid biosynthesis, prolamellar body formation, and photomorphogenesis[J]. Plant Cell, 2002, 14 (2): 321–332. DOI:10.1105/tpc.010302 |
| [10] | Isaacson T, Ronen G, Zamir D, et al. Cloning of tangerine from tomato reveals a carotenoid isomerase essential for the production of beta-carotene and xanthophylls in plants[J]. Plant Cell, 2002, 14 (2): 333–342. DOI:10.1105/tpc.010303 |
| [11] | Chai C, Fang J, Liu Y, et al. ZEBRA2, encoding a carotenoid isomerase, is involved in photoprotection in rice[J]. Plant Molecular Biology, 2011, 75 (3): 211–221. DOI:10.1007/s11103-010-9719-z |
| [12] | Kato M, Ikoma Y, Matsumoto H, et al. Accumulation of carotenoids and expression of carotenoid biosynthetic genes during maturation in citrus fruit[J]. Plant Physiology, 2004, 134 (2): 824–837. DOI:10.1104/pp.103.031104 |
| [13] | Liu Y, Zeng S, Sun W, et al. Comparative analysis of carotenoid accumulation in two goji (Lycium barbarum L. and L. ruthenicum Murr.) fruits[J]. BMC Plant Biology, 2014, 14 (1): 269. DOI:10.1186/s12870-014-0269-4 |
| [14] | Kishimoto S, Ohmiya A. Regulation of carotenoid biosynthesis in petals and leaves of chrysanthemum (Chrysanthemum morifolium)[J]. Physiologia Plantarum, 2006, 128 (3): 436–447. DOI:10.1111/ppl.2006.128.issue-3 |
| [15] | Yamagishi M, Kishimoto S, Nakayama M. Carotenoid composition and changes in expression of carotenoid biosynthetic genes in tepals of Asiatic hybrid lily[J]. Plant Breeding, 2010, 129 (1): 100–107. DOI:10.1111/pbr.2010.129.issue-1 |
| [16] | Sanae K, Akemi O. Carotenoid isomerase is key determinant of petal color of Calendula officinalis[J]. Journal of Biological Chemistry, 2012, 287 (1): 276–285. DOI:10.1074/jbc.M111.300301 |
| [17] | Ohmiya A, Tanase K, Hirashima M, et al. Analysis of carotenogenic gene expression in petals and leaves of carnation (Dianthus caryophyllus L.)[J]. Plant Breeding, 2013, 132 (4): 423–429. DOI:10.1111/pbr.2013.132.issue-4 |
| [18] | Yamamizo C, Kishimoto S, Ohmiya A. Carotenoid composition and carotenogenic gene expression during Ipomoea petal development[J]. Journal of Experimental Botany, 2010, 61 (3): 709–719. DOI:10.1093/jxb/erp335 |
| [19] | Zhang C, Wang Y, Fu J, et al. Transcriptomic analysis and carotenogenic gene expression related to petal coloration in Osmanthus fragrans 'Yanhong Gui'[J]. Trees-Structure and Function, 2016, 30 (4): 1207–1223. DOI:10.1007/s00468-016-1359-8 |
| [20] | Zhang C, Fu J, Wang Y, et al. Identification of suitable reference genes for gene expression normalization in the quantitative real-time PCR analysis of sweet osmanthus (Osmanthus fragrans Lour.)[J]. PLoS One, 2015, 10 (8): e0136355. DOI:10.1371/journal.pone.0136355 |
| [21] | 侯丹. 桂花主要品种花香和花色及其对温度变化的响应[D]. 临安: 浙江农林大学, 2014. |




