干旱已成为遍及全球的气候问题之一,不同生态系统下的自然环境和农业生产均受到影响,而且有研究预测现在的干旱地区未来会遭受到更加频繁的干旱侵害[1]。在自然条件下营固着生长的植物,已进化出多种机制应对干旱胁迫,如瞬时响应土壤水分亏缺或提早开花躲避季节性干旱[2]。植物在缺水时,除了生理、生化等方面发生变化外[3],分子水平上的信号转导和应激相关基因的表达也会发生相应的变化[4]。近年来,随着组学技术的发展,越来越多的研究者在组学水平上研究植物抗逆响应机制[5]。蛋白质是基因表达的产物,能够反映生命活动的本质特点,因此蛋白质组学可为研究生命活动提供更多信息。研究结果显示,干旱胁迫下植物蛋白质组的变化与植物物种、基因型和胁迫强度有关,表现出多样化的特点,但主要集中在与渗透调节、ROS清除、蛋白质合成与信号转导等相关的蛋白方面[6]。
隶属于豆科沙冬青属的蒙古沙冬青(A. mong-olicus)是阿拉善荒漠地区特有的建群种[7]。作为第三纪古热带气候下的孑遗植物和亚洲中部荒漠唯一和特有的常绿阔叶灌木,因其具有耐干旱、耐低温、耐贫瘠等强抗逆性使其具有很高的科研价值和生态价值[8]。以蒙古沙冬青为材料从分子水平上研究其干旱逆境胁迫响应机制和抗逆基因筛选已取得了许多重要成果,如构建根和叶响应干旱的转录组数据库,鉴定了一批参与干旱调节的转录因子[7, 10];克隆抗逆相关基因研究其功能[11, 12];通过miRNAs研究干旱胁迫下蒙古沙冬青基因调控特征,鉴定了一批保守的和新的非保守miRNAs等[13]。但是,目前对于蒙古沙冬青蛋白质组学的研究较少,可能与蛋白质提取相对较难有关。本研究基于前期建立了蒙古沙冬青根蛋白质组双向电泳体系[14],以蒙古沙冬青叶片为研究对象,改进研究方法,从蛋白质组学水平研究干旱胁迫下叶片组织的蛋白质表达特点,旨在为探索耐逆植物蒙古沙冬青应答干旱胁迫的分子机制提供实验依据。
1 材料与方法 1.1 材料 1.1.1 植物材料与胁迫处理蒙古沙冬青(A. mongolicus)种子采自宁夏回族自治区中卫市。在蛭石:珍珠岩:营养土(2:2:1) 的混合基质中种植,萌发8周后以20% PEG 6000处理模拟干旱胁迫处理1 h、72 h,以0 h为对照。取叶片组织速冻于液氮中,用于后续实验。
1.1.2 试剂固相pH梯度线性干胶条(pH 4-7,24 cm)、IPG buffer(pH 3-10) 购自GE公司。尿素(Urea)、CHAPS (3-[(3-Cholamidopropyl) dimethylammonio] propanesulfonate)、二硫苏糖醇(Dithiothreitol,DTT)、碘乙酰胺(Iodoacetamide,IAA)、丙烯酰胺(Acrylamide)、甲叉双丙烯酰胺(Bis-Acrylamide)、过硫酸铵(Ammonium persulphate)、四甲基乙二胺(TEMED)、SDS(sodium dodecyl sulfate)、Triton X-100、甘氨酸、PVPP(Polyvinylpyrrolidone)、Tris-base均购自Amresco公司。考马斯亮蓝R-250(CBB R-250)、AgNO3购自北京普博欣生物科技责任有限公司,Bradford法蛋白定量试剂盒购自北京百泰克生物技术有限公司,4×Tris-HCl(pH 8.8) 购自生工生物工程(上海)股份有限公司。
1.2 方法 1.2.1 蛋白提取与定量采用改进的TCA-丙酮法提取蒙古沙冬青叶片总蛋白质[15]。称取0.3 g叶片组织,加PVPP 50 mg,置液氮中研磨充分。粉末重悬于预冷的丙酮溶液(含10% W/V TCA,0.07% W/V DTT)中,转入2 mL离心管,-20℃孵育1 h。12 000×g 4℃离心30 min,弃上清,加入相同丙酮溶液充分洗涤多次直至上清液澄清。真空干燥沉淀物,每管加入裂解液(9 mol/L Urea,35 mmol/L Tris,4% W/V CHAPS,1% V/V pH4-7 IPG buffer,1% W/V DTT)400 μL,室温震荡混匀30 min,12 000×g常温离心15 min,取上清液。转入新的离心管后加入其4倍体积的预冷的丙酮溶液(含0.07% W/V DTT),-20℃孵育2 h,12 000×g常温离心20 min,弃上清液,干燥沉淀物,重溶于再水化液中(8 mol/L Urea,2% W/V CHAPS,0.5% V/V IPG buffer,20 mmol/L DTT)。采用Bradford法进行蛋白质定量[16]。
1.2.2 双向电泳双向电泳体系为:24 cm IPG胶条水化上样500 μg,12-15 h后在Ettan IPGphor Ⅲ上进行第一向等点聚焦(Isoelectric focusing,IEF)电泳,程序设定为:100 V 1 h,500 V 1.5 h,1 500 V 2 h,8 000 V 70 000 Vh,500 V任意时间。聚焦结束后,胶条转移至平衡液A(50 mmol/L Tris-HCl pH 8.8,6 mol/L Urea,30% glycerol,2% SDS,0.002% bromophenol blue,1% W/V DTT)中平衡15 min,后转入平衡液B(50 mmol/L Tris-HCl pH8.8,6 mol/L Urea,30% glycerol,2% SDS,0.002% bromophenol blue,4% W/V IAA)中平衡20 min。使用Ettan DALTsix电泳系统,采用厚度为1 mm 12.5% SDS-PAGE分离胶进行第二向垂直SDS-PAGE电泳。以AgNO3染色法染色[17]。进行3次生物学重复以获得稳定清晰的电泳图谱。
1.2.3 差异蛋白的筛选使用UMAX Powerlook 2011XL扫描仪软件MagicScan V6.0以400 dpi分辨率采集图像。采用ImageMaster 2D Platinum 7.0(GE Healthcare)软件进行差异点分析,选择表达丰度(% Vol)的Ratio值超过1.5倍变化、经ANOVA分析(P < 0.05) 且重复性好的差异表达蛋白点。
1.2.4 质谱分析从凝胶上挖取差异表达蛋白点,委托上海厚基生物科技有限公司进行蛋白点MALDI-TOF-TOF质谱鉴定。样品经胶内酶解及Ziptip脱盐,用串联飞行时间质谱仪(4800 Plus MALDI TOF/TOF TMAnalyzer)进行质谱分析。一级质谱(MS)扫描范围为800-4000 Da,选择信噪比大于50的母离子进行二级质谱(MS/MS)分析,每个样品点上选择8个母离子,二级质谱(MS/MS)累计叠加2 500次。
1.2.5 数据库检索用Mascot 2.2软件检索本课题组建立的蒙古沙冬青转录组数据库确定蛋白,搜库所用的转录组数据库中的基因数为129 557个。参数为:Database:mongolicus/ SwissProt;下载时间:2016/8/2;Type of search:Combined(MS+ MS/MS);Enzyme:Trypsin;Fixed modifications:Carbamidomethyl(C);Dynamical modifications:Oxidation(M);Mass values:Monoisotopic;Protein Mass:Unrestricted;Peptide MassTolerance:± 100 ppm;Fragment MassTolerance:± 0.4 Da;Peptide Charge State:1+;Max Missed Cleavages:1。
1.2.6 差异表达蛋白的生物信息学分析进一步采用拟南芥数据库TAIR10,以BLAST鉴定所获得的差异表达蛋白。用AgriGO进行Gene Ontology(GO)注释和富集分析[18],用KOBAS进行京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)代谢通路富集分析[19]。对没有获得GO注释的蛋白,基于文献搜索和蛋白质同源物手动注释。因单一工具分类并不能完全诠释蛋白质的功能,本研究参照Bevan的标准对这些蛋白质进行分类[20]。参照Zhao等[21]的方法,采用5种软件(YLoc[22, 23]、LocTree3[24]、Plant-mPLoc[25]、ngLOC[26]和TargetP[27])在线预测所鉴定蛋白质的亚细胞定位,只有在至少2个软件预测一致时才确定结果有效。若该方法仍无法确定亚细胞定位,则根据GO分析的细胞组分分析和文献搜索进行预测。为了解差异表达蛋白质之间的关系,采用STRING v10进行protein-protein interaction(PPI)网络互作分析[28],使用Cytoscape 2.8.3对网络图进行编辑[29]。
2 结果 2.1 蒙古沙冬青叶片干旱胁迫下的双向电泳图谱特征基于前期的生理学和转录组学研究结果,蒙古沙冬青在干旱胁迫1 h和72 h存在较大的差异,而24 h和72 h结果具有相似性[9],故本研究以20% PEG 6000模拟干旱处理蒙古沙冬青,选取1 h及72 h两个时间点,分别取2个处理组以及对照组(0 h)的叶片进行双向电泳分析。每张双向电泳凝胶上可检测到1 500个以上清晰可见的蛋白点(图 1),处理组与对照组相比总体相似,但部分蛋白点的表达量存在一定差异(图 2)。比较不同处理时间的蛋白质表达量,有73个蛋白点的表达具有显著差异(P < 0.05)(图 1)。
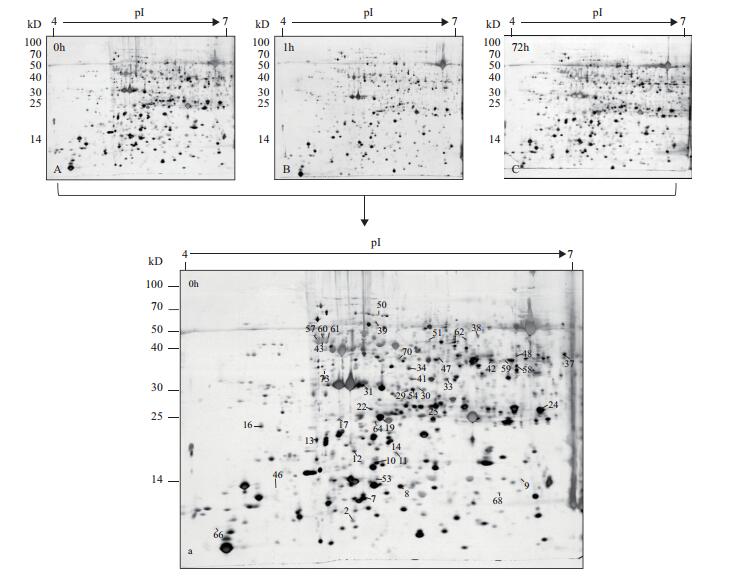
|
| 图 1 蒙古沙冬青叶片蛋白质双向电泳图谱 A:对照组(0 h);B:处理组1 h;C:处理组72 h;73个差异表达蛋白点使用数字标注 |
1 h处理组与对照组相比,有28个蛋白点的表达发生变化:5个点表达量增加,23个点表达量减少;72 h处理组与对照组相比,有40个蛋白点的表达发生变化:24个点表达量增加,16个点表达量减少;72 h与1 h处理组相比,有56个差异表达蛋白点,47个点表达量增加,9个点表达量减少。两个时间点共响应蛋白点13个,其中3个点在两个时间点表达量都增加,7个点在两个时间点表达量都减少,2个点先减少再增加,1个点先增加再减少。
2.2 差异表达蛋白的鉴定和功能分类质谱分析确定了45个差异表达蛋白点的身份,涉及40个差异表达蛋白。GO功能分类分析表明,获得的差异表达蛋白参与的生物过程主要有非生物胁迫应答(Response to abiotic stimulus)(GO:0009628) 和糖代谢过程(carbohydrate metabolic process)(GO:0005975);细胞组分主要有叶绿体(Chloroplast)(GO:0009507) 和细胞质部分(Cytoplasmic part)(GO:0044422);分子功能主要有催化活性(catalytic activity)(GO:0003824) 和结合(binding)(GO:0005488)(图 3)。
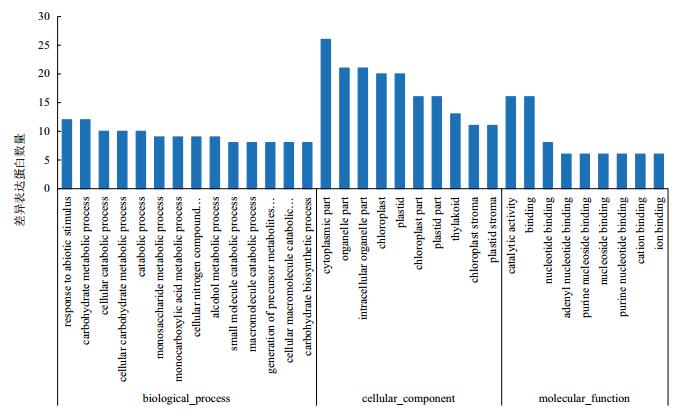
|
| 图 3 差异表达蛋白的GO分析 GO分析可分为生物过程(biological process)细胞组分(cellular component)和分子功能(molecular function)3部分 |
KEGG途径富集分析表明,获得的差异表达蛋白主要参与光合作用中的碳固定代谢(ko00710)、氨基酸的生物合成代谢(ko01230)、代谢(ko01100)、次生代谢物合成(ko01110) 等途径,其中仅碳固定代谢途径具有显著性差异(P < 0.05),该途径包括Rubisco大亚基(RBCL)(点14) 和磷酸丙糖异构酶(TIM)(点9),说明干旱处理下叶片的光合作用受到较大影响。
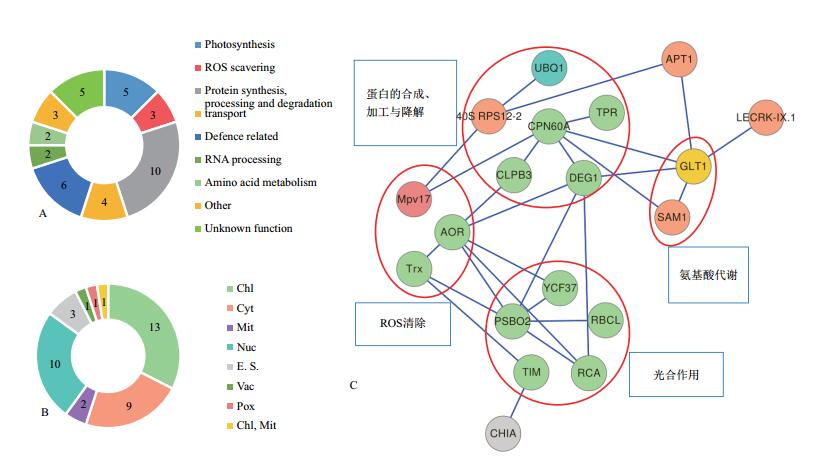
所鉴定的差异表达蛋白可分为9类:光合作用(Photosynthesis),ROS清除(ROS scavering),蛋白的合成、加工与降解(Protein synthesis,processing and degradation),物质运输(Transport),防御相关(Defence related),RNA加工(RNA processing),氨基酸代谢(Amino acid metabolism),其他(Other)和功能未知(Unkown function)蛋白质(图 4-A,表 1)。
亚细胞定位预测结果显示,差异表达蛋白定位于细胞中的7个部位:即叶绿体(Chl)、细胞质(Cyt)、线粒体(Mit)、细胞核(Nuc)、胞外空间(E. S.)、液泡(Vac)和过氧化物酶体(Pox)。此外,1个蛋白质(GLT1) 定位在两个不同的位点(图 4-B,表 1)。
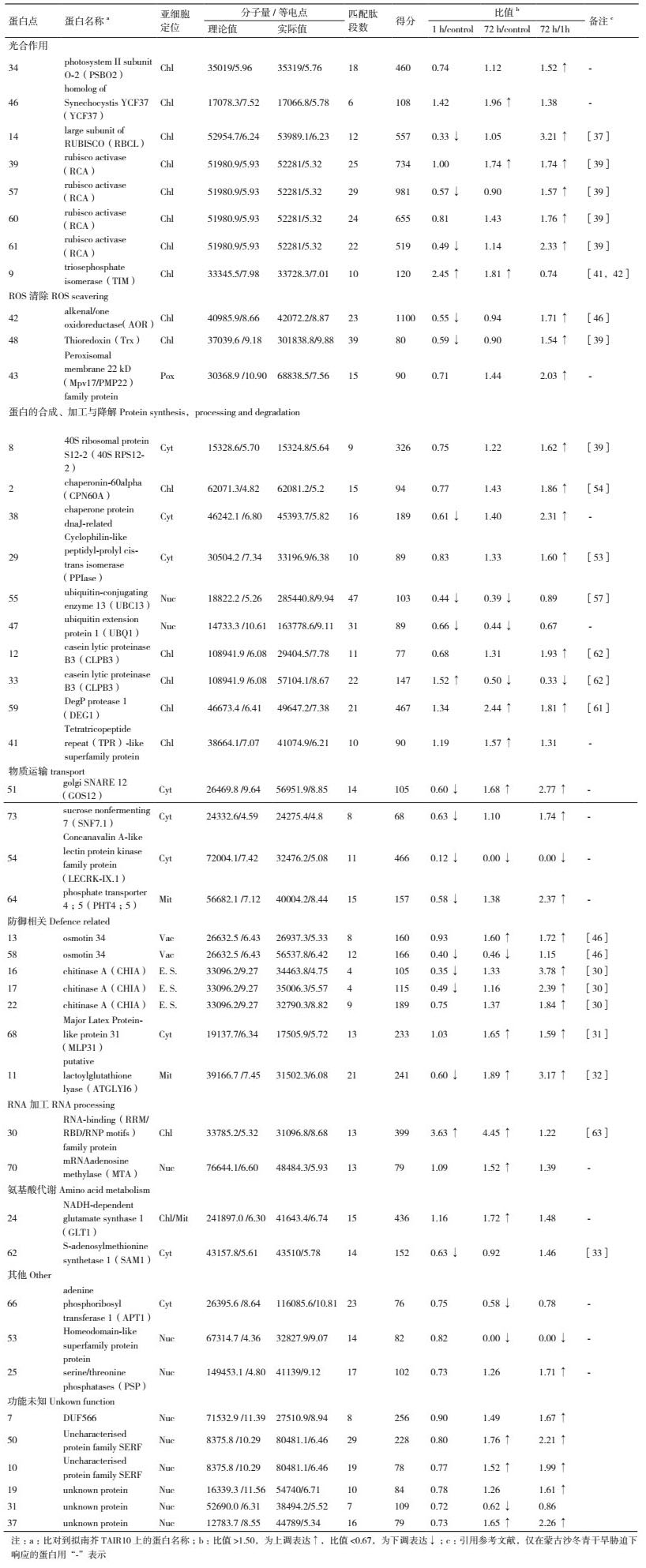
2.3 差异表达蛋白质的互作网络分析PPI分析结果显示,部分表达差异蛋白构成一个结构复杂的多中心互作网络,该网络含19个蛋白质节点,有27种互作关系,各差异表达蛋白质间通过多条通路进行调节。在PPI网络中,相互作用较强的蛋白质主要为光合作用、蛋白的合成、加工与降解、ROS清除、氨基酸代谢的相关蛋白(图 4-C)。
3 讨论植物的叶片组织是植物进行光合作用的重要器官。本研究采用蛋白质组学方法研究了干旱胁迫下耐逆植物蒙古沙冬青叶片蛋白表达特点。对获得的45个差异表达蛋白点分析表明,干旱胁迫影响了蒙古沙冬青叶片的多个生物学过程,其中光合作用受到影响最大,此外ROS清除、蛋白合成、加工与降解等也受到影响。
3.1 光合作用相关蛋白叶绿体的光合作用对植物的生存至关重要。通过亚细胞定位确定了14个差异表达蛋白(共计17个蛋白点)定位于叶绿体中,占所鉴定的差异表达蛋白的比例最高(35%),其中有5个光合作用相关蛋白:光系统Ⅱ(Photosystem Ⅱ,PS Ⅱ)亚基(PSBO2)(点34)、蓝藻同源YCF37(YCF37)(点46)、Rubisco大亚基(RBCL)(点14)、Rubisco活化酶(RCA)(点39,57,60,61) 和磷酸丙糖异构酶(TIM)(点9)。
PSBO2是高等植物PS Ⅱ的外周蛋白,具有维持PS Ⅱ活性的功能[34],且能结合GTP作为GTP酶,推测有类似碳酸酐酶的作用[35]。本研究中PSBO2(点34) 在1h略有减少,72 h恢复并略有提高,表明能够通过调整来维持PSⅡ与碳固定活性。YCF37是参与组装与稳定光系统Ⅰ(PSI)的相关蛋白[36],在本研究中显示持续上调表达,表明干旱胁迫下叶片中的YCF37参与调控PSI的作用增强,有助于稳定光合作用。
Rubisco是光合作用中固定CO2的关键酶。研究表明,干旱胁迫下小麦的Rubisco合成和稳定性受到影响,Rubisco亚基表达降低[37]。本研究结果显示蒙古沙冬青叶片的Rubisco大亚基表达量1 h下调后,72 h恢复到正常水平,说明蒙古沙冬青对干旱环境具有较强的适应性。Rubisco活化酶(RCA)可以改变Rubisco的活性[38],刀豆中的RCA在干旱胁迫后表现为下调表达[39],本研究中有4个蛋白点为RCA,其中3个蛋白点1 h胁迫后下调,72 h恢复到正常水平;另外1个蛋白点1 h胁迫后无变化,72 h后表达量增加,表明干旱胁迫下蒙古沙冬青叶片能够通过调控达到恢复与提高Rubisco的催化活性,以填补Rubisco受胁迫影响而减少的部分,提高耐受能力。磷酸丙糖异构酶(TIM)在叶绿体中负责光合过程的碳固定[40],在干旱胁迫下小麦[41]、杨树[42]叶片的TIM表达TIM上调,本研究的结果与其相同,干旱胁迫下TIM持续上调表达。上述蛋白点的表达变化表明干旱胁迫对蒙古沙冬青叶片的光合作用产生了影响,但其能够采取积极的应答策略以适应逆境胁迫。
3.2 ROS清除相关蛋白干旱胁迫下产生对植物有毒害作用的活性氧ROS,必须通过植物抗氧化系统及时清除[43]。本研究鉴定了3种ROS清除相关蛋白:烯酮/氧化还原酶(AOR)(点42),硫氧还蛋白(Trx)(点48) 和过氧化物酶体膜22 kD(Mpv17 / PMP22) 家族蛋白(点43)。所鉴定的AOR和Trx定位于叶绿体中。AOR主要参与活性羰基化合物解毒,保护PSⅡ组件免受ROS的损害[44]。Trx可清除过氧化氢,在光照条件下通过铁氧还蛋白-硫氧还蛋白还原酶调节光合酶的活性[45]。已有研究显示,干旱胁迫下甜菜的AOR上调表达[46],而刀豆的Trx下调表达[39],本研究中AOR和Trx在胁迫1 h为下调表达,但72 h恢复到正常水平,表明叶绿体内的ROS系统在干旱胁迫下受到影响,并能做出调整得以恢复。本研究鉴定的过氧化物酶体膜22 kD(Mpv17 / PMP22) 蛋白是完全嵌入过氧化物酶体脂质双层中的完整膜蛋白[47],该蛋白在干旱胁迫下在1 h略有减少,72 h恢复并表达增加,表明过氧化物酶体膜蛋白受到胁迫后可以通过调整表达量以稳固过氧化物酶体、积极应对干旱胁迫。
3.3 蛋白的合成、加工与降解相关蛋白已有研究显示干旱胁迫增加蛋白质错误折叠的潜在风险,导致非功能性蛋白质合成[48]。本研究鉴定的差异蛋白中有3类蛋白代谢相关蛋白,分别属于蛋白质合成、加工和降解相关蛋白。
40S核糖体蛋白S12-2(40S RPS12-2)(点8) 是核糖体小亚基的主要组分,在干旱胁迫下在1h略有减少,72 h恢复并略有提高,表明胁迫后可以通过调整稳定蛋白合成能力,研究显示干旱胁迫下刀豆中该蛋白呈现上调表达[39]。
所获得与蛋白质加工相关的蛋白有4个:伴侣蛋白-60α6CPN60A)(点2),dnaJ相关伴侣蛋白(点38),类亲环素肽基脯氨酰顺反异构酶(PPIase)(点29) 和TPR超家族蛋白(点41)。CPN60利用ATP水解帮助新合成、易位或应激变性蛋白质的折叠[49, 50];dnaJ是Hsp40和类Hsp40蛋白与其配体Hsp70之间相互作用所需的主要结合位点,Hsp40会刺激Hsp70水解[51];PPIases是涉及细胞内蛋白质折叠、转运和组装的肽基-脯氨酰顺反异构酶[52]。本研究结果显示,干旱胁迫下蒙古沙冬青叶片中的CPN60A、PPIases、dnaJ皆表现为在1 h处理下表达量不同程度减少,但处理72 h后都恢复并表达提高,表明在干旱胁迫下能够做出调整,积极处理胁迫产生的错误折叠蛋白。干旱胁迫下鹰嘴豆中的PPIases表现为下调表达[53],在西瓜中发现CPN60A、HSP等伴侣蛋白在干旱胁迫下参与叶绿体的发育和维持[54]。
本研究中鉴定的降解蛋白质的相关蛋白有4个:泛素结合酶13(UBC13)(点55)、泛素延伸蛋白1(UBQ1)(点47)、酪蛋白裂解蛋白酶B3(CLPB3)(点12,33) 和蛋白酶DegP 1(DEG1)(点59)。泛素在蛋白降解过程中具有枢纽作用[55],UBC和UBQ是参与泛素化的关键酶,拟南芥中的UBC能通过促进脯氨酸渗透来保护水分匮缺下的植物[56],这个蛋白点未在其他物种的干旱胁迫应答中发现表达变化,但有报道参与低温胁迫,如鹰嘴豆叶片中UBC在低温胁迫下表现为先上调后下调表达[57]。本研究中UBC和UBQ分布于细胞核中,干旱胁迫后表达持续下调。DEG1主要参与PSⅡ中清除错误折叠的蛋白,修复PSⅡ损伤[58, 59],定位于叶绿体中,本研究中表现为持续上调表达。CLPB3介导类囊体膜的形成,可重塑或拆解错误折叠的蛋白质复合体[60],也定位于叶绿体中,2个蛋白点表达变化趋势相反。而DEG1和CLPB3蛋白未在其他物种的干旱胁迫应答中发现表达变化,但有报道显示大麦中的蛋白酶DEG1[61]、甜菜中的CLPB3通过上调表达参与高盐胁迫应答[62]。研究结果表明蒙古沙冬青叶片中定位于不同部位的降解类蛋白采取不同的模式应对干旱胁迫。
此外,本研究发现RNA结合蛋白(点30) 在干旱胁迫下持续显著上调表达,RNA结合蛋白是控制植物生长、发育和应激反应过程中转录后RNA代谢的中心调节因子,某些RNA结合蛋白含量增加有助于增强植物对干旱、低温等胁迫的耐受性[63],本研究结果显示RNA结合蛋白在干旱胁迫下显著上调,有助于增强蒙古沙冬青干旱耐受性。
3.4 蛋白质互作网络呈现的整体调控机制在PPI调控网络中,具有19个蛋白质节点,其中12个蛋白质定位于叶绿体。干旱胁迫下差异表达蛋白中,与光反应相关蛋白(PSBO2、YCF37) 表达量普遍上调,与暗反应相关蛋白RBCL、RCA、TIM等的表达发生了变化。定位于叶绿体中的ROS清除蛋白AOR和Trx、具有蛋白的降解和折叠作用的DEG1等表达也发生了变化。说明干旱胁迫影响了叶绿体,其干旱应答的核心是叶绿体结构和光合作用的维持。此外,定位在细胞质和其他细胞器的蛋白表达也发生了变化[如谷氨酸合成酶(GLT1) 表达上调、胞质内核糖体小亚基40S表达上调],这些蛋白的变化说明蒙古沙冬青叶组织可以通过细胞中各细胞器的不同蛋白的协同变化以应对干旱胁迫。
3.5 干旱胁迫下蒙古沙冬青叶片蛋白质组与转录组结果比较本课题组此前已对干旱胁迫下蒙沙冬青叶开展了转录组学的研究[9],两者都显示72 h胁迫比1 h胁迫得到的差异表达基因/蛋白数量更多,但基因/蛋白在功能分类上差异较大。蛋白质组学发现有大量蛋白质定位于叶绿体中,而转录组学并未发现光合作用相关基因,且GO分析中细胞组分结果也未发现基因在叶绿体显著富集。蛋白质组学研究结果显示蛋白的合成、加工与降解类蛋白数目最多,而转录组结果中次生代谢物的生物合成、激素刺激和植物激素信号转导的反应富集最多。两种方法在研究结果上的差异反映研究水平不同,综合二者获得的数据,可从不同水平、不同角度为蒙古沙冬青抗逆机制的研究提供依据。
4 结论本研究通过20% PEG 6000模拟干旱胁迫、采用蛋白质组学方法研究了蒙古沙冬青叶片的干旱胁迫响应特点,获得了40个差异表达蛋白。结果显示,干旱胁迫影响了蒙古沙冬青叶片中部分蛋白的表达,主要涉及光合作用、ROS清除、蛋白的合成、加工与降解等相关蛋白。表明蒙古沙冬青叶片能够通过改变不同功能蛋白的表达来维持光合作用,以适应干旱胁迫。
| [1] | Gray SB, Brady SM. Plant developmental responses to climate change[J]. Developmental Biology, 2016, 419 (1): 64–77. DOI:10.1016/j.ydbio.2016.07.023 |
| [2] | Basu S, Ramegowda V, Kumar A, et al. Plant adaptation to drought stress[J]. F1000Research, 2016, 5 : 1554. DOI:10.12688/f1000research |
| [3] | Bray EA. Plant responses to water deficit[J]. Trends in Plant Science, 1997, 2 (2): 48–54. DOI:10.1016/S1360-1385(97)82562-9 |
| [4] | Khan MS, Khan MA, Ahmad D. Assessing utilization and environmental risks of important genes in plant abiotic stress tolerance[J]. Frontiers in Plant Science, 2016, 7 : 792. |
| [5] | Debnath M, Pandey M, Bisen PS. An omics approach to understand the plant abiotic stress[J]. Omics:a Journal Of Integrative Biology, 2011, 15 (11): 739–762. DOI:10.1089/omi.2010.0146 |
| [6] | Wang X, Cai X, Xu C, et al. Drought-responsive mechanisms in plant leaves revealed by proteomics[J]. International Journal of Molecular Sciences, 2016, 17 (10): pii:E1706. DOI:10.3390/ijms17101706 |
| [7] | 刘果厚. 阿拉善荒漠特有植物沙冬青濒危原因的研究[J]. 植物研究, 1998, 18(3): 341–345. |
| [8] | 周宜君, 高飞, 冯金朝, 等. 民族地区沙冬青种质资源保护与利用[J]. 安徽农业科学, 2011, 39(10): 5851–5853. DOI:10.3969/j.issn.0517-6611.2011.10.072 |
| [9] | Gao F, Wang J, Wei S, et al. Transcriptomic analysis of drought stress responses in Ammopiptanthus mongolicus leaves using the RNA-Seq technique[J]. PLoS One, 2015, 10 (4): e0124382. DOI:10.1371/journal.pone.0124382 |
| [10] | Wu Y, Wei W, Pang X, et al. Comparative transcriptome profiling of a desert evergreen shrub, Ammopiptanthus mongolicus, in response to drought and cold stresses[J]. BMC Genomics, 2014, 15 (1): 1–16. DOI:10.1186/1471-2164-15-1 |
| [11] | 郭婷, 王茅雁, 董博, 等. 蒙古沙冬青AmDREB2C基因的克隆及表达分析[J]. 植物遗传资源学报, 2015, 16(2): 344–348. |
| [12] | 李章磊, 高飞, 曹玉震, 等. 蒙古沙冬青AmDREB2. 1基因的克隆及表达分析[J]. 生物技术通报, 2015(3): 108–114. |
| [13] | Gao F, Wang N, Li H, et al. Identification of drought-responsive microRNAs and their targets in Ammopiptanthus mongolicus by using high-throughput sequencing[J]. Scientific Reports, 2016, 6 : 34601. DOI:10.1038/srep34601 |
| [14] | 刘楠, 高飞, 周宜君, 等. 蒙古沙冬青根蛋白的提取及双向电泳体系的建立[J]. 北京师范大学学报:自然科学版, 2013, 49(4): 365–368. |
| [15] | Mathesius U, Keijzers G, Natera SHA, et al. Establishment of a root proteome reference map for the model legume Medicago truncatula using the expressed sequence tag database for peptide mass fingerprinting[J]. Proteomics, 2001, 1 (11): 1424–1440. DOI:10.1002/(ISSN)1615-9861 |
| [16] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72 (1-2): 248–254. DOI:10.1016/0003-2697(76)90527-3 |
| [17] | Rabilloud T. Silver staining of 2D electrophoresis gels[J]. Quantitative Methods in Proteomics, 2012, 893 : 61–73. DOI:10.1007/978-1-61779-885-6 |
| [18] | Du Z, Zhou X, Ling Y, et al. agriGO:a GO analysis toolkit for the agricultural community[J]. Nucleic Acids Research, 2010, 38 : W64–W70. DOI:10.1093/nar/gkq310 |
| [19] | Xie C, Mao X, Huang J, et al. OBAS 2. 0:a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 2011, 39 : W316–W322. DOI:10.1093/nar/gkr483 |
| [20] | Bevan M, Bancroft I, Bent E, et al. Analysis of 1. 9 Mb of contiguous sequence from chromosome 4 of Arabidopsis thaliana[J]. Nature, 1998, 391 (6666): 485–488. DOI:10.1038/35140 |
| [21] | Zhao Q, Gao J, Suo J, et al. Cytological and proteomic analyses of horsetail(Equisetum arvense L.)spore germination[J]. Frontiers in Plant Science, 2015, 6 : 441. |
| [22] | Briesemeister S, Rahnenführer J, Kohlbacher O. YLoc-an interpre-table web server for predicting subcellular localization[J]. Nucleic Acids Research, 2010, 38 : W497–W502. DOI:10.1093/nar/gkq477 |
| [23] | Briesemeister S, Rahnenführer J, Kohlbacher O. Going from where to why-interpretable prediction of protein subcellular localization[J]. Bioinformatics, 2010, 26 (9): 1232–1238. DOI:10.1093/bioinformatics/btq115 |
| [24] | Goldberg T, Hecht M, Hamp T, et al. LocTree3 prediction of localization[J]. Nucleic acids research, 2014, 42 (W1): W350–W355. DOI:10.1093/nar/gku396 |
| [25] | Chou KC, Shen HB. Plant-mPLoc:a top-down strategy to augment the power for predicting plant protein subcellular localization[J]. PLoS One, 2010, 5 (6): e11335. DOI:10.1371/journal.pone.0011335 |
| [26] | King BR, Guda C. ngLOC:an n-gram-based Bayesian method for estimating the subcellular proteomes of eukaryotes[J]. Genome Biology, 2007, 8 (5): 1. |
| [27] | Emanuelsson O, Nielsen H, Brunak S, et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology, 2000, 300 (4): 1005–1016. DOI:10.1006/jmbi.2000.3903 |
| [28] | Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10:protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Research, 2014, 43 : D447–D452. |
| [29] | Smoot ME, Ono K, Ruscheinski J, et al. Cytoscape 2. 8:new features for data integration and network visualization[J]. Bioinformatics, 2011, 27 (3): 431–432. DOI:10.1093/bioinformatics/btq675 |
| [30] | Bohler S, Sergeant K, Jolivet Y, et al. A physiological and proteomic study of poplar leaves during ozone exposure combined with mild drought[J]. Proteomics, 2013, 13 (10-11): 1737–1754. DOI:10.1002/pmic.v13.10-11 |
| [31] | Wang Y, Yang L, Chen X, et al. Major latex protein-like protein 43(MLP43) functions as a positive regulator during abscisic acid responses and confers drought tolerance in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2015, 67 (1): 421–434. |
| [32] | Shu L, Lou Q, Ma C, et al. Genetic, proteomic and metabolic analysis of the regulation of energy storage in rice seedlings in response to drought[J]. Proteomics, 2011, 11 (21): 4122–4138. DOI:10.1002/pmic.v11.21 |
| [33] | Zhang S, Chen F, Peng S, et al. Comparative physiological, ultrastructural and proteomic analyses reveal sexual differences in the responses of Populus cathayana under drought stress[J]. Proteomics, 2010, 10 (14): 2661–2677. DOI:10.1002/pmic.200900650 |
| [34] | Offenbacher AR, Polander BC, Barry BA. An intrinsically disordered photosystem II subunit, PsbO, provides a structural template and a sensor of the hydrogen-bonding network in photosynthetic water oxidation[J]. Journal of Biological Chemistry, 2013, 288 (40): 29056–29068. DOI:10.1074/jbc.M113.487561 |
| [35] | Bricker T M, Frankel LK. Auxiliary functions of the PsbO, PsbP and PsbQ proteins of higher plant Photosystem II:a critical analysis[J]. Journal of Photochemistry and Photobiology B:Biology, 2011, 104 (1): 165–178. |
| [36] | Wilde A, Lünser K, Ossenbühl F, et al. Characterization of the cyanobacterial ycf37:mutation decreases the photosystem I content[J]. Biochemical Journal, 2001, 357 (1): 211–216. DOI:10.1042/bj3570211 |
| [37] | Demirevska K, Zasheva D, Dimitrov R, et al. Drought stress effects on Rubisco in wheat:changes in the Rubisco large subunit[J]. Acta Physiologiae Plantarum, 2009, 31 (6): 1129–1138. DOI:10.1007/s11738-009-0331-2 |
| [38] | Ebrahimzadeh H. Drought stress increases the expression of wheat leaf ribulose-1, 5-bisphosphate carboxylase/oxyenase protein[J]. Iranian Journal of Science and Technology(Sciences), 2006, 30 (1): 1–7. |
| [39] | Zadražnik T, Hollung K, Egge-Jacobsen W, et al. Differential proteomic analysis of drought stress response in leaves of common bean(Phaseolus vulgaris L.)[J]. Journal of Proteomics, 2012, 78 (1): 254–272. |
| [40] | Zaffagnini M, Michelet L, Sciabolini C, et al. High-resolution crystal structure and redox properties of chloroplastic triosephosphate isomerase from Chlamydomonas reinhardtii[J]. Molecular Plant, 2014, 7 (1): 101–120. DOI:10.1093/mp/sst139 |
| [41] | Budak H, Akpinar B A, Unver T, et al. Proteome changes in wild and modern wheat leaves upon drought stress by two-dimensional electrophoresis and nanoLC-ESI-MS/MS[J]. Plant Molecular Biology, 2013, 83 (1): 89–103. |
| [42] | Durand T C, Sergeant K, Renaut J, et al. Poplar under drought:comparison of leaf and cambial proteomic responses[J]. Journal of Proteomics, 2011, 74 (8): 1396–1410. DOI:10.1016/j.jprot.2011.03.013 |
| [43] | Ye T, Shi H, Wang Y, et al. Contrasting changes caused by drought and submergence stresses in bermudagrass(Cynodon dactylon)[J]. Frontiers in Plant Science, 2015, 6 : 951. |
| [44] | Yamauchi Y, Hasegawa A, Mizutani M, et al. Chloroplastic NADPH-dependent alkenal/one oxidoreductase contributes to the detoxification of reactive carbonyls produced under oxidative stress[J]. FEBS Letters, 2012, 586 (8): 1208–1213. DOI:10.1016/j.febslet.2012.03.013 |
| [45] | Schürmann P, Jacquot JP. Plant thioredoxin systems revisited[J]. Annual Review of Plant Biology, 2000, 51 (1): 371–400. DOI:10.1146/annurev.arplant.51.1.371 |
| [46] | Hajheidari M, Abdollahian-Noghabi M, Askari H, et al. Proteome analysis of sugar beet leaves under drought stress[J]. Proteomics, 2005, 5 (4): 950–960. DOI:10.1002/(ISSN)1615-9861 |
| [47] | Tugal HB, Pool M, Baker A. Arabidopsis 22-kilodalton peroxisomal membrane protein. Nucleotide sequence analysis and biochemical characterization[J]. Plant Physiology, 1999, 120 (1): 309–320. DOI:10.1104/pp.120.1.309 |
| [48] | Ashoub A, Beckhaus T, Berberich T, et al. Comparative analysis of barley leaf proteome as affected by drought stress[J]. Planta, 2013, 237 (3): 771–781. DOI:10.1007/s00425-012-1798-4 |
| [49] | Jiang Q, Mei J, Gong XD, et al. Importance of the rice TCD9 encoding α subunit of chaperonin protein 60(Cpn60α)for the chloroplast development during the early leaf stage[J]. Plant Science, 2014, 215-216 : 172–179. DOI:10.1016/j.plantsci.2013.11.003 |
| [50] | Dickson R, Weiss C, Howard RJ, et al. Reconstitution of higher plant chloroplast chaperonin 60 tetradecamers active in protein folding[J]. Journal of Biological Chemistry, 2000, 275 (16): 11829–11835. DOI:10.1074/jbc.275.16.11829 |
| [51] | Hennessy F, Nicoll W S, Zimmermann R, et al. Not all J domains are created equal:implications for the specificity of Hsp40-Hsp70 interactions[J]. Protein Science, 2005, 14 (7): 1697–1709. DOI:10.1110/(ISSN)1469-896X |
| [52] | Weisman R, Creanor J, Fantes P. A multicopy suppressor of a cell cycle defect in S. pombe encodes a heat shock-inducible 40 kDa cyclophilin-like protein[J]. Embo Journal, 1996, 15 (3): 447–456. |
| [53] | Bhushan D, Pandey A, Choudhary MK, Datta A, Chakraborty S, Chakraborty N. Comparative proteomics analysis of differentially expressed proteins in chickpea extracellular matrix during dehydration stress[J]. Mol Cell Proteomics, 2007, 6 : 1868–1884. DOI:10.1074/mcp.M700015-MCP200 |
| [54] | Akashi K, Yoshida K, Kuwano M, et al. Dynamic changes in the leaf proteome of a C3 xerophyte, Citrullus lanatus(wild watermelon), in response to water deficit[J]. Planta, 2011, 233 (5): 947–960. DOI:10.1007/s00425-010-1341-4 |
| [55] | 王彦杰, 张超, 王晓庆, 等. 牡丹泛素延伸蛋白基因片段克隆与表达分析[J]. 扬州大学学报:农业与生命科学版, 2013(1): 79–83. |
| [56] | Wan X, Mo A, Liu S, et al. Constitutive expression of a peanut ubiquitin-conjugating enzyme gene in Arabidopsis confers improved water-stress tolerance through regulation of stress-responsive gene expression[J]. Journal of Bioscience and Bioengineering, 2011, 111 (4): 478–484. DOI:10.1016/j.jbiosc.2010.11.021 |
| [57] | Heidarvand L, Maaliamiri R. Physio-biochemical and proteome analysis of chickpea in early phases of cold stress[J]. Journal of Plant Physiology, 2013, 170 (5): 459–469. DOI:10.1016/j.jplph.2012.11.021 |
| [58] | Krojer T, Sawa J, Sch?fer E, et al. Structural basis for the regulated protease and chaperone function of DegP[J]. Nature, 2008, 453 (7197): 885–890. DOI:10.1038/nature07004 |
| [59] | Hau?ühl K, Andersson B, Adamska I. A chloroplast DegP2 protease performs the primary cleavage of the photodamaged D1 protein in plant photosystem II[J]. The EMBO Journal, 2001, 20 (4): 713–722. DOI:10.1093/emboj/20.4.713 |
| [60] | Lee U, Rioflorido I, Hong SW, et al. The Arabidopsis ClpB/Hsp100 family of proteins:chaperones for stress and chloroplast development[J]. The Plant Journal, 2007, 49 (1): 115–127. |
| [61] | Wu D, Shen Q, Qiu L, et al. Identification of proteins associated with ion homeostasis and salt tolerance in barley[J]. Proteomics, 2014, 14 (11): 1381–1392. DOI:10.1002/pmic.v14.11 |
| [62] | Yang L, Zhang Y, Zhu N, et al. Proteomic analysis of salt tolerance in sugar beet monosomic addition line M14[J]. Journal of Proteome Research, 2013, 12 (11): 4931–4950. DOI:10.1021/pr400177m |
| [63] | Lee K, Kang H. Emerging roles of RNA-binding proteins in plant growth, development, and stress responses[J]. Molecules & Cells, 2016, 39 (3): 179–185. |