2. 黑龙江省科学院微生物研究所,哈尔滨 150010
2. Institute of Microbiology, Heilongjiang Academy of Sciences, Harbin 150010
神奇的自然进化造就了CRISPR-Cas系统,它是细菌及古细菌中的一种特异性免疫系统,最早由Ishino发现于大肠埃希菌的iap基因[1]。CRISPR-Cas系统不仅为研究微生物的进化提供了新思路,更为人类对基因的编辑找到了新的突破口。随着科学界对其研究的逐渐深入,该系统现已发展成为一种高效的基因编辑手段,强大的优势使其迅速成为近十年来微生物及相关领域研究的热点。目前,此项技术已经成功应用于包括微生物、植物、动物在内的诸多物种当中[2-5]。本文详细阐述了CRISPR-Cas系统的组成、结构、分型以及CRISPRs技术作用机理、优势等内容,并着重分析了嗜热链球菌中CRISPR基因的碱基排布及其高级结构,旨在为CRISPR技术的进一步研究与应用提供参考。
1 细菌的CRISPR-Cas系统细菌在长期的生存斗争中,进化出多种免疫机制[6],包括已研究较为透彻的限制修饰系统、渗透阻断、流产感染系统及毒素-抗毒素系统等[7]。近几年发现的CRISPR-Cas系统与前三者共同构筑了细菌体内一道“坚不可摧的城墙”,实施干扰和切断外源基因片段的作用。
1.1 CRISPR-Cas系统的组成CRISPRs意为规律成簇的短间距回文重复序列。CRISPR基因簇以“重复序列-间隔区”为单元,为细菌DNA上的一段非编码区。Cas蛋白是一种与CRISPRs基因相关的蛋白质,具有核酸酶和解旋酶活性[1]。CRISPRs基因与Cas蛋白共同组成了CRISPR-Cas系统,该系统最早发现于嗜热古细菌,用于抵御外来DNA(如质粒、噬菌体DNA)的侵染[8]。
典型的CRISPRs基因簇由高度保守的重复序列和间隔片段串联而成,通常含有21-48 bp正向重复序列,重复序列之间又由长度基本一致的26-72 bp非重复间隔子隔开[9]。其5'端上游通常含有一段高AT含量的DNA片段作为前导序列。CRISPR序列的二级结构非常稳定,且两端序列高度保守[8, 9]。由于其部分回文重复,CRISPR序列可形成稳定的发夹结构[10]。
毗邻CRISPR序列的操纵子编码Cas蛋白,cas基因具有编码解旋酶、聚合酶和多种RNA结合蛋白的核酸的特征。Cas蛋白的类型多种多样,它能够参与CRISPR基因位点转录的不同步骤,并协助目标DNA的裂解和新间隔子的整合[11-13]。不同Cas蛋白参与不同类型CRISPR-CAS系统的各个过程,执行不同的功能,因此,Cas蛋白是该系统必不可少的组成部分。
1.2 CRISPR-CAS系统的作用机理CRISPR-CAS系统是细菌及部分古细菌的特异性免疫系统[14],它的作用机理类似于RNA干扰。外源DNA侵入宿主细胞后,部分DNA片段会整合到CRISPR基因簇上;待该序列再次入侵时,宿主细胞在CRISPR-Cas系统的作用下,切断、裂解入侵的DNA[15]。CRISPR-CAS免疫系统对外来DNA的排斥过程主要包括适应、表达和干扰3个阶段[15]。
在适应阶段,外源DNA入侵宿主细胞后,Cas1、Cas2蛋白通过识别前间隔子相邻基序(Protospacer adjacent motif,PAM),将一段外源DNA(称为前间隔子)整合于宿主的CRISPR基因簇前端,形成新的间隔子,由此形成对入侵DNA的“记忆”。在表达阶段,带有外源DNA片段前间隔子的CRISPR基因簇转录出一条单链RNA(前体crRNA),前体crRNA经Cas1、Cas2蛋白及RNA酶的加工、剪切,成为成熟的短链crRNA,它由对应于CRISPR基因簇上的单链间隔子和相邻单链重复序列组成。成熟的crRNA可进一步与相关Cas蛋白或Cascade复合体结合,形成CRISPR效应核蛋白复合体(crRNP)。在干扰阶段,crRNP复合体结合的crRNA作为gRNA引导复合体向目标DNA发起进攻,最终导致外源DNA的降解。在广泛应用的CRISPR/Cas9系统中,成熟crRNA可与tracrRNA(Trans-encodedsmall RNA)互补配对形成双链RNA,dsRNA与Cas9蛋白、RNase Ⅲ结合成为靶向切割复合体,与被识别的目标DNA互补配对,再对其进行切割、裂解[16]。这种高效率、特异性的DNA切割、编辑方式为基因的修饰及突变体的构建提供了新的思路。
1.3 CRISPR-CAS系统的分型现有的生物信息学分析发现,不同物种的CRISPR基因簇可编码大约65个同源Cas蛋白,由此可将其分为8个CRISPR-CAS系统亚型,但复杂的分类方法会阻碍CRISPR-CAS系统的应用。结合系统发育树信息和比较基因组分析,Makarova等[17]提出一种新型的分类系统,主要包括3种不同类型。
Ⅰ型CRISPR-Cas系统基因簇包含cas3基因,可编码一种大分子量的Cas3蛋白,其基因簇上还存在编码一种Cas蛋白复合体——Cascade(CRISPR-associatedcomplex for antiviral defense)的基因,这种复合体包含大量属于重复相关可疑蛋白(Repeat-associated mysterious proteins,RAMP)超家族的蛋白质(如同源性较近的Cas7和Cas8)。Ⅰ型系统中,发挥主要作用的是一种具有RNA酶活性的RAMP蛋白,它能够切割前体-crRNA,进而将其加工为成熟的crRNA。在对外源DNA的降解阶段,目标DNA由Cas3蛋白的HD核酸酶结构域靶向并进行切割[10]。
Ⅱ型系统又称“HNH”系统、链球菌样系统及Nmeni亚型。Ⅱ型系统除具有普遍存在于各型系统的Cas1和Cas2蛋白外,还含有一种特征性的大分子量(包含约1 000个氨基酸)单亚基蛋白——Cas9,其作用为协助加工crRNA以及裂解目标DNA。Cas9蛋白上有两个不相干的核苷酸结构域[1],分别称作RuvC样(RNA酶H折叠)和HNH(McrA样)核酸酶结构域[18],它们分别负责CRISPR基因簇的转录和外源DNA的干扰[1]。Ⅱ型系统可分为3个亚型,Ⅱ-A、Ⅱ-B和Ⅱ-C亚型。Ⅱ-A亚型系统包含一个额外的csn2基因。该蛋白虽不具有干扰目标DNA的功能,但在间隔子的整合阶段发挥一定作用,它编码的Csn蛋白呈四环结构,线性双链DNA可通过中心孔结合于Csn蛋白上[10]。Ⅱ-B亚型系统则由一种属于Cas4蛋白大家族的特殊蛋白发挥主要作用,该蛋白具有5'-单链DNA外切酶活性[1]。Ⅱ-C亚型是已知的CRISPR-CAS系统中仅仅依靠Cas1、Cas2和Cas9三种蛋白发挥作用的系统[19],该系统常用于细菌基因组测序,现已发展成为一种强有力的基因编辑工具[20]。
Ⅲ型系统主要包括Ⅲ-A(又称Mtube和CASS6亚型)和Ⅲ-B(又称聚合酶-RAMP模块)两个亚型,两种亚型都含有一种典型的多结构域Cas10蛋白[21]。Ⅲ型CRISPR-CAS系统往往不编码Cas1和Cas2,而是与Ⅰ型或者Ⅱ型系统共用crRNA[22]。Ⅲ-A亚型具有cas1、cas2和cas6基因,能够靶向DNA[23];而大多数Ⅲ-B亚型缺失这些基因,一般需要依赖同基因组的其它CRISPR-Cas系统,该亚型能够靶向RNA[24]。Cas10蛋白是Ⅲ型系统效应物复合物上的大亚基。每种Ⅲ型位点也编码其他亚基的效应复合物,如小亚基基因、编码Cas5和Cas7大家族RAMP蛋白的基因[25]。
考虑到CRISPR-Cas系统的快速演变,今后将会有更多类型被发掘出来,即使现有的分型系统,也不能涵盖所有物种,如嗜酸氧化亚铁硫杆菌Acidithiobacillus ferrooxidans ATCC 23270菌株的CRISPR-Cas系统便不能纳入上述3种类型,有的分类系统将其归入U型CRISPR-Cas系统[18]。该系统不含Cas1、Cas2蛋白,也无以上3种系统的任何特征。
1.4 嗜热链球菌中的CRISPR-Cas嗜热链球菌是CRISPR-Cas Ⅱ型系统常用的特征模型系统,其gRNA-Cas9复合体是一种传统的CRISPR/Cas基因编辑工具,它能特异性地识别一个包含碱基配对区和PAM的DNA片段,并且可以诱导DNA双链在目标位点断裂。相关研究表明,嗜热链球菌的CRISPR/Cas系统以5'-NNAGAA作为PAM识别序列[26]。长期的免疫过程让嗜热链球菌的CRISPR基因与相关功能单位实现了“进化”,同时CRISPR基因的多样性也更加丰富。
嗜热链球菌的CRISPR基因呈现多态性,按其基因位点的不同可分为4种类型,分别为CRISPR1、CRISPR2、CRISPR3及CRISPR4[27]。除CRISPR2的位点在cas基因的中部外,其它3种类型的CRISPR位点均分布在cas基因的下游[27],这在一定程度上,与CRISPR2是原始革兰氏阳性菌基因的遗留部分有关。
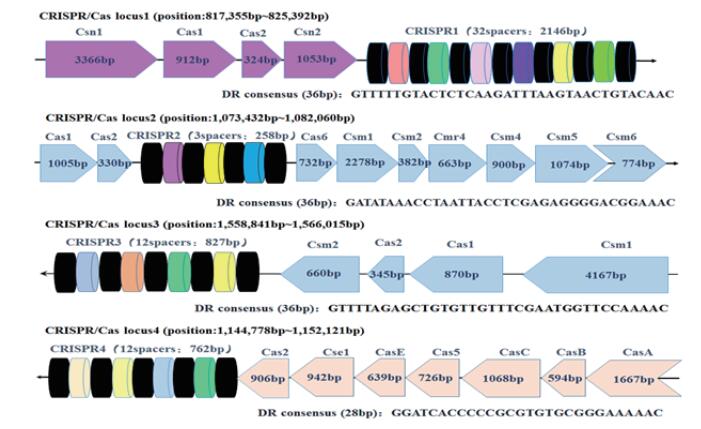
不同CRISPR基因簇的碱基个数以及组成存在差异(图 1)。CRISPR1/Cas基因簇为8 037 bp,其中CRISPR1基因包含32个间隔子,长2 146 bp,这种多重复序列、多间隔子的特点有利于外源DNA片段的整合;CRISPR2/Cas基因簇总长8 628 bp,但CRISPR2基因仅包含3个间隔子,基因总长为258 bp,所含的Cas蛋白基因比重较大,这样的结构不利于新间隔子的整合,使得CRISPR2/Cas发挥的作用相对较小;CRISPR3/Cas基因簇总长7 147 bp,CRISPR3基因包含12个间隔子,基因长度为827 bp;CRISPR4/Cas基因簇总长为7 343 bp,CRISPR4基因长762 bp,包含12个间隔子[27]。
上述4种类型CRISPR基因包含的重复序列大小及碱基构成各不相同(图 2):CRISPR1、CRISP-R2和CRISPR3 3种基因的重复序列(分别以DR1、DR2、DR3表示)包含的碱基个数相同,但碱基排布各不相同,且均包含回文序列,可形成茎环状的二级结构。DR1的碱基序列构成为GTTTTTGTAC-TCTCAAGATTTAAGTAACTGTACAAC(5'-3')[28],头部为21个碱基形成的大环,茎区长6 bp,尾部长4和1 bp[29],如图 2-a所示;DR2的碱基为GATATAAACCTAATTACCTCGAGAGGGGACGGAAAC(5'-3')[28],DR2环头部仅由6个碱基组成,茎区长4 bp,很大一部分碱基集中在了尾部的游离区,分别长16和8 bp[29],如图 2-b所示;DR3序列为GTTTTAGAGCTGTGTTGTTTCGAATGGTTCCAAAAC(5'-3')[28],DR3呈典型的茎环结构,除头部外,均形成了茎区,尾部没有游离碱基[29]。与上述3种CRISPR基因不同,CRISPR4的重复序列DR4仅长28 bp,碱基排布为GGATCACCCCCGCGTGTGCGGGAAAAAC,其二级结构信息尚未有文献报道。
在漫长的防御过程中,CRISPR基因与细菌质粒、病毒DNA等实现了“共进化”[30],研究表明,它们之间有部分同源性较高的序列[31]。CRISPR基因簇的3'末端(尾部末端)在不同菌株间呈现高同源性,而5'末端(前导末端)呈高变异性[28],这与外源DNA在前导序列之后插入密切相关。间隔子数量最多的CRISPR1基因呈现最高的多样性,而仅有3个间隔子的CRISPR2最为保守。Horvath等[28]通过研究表示:CRISPR1是某些嗜热链球菌特有的,CRISPR3在嗜热链球菌CRISPR-Cas系统中的分布更为广泛,而CRISPR2则可能由原始的革兰氏阳性菌DNA退化而产生。
不同类型CRISPR基因簇中包含的cas基因也有一定的区别及相似性,这体现了CRISPR基因与Cas蛋白的联系及功能上的协作。Cas蛋白能够参与CRISPR的产生过程,如Ⅲ型系统中内切酶样的Cas蛋白涉及DNA的追踪,因此噬菌体衍生的间隔子对嗜热链球菌CRISPR结构有一定偏向性[30]。cas1和cas2基因在4种CRISPR / Cas基因座中的分布可能影响其在防御系统中的活性[32]。CRISPR1和CRISPR3的结构较为保守,它们的重复间隔区上游存在有4个cas基因;而CRISPR2基因簇的组成及结构差异性较大,cas基因则位于重复间隔区的两侧[28]。虽然CRISPR1和CRISPR3上cas基因的分布较为相似,但序列相似性低,即使是在蛋白质水平也仅有22%的相似度,仅有cas1是3种基因座之间唯一保守的基因[28]。
2 CRISPRs基因编辑技术研究者利用人为改造过的gRNA-cas9复合体进行基因的靶向编辑,即CRISPRs技术,这一重大突破已成为继ZFN(锌指核酸酶)、TALEN(类转录激活因子效应物核酸酶)以后的第三代基因编辑技术。
2.1 CRISPRs基因编辑技术基因定点编辑是一种针对生物基因组特定的靶位点构建序列特异性核酸酶,从而引导目标位点的DNA双链断裂(DNA double-strand breaks,DSB),随后经生物体内源DNA断裂修复系统实现断裂DNA修复的技术。易错非同源性末端连接(Non-homologous end joining,NETJ)是一种有效的DNA损伤修复机制,但由易错非同源性末端连接(NETJ)介导的DNA双链断裂(DBS)修复有时会伴有缺失突变,从而造成目标位点基因组信息的破环[9]。在Ⅱ型gRNA-cas9复合体的参与下,同源重组修复反应可以通过引入外源供体DNA作为修复模板产生敲入突变,从而缓解基因组破坏[9]。
ZFN、TALEN和CRISPR/Cas9系统都可以通过设计目标位点对复杂的基因组实现定点编辑[16],ZFN和TALEN技术目前仍在广泛使用,而新兴的CRISPRs技术能够基于细菌及古细菌中的CRISPR-CAS系统,将生物自身的DNA识别、加工手段运用于突变的构建,较前者具有更大的优势。
Ⅱ型系统的靶向切割复合物结构最为简单,除tracrRNA、RNase Ⅲ外只需要Cas9一种蛋白的协助[33],因而成为应用最广的CRISPR基因编辑技术。CRISPR/Cas9技术的主要工具为sgRNA-Cas9复合体。Jinek等[34]发现,tracrRNA的5'端能够与成熟crRNA的3'端部分配对形成茎环结构,这是识别靶DNA所必须的,他们将tracrRNA与crRNA表达为嵌合的向导RNA(gRNA)。此外,Cas9蛋白也是CRISPR/Cas9技术中必不可少的部分,Cas9蛋白(800-1,400个氨基酸)包含RuvC和HNH两个核酸酶结构域[18],二者均能参与靶DNA的裂解[35]。此外,还具有α-螺旋组成的识别区(REC)以及位于C端的PAM结合位点[4]。Cas9蛋白的晶体结构解析显示:Cas9具有双叶结构[36],双叶的两环均贡献一个PAM识别位点,因此靶DNA和sgRNA可结合于两叶之间的接口处。Qi等[37]通过研究指出CRISPR/Cas9系统可以有效地沉默细菌基因,进而有效减少脱靶现象。sgRNA-Cas9复合体除具有各自功能外,还能够阻断转录期间RNA聚合酶与DNA的结合[37],从而抑制细菌基因表达,破坏靶DNA稳定性。
CRISPRs技术主要运用于基因定点编辑,其中包括单基因及多基因敲除、精确突变、微缺失插入突变以及单基因的多重编辑。该技术在人、鼠等哺乳动物的基因编辑中应用最为广泛,Mali等[38]将CRISPR/Cas9系统用于诱导人类多能干细胞的多重突变;Zhang等[39]将CRISPRs技术成功运用于小鼠ES细胞中大片段DNA的插入和敲除。Oh等[40]结合CRISPR-Cas9和重组技术,成功地把密码子饱和诱变应用于罗伊氏乳杆菌当中,CRISPR-Cas9还可用于在低重组效率的细菌中鉴定重组细胞。此外,对CRISPR基因簇与Cas蛋白的研究还可为探究种属间亲缘关系及多样性提供新的思路[29]。
CRISPRs基因编辑技术的操作程序主要包括以下几步:首先通过测定转化效率确定CRISPR-Cas自杀活性,接下来对转座子进行诱变,诱变完成后通过CRISPR / Cas系统筛选自杀逃避型突变体,进而排除假阳性,然后运用PCR技术鉴定转座子插入位点,通过“清除”缺失来验证表型,最后补充转导中缺失的片段,即可完成基因编辑[41]。
2.2 gRNA-Cas9进行基因编辑的影响因素当前基于CRISPR-Cas系统的基因编辑主要依赖gRNA-Cas9,其主要受以下因素影响。
2.2.1 gRNA的选择gRNA的引导方式及其tracrRNA长度的设计是CRISPRs基因编辑技术的关键影响因素[42]。gRNA的引导方式有单独引导和双向引导两种方式,单独引导是将与靶DNA互补的crRNA与tracrRNA连接成为一条单链的引导RNA(single guide RNA,sgRNA)。相对于双引导RNA(dual guide RNA),sgRNA不易引起Indel突变,编辑速度也比双引导更快;其次,gRNA上tracrRNA序列的长度也是影响CRISPR系统作用的主要因素,多数研究将gRNA的大小设计为100 nt左右,gRNA 5'端20 nt的区域为DNA互补区、3'端70-80 nt的区域为tracrRNA序列,crRNA区则居于中间位置[42]。
gRNA-Cas9复合体的结合方式也会影响CRISPR/Cas9的作用效率。延伸gRNA上的Cas9结合区能够增强Cas蛋白活性,进而提高编辑效率,主要方法有两种:在gRNA3'端紧邻tracrRNA的区域增加一段5 nt的核苷酸序列,或者于螺旋区增加4-10 bp的碱基对以增强crRNA与tracrRNA的结合[43]。
2.2.2 脱靶效应的显著影响脱靶效应与gRNA和Cas9蛋白的浓度及比例相关,降低gRNA的浓度可以降低脱靶率,但降低Cas9蛋白浓度在降低脱靶率的同时降低了正靶率[44]。鉴于gRNA及Cas9蛋白对脱靶效应的影响尚有争议[44-46],我们可以从gRNA及Cas9的浓度比入手,选择出最佳的gRNA/Cas9比例,以降低脱靶率。sgRNA的结构对脱靶效应也有影响,可以通过对sgRNA进行截短、修饰来提高正靶/脱靶之比,如在5'端识别区之前加额外的GG碱基,或将gRNA的3'末端截短[47]。此外,有研究表明gRNA-Cas9复合体能够耐受靶向序列5'端的突变[48, 49]。再者,通过点突变使RuvC或HNH亚基失活,构建Cas9蛋白的突变体DNA切口酶(又称dCas9,即deadCas9) 亦能够减少脱靶现象。该突变体能够引起DNA单链断裂(SSB),相比于DNA双链断裂,DNA单链断裂能够产生近距离切口,进而引起带有黏性末端的DSB[45],提高打靶效率,同时能够减轻脱靶的不利影响。张锋等人的研究指出,减少mRNA的用量也能够有效地抑制脱靶现象的发生[50]。
2.2.3 突变体位点的选择CRISPR/Cas9对靶点的识别需要PAM(NGG)和紧邻PAM的11 bp种子序列完全保守,所以将突变位点设定在这14 bp之内可以防止突变引起的切割功能丧失[42]。错配序列的出现会导致比较严重的脱靶现象,有研究表明,前半部分序列的单核苷酸错配比后半部分的错配具有更好的耐受性[43],因此对目标位点的合理选择有望能够使脱靶效应的不利影响最小化。
2.3 优势 2.3.1 简单易行的设计操作方法CRISPR/Cas9系统的构造简单,该技术对基因的编辑主要依靠一个sgRNA-Cas9复合体,而无须太多辅助蛋白,编辑成本低,操作更加简便。此外,CRISPR/Cas9识别域的构建相对简单,想要改变靶DNA识别位点仅需要改变一小段20 bp大小的前导子序列[51],载体构建时间明显缩短,大大减少了工作量。
2.3.2 高效率的编辑与更好的通用性相比于ZFN、TALEN等基因编辑工具,CRISPR技术能够更加高效地进行基因编辑。Ding等[42]分别用CRISPR、TALEN对人类多功能干细胞进行编辑,结果显示CRISPR法的敲入克隆效率可达11%,而TALEN的效率仅有1.6%。CRISPR对靶DNA的特异性识别依赖于长度仅为2-5 bp的PAM序列[52],因此能够识别更多的序列,扩大CRISPR的通用性。
2.3.3 更高的打靶特异性利用CRISPR进行定点编辑更加精确。主要原因在于Cas9蛋白有RuvC和HNH两个功能区,它们分别负责DNA两条链的切割,还能利用任意一个功能区的突变将其改造为DNA切口酶,分别切割DNA的两条链并产生黏性末端。D10A及H840双突变dCas9更能够成为一种高特异性的锚定蛋白[53, 54]。
2.3.4可实现同时对多个不同靶DNA序列的编辑CRISPR/Cas9技术更大的优势在于它能够用于同时编辑多个靶基因位点。“重复-间隔”的CRISPR基因座天然结构使其能够同时插入多个新的外源DNA片段,从而实现多位点编辑。Chen等[55]利用CRISPR/Cas9对小鼠模型进行多个靶肿瘤基因的筛查,Zhang等则将多个引导序列编码到单个CRISPR阵列中,以使其能够同时编辑哺乳动物基因组内的多个位点[56]。
2.3.5可运用于对真核生物基因的编辑修饰CRISPR-Cas系统虽来自于细菌和古细菌,却能够被广泛应用于真核细胞的DNA突变中。这一应用的实现只需人为将Cas9蛋白转运到哺乳动物细胞内。Cong和Zhang等[56]研究首次利用CRISPR/Cas9系统实现了对人293T细胞的EMX1和PVALB基因及小鼠Nero2A细胞的Th基因的定点突变,并指出需要在Cas9蛋白的两端加上真核细胞的核定位号NLS,但也有研究指出只需在一端添加NLS即可实现Cas9的高效转运[38]。
3 展望自21世纪初CRISPRs系统被发现以来,CRISPRs基因编辑技术飞速发展,已然成为第三代基因编辑工具,并造就了一批批科研奇迹。该技术的应用,涵盖生物、农业、环境及医学等多个领域。生物学领域中,它常作为构建突变体的重要手段,将为基因工程的进一步应用以及生物体的保护、研究提供更新、更广的思路;农业领域中,在动植物的改造以及病原微生物的防治方面蕴藏着巨大的应用潜力;医疗领域中,CRISPRs不仅可以用于人类遗传性疾病的治疗,还可以用于疾病相关基因的筛查与检测、帮助确定潜在药靶,为某些疑难杂症,如肿瘤、白血病等的治疗寻求突破口。
但目前该技术的应用也存在着多方面的问题。脱靶效应是影响CRISPRs编辑效率的一个重要因素,如何有效地降低脱靶率是一个较为棘手的问题。其次,来源于细菌的CRISPRs系统是否会对人类以及现有的动植物产生危害或者引起排异现象?如何减少错配现象的发生以及如何缓解突变位点的选择限制对靶基因编辑的影响也是亟待解决的问题。未来的研究中,除了CRISPRs进一步应用外,我们必须从解决该编辑方法自身的问题入手,为CRISPRs的发展扫清障碍。
| [1] | Chylinski K, Makarova KS, Charpentier E, et al. Classification and evolution of type Ⅱ CRISPR-Cas systems[J]. Nucleic Acids Research, 2014, 42 (10): 6091–6105. DOI:10.1093/nar/gku241 |
| [2] | 朱金洁. CRISPR-Cas9介导的玉米基因组定点编辑研究[D]: 北京: 中国农业大学, 2015. |
| [3] | Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31 (3): 233–239. DOI:10.1038/nbt.2508 |
| [4] | Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2011, 156 (5): 935–949. |
| [5] | Chang N, Sun C, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in Zebrafish embryos[J]. Cell Research, 2013, 23 (4): 465–472. DOI:10.1038/cr.2013.45 |
| [6] | Sturino JM, Joseph MS, Todd RK. Engineered bacteriophage-defence systems in bioprocessing[J]. Nature Reviews Microbiology, 2006, 4 (5): 395–404. DOI:10.1038/nrmicro1393 |
| [7] | Abedon ST. Bacterial 'immunity' against bacteriophages[J]. Bacteriophage, 2012, 2 (1): 50–54. DOI:10.4161/bact.18609 |
| [8] | 王立人. CRISPR/CAS系统介导的基因组大片段DNA编辑[D]: 上海: 华东师范大学, 2015. |
| [9] | Bondy-Denomy J, Davidson AR. To acquire or resist:the complex biological effects of CRISPR-Cas systems[J]. Trends in Microbiology, 2014, 22 (4): 218–225. DOI:10.1016/j.tim.2014.01.007 |
| [10] | Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biology, 2007, 8 (4): 61. DOI:10.1186/gb-2007-8-4-r61 |
| [11] | van Embden JDA, van Gorkom T, Kremer K, et al. Genetic variation and evolutionary origin of the direct repeat locus of mycobacterium tuberculosis complex bacteria[J]. Journal of Bacteriology, 2000, 182 (9): 2393–2401. DOI:10.1128/JB.182.9.2393-2401.2000 |
| [12] | Demay C, Liens B, Burguière T, et al. SITVITWEB-A publicly available international multimarker database for studying Mycobac-terium tuberculosis genetic diversity and molecular epidemiology[J]. Infection, Genetics and Evolution, 2012, 12 (4): 755–766. DOI:10.1016/j.meegid.2012.02.004 |
| [13] | Zanden AGMVD, Kremer K, Schouls LM, et al. Improvement of differentiation and interpretability of spoligotyping for mycobacterium tuberculosis complex isolates by introduction of new spacer oligonucleotides[J]. Journal of Clinical Microbiology, 2002, 40 (12): 4628–4639. DOI:10.1128/JCM.40.12.4628-4639.2002 |
| [14] | Lillest?l R, Redder P, Garrett RA, et al. A putative viral defence mechanism in archaeal cells[J]. Archaea, 2006, 2 (1): 59–72. DOI:10.1155/2006/542818 |
| [15] | Choi KR, Sang YL. CRISPR technologies for bacterial systems:current achievements and future directions[J]. Biotechnology Advances, 2016, 34 (7): 1180–1209. DOI:10.1016/j.biotechadv.2016.08.002 |
| [16] | Li C, Cao W. Advances in CRISPR/Cas9-mediated gene editing[J]. Chinese Journal of Biotechnology, 2015, 31 (11): 7080–7081. |
| [17] | Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2011, 9 (6): 467–477. DOI:10.1038/nrmicro2577 |
| [18] | Makarova KS. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems[J]. Biology Direct, 2011, 6 (1): 38–38. DOI:10.1186/1745-6150-6-38 |
| [19] | Chylinski K, Le RA, Charpentier E. The tracrRNA and Cas9 families of type Ⅱ CRISPR-Cas immunity systems[J]. RNA Biology, 2013, 10 (5): 726–737. DOI:10.4161/rna.24321 |
| [20] | Wei C, Liu J, Yu Z, et al. TALEN or Cas9 -rapid, efficient and specific choices for genome modifications[J]. Journal of Genetics and Genomics, 2013, 40 (6): 281–289. DOI:10.1016/j.jgg.2013.03.013 |
| [21] | Anantharaman V, Iyer LM, Aravind L. Presence of a classical RRM-fold palm domain in Thg1-type 3' -5' nucleic acid polymerases and the origin of the GGDEF and CRISPR polymerase domains[J]. Biology Direct, 2010, 5 (1): 43. DOI:10.1186/1745-6150-5-43 |
| [22] | Nickel L, Weidenbach K, J?ger D, et al. Two CRISPR-Cas systems in Methanosarcina mazei strain G?1 display common processing features despite belonging to different types Ⅰ and Ⅲ[J]. RNA Biology, 2013, 10 (5): 779–791. DOI:10.4161/rna.23928 |
| [23] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA[J]. Science, 2008, 322 (322): 1843–1845. |
| [24] | Hale CR, Majumdar S, Elmore J, et al. Essential features and rational design of CRISPR RNAs that function with the Cas RAMP module complex to cleave RNAs[J]. Molecular Cell, 2012, 45 (3): 292–302. DOI:10.1016/j.molcel.2011.10.023 |
| [25] | Makarova KS, Koonin EV. Annotation and classification of CRISPR-Cas systems[J]. Methods in Molecular Biology(Clifton, NJ), 2015, 1311 : 47–75. DOI:10.1007/978-1-4939-2687-9 |
| [26] | Fujii W, Kakuta S, Yoshioka S, et al. Zygote-mediated generation of genome-modified mice using Streptococcus thermophilus 1 -derived CRISPR/Cas system[J]. Biochemical & Biophysical Research Communications, 2016, 477 (3): 473–476. |
| [27] | Wu Q, Tun HM, Leung FC, et al. Genomic insights into high exopolysaccharide-producing dairy starter bacterium Streptococcus thermophilus ASCC 1275[J]. Scientific Reports, 2014, 4 (7500): 4974. |
| [28] | Horvath P, Romero DA, Co?témonvoisin AC, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus[J]. Journal of Bacteriology, 2008, 190 (4): 1401–1412. DOI:10.1128/JB.01415-07 |
| [29] | 邓凯波, 霍贵成. 嗜热链球菌中CRISPR序列的检测与同源性分析[J]. 食品科学, 2013, 34(3): 153–157. |
| [30] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats(CRISPRs)have spacers of extrachromosomal origin[J]. Microbiology, 2005, 151 (Pt 8): 2551–2561. |
| [31] | Mojica FJ, Díezvillase?or C, Garcíamartínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. Journal of Molecular Evolution, 2005, 60 (2): 174–182. DOI:10.1007/s00239-004-0046-3 |
| [32] | Goh YJ, Goin C, O'Flaherty S, et al. Specialized adaptation of a lactic acid bacterium to the milk environment:the comparative genomics of Streptococcus thermophilus LMD-9[J]. Microbial Cell Factories, 2011, 10 Suppl 1 (1): S22. |
| [33] | Brouns SJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321 (5891): 960–964. DOI:10.1126/science.1159689 |
| [34] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337 (6096): 81. |
| [35] | Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J]. Nucleic Acids Research, 2011, 39 (21): 9275–9282. DOI:10.1093/nar/gkr606 |
| [36] | Jinek M, Jiang F, Taylor DW, et al. Structures of Cas9 endonucle-ases reveal RNA-mediated conformational activation[J]. Science, 2014, 343 (6176): 124799. |
| [37] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152 (5): 1173–1183. DOI:10.1016/j.cell.2013.02.022 |
| [38] | Mali P, Yang L, Esvelt KM, Aach J, Guell M, Dicarlo JE, Norville JE, Church GM. NA-guided human genome engineering via Cas9[J]. Science, 2013, 339 : 823–826. DOI:10.1126/science.1232033 |
| [39] | Zhang L, Jia R, Palange NJ, et al. Large genomic fragment deletions and insertions in mouse using CRISPR/Cas9[J]. PLoS One, 2015, 10 (3): e0120396. DOI:10.1371/journal.pone.0120396 |
| [40] | Oh JH, van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J]. Nucleic Acids Research, 2014, 42 (17): e131. DOI:10.1093/nar/gku623 |
| [41] | Yosef I, Goren MG, Edgar R, et al. Using the CRISPR-Cas system to positively select mutants in genes essential for its function[J]. Methods in Molecular Biology, 2015, 1311 : 233–250. DOI:10.1007/978-1-4939-2687-9 |
| [42] | Ding Q, Regan SN, Xia Y, et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs[J]. Cell Stem Cell, 2013, 12 (4): 393–394. DOI:10.1016/j.stem.2013.03.006 |
| [43] | Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells[J]. Elife, 2013, 2 (2): e00471. |
| [44] | Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nature Biotechnology, 2013, 31 (9): 839–843. DOI:10.1038/nbt.2673 |
| [45] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes[J]. Nature Biotechnology, 2014, 32 (4): 347–355. DOI:10.1038/nbt.2842 |
| [46] | Fu Y, Foden JA, Khayter C, et al. High frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. NatureBiotechnology, 2013, 31 (9): 822–826. |
| [47] | Cho SW, Kim S, Kim Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Research, 2014, 24 (1): 377–389. |
| [48] | Semenova E, Severinov K. Interference by clustered regularly interspaced short palindromic repeat(CRISPR)RNA is governed by a seed sequence[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108 (25): 10098–10103. DOI:10.1073/pnas.1104144108 |
| [49] | Cradick TJ, Fine EJ, Antico CJ, et al. CRISPR/Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity[J]. Nucleic Acids Research, 2013, 41 (20): 9584–9592. DOI:10.1093/nar/gkt714 |
| [50] | Zhang F. CRISPR/Cas9 for genome editing:progress, implications and challenges[J]. Human Molecular Genetics, 2014, 24 (R6): 40–48. |
| [51] | Gupta RM, Musunuru K. Expanding the genetic editing tool kit:ZFNs, TALENs, and CRISPR-Cas9[J]. The Journal of Clinical Investigation, 2014, 124 (10): 4154–4161. DOI:10.1172/JCI72992 |
| [52] | Shah SA, Shah SA, Erdmann S, et al. Protospacer recognition motifs:mixed identities and functional diversity[J]. RNA Biology, 2013, 10 (5): 891–899. DOI:10.4161/rna.23764 |
| [53] | Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification[J]. Nature Biotechnology, 2014, 32 (6): 577–582. DOI:10.1038/nbt.2909 |
| [54] | Tsai SQ, Wyvekens N, Khayter C, et al. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing[J]. Nature Biotechnology, 2014, 32 (6): 569–576. DOI:10.1038/nbt.2908 |
| [55] | Chen S, Sanjana Neville E, Zheng K, et al. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis[J]. Cell, 2015, 160 (6): 1246–1260. DOI:10.1016/j.cell.2015.02.038 |
| [56] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339 (6121): 819–823. DOI:10.1126/science.1231143 |