基因编辑技术是一种可以对基因组或转录产物进行精确修饰的技术,可完成基因定点突变、片段的敲除或敲入等。在后基因组时代,基因编辑技术已成为生命科学领域的重要研究内容。传统的基因编辑技术以胚胎干细胞和基因重组为基础进行生物基因组定向修饰,但是该技术存在打靶效率低,实验周期长及应用范围窄等缺点[1, 2]。随着基因编辑技术的不断发展,人工核酸酶介导的基因编辑技术开始被广泛应用,该技术通过特异性地识别并裂解靶DNA双链,激发细胞内源性的修复机制实现基因定向改造,与传统基因编辑技术相比,基因编辑新技术打靶效率高、构建成本低、应用范围广,极大地促进了生命科学和医学领域的研究进程[3, 4]。目前基因组编辑新技术主要包括以下几种:人工核酶介导的锌指核酸酶(Zinc finger nucleases,ZFN)技术[5, 6]、类转录激活因子效应物核酸酶(Transcription activator-like effectors nuclease,TALEN)技术[7]、成簇规律的间隔短回文重复相关蛋白9核酸酶(Clustered regularly interspaced short palindromic repeats-associated protein-9 nuclease,CRISPR/Cas9) 技术、CRISPR/Cpf1技术、Ago/gDNA技术以及结构引导的核酸酶(Structure-guided nuclease,SGN)技术。现对上述几种新型技术的作用原理、优缺点、应用等进行比较分析。
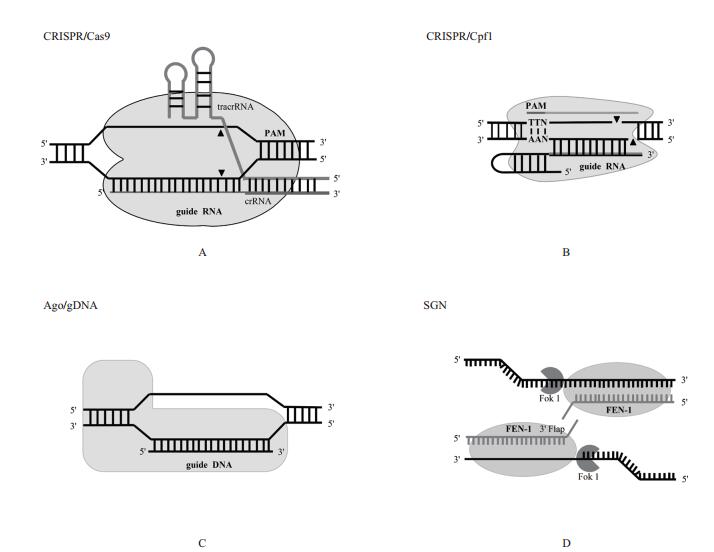
1 CRISPR/Cas9CRISPR/Cas9从对真菌和古菌宿主免疫防御研究的基础上发展而来,属于CRISPR/Cas蛋白家族中的Ⅱ型,该系统结构简单,仅由Cas9蛋白、tracrRNA、crRNA及RNaseⅢ 4种成分组成[8]。其中Cas9蛋白本身既具有核酸内切酶活性,又能与tracrRNA:crRNA复合物结合,该蛋白含有两个独特的活性位点,分别是位于氨基末端的RuvC和蛋白中部的HNH,这两个位点在crRNA的成熟及双链DNA的剪切过程中发挥重要作用。反式激活crRNA(Trans-activating crRNA,tracrRNA)与pre-crRNA的重复序列互补,在pre-crRNA转录的同时转录出来,且促使Cas9利用其活性位点对pre-crRNA进行加工。另外,RNaseⅢ核酸酶也参与pre-crRNA的加工剪切过程。加工得到的crRNA与tracrRNA形成tracrRNA:crRNA复合物并结合于Cas9蛋白组成CRISPR/Cas9系统,tracrRNA:crRNA复合物引导Cas9识别靶序列DNA的PAM序列,随后crRNA与互补链结合形成RNA:DNA杂合链;另一条非互补DNA链游离,最后由Cas9蛋白的RuvC和HNH位点分别切割互补链和非互补链进而引入DNA双链断裂,然后通过体内的DNA修复机制将DNA双链修复完整,目前的CRISPR/Cas9通常将用人工设计的sgRNA替换tracrRNA:crRNA复合物(图 1-A)。尽管Cas9蛋白分子量较大且长短不一,但都是用Ruvc核酸酶结构域和HNH核酸酶结构域行使切割靶序列的功能,使靶DNA双链断裂[9, 10],这样大大降低了CRISPR/Cas9系统的设计难度。
由于CRISPR/Cas9系统构建简单、成本低廉,现已广泛应用于细胞和动物模型的快速制备、转录调控及遗传性疾病的治疗等。Niu等[11]利用CRISPR/Cas9系统成功对食蟹猴进行了精确的基因编辑,在该项研究中,实验人员以食蟹猴胚胎中的Nr0b1、Ppar-r和Rag1等基因作为靶位点进行实验,结果发现Ppar-r和Rag1出现突变,随后经代孕生出的子代孪生猴也被证实存在Ppar-r和Rag1基因突变。Liang等[12]利用CRISPR/Cas9成功编辑人类三核受精卵中的地中海贫血相关基因HBB,这也是首次发表有关人类基因编辑的研究。研究选取了54个三核受精卵(其发育止步于囊胚阶段无法发育为人类胚胎)样本作为实验对象,其中28个样本中的HBB基因被成功编辑,同时也发现某些样本出现了脱靶突变。Kang等[13]利用CRISPR/Cas9对人类三核受精卵引入CCR5 23基因突变,以期建立受试样本对HIV的免疫力。结果表明,26个胚胎中的4个成功获得改造,同时也发现脱靶现象。Zhu等[14]利用配对导向RNA(Paired-guided RNA,pgRNA)和CRISPR/Cas9,在全基因组范围内对人源肝癌细胞中的近700个疾病相关长链非编码RNA(long non-coding RNA,lncRNA)进行基因敲除研究。Lu等首次将CRISPR/Cas9用于临床人体试验,研究人员利用CRISPR/Cas9对从肺癌患者血液中分离出的免疫细胞进行基因编辑,靶向失活其PD-1蛋白,随后又将编辑过的免疫细胞重新注入患者体内用来观察人体对免疫细胞的反应[15]。目前该研究尚处于初步阶段,后续的研究还将陆续展开。
2 CRISPR/Cpf1CRISPR/Cas9系统的发现无疑为整个基因编辑领域带来了一场技术革命,然而张锋团队并不满足于CRISPR/Cas9系统尚不完美的现状,继续在CRISPR家族内寻找更加完善的基因编辑工具酶,最终发现了基因编辑效率较高的Cpf1蛋白,它属于CRISPR/Cas蛋白家族的Ⅴ型[16]。CRISPR/Cpf1系统中的Cpf1蛋白为三角形单体[17],具有结合crRNA和切割靶序列的能力。与CRISPR/Cas9系统不同,CRISPR/Cpf1系统中crRNA的形成不需要tracrRNA和RNase Ⅲ核酸酶的参与。成熟的crRNA一部分形成发卡结构紧密结合于Cpf1蛋白的核酸结合结构域,发卡结构对于靶序列的切割过程具有重要意义;另一部分通过碱基互补的方式结合靶序列从而引导Cpf1到达靶序列并进行切割。CRISPR/Cpf1系统识别靶序列DNA的过程同样需要PAM序列的存在,切割靶序列后留下长度为5 nt的黏性末端,最终通过细胞内的DNA修复途径进行修复[16](图 1-B)。
CRISPR/Cpf1系统与CRISPR/Cas9系统的差别主要体现在:(1) 天然的CRISPR/ Cpf1系统在组成上更加简单,仅仅包含一条42 nt的crRNA,另外Cpf1蛋白比Cas9蛋白要小,使其更易于被设计和输送至细胞内;(2) CRISPR/Cpf1系统以一种不同的方式切割靶DNA。CRISPR/Cas9系统切割靶DNA双链后留下的“平末端”在修复时容易发生突变,因此严格来讲,CRISPR/Cas9系统对靶基因实行的是破坏而不是编辑,CRISPR/Cpf1系统则在切割靶DNA后留下“黏性末端”,这可能有助于外源DNA片段的精确整合,从而实现真正意义上的基因组编辑;(3) 与CRISPR/Cas9系统类似,CRISPR/ Cpf1系统对靶DNA的识别同样依赖PAM序列的存在[18],不过Cas9系统识别的是前间隔序列3'端的NGG PAM序列,而Cpf1系统识别前间隔序列5'端TTN PAM序列;(4) CRISPR/ Cpf1系统会在距离识别位点较远的位置进行切割,这为研究人员提供了更多的编辑位点。同时,已有研究证明CRISPR/ Cpf1系统的脱靶率远远低于CRISPR/ Cas9系统[19]。
3 Ago/gDNAArgonaute(Ago)是一个高度保守的蛋白家族,广泛存在于几乎所有物种体内,作为RNA诱导的沉默复合体(RNA-induced silenceing complex,RISC)的主要成分,在如RNA干扰、microRNA及piRNA介导的转录后沉默等过程中发挥至关重要的作用[20-22]。研究发现,某些原核生物Ago,如TtAgo和PfAgo[20-25],可以通过小干扰核酸gDNA介导外源基因(外源性质粒或噬菌体)的表达抑制。TtAgo以长度13-25 nt,5'P-gDNA为向导,靶向切割外源性单链DNA或双链DNA;PfAgo通过结合长度15-31 nt,5'P-gDNA引起外源性基因靶向突变(图 1-C)。但无论TtAgo还是PfAgo都只在65℃才能进行反应,无法在哺乳动物细胞中发挥作用[24, 25]。
Gao等[26]发现Natronobacterium gregoryi中的Ago蛋白(NgAgo)与TtAgo和PfAgo同源性较高,并用实验证明NgAg/gDNA能够在人类细胞中发挥DNA编辑功能。不过,Qi等[27]研究表明,利用NgAgo/gDNA技术的确能够降低靶基因fabp11a表达水平,进而影响斑马鱼眼睛的正常发育。但测序结果表明,fabp11a基因并未发生改变,而只是其mRNA的表达量下降。同样,对斑马鱼的ta、kdrl、lamal和flt1等基因进行检测,发现NgAgo/gDNA带来的表型改变均与基因编辑无关。基于以上数据,研究人员推测NgAgo/gDNA可能通过基因敲减而非基因编辑发挥其调控作用。与此同时,Burgess等[28]20余名科学家联名发文声称,按照Gao等论文中的实验步骤进行操作,未能发现NgAgo的基因编辑作用。Lee等[29]3个研究团队也同样表示,无法在Hela、HEK293T及U20S等人类细胞系中检测到NgAgo/gDNA的基因编辑现象。
4 SGN在过去几年中,以ZFN和TALEN为代表的序列特异性核酸酶技术以其能够高效率地进行定点基因组编辑,在基础研究、遗传改良和基因治疗等方面展示出巨大的潜力。不过,由于邻近序列效应的存在[30-32],导致ZFN技术脱靶现象较为严重;而TALEN技术中类转录激活因子效应物与碱基一一对应的关系在一定程度上降低了脱靶率,但由于TALEN的组装与模块化设计过程繁琐,极大的限制了其推广应用。ZFN与TALEN都依赖FokⅠ核酸内切酶的存在,FokⅠ是一种存在于细菌Flavobacterium okeanokoites体内的ⅡS型限制性核酸内切酶,含有一个DNA结合区和一个DNA切割区。其中ZFN技术利用锌指蛋白(Zinc finger protein,ZFP)可以通过DNA-蛋白质的原理特异性识别DNA序列的特点,研究人员将锌指DNA结合区与限制性核酸内切酶FokⅠ的切割区进行融合,锌指DNA结合区负责特异性识别靶序列,FokⅠ则负责对靶序列进行切割;在TALEN技术中,负责切割靶序列DNA的功能元件仍是FokⅠ核酸酶的切割区,只不过将特异性识别靶序列的定位元件替换成了一段人造的TALE[9, 33]。片状核酸内切酶1(Flap endonulease Ⅰ,FENⅠ)是拥有5'片状核酸内切酶、缺口依赖性核酸内切酶及核酸外切酶三种酶切活性,可以对DNA复制和修复途径中的多种中间产物进行加工,在冈崎片段成熟长片段碱基切除修复和端粒维持中发挥重要作用[34-36]。
最近,研究人员通过连接子将FokⅠ和FENⅠ进行组装,设计出了一种新型的基因组DNA编辑工具SGN,并成功将其应用于斑马鱼的基因组DNA编辑[37]。SGN的作用原理如下:设计gDNA与靶序列DNA链杂化并形成3'片瓣状结构,随后SGN的靶向部分FENⅠ特异性识别这一特殊结构,引导SGN与靶序列的切割位点(距离gDNA 3'端9-10 nt)结合进而对靶序列实施切割,最后通过DNA修复机制对断裂的双链进行修复(图 1-D)。研究人员利用SGN成功突变了斑马鱼的Znf703和Cyp26b1基因,其中Znf703的突变率为1.04%,被切割的片段为754 bp,另外下游序列还有11 bp被删除;Cyp26b1的突变率为10.3%,被删除的碱基片段则多达2 610 bp。与上述几种基因编辑技术显著不同,SGN对靶序列的切割是大段的,原因可能是:SGN识别切割靶DNA序列后产生缺口,而SGN中的FENⅠ能够识别缺口结构,引导SGN对含有缺口结构的DNA链进行持续切割,最终产生大片段的序列缺失。
5 展望表 1汇总比较了CRISPR/Cas9、CRISPR/Cpf、Ago/gDNA和SGN等4种技术的特点。CRISPR/Cas9作为基因编辑工具,一经问世便受到了基因编辑领域科研工作者的追捧,短短几年时间就已经发展得相当成熟,现已广泛应用到多个物种体内并取得了相当可观的成就,而且人类已经迈出了将CRISPR/Cas9应用于临床治疗的第一步。为了克服脱靶效应获得更高的基因编辑效率,科研人员对CRISPR/Cas9的研究和改进工作仍在进行中。CRISPR/Cpf1被认为是CRISPR/Cas9的升级版,在结构组成上及切割方式上的优势使其具有很大的潜在应用价值。SGN沿用了第一、二代基因编辑技术的功能元件FokⅠ,但在靶向元件设计方面独具匠心,选用了识别特异性结构的FENⅠ。不过就目前的研究来看,SGN的基因编辑效率较低,而且其裂解靶序列后留下的大片段缺失会造成后续DNA修复困难,脱靶效应是否存在还有待考证。目前的研究表明,Ago/gDNA系统还无法用于哺乳动物细胞的基因编辑,建议继续进行以下尝试:可以对TtAgo和PfAgo的结构进行改造,使其可以在37℃下工作。另外,可以继续从非嗜热菌中发掘可以进行基因编辑的Ago蛋白。基因编辑技术极大地推动了整个生命科学领域的研究。随着对各种基因编辑工具的不断改进,人们对生命奥秘的研究也更加深入,不过想要真正将其应用于临床治疗造福人类还需做出更多的努力。
| [1] | Plump AS, Smith JD, Hayek T, et al. Severe hypercholesterolemia and atherosclerosis in apolipoprotein E deficient mice created by homologous recombination in ES cells[J]. Cell, 1992, 71 (2): 343–353. DOI:10.1016/0092-8674(92)90362-G |
| [2] | Capecchi MR. Altering the genome by homologous recombination[J]. Science, 1989, 244 (4910): 1288–1292. DOI:10.1126/science.2660260 |
| [3] | Wood AJ, Lo TW, Zeitler B, et al. Targeted genome editing across species using ZFNs and TALENs[J]. Science, 2011, 333 (6040): 307–307. DOI:10.1126/science.1207773 |
| [4] | Lo TW, Pickle CS, Lin S, et al. Precise and heritable genome editing in evolutionarily diverse nematodes using TALENs and CRISPR/Cas9 to engineer insertions and deletions[J]. Genetics, 2013, 195 (2): 331–348. DOI:10.1534/genetics.113.155382 |
| [5] | Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature, 2005, 435 (7042): 646–651. DOI:10.1038/nature03556 |
| [6] | Miller JC, Holmes MC, Wang J, et al. An improved zinc-finger nuclease architecture for highly specific genome editing[J]. Nat Biotechnol, 2007, 25 (7): 778–785. DOI:10.1038/nbt1319 |
| [7] | Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors[J]. Science, 2009, 326 (5959): 1509–1512. DOI:10.1126/science.1178811 |
| [8] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337 (6069): 816–821. |
| [9] | Hsu PD, Lander ES, Zhang F. Development and applications of crispr-cas9 for genome engineering[J]. Cell, 2014, 157 (6): 1262–1278. DOI:10.1016/j.cell.2014.05.010 |
| [10] | Chylinski K, Le Rhun A, Charpentier E. The tracrRNA and Cas9 families of type Ⅱ CRISPR/Cas immunity systems[J]. RNA Biol, 2013, 10 (5): 726–737. DOI:10.4161/rna.24321 |
| [11] | Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell, 2014, 156 (4): 836–843. DOI:10.1016/j.cell.2014.01.027 |
| [12] | Liang P, Xu Y, Zhang X, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes[J]. Protein Cell, 2015, 6 (5): 363–372. DOI:10.1007/s13238-015-0153-5 |
| [13] | Kang X, He W, Huang Y, et al. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing[J]. J Assist Reprod Gen, 2016, 33 (5): 581–588. DOI:10.1007/s10815-016-0710-8 |
| [14] | Zhu S, Li W, Liu J, et al. Genome-scale deletion screening of human long non-coding RNAs using a paired-guide RNA CRISPR-Cas9 library[J]. Nat Biotechnol, 2016, 34 (12): 1279–1286. DOI:10.1038/nbt.3715 |
| [15] | Cyranoski D. CRISPR gene-editing tested in a person for the first time[J]. Nature, 2016, 539 (7630): 479. DOI:10.1038/nature.2016.20988 |
| [16] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163 (3): 759–771. DOI:10.1016/j.cell.2015.09.038 |
| [17] | Dong D, Ren K, Qiu X, et al. The crystal structure of Cpf1 in complex with CRISPR RNA[J]. Nature, 2016, 532 (7600): 522–526. DOI:10.1038/nature17944 |
| [18] | Mali P, Aach J, Stranges PB, et al. CAS9 transcriptional activators for target specificity screening and paried nickases for cooperative genome engineering[J]. Nat Biotechnol, 2013, 31 (9): 833–838. DOI:10.1038/nbt.2675 |
| [19] | Kim D, Kim J, Hur JK, et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells[J]. Nat Biotechnol, 2016, 34 (8): 863–868. DOI:10.1038/nbt.3609 |
| [20] | Song JJ, Smith KK, Hannon GJ, et al. Crystal structure of Argonaute and its implications for RISC slicer activity[J]. Science, 2004, 305 (5689): 1434–1437. DOI:10.1126/science.1102514 |
| [21] | Yuan YR, Pei Y, Ma JB, et al. Crystal structure of A. aeolicus argonaute, a site-specific DNA-guided endoribonuclease, provides insights into RISC-mediated MRNA cleavage[J]. Mol Cell, 2005, 19 (3): 405–419. DOI:10.1016/j.molcel.2005.07.011 |
| [22] | Junho KH, Ivan O, Alexei AA. Prokaryotic Argonautes defend genomes against invasive DNA[J]. Trend Biochem Sci, 2014, 39 (6): 257–259. DOI:10.1016/j.tibs.2014.04.006 |
| [23] | Meister G. Argonaute protein:Functinal insights and emerging roles[J]. Nat Rev Genet, 2013, 14 (7): 447–459. DOI:10.1038/nrg3462 |
| [24] | Swarts DC, Jore MM, Westra ER, et al. DNA-guided DNA interference by a prokaryotic Argonaute[J]. Nature, 2014, 507 (7491): 258–261. DOI:10.1038/nature12971 |
| [25] | Daan CS, Jorrit WH, Ismael H, et al. Argonaute of the archaeon Pyrococcus furiosus is a DNA-guided nuclease that targets cognate DNA[J]. Nucleic Acids Res, 2015, 43 (10): 5120–5129. DOI:10.1093/nar/gkv415 |
| [26] | Gao F, Shen XZ, Jiang F, et al. DNA-guided genome editing using the Natronobacterium gregoryi Argonaute[J]. Nat Biotechnol, 2016, 34 (7): 768–773. DOI:10.1038/nbt.3547 |
| [27] | Qi J, Dong Z, Shi Y, et al. NgAgo-based fabp11a gene knockdown causes eye developmental defects in zebrafish[J]. Cell Res, 2016, 26 (12): 1349–1352. DOI:10.1038/cr.2016.134 |
| [28] | Burgess S, Cheng L, Gu F, et al. Questions about NgAgo[J]. Protein Cell, 2016, 7 (12): 913–915. DOI:10.1007/s13238-016-0343-9 |
| [29] | Lee SH, Turchiano G, Ata H, et al. Failure to detect DNA-guided genome editing using Natronobacterium gregoryi Argonaute[J]. Nat Biotechnol, 2016, 35 (1): 17–18. |
| [30] | Imanishi M, Nakamura A, Morisaki T, et al. Positive and negative cooperativity of modularly assembled zinc fingers[J]. Biochem Bioph Res Co, 2009, 387 (3): 440–443. DOI:10.1016/j.bbrc.2009.07.059 |
| [31] | Maeder ML, Thibodeau-Beganny S, Osiak A, et al. Rapid "open-source" engineering of customized zinc-finger nucleases for highly efficient gene modification[J]. Mol Cell, 2008, 31 (2): 294–301. DOI:10.1016/j.molcel.2008.06.016 |
| [32] | Juillerat A, Dubois G, Valton J, et al. Comprehensive analysis of the specificity of transcription activator-like effector nucleases[J]. Nucleic Acids Res, 2014, 42 (8): 5390–5402. DOI:10.1093/nar/gku155 |
| [33] | Smith J, Grizot S, Arnould S, et al. A combinatorial approach to create artificial homing endonucleases cleaving chosen sequences[J]. Nucleic Acids Res, 2006, 34 (22): e149. DOI:10.1093/nar/gkl720 |
| [34] | Harrington JJ, Lieber MR. The characterization of a mammalian DNA structure-specific endonuclease[J]. EMBO J, 1994, 13 (5): 1235–1246. |
| [35] | Kaiser MW, Lyamicheva N, Ma W, et al. A comparison of eubacterial and archaeal structure-specific 5' -exonucleases[J]. J Biol Chem, 1999, 274 (30): 21387–21394. DOI:10.1074/jbc.274.30.21387 |
| [36] | Kao HI, Henricksen LA, Liu Y, et al. Cleavage specificity of Saccharomyces cerevisiae flap endonuclease 1 suggests a double-flap structure as the cellular substrate[J]. J Biol Chem, 2002, 277 (17): 14379–14389. DOI:10.1074/jbc.M110662200 |
| [37] | Xu S, Cao S, Zou B, et al. An alternative novel tool for DNA editing without target sequence limitation:the structure-guided nuclease[J]. Genome Biol, 2016, 17 (1): 186. DOI:10.1186/s13059-016-1038-5 |
| [38] | Isabella Wolcott. Could the CRISPR-Cas9 genome editing system be replaced by the NgAgo-gDNA system?[J]. Express Biology, 2016, 1 (1): 10–15. |